PLUG引物和IT引物在頂芒山羊草分子標記建立中的通用性分析
宮文萍 李凱 程敦公 武智民 李洪振 韓冉 李豪圣 劉成 劉建軍

摘要:分子標記是檢測和追蹤小麥背景中近緣物種染色體的重要手段。依據禾本科物種基因共線性原則,基于基因序列設計的引物在不同物種中可能具有通用性。本研究對水稻PLUG引物和簇毛麥IT引物在頂芒山羊草中的通用性進行了分析,并建立了頂芒山羊草染色體特異IT標記。供試的397對PLUG引物和841對IT引物對小麥-頂芒山羊草雙二倍體與中國春的擴增結果顯示,377對(95.0%)PLUG引物和593對(70.5%)IT引物可以在供試材料中擴增出清晰條帶。因此,PLUG引物在頂芒山羊草中的通用性優于IT引物。相比小麥對照,PLUG引物和IT引物能在小麥-頂芒山羊草雙二倍體中擴增出多態性條帶的引物數分別有17對和5對,分別占供試PLUG引物和IT引物的4.3%和0.6%。利用獲得的IT多態性引物對一套小麥-頂芒山羊草附加系和一套小麥-卵穗山羊草附加系進行擴增,結果發現僅引物CINAU1060、CINAU1465和CINAU1517能在相應附加系中擴增出大小分別約為300、480 bp和320 bp的多態性標記,為小麥背景中頂芒山羊草染色質的鑒定提供了新的檢測手段。然而,本研究中IT引物建立頂芒山羊草分子標記的比率僅為0.4%,低于我們之前用PLUG引物建立頂芒山羊草分子標記的比率(8.9%)。
關鍵詞:頂芒山羊草;分子標記;PLUG引物;IT引物;通用性
中圖分類號:S512.103.53文獻標識號:A文章編號:1001-4942(2019)07-0010-07
Abstract Molecular marker plays an important role in detecting and tracking chromosomes of related species in wheat background. According to the principle of gene collinearity in Gramineae species, primers designed based on gene sequences might be universal in different species. In this research, the universality of PLUG primers for Oryza sativa and IT primers for Dasypyrum villosum were analyzed in Aegilops comosa, and chromosome specific IT markers of Ae.comosa were established. The amplification results of 397 pairs of PLUG primers and 841 pairs of IT primers in wheat-Ae.comosa amphiploid and Chinese Spring showed that 377 pairs (95.0%) of PLUG primers and 593 pairs (70.5%) of IT primers could amplify clear bands in the tested materials. Therefore, the universality of PLUG primers in Ae.comosa was better than that of IT primers. Compared with Chinese Spring, the number of PLUG and IT primers that could amplify polymorphic bands in wheat-Ae.comosa amphiploid was 17 pairs and 5 pairs, which accounted for 4.3% and 0.6% of the PLUG and IT tested primers, respectively. In this research, a set of wheat-Ae.comosa addition and a set of wheat-Ae.geniculata addition were amplified with the obtained IT polymorphic primers. The results showed that 300, 480 bp and 320 bp in length polymorphism bands could be amplified by primers CINAU1060, CINAU1465 and CINAU1517 in relative additions, respectively, which provided new method for the identification of Ae.comosa chromatin in wheat background. However, the ratio for molecular marker development of Ae.comosa using IT primers was 0.4%, which was much lower than that of PLUG primers (8.9%).
Keywords Aegilops comosa; Molecular marker; PLUG primer; IT primer; Universality
小麥(Triticum aestivum L.)是世界上播種面積最大、總產量最多和分布范圍最廣的重要糧食作物,但其病害如小麥條銹病、葉銹病和白粉病等嚴重阻礙了小麥生產[1]。小麥近緣物種抗病性優異,從小麥近緣物種中將抗病基因導入小麥是改良小麥抗病性的重要方法之一。小麥近緣物種如山羊草屬(Aegilops)中的頂芒山羊草(Ae. comosa,也作Triticm comosum,2n=2x=14,基因組MM),起源于歐洲南部的希臘和土耳其等國,一年生草本植物,高抗小麥條銹病[2]、稈銹病[2]、葉銹病[3]和白粉病[4],是小麥抗病育種的優異基因源。小麥-近緣物種雙二倍體是向普通小麥轉育頂芒山羊草抗病性的重要橋梁[5,6],目前,本試驗室已鑒定獲得小麥-頂芒山羊草雙二倍體[7]以及該雙二倍體與普通小麥的雜交后代材料,然而,分子標記的缺乏極大地限制了這批雜交后代材料的精確鑒定。
分子標記對小麥背景中近緣物種染色體的鑒定和追蹤起到重要作用。研究證明,可依據禾本科物種基因共線性原則,根據已測序物種的基因序列開發引物用于建立另一未測序物種的分子標記[3,6]。基于水稻和簇毛麥基因序列的PLUG引物[8]和IT引物[9]已經被分別開發出來。目前,PLUG標記不僅已經被用于識別小麥與外源染色體的同源群[10-12],還被用于標記輔助選擇(MAS)、比較基因組學、異源染色體的追蹤和基因分型[10-13]。近年來,IT引物也已被用于鑒定小麥背景中的簇毛麥染色質[9,14]。然而,這兩類引物在頂芒山羊草中的通用性還不清楚。本研究利用PLUG引物和IT引物對小麥-頂芒山羊草雙二倍體及對照小麥進行PCR擴增,比較了兩類引物在頂芒山羊草中的通用性,進而利用一套小麥-頂芒山羊草附加系為材料,對獲得的多態性片段進行染色體定位,建立了頂芒山羊草IT分子標記,為小麥背景中頂芒山羊草染色質的檢測提供了新的手段。
1 材料與方法
1.1 試驗材料
24份供試材料的相關信息列于表1,其中,小麥中國春由電子科技大學(University of Electronic Science and Technology of China, UESTC)楊足君教授提供。序號2—13的材料由英國John Innes Centre(JIC)的Reader S.M.教授提供。序號14—20的材料由美國堪薩斯州立大學小麥遺傳與基因資源中心(Wheat Genetics Resource Center, WGRC)的Friebe B.教授提供。序號21—24的材料由本實驗室(Current Lab, CL)育成。
1.2 試驗方法
1.2.1 DNA提取
供試材料基因組總DNA用快捷型植物基因組DNA提取試劑盒(天根生化科技(北京)有限公司)提取。取約100 mg的葉片放入2 mL離心管(提前加入直徑2 mm的鋼珠)中,液氮中速凍,然后用SCIENTZ-192組織研磨機在30 Hz頻率下將葉片打碎至粉末,加入400 μL緩沖液FP1和6 μL的TNase,漩渦振蕩1 min,室溫放置10 min。然后加入130 μL緩沖液FP2,輕輕混勻,渦旋振蕩1 min。12 000 r·min-1離心5 min,將上清轉移至新的離心管中。向上清液中加入70%體積的異丙醇,充分混勻,12 000 r·min-1離心2 min,棄上清,保留沉淀。加入500 μL 70%乙醇洗滌DNA,12 000 r·min-1離心2 min,棄上清,再次用500 μL 70%乙醇洗滌一次,步驟與第一次相同。晾干后加入TE溶解DNA備用。
1.2.2 ?PLUG引物與IT引物的PCR擴增及電泳分析
397對PLUG引物根據水稻基因序列設計,染色體第一到第七同源群引物分別為51、55、63、54、61、59、54對,引物序列及PCR擴增程序參照文獻[11],PLUG引物由成都瑞信生物公司合成。841對IT引物根據簇毛麥基因內含子區多態性設計[9,14],染色體第一到第七同源群引物分別為135、175、120、89、140、71、111對,擴增程序參照文獻[9]。IT引物由青島擎科天成生物技術有限公司合成。
PLUG-PCR和IT-PCR產物在2%的瓊脂糖凝膠中電泳,電泳后凝膠在紫外凝膠成像儀GDS-Gel Dol 2000下掃描照相。用凝膠成像系統的Quality One軟件進行條帶統計,同時人工統計條帶與軟件統計結果相互核實印證。
2 結果與分析
2.1 PLUG引物和IT引物的擴增結果
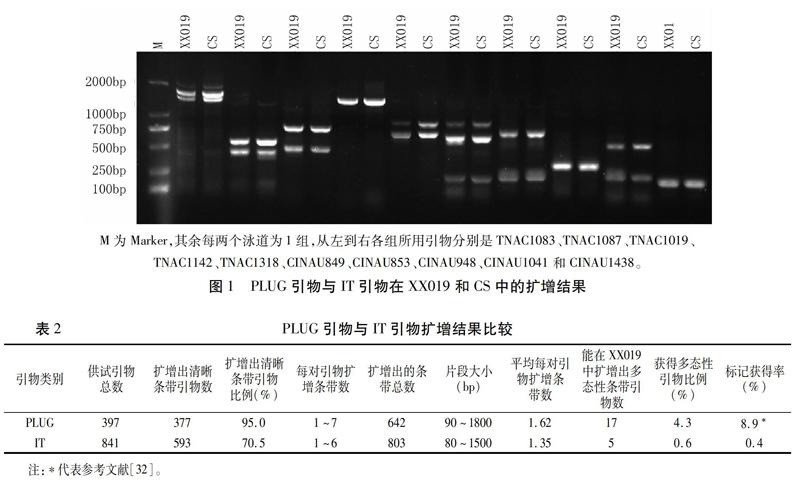
用397對PLUG引物對小麥-頂芒山羊草雙二倍體和中國春基因組DNA進行PCR擴增,結果顯示,377對(95.0%)引物可以在供試材料中擴增出清晰條帶,其中,引物TNAC1083、TNAC1087、TNAC1019、TNAC1142和TNAC1318的擴增效果如圖1所示。每對PLUG引物的擴增條帶數1~7條不等,片段大小90~1 800 bp,共擴增出642條清晰條帶,平均擴增條數為1.62條(表2)。
用841對IT引物對小麥-頂芒山羊草雙二倍體和中國春基因組DNA進行PCR擴增,結果顯示, 593對(70.5%)引物能在供試物種中擴增出清晰條帶,其中,引物CINAU849、CINAU853、CINAU948、CINAU1041和CINAU1438的擴增效果如圖1所示。每對IT引物的擴增條帶數1~6條不等,片段大小80~1 500 bp,共擴增出803條清晰條帶,平均擴增條數為1.35條(表2)。
2.2 頂芒山羊草特異PLUG和IT片段
能在供試材料中擴增出清晰條帶的377對PLUG引物中,相比對照小麥中國春、濟麥23、濟麥44、濟麥229和濟麥262,TNAC1017、TNAC1204和TNAC1364等17對引物能在小麥-頂芒山羊草雙二倍體中擴增出多態性條帶,其中,引物TNAC1296和TNAC1920各擴增出2個多態性條帶,其余引物均擴增出1個多態性條帶,共計獲得19個頂芒山羊草的特異性片段,獲得多態性引物比例為4.3%(17/397)。所獲19個多態性片段大小220~1 400 bp不等,目前這些多態性標記已經被應用于小麥-頂芒山羊草雜交種質的鑒定中[15]。
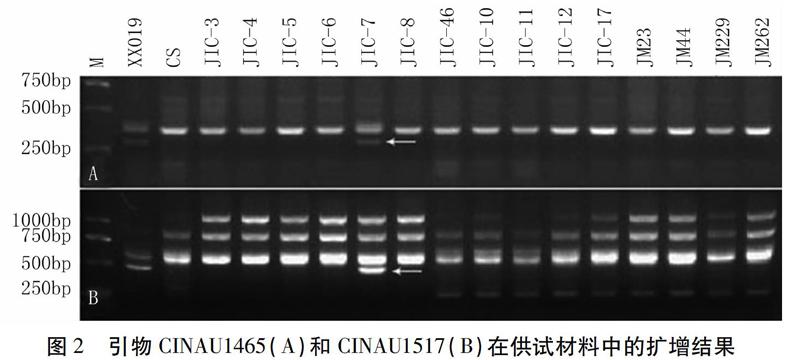
能在供試材料中擴增出清晰條帶的593對IT引物中,相比中國春、濟麥23、濟麥44、濟麥229和濟麥262,CINAU883、CINAU1060、CINAU1067、CINAU1465和CINAU1517共5對引物能在小麥-頂芒山羊草雙二倍體中擴增出多態性條帶,5對引物分別擴增出1個多態性條帶,共獲得5個頂芒山羊草的特異性片段,獲得多態性引物比例為0.6%(5/841)。其中,CINAU1465和CINAU1517的擴增結果如圖2所示。
2.3 頂芒山羊草染色體特異IT分子標記的建立
因為頂芒山羊草的M染色體組是卵穗山羊草Mg染色體組的供體[16],所以本研究同時利用小麥-頂芒山羊草2M-7M附加系和小麥-卵穗山羊草1Mg-7Mg附加系對上述多態性片段外進行染色體定位。利用引物CINAU883、CINAU1060、CINAU1067、CINAU1465和CINAU1517對上述兩套附加系進行擴增,結果發現,根據簇毛麥2VL上基因序列設計的引物CINAU1060僅能在頂芒山羊草2M和卵穗山羊草2Mg染色體擴增出片段長度約為300 bp的目標片段(表3);根據簇毛麥5VS上基因序列設計的引物CINAU1465僅能在頂芒山羊草6M(表3,圖2A)和卵穗山羊草5Mg染色體擴增出片段長度約為480 bp的目標片段(表3);根據簇毛麥6VL上基因序列設計的引物CINAU1517僅能在頂芒山羊草6M(表3,圖2B)和卵穗山羊草5Mg染色體擴增出片段長度約為320 bp的目標片段(表3)。
本研究篩選了841對IT引物,獲得頂芒山羊草2M染色體特異標記1個和6M染色體標記2個(表3),獲得標記比例約為0.4%(3/841),遠低于我們之前利用PLUG引物建立頂芒山羊草染色體特異標記8.9%的比例(表2)。本研究中,CINAU1465和CINAU1517均分別能在頂芒山羊草6M和卵穗山羊草5Mg染色體上擴增出特異目標多態性片段,這表明頂芒山羊草和卵穗山羊草染色體可能存在5/6重組。
3 討論
3.1 水稻PLUG引物在小麥族不同物種中的通用性及分子標記建立中的應用
Ishikawa等[8,16]最早利用水稻單拷貝基因序列開發設計獲得PLUG引物,用于有效分選基因產物,是大規模標記開發的有效工具。其后,Li等[17]建立了長穗偃麥草染色體第七同源群的PLUG標記,用于鑒定小麥-長穗偃麥草附加系材料1—27,分子標記獲得比例為5.4%。Li等[18]建立了帝國黑麥染色體特異PLUG標記79個, 分子標記獲得比例為54.9%。Lang[10]和Hu[19]等分別篩選PLUG引物建立了中間偃麥草特異PLUG標記,用于鑒定小麥-中間偃麥草雜交種質,分子標記獲得比例分別為82.0%和84.9%。Li[20]和Liu[21]等分別建立了多年生簇毛麥染色體不同同源群PLUG標記用于鑒定小麥-多年生簇毛麥染色體系,分子標記獲得比例41.7%和10.7%。Liu等[22]建立了無芒山羊草染色體特異PLUG標記,分子標記獲得比例為6.9%。上述實例表明,PLUG引物在小麥族不同物種中的通用性及分子標記建立獲得率相差較大,上述物種中,PLUG引物在黑麥和偃麥草標記建立方面優于其他物種,且對同一物種而言隨著PLUG引物對用量的多少其建立物種分子標記的獲得比例也可能發生變化。因為本研究發現,供試的397對PLUG引物中,有17對可以用于建立頂芒山羊草染色體特異分子標記,獲得多態性比例僅為4.3%,而篩選更多量的PLUG引物(526對)建立頂芒山羊草分子標記的比例反而增加到了8.9%,這可能是因為水稻PLUG與小麥族物種不同染色體同源群EST同源性有所差異造成的。
3.2 簇毛麥IT引物在頂芒山羊草中的通用性及分子標記建立中的應用
基于基因內含子序列開發IT引物并建立物種分子標記的工作已經在馬鈴薯和龍葵[23]、豌豆[24]、高粱[25]和燕麥[26]等物種中報道。然而,在小麥野生近緣種中卻鮮有報道。近年來,小麥野生近緣種山羊草IT引物[27] 、簇毛麥IT引物[9,14]已逐漸被開發出來。Sheikh等[27]開發了135對IT引物,建立了粘果山羊草和易變山羊草染色體特異標記29個,標記獲得率為21.5%。Zhang[9]等利用開發的1 624對IT引物建立了簇毛麥染色體特異標記841個,標記獲得率為51.8%。Wang[14]等利用流式細胞儀對簇毛麥4VS染色體臂進行分揀并測序,根據測序結果開發了IT引物359對,建立了簇毛麥4VS特異性標記232個,標記獲得率為64.62%。本研究利用上述簇毛麥的841對IT引物對小麥對照和小麥-頂芒山羊草雙二倍體等物種進行擴增,結果發現,這批IT引物能在供試物種中擴增出清晰條帶的占70.5%,然而,僅3對可以用于建立頂芒山羊草染色體特異IT標記,標記獲得率僅為0.4%,這可能是因為本研究所用瓊脂糖凝膠的分辨率遠低于Zhang[9]和Wang[14]等所用聚丙烯酰胺凝膠所致。目前,我們正在開展利用聚丙烯酰胺凝膠建立頂芒山羊草染色體特異IT標記工作。
3.3 頂芒山羊草特異標記的建立
對不同引物進行物種間通用性分析,不僅可以用于評價物種間親緣關系[27],還可用于物種分子標記的建立[15-21]。在頂芒山羊草特異標記建立方面,Teoh等[28]通過C帶技術獲得不同來源頂芒山羊草的標準核型圖,即建立了頂芒山羊草細胞遺傳標記。Wang等[29]從頂芒山羊草中發現一類新的對小麥面團品質具有正面效應的低分子麥谷蛋白基因并開發了分子標記。翁躍進等[30-31]建立了頂芒山羊草M染色體組RFLP標記40個,并將其應用于小麥-頂芒山羊草4M(4D)異代換系的鑒定。Liu等利用水稻PLUG引物對頂芒山羊草染色體組分子標記進行開發,獲得頂芒山羊草染色體特異標記44個[32]。本研究首次利用IT引物對頂芒山羊草進行擴增,獲得頂芒山羊草新型IT分子標記3個,為檢測小麥背景中頂芒山羊草染色質提供了新的檢測方法。
4 結論
PLUG引物在頂芒山羊草中的通用性優于IT引物;頂芒山羊草染色體特異IT標記CINAU1060、CINAU1465和CINAU1517的建立為小麥背景中頂芒山羊草染色質的鑒定提供了新的檢測手段。
參 考 文 獻:
[1] Mcintosh R A, Miller T E, Chapman V.Cytogenetical studies in wheat Ⅻ. Lr28 for resistance to Puccinia recondita and Sr34 for resistance to Puccinia graminis tritici[J].Zeitschrift Pflanzenzuhtung, 1982, 89(4):295-306.
[2] Riley R, Chapman V, Johnson R. Introduction of yellow rust resistance of Aegilops comosa in wheat by genetically induced homoeologous recombination[J]. Nature, 1968, 217(5126):383-384.
[3] 劉成, 宮文萍, 李光蓉, 等.小麥-無芒山羊草創新種質資源的鑒定[C]∥中國作物學會.第六屆全國小麥基因組學及分子育種大會論文集.2015:164.
[4] Eser V. Characterisation of powdery mildew resistant lines derived from crosses between Triticum aestivum and Aegilops speltoides and Ae.mutica[J]. Euphytica, 1998, 100:269-272.
[5] 蔣華仁, 戴大慶, 孫東發. 小麥特異種質資源的創新研究[J]. 四川農業大學學報, 1992, 10(2):255-259.
[6] Gong W P, Li G R, Zhou J P, et al. Cytogenetic and molecular markers for detecting Aegilops uniaristata chromosomes in a wheat background[J]. Genome, 2014, 57(9):489-497.
[7] 宮文萍, 韓冉, 宋健民, 等. 頂芒和無芒山羊草育種價值及細胞學標記[J]. 核農學報, 2017, 31(10):1889-1895.
[8] Ishikawa G, Nakamura T, Ashida T, et al. Localization of anchor loci representing five hundred annotated rice genes to wheat chromosomes using PLUG markers[J]. Theoretical and Applied Genetics, 2009, 118(3):499-514.
[9] Zhang X D, Wei X, Xiao J, et al. Whole genome development of intron targeting (IT) markers specific for Dasypyrum villosum chromosomes based on next-generation sequencing technology[J]. Molecular Breeding, 2017, 37:115.
[10]Lang T, La S X, Li B, et al. Precise identification of wheat-Thinopyrum intermedium translocation chromosomes carrying resistance to wheat stripe rust in line Z4 and its derived progenies[J]. Genome, 2018, 61(3):177-185.
[11]Lei M P,Li G R,Zhang S F,et al.Molecular cytogenetic characterization of a new wheat Secale africanum 2Ra(2D) substitution line for resistance to stripe rust[J].Journal of Genetics,2011,90(2):283-287.
[12]Zhan H X, Li G R, Chang Z J, et al. Molecular identification of a new wheat-Thinopyrum intermedium cryptic translocation line for resistance to powdery mildew[J]. IJBBB,2013, 4(3):376-378.
[13]Hu L J, Li G R, Zeng Z X, et al. Molecular cytogenetic identification of a new wheat-Thinopyrum substitution line with stripe rust resistance[J]. Euphytica, 2011, 177(2):169-177.
[14]Wang H Y, Dai K L, Xiao J, et al. Development of intron targeting (IT) markers specific for chromosome arm 4VS of Haynaldia villosa by chromosome sorting and next-generation sequencing[J]. BMC Genomics, 2017, 18:167-175.
[15]Schneider A, Molnar I, Molnar L M. Utilisation of Aegilops (goatgrass) species to widen the genetic diversity of cultivated wheat[J]. Euphytica, 2008, 163(1):1-19.
[16]Ishikawa G, Yonemaru J, Saito M, et al. PCR-based landmark unique gene (PLUG) markers effectively assign homoeologous wheat genes to A, B and D genomes[J]. BMC Genomics, 2007, 8(1):135.
[17]Li X J,Hu X G,Hu T Z,et al.Identification of a novel wheat-Thinopyrum ponticum addition line revealed with cytology, SSR, EST-SSR, EST-STS and PLUG markers[J]. Cereal Research Communications, 2015, 43(4):1-10.
[18]Li J J, Endo T R, Saito M, et al. Homoeologous relationship of rye chromosome arms as detected with wheat PLUG markers[J]. Chromosoma, 2013, 122(6):555-564.
[19]Hu L J, Li G R, Zhan H X, et al. New St-chromosome specific molecular markers for identifying wheat-Thinopyrum intermedium ?derivative lines[J]. J.Genet., 2012, 91:e69-e74.
[20]Li G R, Zhao J M, Li D H, et al. A novel wheat-Dasypyrum breviaristatum substitution line with stripe rust resistance[J]. Cytogenetic and Genome Research, 2014, 143(4):280-287.
[21]Liu C, Li G R, Yan H F, et al. Molecular and cytogenetic identification of new wheat-Dasypyrum breviaristatum additions conferring resistance to stem rust and powdery mildew[J]. Breeding Science, 2011, 61(4):366-372.
[22]Liu C, Li G R, Gong W P, et al. Molecular and cytogenetic characterization of a powdery mildew-resistant wheat-Aegilops mutica partial amphiploid and addition line[J]. Cytogenetic and Genome Research, 2016, 147(2/3):186-194.
[23]Poczai P, Cernak I, Gorji A M, et al. Development of intron targeting (IT) markers for potato and cross-species amplification in Solanum nigrum (Solanaceae) [J]. American Journal of Botany, 2010, 97(12):e142-e145.
[24]Jain S L, Weeden N F, Portwe L D, et al. Finding linked markers to En for efficient selection of pea enation mosaic virus resistance in pea[J]. Crop Science, 2013, 53(6):2392-2399.
[25]Jaikishan I, Rajendrakumar P, Madhusudhana R,et al. Development and utility of PCR-based intron polymorphism markers in sorghum [Sorghum bicolor (L.) Moench][J]. Journal of Crop Science and Biotechnology, 2015, 18(5):309-318.
[26]Holland J B, Hekkand S J, Sharopova N, et al. Polymorphism of PCR-based markers targeting exons, introns, promoter regions, and SSRs in maize and introns and repeat sequences in oat[J]. Genome, 2001, 44(6):1065-1076.
[27]Sheikh I, Sharma P, Verma S K, et al. Development of intron targeted amplified polymorphic markers of metal homeostasis genes for monitoring their introgression from Aegilops species to wheat[J]. Molecular Breeding, 2018, 38(4):47.
[28]Teoh S B,Miller T E,Readet S M.Intraspecific variation in C-banded chromosomes of Aegilops comosa and Aegilops speltoides[J]. Theoretical and Applied Genetics, 1983, 65(4):343-348.
[29]Wang K, Gao L Y, Wang S L, et al. Phylogenetic relationship of a new class of LMW-GS genes in the M genome of Aegilops comosa[J]. Theoretical and Applied Genetics, 2011, 122(7):1411-1425.
[30]翁躍進, 賈繼増, 董玉琛. 小麥M染色體組的RFLP標記[J]. 農業生物技術學報,1997(3):211-215.
[31]翁躍進, 賈繼增, 董玉琛. 利用RFLP分子標記鑒定小麥-頂芒山羊草異代換系[J]. 遺傳學報, 1997, 24(3):248-254.
[32]Liu C, Gong W P, Han R, et al. Characterization, identification and evaluation of a set of wheat-Aegilops comosa chromosome lines[J]. Scientific Reports, 2019, 9(1):4773.

