青藏高原地區山生柳遺傳多樣性研究
吳錦 楊茜 王久利 高慶波 張發起 陳世龍


摘要:? 利用葉綠體非編碼區片段研究分布于青藏高原地區的山生柳居群遺傳多樣性,對未來山生柳生態環境和青藏高原地區物種豐富度的保護具有指導意義。該研究設計并篩選出cpDNA引物5′trnG2G-3′trnG(UUC)和5′rpS12-rpL20,用擴增出的片段和對應的聯合片段進行后續的遺傳多樣性分析。結果表明:通過山生柳的聯合片段檢測到35種單倍型,單倍型多態性0.626,核苷酸多態性0.000 85。中性檢驗Tajima’s D(-2.286 70, P<0.01)和Fu’s Fs(-5.298 05, P<0.02)都是顯著負值,推測山生柳個體數近期經歷過擴張。AMOVA分析顯示,居群內和居群間遺傳變異分別為93.70%和6.30%,表明居群內的變異是山生柳遺傳變異的主要來源。居群間遺傳分化程度中等偏低(FST=0.063),基因流(Nm)為7.439,說明山生柳各居群的基因交流非常頻繁,不同地理居群間存在一定的基因流動。遺傳分化系數NST(0.075)大于GST(0.068)和基于遺傳距離和單倍型的UPGMA聚類分析,表明山生柳12個居群分為4組且與居群的地理分布沒有明顯相關性。山生柳是進行有性繁殖還是無性繁殖主要受環境因素的影響,居群內變異是山生柳遺傳變異性的主要來源,居群間基因交流頻繁。
關鍵詞: 山生柳, 基因流, 青藏高原, 葉綠體DNA, 遺傳多樣性
中圖分類號:?; Q943文獻標識碼:? A文章編號:? 1000-3142(2019)09-1180-11
Abstract:? We studied the genetic diversity of Salix oritrepha in the Qinghai-Tibetan Plateau using the non-coding region of chloroplast DNA fragments. The study will have? guiding significance for the protection of ecological environment and animal husbandry of S. oritrepha and species richness in Qinghai-Tibet Plateau in the future. We designed two useful primers 5′trnG2G-3′trnG (UUC) and 5′rpS12-rpL20. The amplified fragments and their combined fragments were used for subsequent genetic diversity analysis of S. oritrepha. The results showed that we detected 35 haplotypes by the combined sequences, haplotype diversity (Hd) was 0.626, nucleotide diversity (Pi) was 0.000 85. Neutrality tests of Tajima’s D (-2.286 70, P<0.01) and Fus Fs (-5.298 05, P<0.02) showed significantly negative values, which meant that S. oritrepha had expansion recently of its individual numbers. Analysis of molecular variance showed that within-population variance was described as 93.70% of the total variance, while among-population variance was 6.30%, which meant that within-population variance was the main resource of genetic variation of S. oritrepha. The fi-xation index (FST) was 0.063 and number of migrants (Nm) was 7.439, which meant that gene flow was very frequent among different geographical populations. An estimation of non-signification NST (0.075) was bigger than gene differentiation coefficient GST (0.068). UPGMA tree based on genetic distance and haplotypes suggested that all populations were divided into four groups and had no significant correlation with the geographical distribution of the population. The mode of reproduction (sexual or asexual) of S. oritrepha mainly depended on environmental factors. The genetic variability mainly came from within-population and it has a frequently gene flow among populations.
Key words:? Salix oritrepha, gene flow, Qinghai-Tibetan Plateau, chloroplast DNA, genetic diversity
山生柳(Salix oritrepha)是中國特有物種,落葉矮小灌木,高10~120 cm,藏藥中稱朗瑪,生于海拔3 000~4 700 m的高山地帶,繁衍方式以有性生殖為主,無性繁殖為輔,是青藏高原地區高寒柳灌叢的建群種和優勢種,具有極高的藥用價值,對生態環境的保護也尤為主要。一方面對山生柳化學成分的研究,證明其可解熱、消炎止痛、治療婦科疾病和提高機體免疫力(封士蘭等,2001);另一方面山生柳是深根性灌木,根深長度達到植株縱向高度的4 ~ 5倍,故可以抵抗干旱和低溫。由于耐高鹽高堿且生長繁殖快速,故能涵養水源、治沙和防沙,使其起到河岸防護、水土保持的作用(陳文業等,2008)。常與高山繡線菊(Spiraea alpina)、金露梅(Potentilla fruticosa)、沙棘(Hippophae rhamnoides)、窄葉鮮卑花(Sibiraea angustata)、大苞柳(Salix pseudospissa)等組成高寒灌叢(王芳等,2010),是藏民牲畜的夏季放牧地。
遺傳多樣性分析是評價和保護瀕危物種的主要指標(Cires et al., 2011),種內遺傳多樣性越豐富,物種對環境的適應能力就越強,保護了物種和生態系統的多樣化,也能減慢因為適應、進化所致的滅亡進程。近幾十年來,對青藏高原地區植被的遺傳多樣性的研究越來越深入(Duan et al., 2011; Khan et al., 2014),這些都對保護青藏高原地區植被豐富度具有重要的指導意義。研究植物遺傳多樣性的方法中,葉綠體基因組DNA被廣泛使用(Demesure et al., 1996;Rungis et al., 2017;Wu, 2016)。葉綠體DNA的結構緊湊簡單、分子量小,可從分子角度作比較精準的分析,其比核DNA存在更加穩定、進化更加緩慢且編碼序列也保守,故廣泛應用于植物遺傳多樣性的剖析研究(Shaw et al., 2014)。葉綠體DNA在大多數被子植物中為母系遺傳,只通過種子進行遺傳,不受花粉遺傳的影響(Mogensen, 1996)。
目前,對高寒山生柳灌叢的群落特征、在畜牧業中的作用以及在防止沙漠化中起到的作用都有研究(Kang et al., 2015;王緯和王盛,2015;楊路存,2013),但對山生柳遺傳多樣性的研究較少,郭敏等(2012)的研究表明山生柳種群具有較高的遺傳多樣性且隨著海拔梯度的升高而呈現升高趨勢,且遺傳變異主要存在于居群內,因其所研究的地理分布范圍僅限祁連山,不能準確分析出山生柳遺傳結構及種群動態。本研究采集了青藏高原大部分地區的山生柳,設計并篩選出兩對特異性針對山生柳的cpDNA引物,用葉綠體非編碼區的聯合片段對山生柳進行遺傳多樣性研究,將對山生柳生殖系統的研究、種質資源的保護和生態環境的保護都有重要意義。
1材料與方法
1.1 材料
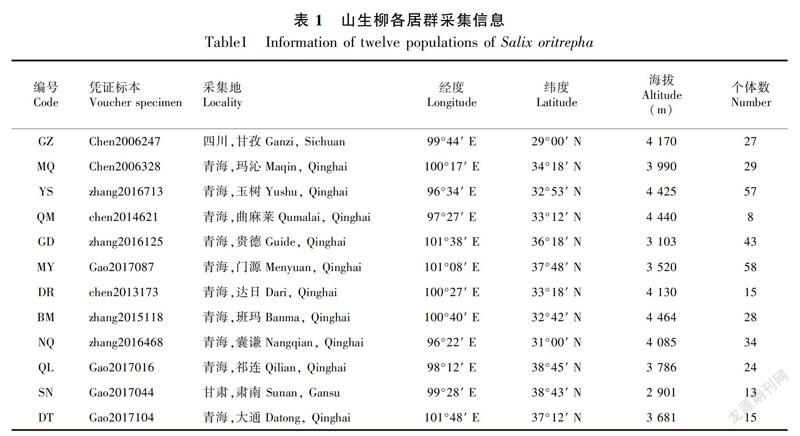
2006—2017年對分布于青藏高原地區的山生柳進行廣泛的野外采集。對每個野外自然居群選取8~58個個體,個體間距超過100 m,共收集山生柳12個居群351個個體(表1)。采集山生柳生長良好的新鮮幼嫩葉片,迅速用硅膠進行干燥,之后放于-20 ℃ 長期保存。所采集的憑證標本存放于中國科學院西北高原生物研究所青藏高原生物標本館(HNWP)中。
1.2 DNA提取與PCR擴增
選取改良的CTAB法從干燥處理的山生柳嫩葉中提取基因組DNA(Porebski et al., 1997)。參照序列選取已上傳到NCBI上的Salix arbutifolia、S. babylonica、S. interior、S. oreinoma、S. purpurea、S. suchowensis和S. tetrasperma等柳屬植物葉綠體基因組序列,引物片段的選取參考之前的研究(Hamilton, 1999; Scarcelli et al., 2011; Shaw et al., 2005),用軟件Primer Premier 5.0(Singh et al., 1998)、BioXM 2.6(黃驥和張紅生,2004)和SnapGene 3.2.1(McKeone et al., 2014)進行引物設計,共設計了21對引物用于PCR擴增。擴增反應體系為25 μL:2.5 μL的10×PCR Buffer(含1.5 mmol·L-1 MgCl2)、0.5 μL的10 mmol·L-1 dNTP、正反引物各0.5 μL(10 pmol·L-1)、1個單位的Taq DNA酶(TaKaRa,大連)、15~50 ng的模板DNA,去離子水補齊到25 μL。PCR反應擴增程序:94 ℃預變性3 min;94 ℃變性45 s;60 ℃退火45 s;72 ℃延伸30 s;35個循環;72 ℃延伸5 min;4 ℃保存。PCR反應產物送至生物工程(上海)股份有限公司測序。將獲得21對引物在12個居群24個個體中進行PCR擴增預試驗,篩選出獲得兩對遺傳多態性較高的引物 [5′trnG2G-3′trnG(UUC)和5′rpS12-rpL20]用于所有個體的擴增測序(表2)。
1.3 數據分析
運用Chromas軟件(http://technelysium.com.au/wp/chromas/)和MEGA 7.0.26(Kumar et al., 2018)對所有測序序列進行人工校對,并用MEGA軟件對校對好的所有個體的擴增片段進行對位排列。利用DnaSP 6.10.04(Rozas et al., 2017)軟件確定山生柳單倍型,并統計中性檢驗中的Tajima’s D和Fu’s Fs。運用SPSS(George & Mallery, 2003)軟件獲得基于遺傳距離和單倍型分布的UPGMA聚類分析圖。利用Arlequin軟件包(Excoffier & Lischer, 2010)中的分子變異分析方法(analysis of molecular variance, AMOVA)檢測居群內和居群間的遺傳變異水平,并對單倍型分布的FST進行評價(1 000次重復置換檢測)。根據基因流(Nm)與遺傳分化系數(FST)之間的關系計算Nm值和居群平均等位基因數。失配分布(mismatch distribution)分析也在Arlequin中完成。通過PERMUT軟件計算居群內平均遺傳多樣性(average gene diversity within populations,hS)、總遺傳多樣性(total gene diversity,hT)、居群間遺傳分化系數(GST)和(NST),并用U-統計方法對GST和NST進行比較(1 000次重復置換檢測)以檢測山生柳單倍型的分布特點(Caraux & Pinloche, 2005)。利用TCS軟件(Clement et al., 2000)來建立山生柳單倍型之間的TCS簡約網絡關系圖。
2結果與分析
2.1 山生柳遺傳多樣性分析
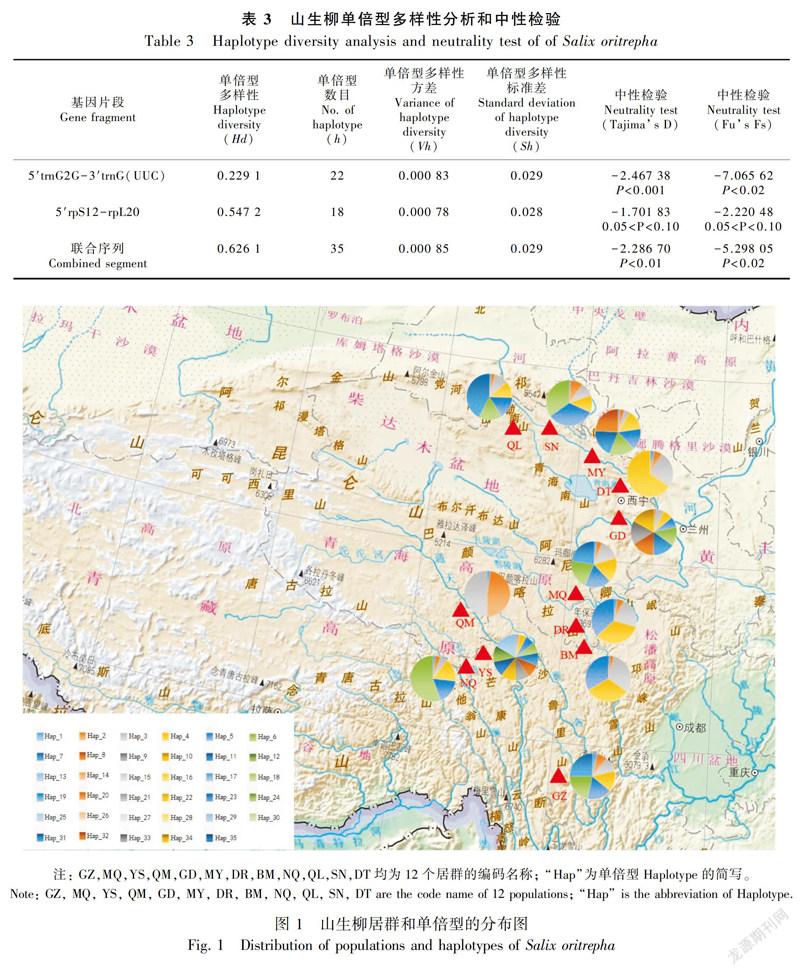
對351個個體的擴增產物進行單向測序,利用MEGA軟件對位排列且刪除poly結構后,5′trnG2G-3′trnG(UUC)和5′rpS12-rpL20片段矩陣長度分別為481 bp、730 bp,共檢測到36個變異位點,其中17個為簡約性信息位點,變異率為2.97%。從山生柳的聯合片段中檢測出35個單倍型(Haplotype, H01~H35),將5′trnG2G-3′trnG(UUC)片段的單倍型序列和5′rpS12-rpL20片段的單倍型序列提交至GeneBank(MK602662~MK602696和MK606373~MK606407)。聯合片段的單倍型多樣性為0.626 1,單倍型多樣性標準差為0.029(表3)。各居群聯合片段之間的多態性表現不同,居群的平均等位基因數(Na)依次為1.067、1.078、1.278、1.022、1.656、1.089、1.067、1.100、1.056、1.078、1.089、1.033、1.134。遺傳多樣性最豐富的是居群YS,其具有最高的核苷酸多樣性(0.001 45)、平均核苷酸差異(1.722)和最多的單倍型數目(13)(表4)。
由各居群的單倍型分布可知,僅H01被全部12個居群所共享;H04、H05、H06和H07是GZ所擁有的特有單倍型;H11是MQ所擁有的特有單倍型;H17、H18、H19、H20、H21和H32是YS所擁有的特有單倍型;H27、H28、H29和H30是GD所擁有的特有單倍型;H16是MY所擁有的特有單倍型;H15是DR所擁有的特有單倍型;H24、H25和H26是BM所擁有的特有單倍型;H31是NQ所擁有的特有單倍型;H33是QL所擁有的特有單倍型;H34和H35是SN所擁有的特有單倍型;H02,H03,H08,H09,H10,H12,H13,H14,H16,H17,H22,H23是2~10個居群所共有的單倍型(圖1,表4)。
2.2 山生柳遺傳結構分析
分子變異分析(AMOVA)結果表明,居群間遺傳變異為6.30%,居群內遺傳變異為93.70%(P<0.01),說明居群內遺傳變異為山生柳遺傳變異的主要來源(表5)。遺傳分化系數FST=0.062 98,居群間遺傳分化很小。根據基因流Nm與FST之間的關系(Kirk & Freeland, 2011),Nm =(1-FST)/2FST,得到Nm為7.439, 基因流很強。用PERMUT和DnaSP軟件計算得出山生柳居群內平均遺傳多樣山生柳各居群間的遺傳距離大部分都較小,其中QM和NQ之間出現了最大遺傳距離0.977,YS和SN出現了最小遺傳距離0.000,大多數居群之間的遺傳距離差異性不大,總的平均遺傳距離為0.424;居群間的遺傳分化指數(FST)顯示有18個負值,占27.28%。其中QM和NQ之間出現了最大的FST=0.255 0(表6)。
山生柳12個居群的遺傳距離的范圍為0.000~1.234,表明山生柳居群內遺傳多樣性豐富。基于遺傳距離的UPGMA聚類分析,得到山生柳的12個居群分為I(YS, SN, MY, DT, QL, BM)、Ⅱ(GZ, MQ, DR)、Ⅲ(GD, NQ)和IV(QM)共四組(圖2)。基于單倍型的UPGMA聚類分析,得到12個居群分為I(YS, GD, MY)、II(NQ)、III(BM, GZ)和IV(QM, SN, QM, QL, DT, DR)共四組(圖3)。兩者均沒有表現出與12個居群的地理分布相對應的聚類關系。
2.3 山生柳種群動態
中性檢驗結果表明,山生柳各個引物片段和聯合片段的Tajima’s D(-2.286 70, P<0.01)和Fu’s Fs(-5.298 05, P<0.02)均為顯著的負值(表3)。失配分析結果顯示,在山生柳分布范圍內其分布曲線整體呈現單峰分布, 其SSD(sum of square devia-tions)值 (0.003 8, P=0.68)和HRag (Harpending’s raggedness index)值(0.047 9, P=0.72)均不顯著(P>0.05),這與中性檢驗的結果相一致(圖4)。
用山生柳葉綠體基因所檢測出的35個單倍型,建立單倍型多樣性的簡約網絡圖(圖5)。回路結構是趨同演化的結果,矩形框代表祖先(或根部)單倍型即H01(Saltonstall, 2002)。圖5中略去了單倍型的頻率數值。圖5中“o”表示理論分析出的,但沒有實際觀測到的單倍型。除YS(H32)和DG(H27,H29)的單倍型同其它單倍型關系較遠外,大部分單倍型之間僅有一到兩步突變,演化關系呈現星狀輻射結構(楊玉慧等,2004)。
3討論
3.1 山生柳的居群遺傳多樣性
篩選出的山生柳特異性條帶為山生柳無性系鑒定提供可操作性。山生柳的聯合片段共檢測到36個變異位點和17個簡約性的信息位點,插入缺失變異位點19個,占總變異的52.78%。插入缺失是山生柳變異位點出現的主要來源,與DNA在進行復制時存在高頻率的點突變有關。在殘酷的高山圖 4基于山生柳單倍型的失配分析山地冰雪寒溫帶生存環境中,山生柳的DNA復制發生堿基錯配的可能性更大,機體的自我修復功能會受到一定阻礙,說明惡劣的生存環境可能在一定程度上影響DNA復制過程(Baccarelli & Bollati, 2009; Chinnusamy & Zhu, 2009; Dubrova, 2003)。
對山生柳居群的聯合片段進行數據分析,檢測到35個單倍型。數據分析的結果總體呈現出較低的遺傳多樣性(Hd=0.626 1, Pi=0.000 85)。山生柳每個居群間的單倍型多樣性(Hd)表現不同,推測可能是由于在青藏高原地區的山生柳分布范圍廣,各居群所在的自然生存環境條件具有差異,經過長期不同的生態環境和自然選擇,會導致具有一定地理隔離的山生柳居群存在差異的遺傳多樣性。根據之前對山生柳在祁連山上不同海拔種群的遺傳多樣性水平的研究結論(郭敏等,2012),推測不同的地理位置、生存環境和海拔高度都體現山生柳不顯著的分子水平的差異性,但是差異性不會太大。
3.2 山生柳的居群遺傳結構
AMOVA分子變異結果表明,山生柳居群內的遺傳變異占總變異的93.70%(P<0.01),居群間的遺傳變異僅為6.30%,揭示了山生柳居群內的遺傳變異比居群間的遺傳變異要大,居群內的遺傳變異是山生柳遺傳變異的主要來源。推測的原因:(1)35個單倍型中僅有H01為12個居群都擁有的共享單倍型,共有208個個體擁有,所占比例為59.26%,由于H01在居群中是無規律分布,因此山生柳居群間的遺傳差異并不明顯;(2)35個單倍型中有22個單倍型是各個居群所獨有,甚至某一居群會同時固定多個特有單倍型,這就說明山生柳的某些居群內部會有可辨別的遺傳變異。特有單倍型比例較高的物種往往不能揭示出明顯的譜系地理格局(Geng et al., 2018)。繁育系統和遺傳變異分布之間存在著密切的聯系(Hogbin & Peakall, 1999),即異交物種的遺傳變異大多分布在群體之內,群體間的遺傳變異通常占27%以下(趙景奎等,2008),與本研究結果相符。
植物居群的遺傳結構受群體進化歷史、基因流等影響,群體進化歷史一般通過遺傳分化指數(FST)來反映,其大小可在一定程度上揭示種群間基因流和遺傳漂變的程度(楊艷婷等,2018)。一般來講,當FST∈[0,0.05),群體間沒有遺傳分化;當FST∈[0.05,0.15),群體分化程度中等;當FST∈[0.15,0.25),群體間高度分化;FST∈[0.25,1],群體間分化程度非常高(Wright,1950,1970)。山生柳自然居群的遺傳分化指數(FST)為0.062 98,表明居群分化程度為中等。山生柳各居群間的遺傳分化指數(FST)有19個負值,占28.79%(正常情況下FST∈[0,1]),表明少部分居群間沒有分化,但總體上反映出的是大部分居群間出現非常普遍的基因交流(Wright, 1950, 1970, 1982),其中QM和NQ之間的FST值最大為0.255 0。山生柳一般為有性生殖,花粉量大隨風飄散,但在嚴酷的生存環境下會進行無性繁殖,本研究所選的山生柳材料地理分布較廣、地理環境高度復雜,導致居群間遺傳變異的差異性較大,有的居群幾乎無遺傳分化(QM, DT),有的居群分化程度非常高(QL),但總體還是表現為分化程度中等偏低。
根據基因流公式,算得Nm=7.438,當Nm=7.438>1,屬于較高水平(Wright,1982),表明不同的居群之間存在頻繁的基因流動,增大群體間遺傳差異的表現一般為自然選擇以及遺傳漂變,而基因流可以“弱化”群體之間不易察覺的遺傳差異使居群間趨向統一。
對山生柳單倍型變異的地理結構檢驗結果為GST值(0.068)和NST值(0.075)(NST>GST,P>0.05),表明山生柳不同居群間出現了大量的親緣關系靠近的單倍型,可以證明山生柳不存在明顯的譜系地理學的關系。基于遺傳距離和單倍型的UPGMA聚類分析,得到山生柳的12個居群各分為四組且與12個居群地理分布沒有明顯相關性,進一步驗證了山生柳居群不具有譜系地理結構。
3.3 山生柳的種群擴張
基因流分析結果證實山生柳的12個居群存在頻繁的基因交流,山生柳居群間不存在明顯的基因流限制。通過對所有地理居群單倍型進行失配分析,所有的山生柳單倍型呈現只有一個明顯頂峰的單峰型曲線,表示不能拒絕居群擴張的假說。中性檢驗的結果表明,Tajima’s D (-2.286 70, P<0.01)和Fu’s Fs(-5.298 05, P<0.02)均為顯著的負值,這與失配分析的單峰型曲線結果相一致。TCS網絡圖顯示H01單倍型為祖先單倍型,由于H01為種群的共享單倍型,故不能確定山生柳種群的擴散中心。TCS網絡圖呈星狀輻射分布,且新衍生出的單倍型與原始單倍型組成一個“星狀”輻射結構,說明該群體曾經在遇到瓶頸后迅速擴增(Arbogast, 2000)。通過單倍型多樣性與核苷酸多樣性分析得到單倍型多樣性高(Hd>0.5)而核苷酸多樣性低(Pi<0.005),預示著群體曾經歷過瓶頸效應后,伴隨了迅速的種群擴張與變異的積累。綜上所述,推測山生柳在其分布范圍或者其個體數近期經歷過擴張。
對青藏高原地區相同生境的高山繡線菊(Khan et al., 2014)、金露梅(Ma et al., 2014)、沙棘(Bartish et al., 2006)、窄葉鮮卑花(Duan et al., 2011)等灌木的種群動態研究表明其遺傳分組曾經歷過快速的種群擴張事件。這與山生柳的種群擴張結果一致,推測物種生境和繁育方式的相似性會出現物種為了營養、繁殖、保護、擴散以及對環境的適應出現協同進化的現象(Morgan et al., 2005; Thompson & Burdon, 1992)。有研究表明山生柳種群可能以有性和無性兩種繁殖方式進行更新以鞏固其優勢地位(李毅等,2002),年齡結構分布圖呈正三角形,種群動態表現為增長型(李毅等,2002),這與本研究中居群近期出現過擴張相吻合。花粉和種子的擴散是自然植物種群最主要的基因流方式(李海生和陳桂珠,2004),故山生柳生殖方式的多樣化會導致其基因交流頻繁,預期未來山生柳居群會處于進一步擴張狀態。
山生柳頻繁的基因交流和經歷過近期擴張,猜測此結果可能的原因如下:(1)cpDNA是母系遺傳,估算的基因流大小與花粉傳播無關,與其多樣化的繁育方式有關;(2)山生柳一般以有性生殖為主,其生存環境是長期積雪(低溫)、干燥風大等氣象特點,使其種子可以隨處傳播;(3)由于種子的傳播距離遠,故青藏高原地區和橫斷山脈錯綜復雜地形也只能在很小的程度上阻隔山生柳居群間的基因交流;(4)在嚴酷環境壓力下,山生柳種子活力極低,幼苗補充量受限,從而有性生殖受限,此時主要依托無性繁殖來鞏固其物種數量的穩定(陳文業等,2008)。
參考文獻:
ARBOGAST BS, 2000. Phylogeography: The history and formation of species [J]. Integr Comp Biol, 41(1): 134-135.
BACCARELLI A, BOLLATI V, 2009. Epigenetics and environmental chemicals [J]. Curr Opin Pediatr, 21(2): 243-251.
BARTISH IV, KADEREIT JW, COMES HP, 2006. Late Quaternary history of Hippophae rhamnoides L. (Elaeagnaceae) inferred from chalcone synthase intron (Chsi) sequences and chloroplast DNA variation [J]. Mol Ecol, 15(13): 4065-4083.
CARAUX G, PINLOCHE S, 2005. PermutMatrix: A graphical environment to arrange gene expression profiles in optimal linear order [J]. Bioinformatics, 21(1): 1280-1281.
CHEN WY, CHEN YX, ZHENG HP, et al., 2008. Ecological habits and seedling raising techniques of Salix oritrepha [J]. Prot For Sci Technol, (3): 135-137.? [陳文業, 陳永霞, 鄭華平, 等, 2008. 山生柳的生態習性與育苗技術 [J]. 防護林科技, (3):135-137.]
CHINNUSAMY V, ZHU JK, 2009. Epigenetic regulation of stress responses in plants [J]. Curr Opin Plant Biol, 12(2): 133-139.
CIRES E, SAMAIN MS, GOETGHEBEUR P, et al., 2011. Genetic structure in peripheral Western European populations of the endangered species Cochlearia pyrenaica (Brassicaceae) [J]. Plant Syst Evol, 297(1-2): 75-85.
CLEMENT M, POSADA D, CRANDALL KA, 2000. TCS: A computer program to estimate gene genealogies [J]. Mol Ecol, 9(10): 1657-1659.
DEMESURE B, COMPS B, PETIT RJ, 1996. Chloroplast DNA phylogeography of the common beech (Fagus sylvatica L.) in Europe [J]. Evolution, 50(6): 2515.
DUAN YZ, GAO QB, ZHANG FQ, et al., 2011. Phylogeographic analysis of the endemic species Sibiraea angustata reveals a marginal refugium in the Qinghai-Tibet Plateau [J]. Nord J Bot, 29(5): 615-624.
DUBROVA YE, 2003. Radiation-induced transgenerational instability [J]. Oncogene, 22(45): 7087-7093.
EXCOFFIER L, LISCHER H, 2010. Arlequin suite ver 3.5: A new series of programs to perform population genetics analyses under Linux and Windows [J]. Mol Ecol Resour, 10(3): 564-567.
FENG SL, SU C, DUAN SY, et al., 2001. Chemical constituents from Salix oritrepha [J]. Chin J Chin Mat Med, (9): 31-32.? [封士蘭, 蘇策, 段生玉, 等, 2001. 藏藥山生柳的化學成分研究 [J]. 中國中藥雜志, (9):31-32.]
GENG JZM, LI Y, JIA LK, et al., 2018. Phylogeography of Saxifraga tangutica Engl. (Saxifragaceae)? [J]. Acta Bot Boreal-Occident Sin, 38(2): 370-380.? [更吉卓瑪, 李彥, 賈留坤, 等, 2018. 唐古特虎耳草譜系地理學研究 [J]. 西北植物學報, 38(2):370-380.]
GEORGE D, MALLERY P, 2003. SPSS for Windows step by step: A simple guide and reference, 16.0 update [J]. Comp Softw, (100): 357.
GUO M, MA YJ, LI Y, 2012. Analysis of genetic diversity in Salix oritrepha from different altitudes in Gansu Qilian mountains using SSR markers [J]. Acta Pratac Sin, 21(5): 114-121.? [郭敏, 馬彥軍, 李毅, 2012. 祁連山不同海拔梯度山生柳遺傳多樣性的SSR分析 [J]. 草業學報, 21(5):114-121.]
HAMILTON MB, 1999. Four primer pairs for the amplification of chloroplast intergenic regions with intraspecific variation [J]. Mol Ecol, 8(3): 521-523.
HOGBIN PM, PEAKALL R, 1999. Evaluation of the contribution of genetic research to the management of the endangered plant Zieria prostrata [J]. Conserv Biol, 13(3): 514-522.
HUANG Y, ZHANG HS, 2004. Development of nucleotide sequence analysis software based on Windows [J]. Chin J Bioinform, 2(1): 13-17. [黃驥, 張紅生, 2004. 基于Windows的核酸序列分析軟件的開發 [J]. 生物信息學, 2(1):13-17.]
KANG JJ, ZHAO WZ, ZHAO M, et al., 2015. The cutting reproduction technique of Salix oritrepha and its application on degraded grasslands restoration [J]. J Soil Water Conserv, 70(1): 45-53.
KHAN G, ZHANG FQ, GAO QB, et al., 2014. Molecular phylogeography and intraspecific divergence of Spiraea alpina (Rosaceae) distributed in the Qinghai-Tibetan Plateau and adjacent regions inferred from nrDNA [J]. Biochem Syst Ecol, 57: 278-286.
KIRK H, FREELAND J R, 2011. Applications and implications of neutral versus non-neutral markers in molecular ecology [J]. Int J Mol Sci, 12(6): 3966-3988.
KUMAR S, STECHER G, LI M, et al., 2018. MEGA X: Molecular evolutionary genetics analysis across computing platforms [J]. Mol Biol Evol, 35(6): 1547-1549.
LI HS, CHEN GZ, 2004. Genetic diversity of mangrove plant Sonneratia caseolaris in Hainan Island based on ISSR analysis [J]. Acta Ecol? Sin, 24(8): 1657-1663.? [李海生, 陳桂珠, 2004. 海南島紅樹植物海桑遺傳多樣性的ISSR分析 [J]. 生態學報, 24(8):1657-1663.]
LI Y, HU ZZ, WANG ZT, 2002. Populations distribution patterns of alpine Salix Oritrepha in East Qilian Mountains [J]. Acta Pratac Sin, (3): 48-54.? [李毅, 胡自治, 王志泰, 2002. 東祁連山高寒地區山生柳種群分布格局研究 [J]. 草業學報, (3):48-54.]
MA YZ, LI ZH, WANG X, et al., 2014. Phylogeography of the genus Dasiphora (Rosaceae) in the Qinghai-Tibetan Plateau: Divergence blurred by expansion [J]. Biol J Linn Soc, 111(4): 777-788.
MCKEONE R, WIKSTROM M, KIEL C, et al., 2014. Assessing the correlation between mutant rhodopsin stability and the severity of retinitis pigmentosa [J]. Mol Vis, 20: 183-199.
MOGENSEN HL, 1996. Invited special paper: The hows and whys of cytoplasmic inheritance in seed plants [J]. Am J Bot, 83(3): 383-404.
MORGAN MT, WILSON WG, KNIGHT TM, 2005. Plant population dynamics, pollinator foraging, and the selection of self-fertilization [J]. Am Nat, 166(2): 169-183.
POREBSKI S, BAILEY LG, BAUM BR, 1997. Modification of a CTAB DNA extraction protocol for plants containing high polysaccharide and polyphenol components [J]. Plant Mol Biol Rep, 15(1): 8-15.
ROZAS J, FERRER-MATA A, SANCHEZ-DELBARRIO JC, et al., 2017. DnaSP 6: DNA sequence polymorphism analysis of large data sets [J]. Mol Biol Evol, 34(12): 3299-3302.
RUNGIS D, LAIVINS M, GAILITE A, et al., 2017. Genetic analysis of Latvian Salix alba L. and hybrid populations using nuclear and chloroplast DNA markers [J]. iForest, 10(2): 422-429.
SALTONSTALL K, 2002. Cryptic invasion by a non-native genotype of the common reed, Phragmites australis, into North America [J]. P Natl Acad Sci USA, 99(4): 2445-2449.
SCARCELLI N, BARNAUD A, EISERHARDT W, et al., 2011. A set of 100 chloroplast DNA primer pairs to study population genetics and phylogeny in monocotyledons [J]. PLoS ONE, 6(5): e19954.
SHAW J, LICKEY EB, BECK JT, et al., 2005. The tortoise and the hare II: Relative utility of 21 noncoding chloroplast DNA sequences for phylogenetic analysis [J]. Am J Bot, 92(1): 142.
SHAW J, SHAFER HL, LEONARD OR, et al., 2014. Chloroplast DNA sequence utility for the lowest phylogenetic and phylogeographic inferences in angiosperms: The tortoise and the hare IV [J]. Am J Bot, 101(11): 1987-2004.
SINGH VK, MANGALAM AK, DWIVEDI S, et al., 1998. Primer premier: Program for design of degenerate primers from a protein sequence [J]. Biotechniques, 24(2): 318-319.
THOMPSON JN, BURDON JJ, 1992. Gene-for-gene coevolution between plants and parasites [J]. Nature, 360(6400): 121-125.
WANG F, CHEN WY, CAI RZM, et al., 2010. Gray system theory based analysis of hard branch cuttage of Salix oritrepha in alpine area [J]. Pratac Sci, (9): 86-90.? [王芳, 陳文業, 才讓卓瑪, 等, 2010. 高寒區山生柳硬枝扦插灰色系統理論分析研究 [J]. 草業科學, (9):86-90.]
WANG W, WANG S, 2015. Analysis of plant community diversity of Salix oritrepha shrub meadow in Maqu County [J]. For Gansu, (6): 28-30.? [王瑋, 王盛, 2015. 瑪曲縣山生柳灌叢草甸植物群落多樣性分析 [J]. 甘肅林業, (6):28-30.]
WRIGHT S, 1950. Genetic structure of populations [J]. Brit Med J, 2(4669): 36.
WRIGHT S, 1970. A Second Installment. (Book Reviews: Evolution and the genetics of populations. The theory of gene frequencies) [J]. J Biosoc Sci, 168(2): 722-723.
WRIGHT S, 1982. The shifting balance theory and macroevolution [J]. Ann Rev Genet, 16(1): 1-20.
WU ZQ, 2016. The whole chloroplast genome of shrub willows (Salix suchowensis) [J]. Mitochondrial DNA A, 27(3): 1-2.
YANG LC, ZHONG ZB, LIU HC, et al., 2013. Community characteristics of alpine shrub Salix oritrepha [J]. Bull Bot Res, 33(6): 653-658.? [楊路存, 鐘澤兵, 劉何春, 等, 2013. 高寒山生柳灌叢的群落特征研究 [J]. 植物研究, 33(6):653-658.]
YANG YH, ZHANG DX, LI YM, et al., 2004. Mitochondrial DNA diversity and preliminary biogeographic inference of the evolutionary history of the black-spotted pond frog Rana nigromaculata populations in China [J]. Acta Zool Sin, 50(2): 193-201.? [楊玉慧, 張德興, 李義明, 等, 2004. 中國黑斑蛙種群的線粒體DNA多樣性和生物地理演化過程的初探 [J]. 動物學報, 50(2):193-201.]
YANG YT, HOU XY, WEI ZW, et al., 2018. Screening and genetic diversity analysis of chloroplast non-coding region in Leymus chinensis [J]. Acta Pratac Sin, 27(10): 147-157.? [楊艷婷, 侯向陽, 魏臻武, 等, 2018. 羊草葉綠體非編碼區多態性標記篩選及群體遺傳多樣性 [J]. 草業學報, 27(10):147-157.]
ZHAO JK, XU LA, JIE HF, et al., 2008. RAPD analysis of population genetic diversity of Tamarix chinensis in Yellow River delta [J]. J Nanjing For Univ (Nat Sci Ed), 32(5): 56-60.? [趙景奎, 徐立安, 解荷峰, 等, 2008. 黃河三角洲檉柳群體遺傳多樣性RAPD分析 [J]. 南京林業大學學報(自然科學版), 32(5):56-60.]

