水稻條斑病菌內源質粒分析
曾晨 李康佳 尹朝群 牛祥娜 趙嘉城 朱平川 姜偉 何勇強


摘要:【目的】明確水稻條斑病菌(Xanthomonas oryzae pv. oryzicola, 簡稱Xoc)內源質粒分布狀況、類型及其含有的主要功能基因,為下一步開展該菌與質粒相關的毒力進化研究、抗逆機制分析及穿梭載體構建打下基礎。【方法】采用數據庫檢索法和質粒分離鑒定法,調查水稻條斑病菌內源質粒的分布狀況;以GenBank數據庫中Xoc菌株基因組序列為研究對象,開展質粒信息檢索,并對已發表的Xoc內源質粒序列進行系統進化分析;以Xoc中國菌株菌種庫為研究對象,采用堿裂解法提取質粒,進行質粒酶切帶型分析和主要功能基因鑒定。【結果】GenBank全基因組數據庫中Xoc菌株內源質粒檢出率為11.76%,系統進化分析結果表明這些質粒屬于不同的進化分支。在257株Xoc中國菌株中,有29株菌株含有質粒,質粒檢出率為11.28%,帶有內源質粒的菌株均來自于華南稻區,且大部分來自廣西。通過質粒酶切圖譜比較,確認新發現的質粒屬于9種新型Xoc內源質粒。按照已知Xoc內源質粒pXOCgx01上主要功能基因序列設計引物,PCR擴增結果表明新鑒定的Xoc質粒含有重金屬抗性和DNA轉移相關的基因,但在菌株間存在明顯的多樣性。【結論】部分Xoc菌株中含有一定數量的內源質粒,質粒類型和所含功能基因等具有一定的多樣性,攜帶內源質粒的Xoc菌株有明顯地域性。
關鍵詞: 水稻條斑病菌;內源質粒;調查;酶切圖譜;抗逆基因
中圖分類號: S435.111.49? ? ? ? ? ? ? ? ? ? ? ? ? 文獻標志碼: A 文章編號:2095-1191(2019)08-1718-10
Analysis of indigenous plasmids in Xanthomonas oryzae
pv. oryzicola
ZENG Chen, LI Kang-jia, YIN Chao-qun, NIU Xiang-na, ZHAO Jia-cheng,
ZHU Ping-chuan, JIANG Wei, HE Yong-qiang*
(State Key Laboratory for Conservation and Utilization of Subtropical Agro-bioresources/College of Life Science and Technology, Guangxi University, Nanning? 530004, China)
Abstract:【Objective】The aim was to fully understand the distribution, types and biological function of the indigenous plasmids of Xanthomonas oryzae pv. oryzicola(Xoc), and lay the foundation for the future study of virulence evolution analysis, mechanism of resistance and construction of shuttle vector. 【Method】The distribution of endogenous plasmids in Xoc strains was investigated by database mining and plasmid isolation. With Xoc strain genome sequence in GenBank as the research target, plasmids information searching was conducted, and phylogenetic analysis on published Xoc indigenous plasmids sequence was also carried out. With Xoc Chinese strains database as the research target, the plasmids were extracted by alkaline lysis, plasmidrestriction endonuclease pattern was analyzed and main functional genes were identified. 【Result】The detection rate of Xoc strainindigenous plasmids in GenBank whole genome database was 11.76%. The phylogenetic analysis indicated that these plasmids were clustered into different phylogenetic branches. Among the 257 Xoc Chinese strains, 29 contained plasmids with a detection rate of 11.28%. The strains containing indigenous plasmid all came from southern China, and most of them were from Guangxi. Nine novel indigenous plasmids of Xoc were confirmed by restriction endonuclease pattern. The primers were designed based on main functional gene sequence of the known? indigenous plasmid pXOCgx01. The PCR amplification analysis indicated that the novel plasmids contained heavy metal tolerance and DNA transposition related genes, but there was diversity among the strains. 【Conclusion】Partial Xoc strains contain indigenous plasmids, with diversities in plasmid types, functional genes(DNA transposition and heavy metal resistance), and the Xoc strains with indigenous plasmids have obvious geographical origins.
Key words: Xanthomonas oryzae pv. oryzicola; indigenous plasmid; general survey; restriction pattern; resistance gene
0 引言
【研究意義】質粒是獨立于染色體外可自主復制的遺傳物質,多為環狀雙鏈DNA分子,是一些細菌基因組的組成部分(Thomas et al.,2017)。質粒常攜帶有抗生素抗性、重金屬耐性及功能蛋白分泌系統等相關基因,可幫助宿主菌適應生存環境,提高致病能力(Vivian et al.,2001;Sundin,2007)。質粒也是基因水平轉移的主要載體,在細菌抗藥、抗逆,乃至毒力進化中發揮重要作用(De Feyter and Gabriel,1991;Carattoli,2013;Suhartono et al.,2018)。水稻條斑病菌也稱水稻細菌性條斑病菌,學名為Xanthomonas oryzae pv. oryzicola,簡稱Xoc(Ni?o-Liu et al.,2006),漢譯為水稻黃單胞菌稻生致病變種或水稻黃單胞菌棲稻致病變種(肖永勝等,2011)。該菌引起的水稻細菌性條斑病(Bacterial leaf streak,BLS)廣泛發生在熱帶、亞熱帶稻作區,可導致水稻減產20%~60%(Ni?o-Liu et al.,2006)。近年來,該病在我國多個稻區發病較重,至今尚未發現水稻抗BLS的主效基因,也缺乏高效防治該病的方法(馬路等,2018)。明確病原菌的致病機理和環境適應機理是發展高效、有針對性、環境友好的病害防治藥物和方法的基礎。最近,人們在Xoc菌株中發現了內源質粒(Niu et al.,2015;Wilkins et al.,2015),研究表明,Xoc的內源質粒能提高受體菌的抗逆性(Niu et al.,2015)。因此,開展Xoc內源質粒的調查和分析,不僅有助于明確Xoc內源質粒分布狀況、質粒類型,揭示內源質粒在病菌毒力、抗脅迫和環境適應等方面的生物學功能,還能為闡明病原菌毒力因子進化、轉移等方面的機理提供參考。【前人研究進展】前期本課題組從廣西賀州分離的一株Xoc菌株中分離鑒定出一個內生質粒pXOCgx01(Niu et al.,2015),該質粒全長53206 bp,注釋64個編碼基因,包括3個重要的功能基因簇:(1)czcCBA基因簇(簡稱czc基因簇,包括czcA、czcB和czcC 3個基因),編碼鈷—鋅—鎘抗性蛋白(Cobalt-zinc-cadmium resistance protein),在金屬抗逆性方面發揮作用(Nies,1995;Niu et al.,2015);(2)復合轉座系統tnpA-tnpS-tnpT cassette結構,簡稱tnp基因簇,推測在毒力因子基因水平轉移方面發揮重要作用(Ferreira et al.,2015);(3)virB/virD4基因簇,簡稱vir基因簇,在DNA轉運、vir毒力因子分泌等方面發揮作用(Voth et al.,2012)。將pXOCgx01導入不含質粒的水稻白葉枯病菌(學名X. oryzae pv. oryzae,簡稱Xoo;譯為水稻黃單胞菌水稻致病變種)PXO99A菌株,能顯著提高該菌株對重金屬的抗性(Niu et al.,2015)。DNA限制修飾系統在不同物種,甚至是同一物種的不同個體間存在明顯差異,內源質粒的分布狀況非常復雜。已有研究表明,絕大多數水稻黃單胞菌排斥外源質粒,被轉化率僅為6%左右(吳神怡等,2010)。迄今為止,已分離和鑒定了近百株水稻條斑病菌亞洲和非洲分離株(Gonzalez et al.,2007;Ji et al.,2014),一部分菌株如Xoc BLS256等已完成基因組測序(Bogdanove et al.,2011;Wilkins et al.,2015),但僅從兩個Xoc菌株中發現內源質粒,分別為pXOCgx01(Niu et al.,2015)和pXOC2286(Wilkins et al.,2015)。目前,人們對水稻條斑病菌內源質粒的存在狀況、種類、功能等缺乏了解。【本研究切入點】長期以來,本課題組持續開展黃單胞菌致病分子機理研究,建立了黃單胞菌菌種庫,從全國多個省份分離獲得數百株Xoc菌株。在Xoc的其他菌株是否存在有質粒,如果存在質粒,這些質粒會有怎樣的基因組成、行使何種生物學功能,這些質粒在與其他黃單胞菌中的質粒可能會有怎樣的進化關系,以及這些質粒與Xoc核染色體間是否存在協同進化關系等均有待進一步探究。【擬解決的關鍵問題】通過大規模GenBank數據庫搜索與大規模質粒提取分離鑒定相結合檢測Xoc內源質粒,并對獲得的Xoc內源質粒進行酶切圖譜分析和功能基因PCR法檢測,以期對水稻條斑病菌內源質粒的分布狀況、質粒類型和潛在的生物功能有一個全新的了解,為下一步開展該菌與質粒相關的毒力進化、逆境適應機制等研究及穿梭載體構建打下基礎。
1 材料與方法
1. 1 試驗材料
1. 1. 1 菌株、質粒和引物
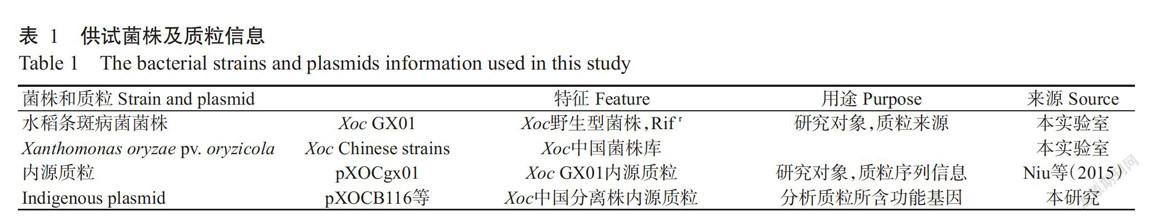
1. 1. 1. 1 菌株來源和質粒 為研究水稻條斑病菌的致病分子機理,廣西大學生命科學與技術學院植物病原細菌功能基因組學實驗室(本課題組屬于該實驗室,以下簡稱本實驗室)建立了水稻黃單胞菌菌種庫,本研究涉及的水稻條斑病菌中國菌株分別從來自于安徽、江蘇、浙江、福建、廣東、海南、云南和廣西等地的水稻病株中分離獲得,共257株菌株。其他菌株和質粒信息見表1。
1. 1. 1. 2 生物試劑 限制性內切酶購自Promega公司、Taq酶購自TaKaRa公司。化學試劑為分析純級(AR)、培養基材料為生化實驗級(BR)。PCR原料dNTP Mix購自索萊寶公司。用于DNA樣品凝膠電泳的染料Gel Red(膠紅)購自美國Biotium公司。
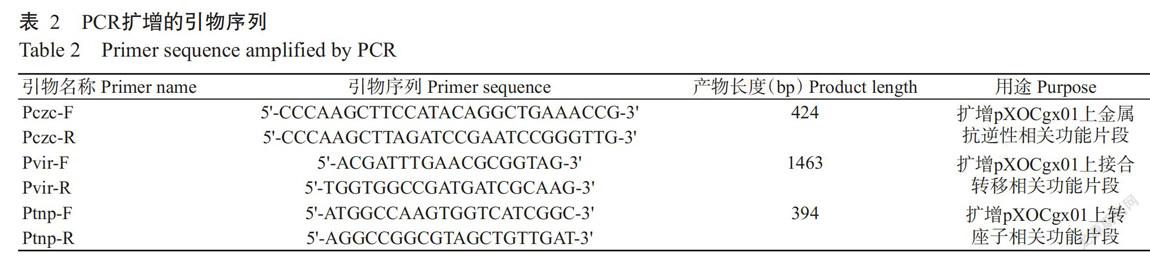
1. 1. 1. 3 PCR擴增引物 采用Vector NTI 11.5軟件,按照pXOCgx01質粒(Niu et al.,2015)中目標基因的序列設計引物(表2),引物合成和DNA測序由深圳華大基因股份有限公司完成。
1. 1. 1. 4 質粒提取試劑 Solution I配方:45.0 mL(50 mmol/L)20%(w/v)葡萄糖、2.0 mL(10 mmol/L)0.5 mol/L EDTA(pH 8.0)、2.5 mL(25 mmol/L)1 mol/L Tris-HCl(pH 8.0)。用于細菌質粒提取第一步,重懸菌體,高壓滅菌,4 ℃短期保存。Solution II配方:10.0 mL(0.2 mol/L)2 mol/L NaOH、10.0 mL(1%,w/v)10%(w/v)SDS。用于細菌質粒提取第二步,裂解菌體,需現配現用。Solution III配方:60.0 mL的5 mol/L醋酸鉀、11.5 mL醋酸。用于細菌質粒提取第三步,沉淀蛋白等雜質,高壓滅菌,4 ℃短期保存(薩姆布魯克和拉塞爾,2002)。
1. 1. 1. 5 電泳緩沖液和上樣緩沖液 電泳緩沖液TBE(10×)配方: 5.5 g(5.5%,w/v)Boric acid(硼酸)、4.0 mL(20 mmol/L) 0.5 mol/L EDTA(pH 8.0)和2.5 mL(25 mmol/L)Tris堿(pH 8.0),可作為DNA樣品電泳緩沖液,制備瓊脂糖凝膠,或作為母液制備0.5×TBE。高壓滅菌,室溫保存(薩姆布魯克和拉塞爾,2002)。上樣緩沖液Loading Buffer(10×)配方:0.02 g (0.2%,w/v)二甲苯青FF、0.02 g(0.2%,w/v)溴酚藍、40.0 mL(200 mmol/L) 0.5 mol/L EDTA(pH 8.0)、1.0 mL(0.1%,w/v)10%(w/v)SDS和50.0 mL(50%)甘油,用于DNA樣品凝膠電泳,可于室溫保存(薩姆布魯克和拉塞爾,2002)。
1. 1. 2 培養基及培養條件
1. 1. 2. 1 培養基 水稻條斑病菌各參試菌株均采用NB(Nutrient broth)培養基(配方:5.0 g/L高聚蛋白胨、3.0 g/L牛肉膏、1.0 g/L酵母提取物、10.0 g/L蔗糖、pH 7.0)進行液體培養,采用NA(Nutrient agar)培養基進行固體培養[在NB培養基配方的基礎上加入1%(w/v)瓊脂粉](Niu et al.,2015)。
1. 1. 2. 2 培養條件 水稻條斑病菌的最適培養溫度為28 ℃。固體培養時,培養基倒置于培養箱中,24~48 h后可出現菌落;液體培養時,搖床轉速為200 r/min,培養16~20 h。
1. 2 試驗方法
1. 2. 1 DNA操作
1. 2. 1. 1 堿裂解法提取質粒 新鮮活化的Xoc菌株接種到含有10.0 mL NB培養基的30 mL通用培養瓶中,于28 ℃下200 r/min搖床培養16~20 h。收集1.5 mL菌液于1.5 mL EP管中,以12000 r/min離心1 min,棄去上清。加入200 μL保存在4 ℃冰箱的Solution I,充分混勻。加入400 μL新鮮配制的Solution II,輕柔混勻。立刻加入300 μL保存在4 ℃冰箱的Solution III,輕柔混勻,白色或淡黃色絮凝狀沉淀產生。1200 r/min離心10~15 min,至沉淀聚集成塊在EP管底部。吸取700~750 μL上清液移至另一新的1.5 mL離心管中,12000 r/min離心10 min。吸取500~600 μL上清液移至另一新的1.5 mL離心管中,加入2倍體積無水乙醇后混勻,于-20 ℃下靜置20 min以上。靜置后的混合液12000 r/min離心10 min使質粒DNA析出,棄上清液。加入500 μL 75%乙醇洗滌質粒DNA,上下晃動使乙醇溶液覆蓋整個EP管內壁,12000 r/min離心3~5 min,棄上清液。用真空干燥器將質粒DNA干燥3~5 min,或將EP管倒置在吸水紙上,自然干燥10~15 min。在干燥后的DNA中加入40~50 μL滅菌的去離子水,待充分溶解后-20 ℃保存備用。
1. 2. 1. 2 限制性酶切體系 分離純化質粒DNA,用不同的限制性內切酶(BamH I、EcoR I、Sma I、Kpn I和Pst I)分別進行單酶切。以20.0 μL酶切體系為例,單酶切時各組分添加比例如下:2.0 μL 10×Buffer、0.2 μL乙酰化BSA(10 μg/μL)、1.0 μL質粒DNA(1.0 μg/μL)、16.0 μL雙蒸水、0.8 μL內切酶(10.0 U/μL)。各反應組分均加入后用移液器吸打使充分混勻,短暫離心,在相應的酶切溫度(推薦溫度)下反應4 h。
1. 2. 1. 3 瓊脂糖凝膠電泳 根據DNA樣品的分子量,確定瓊脂糖凝膠的濃度與大小,本研究用1.0%瓊脂糖,0.5×TBE緩沖液。將DNA樣品和標度DNA(Marker)分別與已混有Gel Red的10×上樣緩沖液混勻后,加入上樣孔。打開電泳儀,100 V電泳至上樣緩沖液移動到凝膠的3/4處。停止電泳,取出凝膠放入凝膠成像系統,拍照記錄結果,用于酶切圖譜分析。
1. 2. 1. 4 PCR擴增 反應體系10.00 μL:50 ng/μL DNA模板0.25 μL,10 μmol/L正、反向引物各0.50 μL,10 mmol/L dNTP 0.25 μL,Taq Buffer 1.00 μL,稀釋Taq酶(1 U)1.00 μL,雙蒸水6.50 μL。擴增程序:95 ℃預變性5 min;95 ℃ 30 s,退火溫度60 ℃(根據引物而定,見表2)下退火45 s,72 ℃ 2 min,進行30個循環;72 ℃延伸2 min。在BIOER PCR儀上完成相關基因的擴增。反應結束后,取1.00 μL瓊脂糖凝膠電泳、凝膠成像、拍照記錄。
1. 2. 2 生物信息學分析 采用Local BLAST對相關水稻黃單胞菌質粒序列進行比對,通過MEGAX進行全序列數據分析,采用Neighbor-joining法構建系統發育進化樹(陳銘,2018),并用Evolview進行標注,采用圓形分支圖進行呈現(He et al.,2016)。已知序列的pXOCgx01和pXOC2286的電子酶切圖在Vector NTI所帶的Gel Analysis組件中完成。
2 結果與分析
2. 1 水稻條斑病菌內源質粒的序列信息和特征
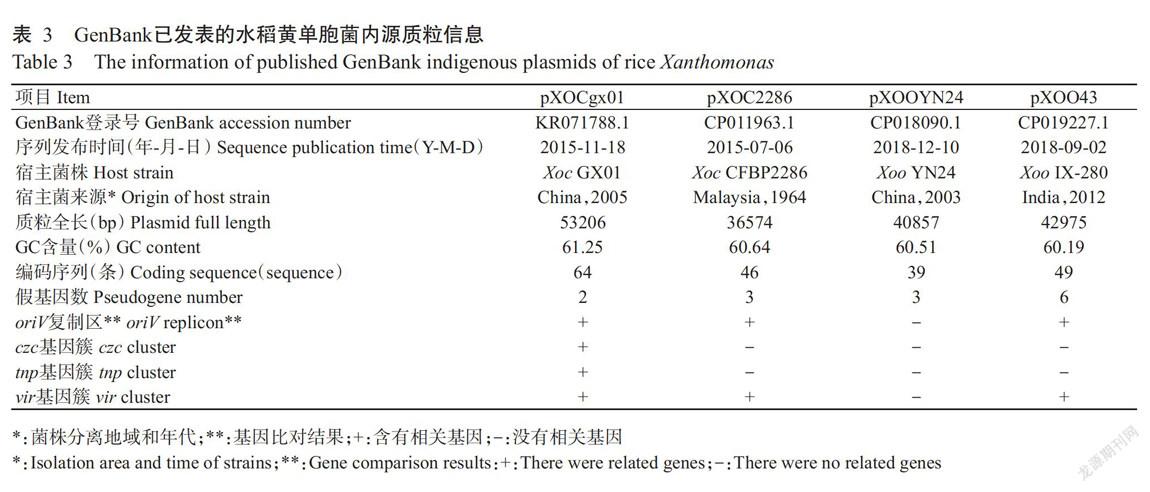
為掌握現有Xoc內源質粒的分布狀況和序列信息,對GenBank數據庫中公布的已完成或正在開展的Xoc全基因組測序數據庫進行分析,結果(表3)顯示已完成Xoc全基因組測序的17株水稻條斑病菌中只有2株菌株含有內源質粒,其中,pXOCgx01來源于中國Xoc GX01菌株,pXOC2286來源于馬來西亞的Xoc CFBP2286菌株。數據庫檢索結果表明,Xoc內源質粒檢出率為11.76%。同時發現43株水稻白葉枯病菌(同屬于水稻黃單胞菌)中也僅有2株菌株含有內源質粒(表3),Xoo內源質粒檢出率僅為4.65%。
質粒序列信息分析結果(表3)顯示,水稻黃單胞菌4種質粒的GC含量在60.19%~61.25%,低于水稻黃單胞菌染色體DNA的GC含量(63.00%~64.00%),但差異不明顯。基因比對分析結果表明,pXOCgx01、pXOC2286和pXOO43的基因具有一定的同源性,序列中存在較長的共線性片段;pXOC2286和pXOO43均含有完整的oriV復制區和vir基因簇,但沒有czc基因簇和tnp基因簇;pXOOYN24質粒與其他3種質粒的基因構成完全不同,不僅沒有oriV復制區,也沒有pXOCgx01質粒攜帶的其他功能基因簇(Niu et al.,2015)。
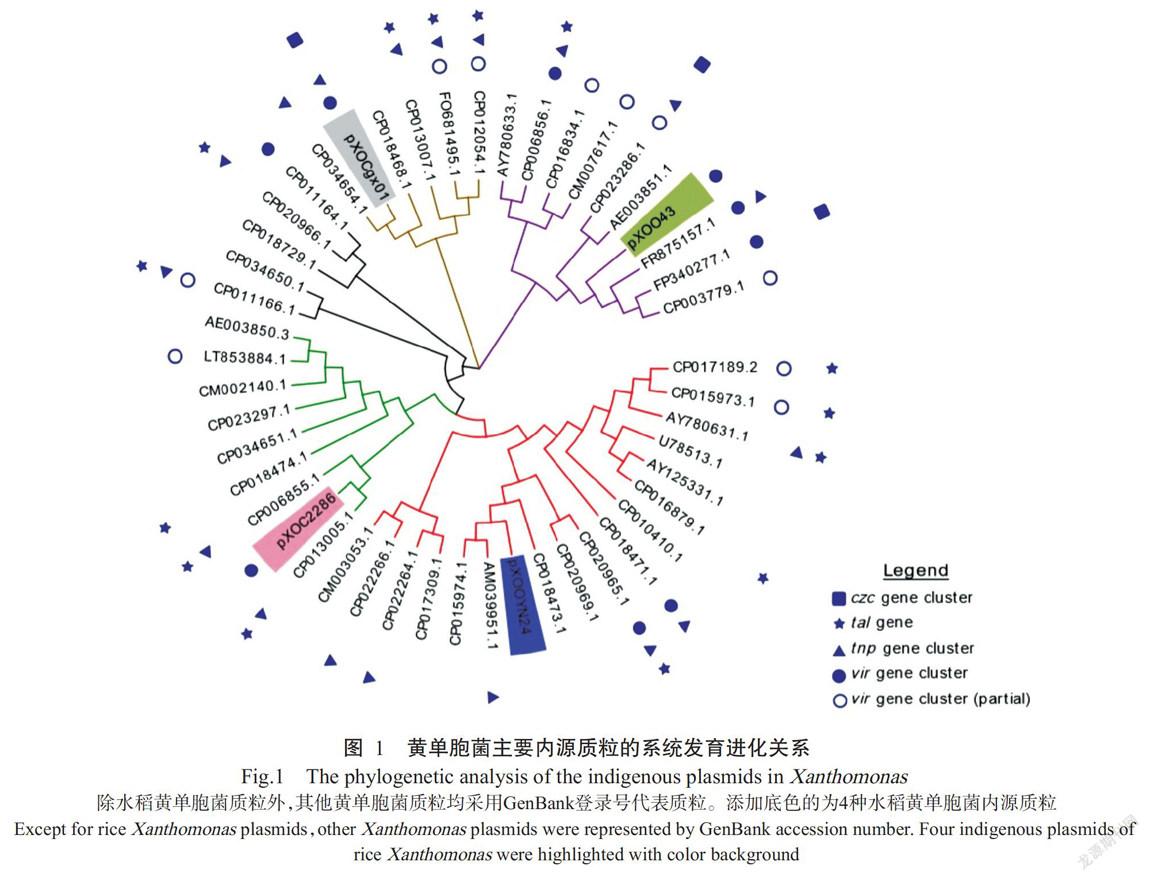
系統發育進化分析結果(圖1)表明,4株水稻黃單胞菌的內源質粒分屬于黃單胞菌內源質粒不同的進化分支。水稻條斑病菌中的2種質粒pXOCgx01和pXOC2286也存在較遠的進化距離。同一分支上的質粒具有類似的序列特點及基因構成等,但這些質粒來源于不同種的黃單胞菌。這一結果提示黃單胞菌在屬水平上來看,接納的質粒差異性很大,質粒是否能在黃單胞菌中存在,與菌株本身的限制修飾系統及質粒類型密切相關。從攜帶的基因來看,相當一部分質粒帶有vir基因簇(包括缺少部分基因的非完整vir基因簇)、tnp基因簇和tal基因簇,少部分質粒帶有czc基因簇。
2. 2 水稻條斑病菌中國菌株中內源質粒的鑒定結果
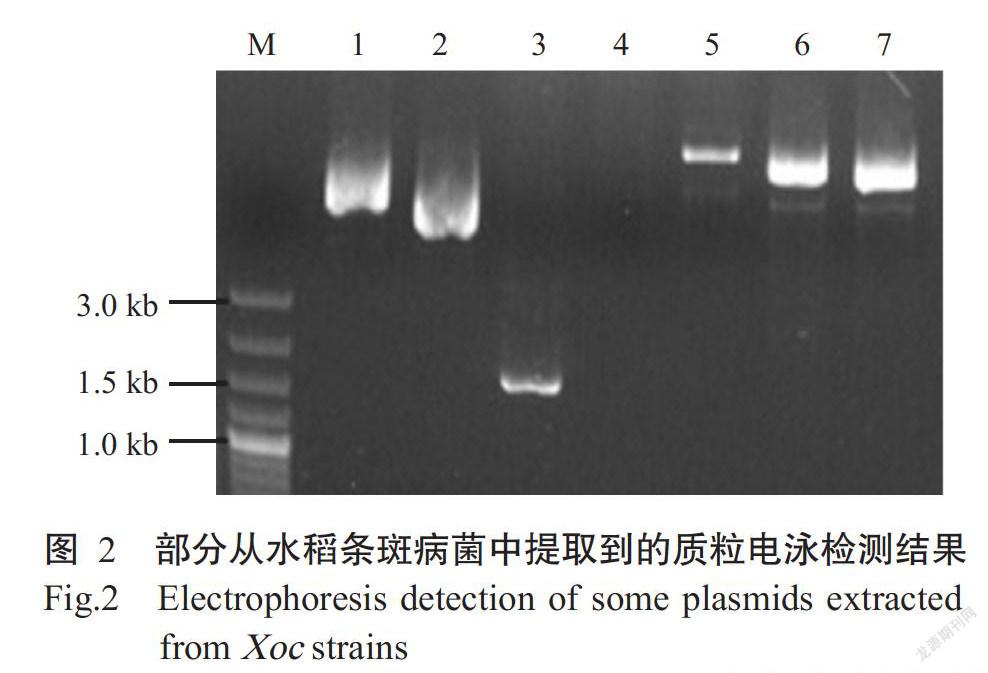
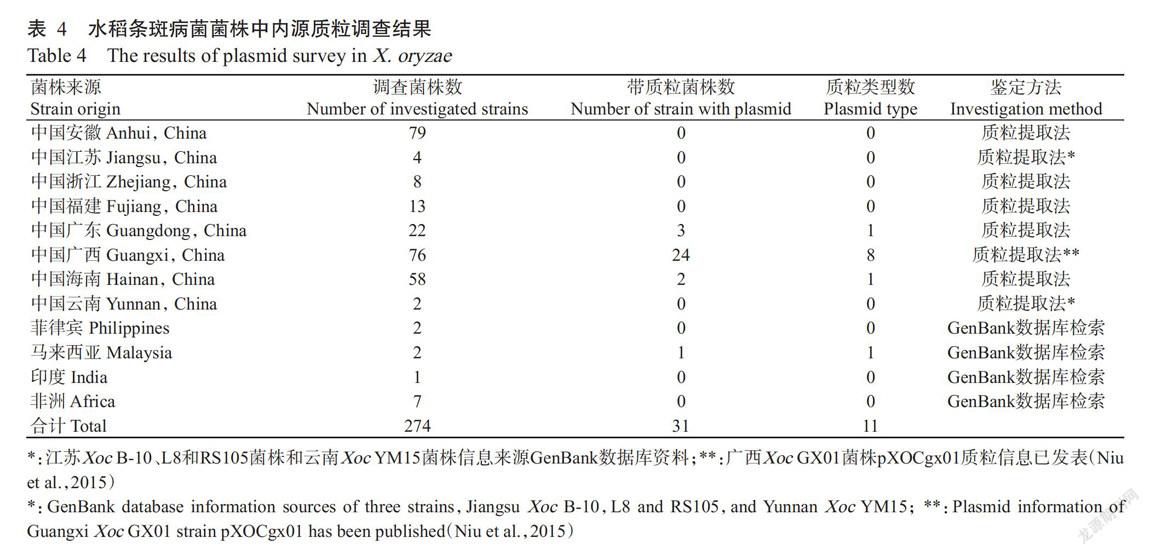
為掌握水稻條斑病菌中國菌株中內源質粒的分布情況,采用堿裂解法提取質粒,再凝膠電泳,對本實驗室分離保存的257株Xoc菌株(其中79株來自安徽、75株來自廣西、58株來自海南、22株來自廣東、13株來自福建、8株來自浙江、1株來自江蘇、1株來自云南)進行質粒鑒定,凡是電泳后經DNA染色有質粒條帶的表示該菌株含有質粒(見圖2中的質粒代表),沒有質粒條帶的確定為不含質粒。結果(表4)表明,在257株水稻條斑病菌中國分離株中共檢測到29株含內源質粒,質粒檢出率為11.28%;從分離菌株的地域來看,帶有內源質粒的菌株均來自于華南地區,且大部分來自廣西(質粒檢出率為31.58%),其原因正在進一步研究。
2. 3 水稻條斑病菌中國菌株內源質粒酶切帶型分析結果
分別采用BamH I、Kpn I、Pst I、EcoR I和Sma I等常用內切酶對pXOCgx01及其他被分離的質粒進行酶切,電泳圖譜見圖3。通過合并因酶切帶型一致而被認為是同種質粒的樣品后,共得到9種不同類型的新內源質粒,分別根據菌株名命名為pXOCgx02、pXOCB116、pXOCB209、pXOCB216、pXOCB220、pXOCB409、pXOC709、pXOC820和pXOC905。從酶切帶型初步判斷pXOC209與pXOCgx01、pXOCgx02與pXOC905、pXOCB216與pXOC709較類似。pXOCB409的長度最短,而pXOCB220的長度可能最長。
比較pXOCgx01質粒的酶切和電子酶切圖譜,結果發現pXOCgx01測序組裝正確,說明電子酶切圖譜可用于DNA帶型比較。從電子酶切圖譜來看,pXOC2286與其他質粒明顯不同。本研究結果表明,目前從水稻條斑病菌共發現11種類型的內源質粒,除pXOCgx01和pXOC2286外,其他9種質粒屬于新型質粒。
2. 4 水稻條斑病菌中國菌株內源質粒主要功能區段的PCR鑒定結果
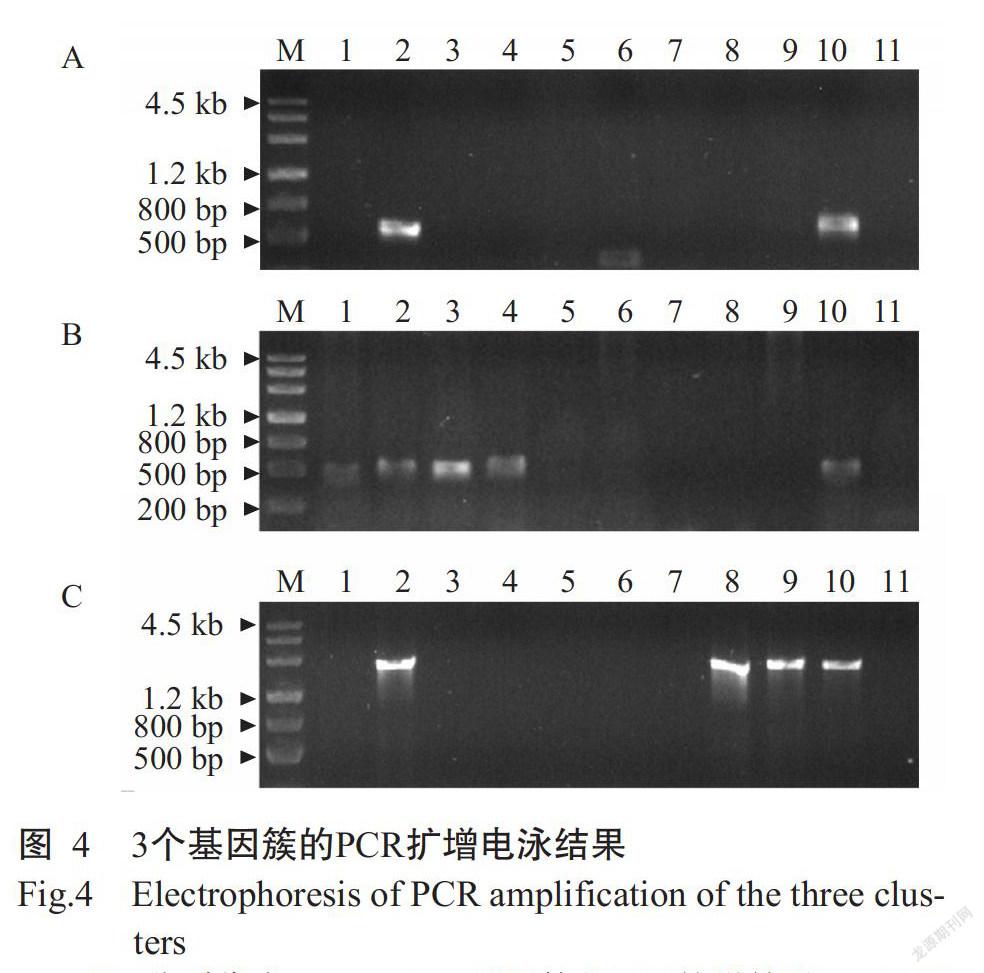
基于pXOCgx01質粒中含有的功能基因簇進行序列比對分析,結果顯示在已完成測序的幾個水稻黃單胞菌內源質粒中,這些基因簇的分布存在明顯的多樣性,值得對中國Xoc菌株內源質粒進行相關基因簇鑒定。根據pXOCgx01中3個基因簇的保守基因設計3對引物(表2),分別以分離的內源質粒為模板進行PCR擴增。電泳結果(圖4)顯示,只有pXOCB209質粒能擴增出3條條帶;pXOCB116、pXOCB216和pXOCB220質粒中能擴增出tnp基因帶;pXOC905和pXOCgx02質粒中能擴增出vir基因帶;pXOC709中能擴增出1條明顯小于預計產物的條帶;pXOCB409和pXOC820中未擴增出任何條帶。PCR擴增電泳結果提示pXOCB209質粒含有相應的3個基因簇(表5),pXOCB116、pXOCB216和pXOCB220質粒含有tnp基因簇,pXOC905和pXOCgx02質粒含有vir基因簇,pXOC709中可能含有變異的czc基因,而pXOCB409和pXOC820可能不含相關的3個基因簇,詳細結果待完成序列測定。
本研究初步鑒定到9種新類型的Xoc內源質粒,加上已報道的2種質粒,目前已從Xoc中發現11種內源質粒,這些質粒在大小和攜帶的功能基因方面呈現出多樣性。酶切圖譜(圖3)與PCR擴增電泳結果關聯分析(圖4和表5)可初步將這些質粒進行分組:(1)Xoc B209菌株攜帶的pXOCB209質粒,與pXOCgx01具有接近的酶切帶型和PCR產物。推測pXOCB209具有類似于pXOCgx01的質粒結構和功能基因;(2)pXOCB116、pXOCB216和pXOCB220在酶切帶型和PCR產物條帶上也非常相似,均具有tnp基因簇;(3)pXOCgx02和pXOC905兩個質粒均含有vir基因簇,與pXOC2286(馬來西亞)所帶功能基因相似(表3);(4)pXOCB409和pXOC820均未擴增出功能基因條帶;(5)pXOC709能擴增出1條小于目標基因的條帶,推測該同源基因內部發生了缺失變異。
3 討論
質粒作為染色體外的遺傳物質,具有可移動、易變異等特點,盡管質粒較少參與細菌的基本代謝,但在宿主菌的環境適應和毒力進化方面發揮重要作用(Carroll and Wong,2018)。近年來,質粒在人類和動物超級病菌進化、傳播等方面的作用機理研究已成為人們關注的熱點問題之一(Wang et al.,2018),而質粒在植物病原細菌方面的研究工作相對較少(Niu et al.,2015;Gochez et al.,2018)。本研究分別采用數據庫檢索和質粒提取兩種方式對Xoc不同地域分離菌株中的內源質粒進行調查,兩種方式的質粒檢出率分別為11.76%和11.28%。Xoc群體中含有內源質粒的菌株比例為11.52%,大致為1∶10的關系,推測質粒對Xoc正常的致病機制或環境適應具有一定貢獻,但不是主要因素。在黃單胞菌屬中,不同菌種帶有內源質粒的比例相差很大,據不完全統計,幾乎所有的柑橘黃單胞菌(Xanthomonas citri)菌株均含有內源質粒,每個菌株含2~4個不同型質粒,柑橘黃單胞菌在致病機制或環境適應方面對內源質粒具有較大的依賴(Gochez et al.,2018)。從黃單胞菌質粒攜帶的基因來看,vir基因、tnp基因和tal基因最常見,少部分質粒帶有czc基因,這些基因在內源質粒中的差異是否會影響黃單胞菌的寄主特異性目前尚不清楚,值得進一步關注。
系統進化分析結果顯示,Xoc中已發表的2種質粒pXOCgx01和pXOC2286位于不同的進化分支,表明質粒間存在較大進化差異,提示同類菌株可攜帶不同類型的質粒。本研究也發現同一分支上的質粒卻來源于不同種的黃單胞菌,提示黃單胞菌在屬水平上質粒接納的菌株特異性不高,質粒是否能在黃單胞菌中存在,與菌株個體本身的限制修飾系統及質粒類型密切相關。
從GenBank數據庫基因組信息可看出,除了中國和馬來西亞2株Xoc菌株含有內源質粒外,世界其他稻區(非洲、印度、日韓和菲律賓等國家和地區)分離的Xoc菌株均未發現攜帶內源質粒。Xoc中國分離株的內源質粒提取結果也顯示出,帶有內源質粒的Xoc菌株分布具有明顯地域特征,均來源于華南稻區,尤其是廣西稻區,內源質粒檢出率高達31.58%。華東地區(4省)104株被檢菌株中均無內源質粒檢出。從被檢菌株的數量來看,質粒分布的這種不均衡性似乎不是人為取樣偏差造成,有關廣西菌株中擁有內源質粒比例相對較高的原因有待進一步探究。廣西是世界范圍內野生稻資源最豐富的地區之一,全基因組關聯分析(Genome-wide association study,GWAS)結果表明,廣西野生稻可能是栽培稻的最近祖先,也是主要的栽培稻馴化地區之一(Huang et al.,2012);同時,廣西屬于多民族地區,水稻耕作方式各異,常規稻品種繁多,推測Xoc菌株面對多樣的水稻品種、復雜的地理環境等因素,在長期的進化過程中,一方面自身在不斷變異,限制系統發生變化,降低了對質粒的排斥作用;另一方面,通過接納質粒獲得水平轉移來的基因,增強自身的生存能力。前期本課題組鑒定的Xoc第一例內源質粒pXOCgx01攜帶有完整的czcCBA基因簇,編碼鈷—鋅—鎘抗性蛋白,在金屬抗逆性方面發揮作用(Niu et al.,2015;Hülter et al.,2017)。該質粒分離自廣西賀州市,該市是我國著名的有色金屬之鄉,多處農田重金屬超標(李忠義等,2009)。推測pXOCgx01攜帶有完整的czcCBA基因簇有利于宿主菌在高污染環境下生存。質粒能提高宿主菌重金屬抗性的研究在多種細菌中已有報道(范麗平等,2010)。從質粒酶切圖譜和PCR擴增電泳結果來看,來自于廣西梧州市藤縣的Xoc B209菌株攜帶的pXOCB209質粒與pXOCgx01具有接近的酶切帶型和PCR產物,推測pXOCB209具有類似于pXOCgx01的質粒結構和功能基因。這兩株菌株的分離地點原屬于同一行政區,不能排除是同一菌株隨種子等在該地區擴散引起。質粒在不同菌株間自主轉移的問題,也可能是同型質粒出現在不同地域的原因之一(Yanagiya et al.,2018)。pXOCB116、pXOCB216和pXOCB220在酶切帶型和PCR產物條帶上也非常相似,均具有tnp基因簇,而且這些質粒的宿主菌來自于廣西玉林和博白,兩地相鄰,也有可能是同一菌株擴散的結果。pXOCgx02(廣西靈川)和pXOC905(廣東英德)兩個質粒均含有vir基因簇,與pXOC2286(馬來西亞)所帶功能基因相似(Wilkins et al.,2015)。本研究結果還顯示,部分質粒沒有擴增出相應基因的條帶,可能不含相關基因,也可能是由于引物問題造成的假陰性,尚有待進一步探究。
4 結論
部分Xoc菌株中含有一定數量的內源質粒,質粒類型、所含功能基因等具有一定的多樣性,主要包括vir毒力因子、DNA轉移及重金屬抗性基因等。攜帶內源質粒的Xoc菌株有明顯的地域性,主要集中在華南稻區,特別是廣西稻區。本研究中鑒定的一系列內源質粒將為構建一系列穿梭質粒載體提供材料,同時這些質粒將在植物病害流行病學中跟蹤毒性菌株的流行路徑、菌體密度等方面發揮重要作用。
參考文獻:
陳銘. 2018. 生物信息學[M]. 第3版.北京:科學出版社. [Chen M. 2018. Bioinformatics[M]. The 3rd Edition. Beijing: Science Press.]
范麗平,王亞峰,霍貴成. 2010. 乳酸乳球菌鎘抗性質粒的鑒定及鎘抗性功能片段的獲取[J]. 中國食品學報,10(3): 205-212. [Fan L P,Wang Y F,Huo G C. 2010. Identification of Lactococcus lactis cadmium resistance plasmid and acquisition of the cadmium resistance functional fragment[J]. Journal of Chinese Institute of Food Science and Technology,10(3): 205-212.]
J. 薩姆布魯克,D. W. 拉塞爾. 2002. 分子克隆實驗指南[M]. 黃培堂譯. 第3版. 北京:科學出版社. [Sambrook J,Ru-ssell D W. 2002. Molecular cloning laboratory manuals[M]. Translated by Huang P T. The 3rd Edition. Beijing: Science Press.]
李忠義,張超蘭,鄧超冰,顧明華,李磊,劉敏. 2009. 鉛鋅礦區農田土壤重金屬有效態空間分布及其影響因子分析[J]. 生態環境學報,18(5): 1772-1776. [Li Z Y,Zhang C L,Deng C B,Gu M H,Li L,Liu M. 2009. Analysis on spatial distribution of soil available heavy metals and its influential factors in a lead-zinc mining area of Guangxi,China[J]. Ecology and Environmental Sciences,18(5): 1772-1776.]
馬路,方媛,肖颯清,周純,金哲倫,葉雯瀾,饒玉春. 2018. 水稻條斑病抗性QTL的挖掘及相關基因的表達[J]. 植物學報,53(4): 468-476. [Ma L,Fang Y,Xiao S Q,Zhou C,Jin Z L,Ye W L,Rao Y C. 2018. QTL exploration of bacterial leaf streak and their gene expression in rice[J]. Chinese Bulletin of Botany,53(4): 468-476.]
吳神怡,劉書言,劉君梁,段承杰,馮家勛. 2010. 一株能通過接合導入外源質粒的水稻黃單胞菌水稻致病變種菌株的獲得及鑒定[J]. 廣西農業科學,41(9): 873-876. [Wu S Y,Liu S Y,Liu J L,Duan C J,Feng J X. 2010. Isolation and identification of a Xanthomonas oryzae pv. oryzae strain capable of efficiently accepting exogenous plasmid by conjugation[J]. Guangxi Agricultural Sciences,41(9): 873-876.]
肖永勝,韋雪雪,郜惠蘋,牛祥娜,岑貞陸,黃萍,吳玥,汪嶸,何勇強. 2011. 一株水稻細菌性條斑病菌的鑒定與遺傳操作系統的建立[J]. 基因組學與應用生物學,30: 1211-1217. [Xiao Y S,Wei X X,Gao H P,Niu X N,Cen Z L,Huang P,Wu Y,Wang R,He Y Q. 2011. Characterization of a Xanthomonas oryzae pv. oryzicola strain and the establishment of its genetic manipulation system[J]. Genomics and Applied Biology,30: 1211-1217.]
Bogdanove A J,Koebnik R,Lu H,Furutani A,Angiuoli S V,Patil P B,Van Sluys M A,Ryan R P,Meyer D F,Han S W,Aparna G,Rajaram M,Delcher A L,Phillippy A M,Puiu D,Schatz M C,Shumway M,Sommer D D,Trapnell C,Benahmed F,Dimitrov G,Madupu R,Radune D,Sullivan S,Jha G,Ishihara H,Lee SW,Pandey A,Sharma V,Sriariyanun M,Szurek B,Vera-Cruz C M,Dorman KS,Ronald P C,Verdier V,Dow J M,Sonti R V,Tsuge S,Brendel V P,Rabinowicz P D,Leach J E,White F F,Salzberg S L. 2011. Two new complete genome sequen-ces offer insight into host and tissue specificity of plant pathogenic Xanthomonas spp.[J]. Journal of Bacteriology,193(19): 5450-5464.
Carattoli A. 2013. Plasmids and the spread of resistance[J]. International Journal of Medical Microbiology,303(6-7): 298-304.
Carroll A C,Wong A. 2018. Plasmid persistence: Costs,benefits,and the plasmid paradox[J]. Canadian Journal of Microbiology,64(5): 293-304.
De Feyter R,Gabriel D W. 1991. At least six avirulence genes are clustered on a 90-kilobase plasmid in Xanthomonas campestris pv. malvacearum[J]. Molecular Plant-Microbe Interactions,4(5): 423-432.
Ferreira R M,de Oliveira A C,Moreira L M,Belasque J Jr,Gourbeyre E,Siguier P,Ferro M I,Ferro J A,Chandler M,Varani A M. 2015. A TALE of transposition: Tn3-like transposons play a major role in the spread of pathogenicity determinants of Xanthomonas citri and other Xanthomonads[J]. MBIO,6(1): e02505-02514.
Gochez A M,Huguet-Tapia J C,Minsavage G V,Shantaraj D,Jalan N,Strau? A,Lahaye T,Wang N,Canteros B I,Jones J B,Potnis N. 2018. Pacbio sequencing of copper-tolerant Xanthomonas citri reveals presence of a chimeric plasmid structure and provides insights into reassortment and shuffling of transcription activator-like effectors among X. citri strains[J]. BMC Genomics,19(1): 16.
Gonzalez C,Szurek B,Manceau C,Mathieu T,Séré Y,Verdier V. 2007. Molecular and pathotypic characterization of new Xanthomonas oryzae strains from west Africa[J]. Molecular Plant-Microbe Interactions,20(5): 534-546.
He Z L,Zhang H K,Gao S H,Lercher M J,Chen W H,Hu S. 2016. Evolview v2: An online visualization and ma-nagement tool for customized and annotated phylogenetic trees[J]. Nucleic Acids Research,44(1):236-241.
Huang X H,Kurata N,Wei X H,Wang Z X,Wang A,Zhao Q,Zhao Y,Liu K Y,Lu H Y,Li W J,Guo Y L,Lu Y Q,Zhou C C,Fan D L,Weng Q J,Zhu C R,Huang T,Zhang L,Wang Y C,Feng L,Furuumi H,Kubo T,Miyabayashi T,Yuan X P,Xu Q,Dong G J,Zhan Q L,Li C Y,Fujiyama A,Toyoda A,Lu T T,Feng Q,Qian Q,Li J Y,Han B. 2012. A map of rice genome variation reveals the origin of cultivated rice[J]. Nature,490(7421):497-501.
Hülter N,Ilhan J,Wein T,Kadibalban A S,Hammerschmidt K,Dagan T. 2017. An evolutionary perspective on plasmid lifestyle modes[J]. Current Opinion in Microbiology,38:74-80.
Ji Z Y,Zakria M,Zou LF,Xiong L,Li Z,Ji G H,Chen G Y. 2014. Genetic diversity of transcriptional activator-like effector genes in Chinese isolates of Xanthomonas oryzae pv. oryzicola[J]. Phytopathology,104(7): 672-682.
Nies D H. 1995. The Cobalt,Zinc,Cadmium efflux system CzcABC from Alcaligenes eutrophus functions as a ca-tion-proton antiporter in Escherichia coli[J]. Journal of Bacteriology,177(10): 2707-2712.
Ni?o-Liu D O,Ronald P C,Bogdanove A J. 2006. Xanthomonas oryzae pathovars: Model pathogens of a mo-del crop[J]. Molecular Plant Pathology,7(5): 303-324.
Niu X N,Wei Z Q,Zou H F,Xie G G,Wu F,Li K J,Jiang W,Tang J L,He Y Q. 2015. Complete sequence and detailed analysis of the first indigenous plasmid from Xanthomonas oryzae pv. oryzicola[J]. BMC Microbiology,15(1): 233-247.
Suhartono S,Savin M C,Gbur E E. 2018. Transmissible plasmids and integrons shift Escherichia coli population toward larger multiple drug resistance numbers[J]. Microbial Drug Resistance,24(3):244-252.
Sundin G W. 2007. Genomic insights into the contribution of phytopathogenic bacterial plasmids to the evolutionary history of their hosts[J]. Annual Review of Phytopathology,45: 129-151.
Thomas C M,Thomson N R,Cerde?o-Tárraga A M,Brown C J,Top E M,Frost L S. 2017. Annotation of plasmid genes[J]. Plasmid,91: 61-67.
Vivian A,Murillo J,Jackson R W. 2001. The roles of plasmids in phytopathogenic bacteria: Mobile arsenals[J]. Microbiology,147(4): 763-780.
Voth D E,Broederdorf L J,Graham J G. 2012. Bacterial Type IV secretion systems: Versatile virulence machines[J]. Future Microbiology,7(2): 241-257.
Wang R,van Dorp L,Shaw LP,Bradley P,Wang Q,Wang X,Jin L,Zhang Q,Liu Y,Rieux A,Dorai-Schneiders T,Weinert L A,Iqbal Z,Didelot X,Wang H,Balloux F. 2018. The global distribution and spread of the mobilized colistin resistance gene mcr-1[J]. Nature Communication,9(1): 1179.
Wilkins K E,Booher N J,Wang L,Bogdanove A J. 2015. TAL effectors and activation of predicted host targets distinguish Asian from African strains of the rice pathogen Xanthomonas oryzae pv. oryzicola while strict conservation suggests universal importance of five TAL effectors[J]. Frontiers in Plant Science,6: 536-552.
Yanagiya K,Maejima Y,Nakata H,Tokuda M,Moriuchi R,Dohra H,Inoue K,Ohkuma M,Kimbara K,Shintani M. 2018. Novel self-transmissible and broad-host-range plasmids exogenously captured from anaerobic granules or cow manure[J]. Frontiers in Microbiology,9: 2602.
(責任編輯 麻小燕)

