OsRhoGDI2過表達轉基因水稻的篩選鑒定及外源基因拷貝數(shù)的初步分析
杜京堯 尚飛 王高華
摘要:水稻Rho GDP解離抑制基因OsRhoGDI2是從幼穗中分離出的功能未知基因。為鑒定該基因的功能,筆者所在實驗室前期構建了植物過表達載體pCAMBIA1302-OsRhoGDI2-GFP,并對水稻進行了遺傳轉化。對OsRhoGDI2過表達轉基因水稻T2代進行篩選和鑒定,采用PCR技術鑒定轉基因植株,采用半定量RT-PCR和實時熒光定量PCR檢測OsRhoGDI2在轉基因水稻中的表達水平,結果顯示,其中6個株系為過表達轉基因植株,OsRhoGDI2表達水平上調1.69~13.35倍。為檢測外源基因在轉基因水稻中的拷貝數(shù),分別以蔗糖磷酸合成酶基因SPS和潮霉素抗性基因HYG為內參基因和標記基因,采用實時熒光定量PCR(qPCR)技術結合內參基因和標記基因的標準曲線進行分析,結果顯示在所檢測的6個轉基因株系中,外源基因的拷貝數(shù)均為1,提示已經獲得穩(wěn)定遺傳的OsRhoGDI2過表達轉基因水稻,為后續(xù)OsRhoGDI2基因的功能研究奠定基礎。
關鍵詞:OsRhoGDI2;轉基因水稻;表達分析;拷貝數(shù);外源基因
中圖分類號: S511.03 ?文獻標志碼: A ?文章編號:1002-1302(2019)14-0050-04
植物Rho/Rac也稱為Rop(Rho of plant),是植物中唯一與信號轉導有關的小GTP結合蛋白,功能涉及細胞極性生長[1-3]、肌動蛋白細胞骨架重組[4-5]、次生壁形成[6]、分生組織信號傳遞[7]以及抗逆和激素應答[8-11]等。作為信號通路的分子開關,Rho蛋白通過與靶蛋白的相互作用,實現(xiàn)對細胞生理活動的調控和影響,已明確的一類相互作用蛋白是Rho GDP解離抑制因子(Rho GDP dissociation inhibitors,RhoGDIs),功能是調節(jié)Rho的胞內定位,通過阻止GTP與Rho的結合,抑制Rho的活化,因而是Rho的負調控因子[12-14]。但是目前對植物RhoGDIs研究的報道較少,雖有從擬南芥、煙草、水稻等植物中篩選到RhoGDIs的報道[15-17],但是對其在植物生長發(fā)育等過程中生物學功能的研究非常有限。Carol等對擬南芥突變體scn1的研究發(fā)現(xiàn),一種擬南芥的RhoGDI基因SCN1/AtrhoGDI1通過控制局部RHD2/AtrbohC NADPH氧化酶的激活,調控根毛細胞的生長[18];Wu等的研究發(fā)現(xiàn),擬南芥AtRhoGDI1通過ROP信號通路,介導對幼苗和葉表皮細胞形態(tài)建成的調節(jié),而且AtRhoGDI1被CPK3磷酸化是執(zhí)行其功能的前提[19],但是至今尚未見水稻RhoGDIs基因功能的報道。
水稻Rho GDP解離抑制基因OsRhoGDI2是通過酵母雙雜交篩選,從雌雄蕊形成期水稻幼穗中分離的功能未知基因。對該基因編碼蛋白的生物信息學分析和亞細胞定位檢測發(fā)現(xiàn),該基因編碼蛋白在活細胞中廣泛分布[20]。對OsRhoGDI2基因啟動子轉基因功能鑒定的研究表明,該基因2 100 bp的上游調控序列即可以啟動GUS在穎花、花藥和花粉中的特異表達,該基因的表達受到生長素(IAA)等多種激素的脅迫和誘導[21]。為了進一步鑒定OsRhoGDI2的功能,本研究基于實驗室的前期工作,對OsRhoGDI2過表達轉基因水稻T2代進行篩選和鑒定,分別采用PCR、RT-PCR和qRT-PCR技術在DNA和RNA水平上進行檢測,并通過qPCR技術鑒定OsRhoGDI2在轉基因株系中的拷貝數(shù),旨在為后續(xù)分析OsRhoGDI2基因的生物學功能奠定基礎。
1 材料與方法
1.1 試驗材料
1.1.1 植物材料 水稻粳稻品種日本晴(Oryza sativa L.japonica.cv.Nipponbare),簡稱WT,由山東省農業(yè)科學院作物與分子育種實驗室提供;OsRhoGDI2過表達轉基因水稻,簡稱OE-OsRhoGDI2,由筆者所在實驗室前期構建,已篩選獲得T1代種子,遺傳背景為日本晴。
1.1.2 載體 植物過表達載體pCAMBIA1302-OsRhoGDI2-GFP由筆者所在實驗室前期構建。
1.1.3 主要試劑 總RNA提取試劑RNAiso Plus、PrimeSTAR HS DNA Polymerase、PrimeScriptTM RT reagent Kit with gDNA Eraser (Perfect Real Time)、SYBR Premix Ex TaqTM Ⅱ購自寶生物工程(大連)有限公司,2×Es Taq MasterMix (Dye)購自北京康為世紀生物科技有限公司。
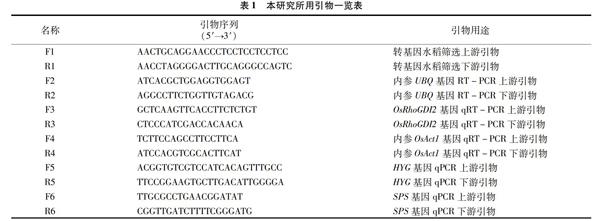
1.1.4 引物設計 本試驗所用引物(表1)均由蘇州金維智生物科技有限公司合成。
1.2 試驗方法
1.2.1 水稻取材 選取適量的對照(WT)水稻種子和OE-OsRhoGDI2水稻T1代種子,2016年5月1日播種于河南師范大學試驗園地(113.90°E、35.32°N)。水稻生長至 20 d (幼苗期)、90 d(抽穗期)時,分別取其植株葉片,液氮冷凍處理,-80 ℃ 保存?zhèn)溆谩?/p>
1.2.2 OsRhoGDI2轉基因植株的篩選 取幼苗期水稻嫩葉,采用十六烷基三甲基三甲基溴化銨(CTAB)法提取基因組DNA。以F1/R1為引物,基因組DNA為模板進行PCR反應,按照2×Es Taq MasterMix說明書配制反應體系(10 μL):2×Taq Master Mix 5.0 μL、F1(10 μmol/L)0.4 μL、R1(10 μmol/L)0.4 μL、基因組DNA 0.2 μL 和無菌水4.0 μL。PCR反應條件為94 ℃預變性 2 min;94 ℃變性30 s,58 ℃退火30 s,72 ℃延伸1 min,30個循環(huán);72 ℃延伸2 min。擴增產物采用1%瓊脂糖凝膠電泳檢測。
1.2.3 OsRhoGDI2基因在轉基因水稻中表達水平的檢測
1.2.3.1 總RNA的提取和cDNA的制備 取抽穗期水稻葉片,根據(jù)寶生物工程(大連)有限公司RNAiso Plus試劑說明書,分別提取對照(WT)水稻和OE-OsRhoGDI2水稻T2代幼苗葉片總RNA,然后按照PrimeScriptTM RT reagent Kit with gDNA Eraser (Perfect Real Time)試劑盒說明書進行反轉錄,制備cDNA。
1.2.3.2 半定量RT-PCR 以“1.2.3.1”節(jié)獲得的cDNA為模板,按照PrimeSTAR HS DNA Polymerase說明書配制半定量RT-PCR反應體系(50 μL),其中內參基因UBQ的PCR擴增體系為5×PrimeSTAR Buffer(Mg2+ Plus)10.0 μL、dNTP(2.5 mmol/L)4.0 μL、UBQ引物F2(10 μmol/L)1.0 μL、UBQ引物R2(10 μmol/L)1.0 μL、PrimeSTAR HS DNA Polymerase 0.5 μL、cDNA模板2.0 μL和無菌水31.5 μL。PCR反應條件為98 ℃變性30 s,55 ℃退火15 s,72 ℃延伸1 min,30個循環(huán)。OsRhoGDI2的擴增體系引物采用F1/R1,其余同UBQ的PCR擴增體系。PCR反應退火溫度為58 ℃,其余參數(shù)同UBQ的PCR反應條件。擴增產物經1%瓊脂糖凝膠電泳檢測。
1.2.3.3 實時熒光定量PCR 取“1.2.3.1”節(jié)制備的cDNA為模板,以F3/R3為引物進行OsRhoGDI2的qRT-PCR反應,以水稻OsAct1基因為內參,按照SYBR Premix Ex TaqTM Ⅱ說明書配制反應體系(20 μL):2×SYBR Premix Ex Taq 10.0 μL、PCR Forward primer(10 μmol/L)0.8 μL、PCR Reverse primer(10 μmol/L)0.8 μL、cDNA 2.0 μL、滅菌水 6.4 μL。采用2步法進行PCR擴增:第1步95 ℃預變性 30 s;第2步PCR反應95 ℃變性5 s,60 ℃延伸30 s,40個循環(huán)。
每組試驗做3次重復,利用Roche LightCycler 96自帶軟件LightCycler 96 v1.1進行數(shù)據(jù)分析,以2-ΔΔCT法[22]分析OsRhoGDI2基因表達水平。
1.2.4 OsRhoGDI2基因在轉基因水稻中拷貝數(shù)的鑒定 HYG基因標準曲線的制作參考楊立桃等的方法[23],以含有HYG基因的pCAMBIA1302-OsRhoGDI2-GFP質粒DNA溶液(107、106、105、104、103拷貝/μL)為模板,以F5/R5為引物進行qPCR反應,按照SYBR Premix Ex TaqTM Ⅱ說明書進行配制,反應體系同“1.2.3.3”節(jié)。采用2步法進行PCR擴增,進行45個循環(huán),其余參數(shù)同“1.2.3.3”節(jié)。得到等梯度拷貝數(shù)對應的CT值。依據(jù)CT值與起始模板數(shù)的對數(shù)值之間的一定的線性關系來制作標準曲線。水稻內參基因SPS標準曲線的制作參考Ding等的方法[24]。
以6個陽性OE-OsRhoGDI2水稻基因組DNA為模板,分別以F5/R5和F6/R6為引物進行qPCR反應,按照SYBR Premix Ex TaqTM Ⅱ說明書進行配制,反應體系同“1.2.3.3”節(jié)。采用2步法進行PCR擴增,進行45個循環(huán),其余參數(shù)同“1.2.3.3”節(jié)。分別獲得6個植株HYG與SPS基因的CT值,并由HYG與SPS基因的相關性方程計算其起始模板數(shù)。用純合二倍體水稻SPS作為內參基因,將H0與S0的比值乘以2得到OsRhoGDI2基因在轉基因水稻中的拷貝數(shù)。
2 結果與分析
2.1 OsRhoGDI2轉基因水稻的篩選
以水稻基因組DNA為模板,用引物F1/R1進行PCR擴增,擴增產物經瓊脂糖凝膠電泳檢測,確定陽性轉基因植株。由于OsRhoGDI2轉基因表達載體內插入的目的基因長度為795 bp,所以從陽性轉基因水稻基因組DNA中能擴增出 795 bp 特異片段,而對照(WT)植株中不能擴增出該大小的條帶。電泳結果顯示,在檢測的15個樣品中,除了3、8、11號之外,都可以擴增得到預期大小的片段,且與陽性對照條帶位置一致(圖1),說明OsRhoGDI2基因已經整合到水稻基因組中。
2.2 OsRhoGDI2在轉基因水稻中表達水平的檢測
選取PCR檢測為陽性的轉基因水稻,提取葉片總RNA,采用半定量RT-PCR檢測該基因的表達水平,內參采用水稻泛素(UBQ)基因,擴增產物經瓊脂糖凝膠電泳檢測,結果顯示,在對照(WT)水稻葉片中,OsRhoGDI2基因的表達水平較低,而在所檢測的7株轉基因水稻中,OsRhoGDI2基因的表達水平均有不同程度的提高,尤其是7、9、10、12、16號這5株的表達量有顯著的增高(圖2),說明這5株水稻是OE-OsRhoGDI2陽性植株。
進一步采用實時定量PCR方法檢測OsRhoGDI2基因在轉基因水稻中的表達水平。由圖3可知,除2號外,在其余6株轉基因水稻中OsRhoGDI2基因的表達水平均比對照(WT)高,基因表達量極顯著上調的有6號(1.69倍)、16號(2.69倍)、10號(4.02倍)、12號(5.98倍)、9號(9.72倍)和7號(13.35倍),且與半定量RT-PCR檢測結果一致,證明OsRhoGDI2基因在這些轉基因水稻中實現(xiàn)了過量表達。
2.3 HYG基因標準曲線的制作
以含有HYG基因的pCAMBIA1302-OsRhoGDI2-GFP質粒DNA溶液(107、106、105、104、103拷貝/μL)為模板,基于qPCR數(shù)據(jù)獲得了HYG基因的標準曲線(圖4),其相關系數(shù) r=0.999 7,說明相關性高。其CT值與起始模板數(shù)(H0)之間的相關性方程為:H0=10(-0.297CT+12.356)。
2.4 OsRhoGDI2在轉基因水稻中拷貝數(shù)的測定
以上述鑒定的6株陽性OE-OsRhoGDI2水稻基因組DNA為模板,分別以F5/R5和F6/R6引物組合,對HYG基因與SPS基因進行qPCR,獲得擴增曲線與待測樣品的CT值(表2),并由HYG與SPS基因的相關性方程計算其起始模板數(shù)。
OsRhoGDI2基因在轉基因水稻中的拷貝數(shù)的計算結果顯示,對照水稻拷貝數(shù)為0,其余有6個OE-OsRhoGDI2水稻拷貝數(shù)均為1(表3)。
3 結論與討論
植物遺傳轉化是農作物改良和基因功能研究的常用試驗方法。本研究在實驗室前期工作的基礎上,對OsRhoGDI2轉基因水稻T2代進行了篩選和鑒定,獲得6個陽性轉基因水稻株系。現(xiàn)有的研究認為,過表達后代中外源基因拷貝數(shù)是影響目的基因表達水平和遺傳穩(wěn)定性的主要因素。據(jù)統(tǒng)計分析顯示,外源基因拷貝數(shù)為1或2時能夠穩(wěn)定遺傳高表達,而外源基因拷貝數(shù)過多(達4~5)往往使外源基因不表達甚至導致基因沉默[25-26]。因此在獲得陽性過表達轉基因植株后,對其外源基因拷貝數(shù)進行鑒定分析是必要的。
本研究對外源基因拷貝數(shù)的檢測所采用的內參基因為水稻特有的單拷貝基因SPS[24],轉基因植株的標志基因是植物表達載體的標志基因HYG,也是轉基因植物篩選常用的標志。檢測結果顯示,OsRhoGDI2在過表達轉基因水稻中均為單拷貝,提示基因能夠穩(wěn)定遺傳和表達,這與筆者所在實驗室對其T1代和T2代的連續(xù)篩選結果是一致的,陽性率均在85%以上。
本研究采用基因組DNA擴增的方法,首先檢測了OsRhoGDI2過表達植物載體是否穩(wěn)定整合到水稻基因組中,進而采用半定量RT-PCR、實時熒光定量PCR技術在轉錄水平檢測了OsRhoGDI2在轉基因水稻中的表達水平,并通過外源基因拷貝數(shù)的測算,證實外源OsRhoGDI2基因整合到水稻基因組中的拷貝數(shù)均為1。本研究篩選和鑒定了6個獨立的OsRhoGDI2過表達轉基因株系,為后續(xù)以這些材料開展對OsRhoGDI2的功能鑒定奠定了良好的基礎。
參考文獻:
[1]Kost B,Lemichez E,Spielhofer P,et al. Rac homologues and compartmentalized phosphatidylinositol 4,5-bisphosphate act in a common pathway to regulate polar pollen tube growth[J]. Journal of Cell Biology,1999,145(2):317-330.
[2]Molendijk A J,Bischoff F,Rajendrakumar C S,et al. Arabidopsis thaliana Rop GTPases are localized to tips of root hairs and control polar growth[J]. EMBO Journal,2001,20(11):2779-2788.
[3]Jones M A,Shen J J,F(xiàn)u Y,et al. The Arabidopsis Rop2 GTPase is a positive regulator of both root hair initiation and tip growth[J]. The Plant Cell,2002,14(4):763-776.
[4]Fu Y,Wu G,Yang Z B. Rop GTPase-dependent dynamics of tip-localized F-actin controls tip growth in pollen tubes[J]. Journal of Cell Biology,2001,152(5):1019-1032.
[5]Fu Y,Li H,Yang Z B. The ROP2 GTPase controls the formation of cortical fine F-actin and the early phase of directional cell expansion during Arabidopsis organogenesis[J]. The Plant Cell,2002,14(4):777-794.
[6]Delmer D P,Pear J R,Andrawis A,et al. Genes encoding small GTP-binding proteins analogous to mammalian rac are preferentially expressed in developing cotton fibers[J]. Molecular & General Genetics,1995,248(1):43-51.
[7]Trotochaud A E,Hao T,Wu G,et al. The CLAVATA1 receptor-like kinase requires CLAVATA3 for its assembly into a signaling complex that includes KAPP and a Rho-related protein[J]. The Plant Cell,1999,11(3):393-406.
[8]Kawasaki T,Henmi K,Ono E,et al. The small GTP-binding protein rac is a regulator of cell death in plants[J]. Proceedings of the National Academy of Sciences of the United States of America,1999,96(19):10922-10926.
[9]Kawasaki T,Koita H,Nakatsubo T,et al. Cinnamoyl-CoA reductase,a key enzyme in lignin biosynthesis,is an effector of small GTPase Rac in defense signaling in rice[J]. Proceedings of the National Academy of Sciences of the United States of America,2006,103(1):230-235.
[10]Fujiwara M,Umemura K,Kawasaki T,et al. Proteomics of Rac GTPase signaling reveals its predominant role in elicitor-induced defense response of cultured rice cells[J]. Plant Physiology,2006,140(2):734-745.
[11]Schultheiss H,Hensel G,Imani J,et al. Ectopic expression of constitutively activated RACB in barley enhances susceptibility to powdery mildew and abiotic stress[J]. Plant Physiology,2005,139(1):353-362.
[12]Dermardirossian C,Bokoch G M. GDIs:central regulatory molecules in Rho GTPase activation[J]. Trends in Cell Biology,2005,15(7):356-363.
[13]Dovas A,Couchman J R. RhoGDI:multiple functions in the regulation of Rho family GTPase activities[J]. The Biochemical Journal,2005,390:1-9.
[14]Klahre U,Becker C,Schmitt A C,et al. Nt-RhoGDI2 regulates Rac/Rop signaling and polar cell growth in tobacco pollen tubes[J]. Plant Journal,2006,46(6):1018-1031.
[15]Bischoff F,Vahlkamp L,Molendijk A,et al. Localization of AtROP4 and AtROP6 and interaction with the guanine nucleotide dissociation inhibitor AtRhoGDI1 from Arabidopsis[J]. Plant Molecular Biology,2000,42(3):515-530.
[16]Kieffer F,Elmayan T,Rubier S,et al. Cloning of Rac and Rho-GDI from tobacco using an heterologous two-hybrid screen[J]. Biochimie,2000,82(12):1099-1105.
[17]梁衛(wèi)紅,唐朝榮,吳乃虎. 兩種水稻GDP解離抑制蛋白基因的分離及特征分析[J]. 中國生物化學與分子生物學報,2004,20(6):785-791.
[18]Carol R J,Takeda S,Linstead P,et al. A RhoGDP dissociation inhibitor spatially regulates growth in root hair cells[J]. Nature,2005,438(770):1013-1016.
[19]Wu Y X,Zhao S J,Tian H,et al. CPK3-phosphorylated RhoGDI1 is essential in the development of Arabidopsis seedlings and leaf epidermal cells[J]. Journal of Experimental Botany,2013,64(11):3327-3338.
[20]彭威風,梁衛(wèi)紅. 水稻OsRhoGDI2蛋白生物信息學分析及亞細胞定位研究[J]. 生物技術通報,2010(5):82-86,92.
[21]Huang J J,Zhang J,Hao Y F,et al. Distinct expression patterns of the GDP dissociation inhibitor protein gene (OsRhoGDI2) from Oryza sativa during development and abiotic stresses[J]. Biologia,2016,71(11):1230-1239.
[22]Livak K J,Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods,2001,25(4):402-408.
[23]楊立桃,趙志輝,丁嘉羽,等. 利用實時熒光定量PCR方法分析轉基因水稻外源基因拷貝數(shù)[J]. 中國食品衛(wèi)生雜志,2005,17(2):140-144.
[24]Ding J Y,Jia J W,Yang L T,et al. Validation of a rice specific gene,sucrose phosphate synthase,used as the endogenous reference gene for qualitative and real-time quantitative PCR detection of transgenes[J]. Journal of Agricultural&Food Chemistry,2004,52(11):3372-3377.
[25]Flavell R B. Inactivation of gene expression in plants as a consequence of specific sequence duplication[J]. Proceedings of the National Academy of Sciences of the United States of America,1994,91(9):3490-3496.
[26]Vaucheret H,Béclin C,Elmayan T,et al. Transgene-induced gene silencing in plants[J]. The Plant Journal,1998,16(6):651-659.

