吉林省四平地區粉黏壤土微生物群落結構與環境因子的關系
黃冠南 丁美月 馬金才


摘要:微生物作為土壤中的重要組成部分,對土壤生態系統中的物質循環和能量流動有著至關重要的作用。吉林四平地區是東北地區三大糧倉之一,該地區粉黏壤土的微生物群落結構情況雖然重要,卻鮮有人進行研究。在該地區的農業土地區域采集6個土壤樣品點,應用二代高通量測序技術進行分析,并利用樣品中的優勢類群與土壤理化性質的Pearson相關性分析、典范對應分析(CCA)以及方差分解分析(VPA),以確定該地區微生物群落結構與環境因子的關系。分析結果顯示,共同影響細菌、真菌優勢類群的環境因素是pH值和銨態氮含量。方差分解分析(VPA)結果則又進一步指出,銨態氮含量、土壤pH值及土壤黏粒程度分別解釋了15.73%、22.84%、16.81%的細菌群落結構組成變化原因;銨態氮含量、土壤pH值、溶解性有機碳含量分別解釋了24.32%、20.74%、22.37%的真菌群落結構組成變化原因。通過二代高通量測序技術以及統計學研究,可以為東北地區粉黏壤土中的微生物群落結構與環境因子的關系研究提供一些新的理論依據。
關鍵詞:Illumina MiSeq高通量測序;粉黏壤土;細菌;真菌;優勢類群
中圖分類號: X172 ?文獻標志碼: A ?文章編號:1002-1302(2019)14-0306-04
土壤作為生態系統中的重要組成部分之一,是人類與動植物生存繁衍的基礎,也是土壤微生物的附著生境,并為其提供了較為豐富的食物。微生物在調節地球生物化學營養物質的循環和生態系統功能等方面起著至關重要的作用[1-2]。由于近幾年來我國對于糧食產量的需求變大,東北農作耕地作為我國重點保護的土壤生態資源,其墾殖面積不斷擴增,濫用施肥的情況也日漸凸顯,使得黑土資源快速衰退的情況愈發惡化[3]。土壤微生物作為土壤結構中極其重要的組成元素,也同時依賴于土壤的生態環境。近幾年來,高通量測序技術的日臻完善與成熟發展,為土壤微生物群落結構及多樣性的研究搭建了更為寬廣的平臺和途徑,使得人們對于土壤中細菌以及真菌的認識愈加豐富[4]。
Illumina MiSeq高通量測序技術[5]具有低成本、低錯誤率、高通量等優點,目前已經成為分析自然界中細菌、真菌等微生物的群落結構組成以及相對豐度的重要工具。由于目前東北四平地區農耕地中的微生物群落結構組成和多樣性以及它們受到環境因子的影響情況鮮有人報道。本研究旨在探究吉林四平地區粉黏壤土[6]的微生物群落結構及其與環境因子的關系。
1 材料與方法
1.1 研究區域概況與樣品采集
四平市是吉林省的第三大城市,也是東北地區的農業重點區域,地處松遼平原中部腹地。四平市在地理上可劃分為平原、丘陵2種地帶,該地區的土地類型有著土地資源多樣化、地貌結構復雜化、地域差異顯著化等特征[7]。
土壤樣品于2017年10月采集于吉林省四平地區的農作耕地區域。采用五點取樣法采樣,將樣品置于冰袋中冷藏,并迅速帶回實驗室,于-80 ℃條件下保存,以便后續進行土壤物理化學性質的測定。土壤樣品的基本信息如表1所示。
1.2 土壤理化性質的測定
土壤樣品經過風干以及過篩后,進行以下理化性質的分析:pH值,使用pH計進行測定;水溶性有機碳(簡稱DOC)含量(mg/kg),使用總有機碳分析儀(SSM-5000A,日本)測定;總氮(簡稱TN)含量(mg/kg),通過過硫酸鉀(K2S2O8)氧化分光光度法測定;銨態氮(簡稱NH4+-N)含量(mg/kg)、硝態氮(簡稱NO3--N)含量(mg/kg),分別使用納氏試劑分光光度法和雙波長比色法測定;總溶解性磷(簡稱TDP)含量(mg/kg),采用鉬銻抗比色法測定;土壤質地(黏粒、粉粒、沙粒含量,%),采用激光粒度分布儀(Bettersize 2000,丹東市百特儀器有限公司)測定。
1.3 總DNA的提取及16S rRNA基因的PCR擴增
土壤總DNA基因組的提取是采用DNA提取試劑盒進行的。目標片段的PCR擴增通常以微生物核糖體RNA等能夠反映菌群組成和多樣性的目標序列為靶點,根據序列中的保守區域設計相應引物,并添加樣本特異性條形碼序列,進而對rRNA基因可變區(單個或連續多個)或特定基因片段進行PCR擴增。PCR擴增采用NEB公司的Q5高保真DNA聚合酶,并嚴格控制擴增循環數,使循環數盡可能低,同時也保證同一批樣本的擴增條件一致。
1.4 統計學分析
應用軟件Mothur進行統計并計算4種常用的生物多樣性指數。將優勢菌門與土壤理化因子結合進行Pearson相關分析。將細菌、真菌門水平上的分類信息用Origin軟件制圖以進行展示,進一步繪制優勢菌門及其優勢綱的相對豐度圖,細菌、真菌的分類信息用Origin 9.0制圖。用軟件R(Pheatmap)繪制CCA、VPA圖。使用SPSS 19.0(IBM,美國)開展優勢類群與土壤理化性質的Pearson相關分析。應用 R 3.3.2 調用vegan程序包進行相關統計學分析。
2 結果與分析
2.1 土壤理化性質
經土壤質地識別系統軟件(國際制標準)測定,吉林四平地區的土壤類型均為粉黏壤土。本研究采集的6個土壤樣品的基本理化性質如表2所示。
2.2 微生物多樣性指數分析
微生物多樣性指數分析是研究土壤微生物多樣性的重要手段之一。對于常用的幾種多樣性指數,一般來說,Chao1或ACE指數與群落的豐富度呈正相關,即Chao1或ACE指數越大,表明該群落的豐富度越高[8]。在本研究中采集到的土壤樣品的微生物多樣性指數如表3所示。其中細菌中SP5樣品的ACE指數高達3 049.55,Chao1指數高達2 906.50,說明在SP5號樣品中的細菌群落豐富度最高。SP4號樣品真菌群落的Chao1指數、ACE指數分別為553.01、553.58,均是同類中最高的,說明SP4號樣品真菌群落的豐富度最高。細菌群落中的SP3、SP5,真菌群落中的SP2、SP3的群落豐富度、均勻度都相對較好。細菌和真菌群落SP2、SP6樣品的Chao1指數或ACE指數及Shannon指數都較低,說明它們與其他樣品相比,群落豐富度和均勻度都是較低的。一般而言,Simpson指數對群落的均勻度以及優勢OTU(最小操作分類單元)更為敏感,而Shannon指數則是對群落的豐富度及稀有OTU更為敏感[9]。
2.3 土壤微生物的群落結構組成與分析
將采集的土壤樣品進行處理后,共獲得細菌25門97綱118目402科801屬和未確定類群。細菌優勢門的相對豐度如圖1所示,可以看出,優勢細菌門有放線菌門(Actinobacteria,相對豐度為32.95%)、變形菌門(Proteobacteria,相對豐度為29.73%),這二者在細菌優勢菌門中占據主導地位,而其余優勢菌門中,芽單胞菌門(Gemmatimonadetes)的相對豐度為11.15%,酸桿菌門(Acidobacteria)則為8.87%。
放線菌門亞綱水平上的相對豐度如圖2所示。可以看出,放線菌門下優勢亞綱中放線菌亞綱(Actinobacteria)的相對豐度達到了58.43%,在放線菌門中的亞綱水平上占據主導地位,嗜熱油菌綱(Thermoleophilia)的相對豐度為 16.71%,醋酸菌亞綱(Acidimicrobiia)的相對豐度為12.26%,紅細菌亞綱(Rubrobacteria)的相對豐度為8.26%。其中放線菌亞綱(Actinobacteria)為最優勢亞綱,其相對豐度也最高。
變形菌門亞綱水平上相對豐度如圖3所示。可以明顯看出,α-變形菌綱(α-alphaproteobacteria)最具優勢,相對豐度達到62.89%。而變形菌門亞綱水平上的其他亞綱排序依次為β-變形菌綱(β-betaproteobacteria)、δ-變形菌綱(δ-deltaproteobacteria)、γ-變形菌綱(γ-gammaproteobacteria),相對豐度分別為14.90%、12.28%、9.92%。
由圖4可見, 采集的土壤樣品經處理后共獲得真菌7門31綱92目190科326屬和未確定類群。其中優勢的真菌門有子囊菌門(Ascomycota),相對豐度為89.20%,在優勢真菌門中占據主導地位。而擔子菌門(Basidiomycota)則占真菌總體的8.16%。
由于子囊菌門的相對豐度達到了89.20%,筆者對于其下 屬的優勢亞綱進行分析,發現其中糞殼菌綱(Sordariomycetes)的相對豐度達到了51.32%,從圖5可以明顯看出。但是其余真菌優勢門在采集的樣品中的比例體現得并不相對均勻,經過分析統計得出,散囊菌綱(Eurotiomycetes)占13.40%,座囊菌綱(Dothideomycetes)占11.45%,未知菌綱占20.60%。
2.4 優勢類群與土壤理化性質的Pearson相關性分析
將細菌(相對豐度大于5%)和真菌(相對豐度大于1%)的優勢菌門與土壤中各項理化性質作Pearson相關性分析。表4結果顯示,細菌群落中的酸桿菌門(Acidobacteria)與 NH4+-N含量呈顯著負相關,芽單胞菌門(Gemmatimonadetes)與pH值呈顯著負相關,其余的相關性均不顯著。而在真菌群落中,子囊菌門(Ascomycota)與NH4+-N含量呈顯著正相關,而擔子菌門(Basidiomycota)則與 NH4+-N 含量呈顯著負相關(表5)。
2.5 土壤理化性質對微生物群落結構的影響
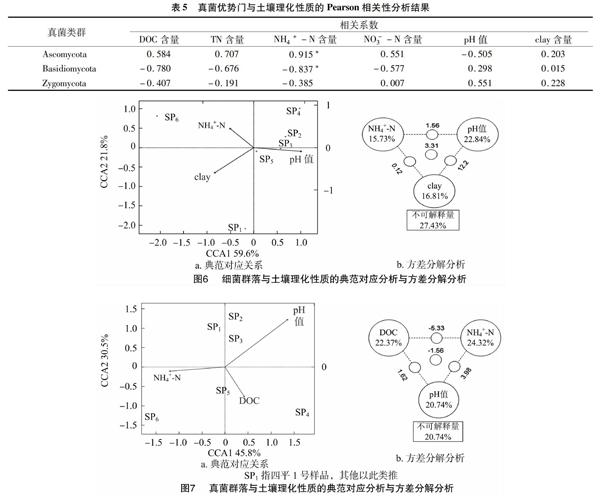
對于微生物群落組成與環境因子之間的關系,筆者采用典范對應分析(CCA)進行研究[10]。其中主要環境因子包括pH值、TN含量、DOC含量、NH4+-N含量、NO-3-N含量和clay含量等。通過典范對應分析,得到對細菌群落組成影響較大的因素分別有NH4+-N含量、pH值和clay含量(圖6),對真菌群落組成影響較大的因素則為DOC含量、pH值、NH4+-N含量(圖7)。
結合Pearson相關分析結果和細菌、真菌的CCA結果,選取這3個主要環境因素進行方差分解分析(VPA),以進一步量化主要環境因素對微生物群落結構組成影響的具體貢獻。在細菌群落中,NH4+-N含量、pH值和黏粒含量這3個變量共解釋了72.57%的細菌群落結構變化原因,其中 NH4+-N 含量、pH值和黏粒含量分別解釋了15.73%、22.84%、16.81%的細菌群落結構變化原因。在真菌微生物群落中,DOC含量、NH4+-N含量和pH值這3個變量共解釋了79.26%的真菌群落結構變化原因,DOC含量、pH值、NH4+-N 含量則分別解釋了22.37%、20.74%、24.32%的真菌群落結構變化原因。
3 結論與討論
微生物組成與土壤理化性質的Pearson相關性分析結果顯示,影響細菌各優勢類群相對豐度的環境因素主要有NH4+-N含量、pH值和黏粒含量等,影響真菌各優勢類群相對豐度的環境因素主要有DOC含量、pH值、NH4+-N含量。而反映微生物群落的總體水平受環境因素影響的CCA結果和Pearson相關性分析表明,NH4+-N含量、pH值是影響沙土中細菌、真菌群落結構組成的共同環境因素。此外,細菌微生物群落結構組成還受黏粒含量的影響,真菌微生物群落結構組成還受DOC影響。
VPA結果又進一步指出3個主要因素在細菌微生物群落、真菌微生物群落多樣性變化中的具體貢獻。其中pH值可能對細菌群落結構產生影響,也可能通過改變其他土壤理化性質,包括陽離子金屬溶解度、有機碳含量、土壤水分狀況和電導率等間接影響微生物群落結構。但是,由于本研究中pH值的分布范圍較窄,所以得出的結果應謹慎對待。
本研究應用二代高通量測序技術對從吉林四平地區粉黏壤土采集的6個土壤樣品的微生物多樣性進行了16S rRNA基因測序及整理,并運用統計學方法進行了分析[11-14]。本研究初步掌握了吉林四平地區粉黏壤土中細菌和真菌微生物群落結構組成和多樣性結構及其受環境因子影響的情況以及與主要影響因子之間的關系,并進一步進行相關性分析,從而為該地區農業耕地微生物群落結構與環境因子的關系提供更為重要的依據。
參考文獻:
[1]馮 瑞. 三峽庫區消落帶的微生物群落結構、碳氮礦化和硝化作用的研究[D]. 重慶:西南大學,2016.
[2]汪海燕,王 輝,董元華,等. 土壤生態環境質量評價指標及方法研究概述[J]. 安徽農業科學,2013,41(20):8575-8577,8652.
[3]崔 明,蔡強國,范昊明. 東北黑土區土壤侵蝕研究進展[J]. 水土保持研究,2007,14(5):29-34.
[4]劉玉莉,魏 霜,劉碧琳,等. 第二代高通量測序技術用于進口細菌型微生物肥料菌群分析[J]. 安徽農業科學,2017,45(28):146-147,150.
[5]李 橋. 基于高通量測序技術下土壤微生物群落結構的研究[D]. 濟南:山東師范大學,2014.
[6]郭彥彪,戴 軍,馮 宏,等. 土壤質地三角圖的規范制作及自動查詢[J]. 土壤學報,2013,50(6):1221-1225.
[7]秦天柱,陳長勝,李德恒. 吉林省四平地區農業氣候資源特征分析[J]. 氣象災害防御,2017,24(2):43-48.
[8]Gasc A,Pavoine S,Lellouch L,et al. Acoustic indices for biodiversity assessments:analyses of bias based on simulated bird assemblages and recommendations for field surveys[J]. Biological Conservation,2015,191:306-312.
[9]Bandeira B,Jamet J L,Jamet D. Mathematical convergences of biodiversity indices[J]. Ecological Indicators,2013,29:522-528.
[10]蔡樹美,徐四新,張翰林,等. 灘涂土壤固氮菌群落與環境因子的典范對應分析[J]. 土壤,2017,49(6):1159-1165.
[11]夏圍圍,賈仲君. 高通量測序和DGGE分析土壤微生物群落的技術評價[J]. 微生物學報,2014,54(12):1489-1499.
[12]張彩霞. 新一代高通量測序技術研究土壤微生物群落結構對環境條件的響應[D]. 南京:南京農業大學,2012.
[13]王佩雯,朱金峰,陳 征,等. 高通量測序技術下連作植煙土壤細菌群落與土壤環境因子的耦合分析[J]. 農業生物技術學報,2016,24(11):1754-1763.
[14]Chen Z B,Xia T Y,Wang D K,et al. Diversity analysis of endophytic fungi in Mentha using illumina MiSeq high-throughput sequencing technology[J]. Medicinal Plant,2016,7(1/2):46-50.

