應用PacBio SMRT測序技術分析蒙古傳統奶酪細菌多樣性
任冬艷,莫藍馨,靳昊,潘琳,馬騰,于潔,孟和畢力格
(內蒙古農業大學乳品生物技術與工程教育部重點實驗室,農業農村部奶制品加工重點實驗室內蒙古自治區乳品生物技術與工程重點實驗室,呼和浩特010018)
0 引 言
傳統工藝制作的蒙古奶酪制品是蒙古民族人們日常飲食中不可缺少的部分。蒙古傳統奶酪的制作工藝不同于歐洲的酶凝乳奶酪,將鮮乳置于室溫下自然發酵酸化凝固,然后加熱凝乳排出乳清后放進模具或木盤中擠壓成型,自然干燥后即可食用[1]。在奶酪制作過程中,乳酸菌等多種微生物參與其中,對奶酪的口感、風味、質地、顏色、營養特性和食用安全性都有很大的影響[2]。因此,了解蒙古傳統奶酪的細菌多樣性對于傳統奶酪的品質改良及其食用安全評估具有重要意義。
本研究通過PacBio SMRT 測序技術分析蒙古奶酪樣品中的細菌多樣性。并從公共數據庫中下載了19 份來自布里亞特和意大利傳統奶酪的16S rRNA 基因序列,與蒙古奶酪進行了比較分析,旨在揭示不同地區奶酪樣品的細菌群落結構差異性。
1 實 驗
1.1 材料與儀器
(1)樣品采集。本研究所用的9 份傳統奶酪樣品(R1-R9)采集自蒙古地區的9 個牧民家庭,樣品采集后立即加入保護劑,迅速冷凍、密封,并盡快運回實驗室進行后續試驗。
(2)試劑。OMEGA eZNA Soil DNA Kit 試劑盒,美國OMEGA 公司;蛋白酶K,北京全式金生物技術有限公司;KAPA HiFiHot Start Ready Mix PCR Kit,美國KAPA 公司;PCR 試劑,大連TaKaRa 公司;5×TBE 電泳緩沖液(Tris 堿54 g、Na2EDTA·2H2O 3.72 g、硼酸27.5 g,定容至1 000 mL,pH 8.0)、1.0%的瓊脂糖膠(1.0 g 瓊脂糖溶于100 mL 0.5×TBE 緩沖液),天津基準化學試劑公司。
(3)儀器。Eppendorf 5810R 臺式高速冷凍離心機,德國Eppendorf公司;CDS8000型UPV 凝膠成像分析系統,北京賽智創業科技有限公司;DYY-12 電泳儀,北京六一儀器廠;ND-1000 型微量紫外分光光度計,美國Nano Drop 公司;AB/Life 梯度PCR 儀,美國AB 公司;Pacifico SMRT RS II 測序平臺,美國PB 公司。
1.2 方法
1.2.1 樣品DNA 提取
取2 g 碾碎的奶酪樣品,用OMEGA eZNA Soil DNA Kit 試劑盒提取樣品中的基因組DNA。對樣品DNA 的完整度、純度以及濃度進行檢驗,經檢驗合格的DNA 用于后續實驗。
1.2.2 樣品16SrRNA 基因片段擴增
DNA 擴 增 采 用 細 菌 16S 通 用 引 物27F(5-AGAGTTTGATCMTGGCTCAG-3’)和1492R(5’-ACCTTGTTACGACTT-3')。為了區別不同樣品將每對引物分別加有16個堿基長度的barcode。
PCR 反應擴增體系為50 μL:正、反向引物(濃度10 μmol/L)各2 μL,模板DNA(<100 ng)1 μL,1×PCRBIO buffer 10 μL,Taq DNA polymerase 1 μL,ddH2O 34 μL;
擴增條件:95 ℃預變性5 min,95 ℃變性30 s,60 ℃退火45 s,72 ℃延伸1 min,30 個循環,72 ℃末端延伸7 min。
1.2.3 PacBio SMRT 三代測序
PCR 反應產物經Agilent DNA 1000 Kit 和Agi?lent 2100 Bioanalyser 檢驗合格后,使用Pacific Biosci?ences Template Prep Kit 2.0 試劑盒構建文庫,用PacBio RS II儀器P6-C4試劑上機測序。
1.2.4 序列質控
使用RS_ReadsOfinsert.1 對原始下機數據進行測序質量控制,要求:(1)插入片段重復測序的次數≥5;(2)最小預測精確度為90;(3)最小插入序列長度為1 400 nt 為生物學中描述DNA 常用的單位,指核苷酸數,通常用于描述單鏈,如RDA,primer等。(4)最大序列長度為1 800 nt[3]。
1.2.5 生物信息學分析
質控合格的序列根據barcode 序列拆分不同的樣本,并去除barcode 和引物序列以進行后續的分析。篩選意大利和布里亞特樣品的16S rRNA 基因序列超過450 bp 的基因片段。應用QIIME 平臺(v1.70),對高質量的序列進行生物信息學分析,主要包括:(1)PyNAST 校準并排齊序列,在100%相似性下進行UCLUST 歸并后,在97%相似性下聚類獲得分類操作單元(Operational taxonomic units,OTU)[4];(2)通過chimeraslayer 去除嵌合體序列后,基于RDP(ribosomal database project, release11.5) 和 Greengenes(Release 13.8)數據庫對OTU 代表性序列進行同源性比對,確定每個OTU 分類學地位[5];(3)使用FastTree 軟件繪制系統發育進化樹[6];(4)以所有樣品中測序量最低的樣品測序量為基準,計算各樣本香農指數(Shan?non-Wiener index)、辛普森指數(Simpson index)、超1指數(Chao1 index)和發現的物種數量指數(Observed species)用于評估各樣本α 多樣性。(5)使用UniFrac距離進行加權(weighted)和非加權(unweighted)的主坐標分析,揭示各個樣本之間菌群結構的差異。
1.2.6 數據處理
分別采用Wilcoxon 秩和檢驗和Kruskal-Wallis 多組檢驗比較樣品間差異。使用Origin 8.6 軟件和R 語言軟件對QIIME產生的結果進行分析和可視化處理。
2 結果與分析
2.1 蒙古奶酪中細菌豐度及多樣性
本研究通過超1 指數、發現物種數、香農指數和辛普森指數評估蒙古奶酪樣品細菌豐度和多樣性。樣品的測序序列信息和α 多樣性指數如表1 所示,樣品R1 細菌豐度及多樣性最高,R4 和R5 細菌豐度及多樣性最低。通過對測序所得序列進行篩選后,9 個蒙古奶酪樣品共獲得64,926 條高質量的序列,平均每個樣品產生7,214 條。按序列97%的相似度進行操作分類單元劃分及嵌合體檢查去除,將剩余的4,243 個OTUs 納入下一步分析,平均每個樣品剩余471 個OTUs。
為了直觀反映當前測序量和97%相似度水平上各樣品的生物多樣性,本試驗用稀疏曲線和香農多樣性曲線對各樣品文庫進行分析。從圖1 結果發現,在測序量較少時,發現OTU 的數量和香農多樣性指數隨著測序量的增加而增加,但隨著測序量繼續增大時,圖1(a)稀疏曲線中新增OTU 的數量逐漸變緩且最終沒有達到平衡狀態,說明隨著測序量增加有可能會有新的細菌種系型被發現。而從圖1(b)香農曲線中看,隨著測序量增加香農曲線已進入平臺期,說明在該測序深度下,可以充分展示樣品的細菌多樣性。因此本研究中細菌測序量滿足后續生物信息學分析的要求。
2.2 蒙古奶酪中細菌群落結構
在門水平上,9 份蒙古奶酪中共檢測到4 個細菌菌門,主要菌門是變形菌門(Proteobacteria)和厚壁菌門(Firmicutes),平均相對含量分別是85.59%和14.13%。
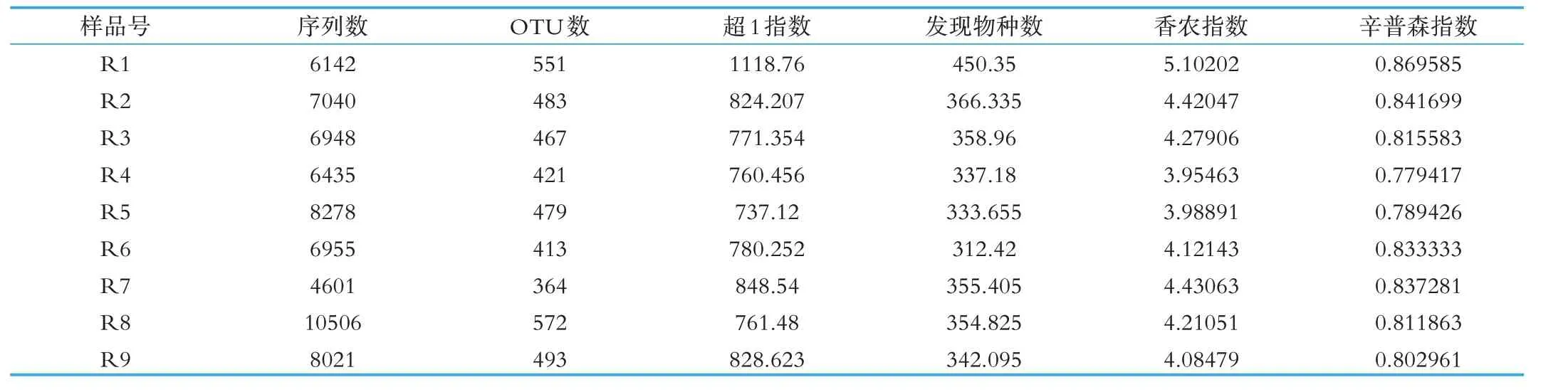
表1 蒙古奶酪樣品的測序序列信息和α 多樣性指數
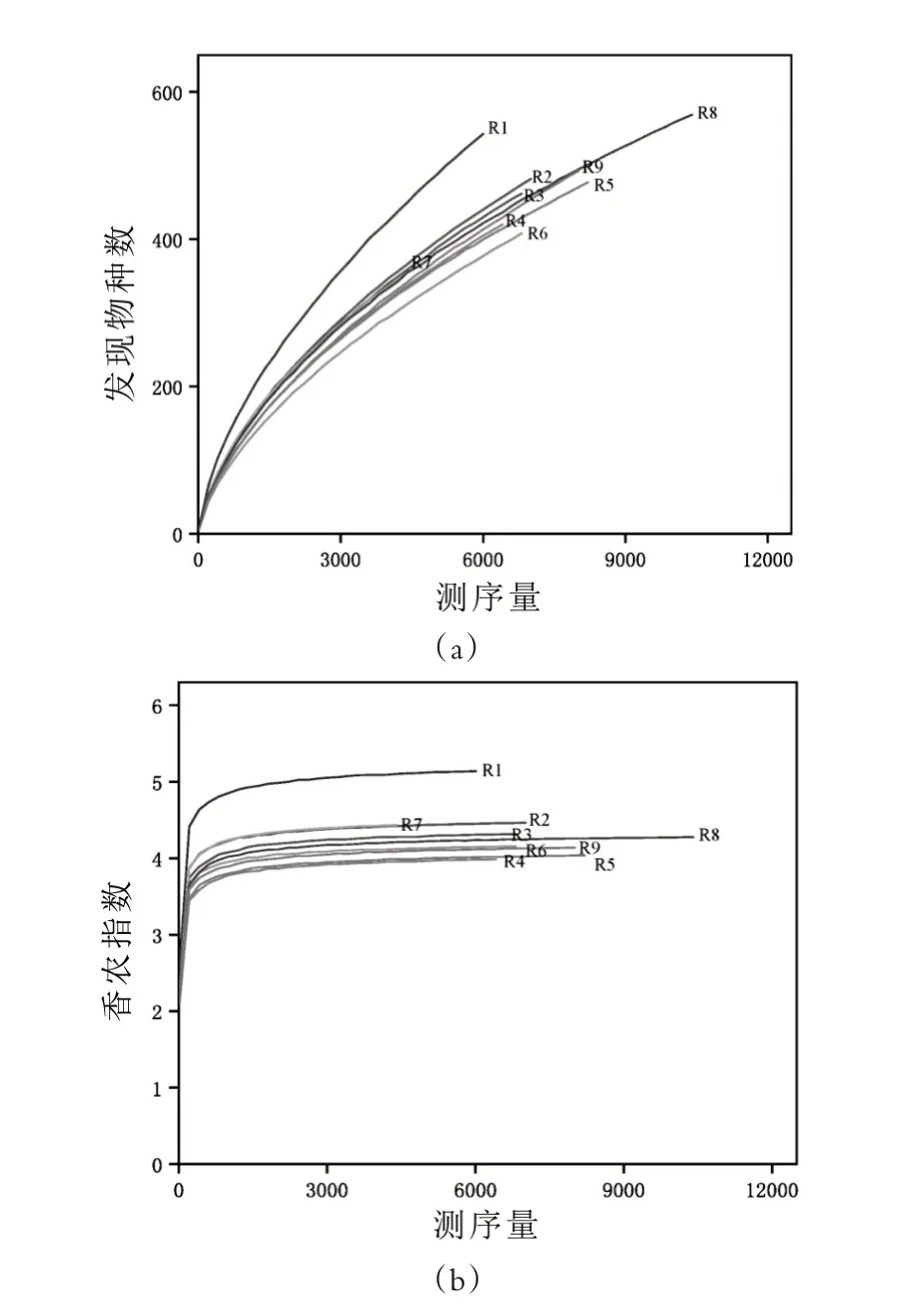
圖1 蒙古奶酪樣品稀疏曲線和香農曲線
在屬水平上,共鑒定到60 個細菌菌屬,主要菌屬有腸桿菌屬(Enterobacter)、鏈球菌屬(Strepto?coccus)、乳球菌屬(Lactococcus)、厭氧芽孢桿菌屬(Anoxybacillus)、乳桿菌屬(Lactobacillus)和芽孢桿菌屬(Bacillus),這些屬的平均相對含量分別為82.34%,5.74%,3.19%,2.27%,1.35%和1.17%,如圖2 所示。在屬水平上,9 份奶酪樣品的細菌群落結構表現出了一定的差異性。
在種水平上,共鑒定到76 個細菌菌種,主要菌種有霍氏腸桿菌(Enterobacter hormaechei)、嗜熱鏈球菌(Streptococcus thermophilus)、乳酸乳球菌(Lacto?coccus lactis)、蠟樣芽孢桿菌(Bacillus cereus)、瑞士乳桿菌(Lactobacillus helveticus)和德氏乳桿菌(Lactobacillus delbrueckii),上述菌種的平均相對含量分別為81.46%,5.65%,2.98%,1.05%,0.81%和0.51%,如圖3 所示。其中霍氏腸桿菌為所有樣品的優勢菌種,尤其在R4 和R5 樣品中相對含量最高,而嗜熱鏈球菌在R2,R3 和R8 樣品中相對含量最高,乳酸乳球菌在R6 和R7 樣品中相對含量最高,瑞士乳桿菌主要是在R1 樣品中,德氏乳桿菌主要在R8和R9樣品中相對含量最高。
2.3 蒙古奶酪與意大利和布里亞特奶酪細菌多樣性的比較
為了探究不同地區傳統奶酪中細菌菌群結構的差異,本研究從數據庫中下載了布里亞特和意大利奶酪的序列與蒙古奶酪進行了比較分析。首先,對三個地區奶酪樣品的豐度和多樣性進行了比較,發現意大利地區奶酪樣品的豐度和多樣性最高,而蒙古地區奶酪樣品的豐度和多樣性最低(如圖4)。
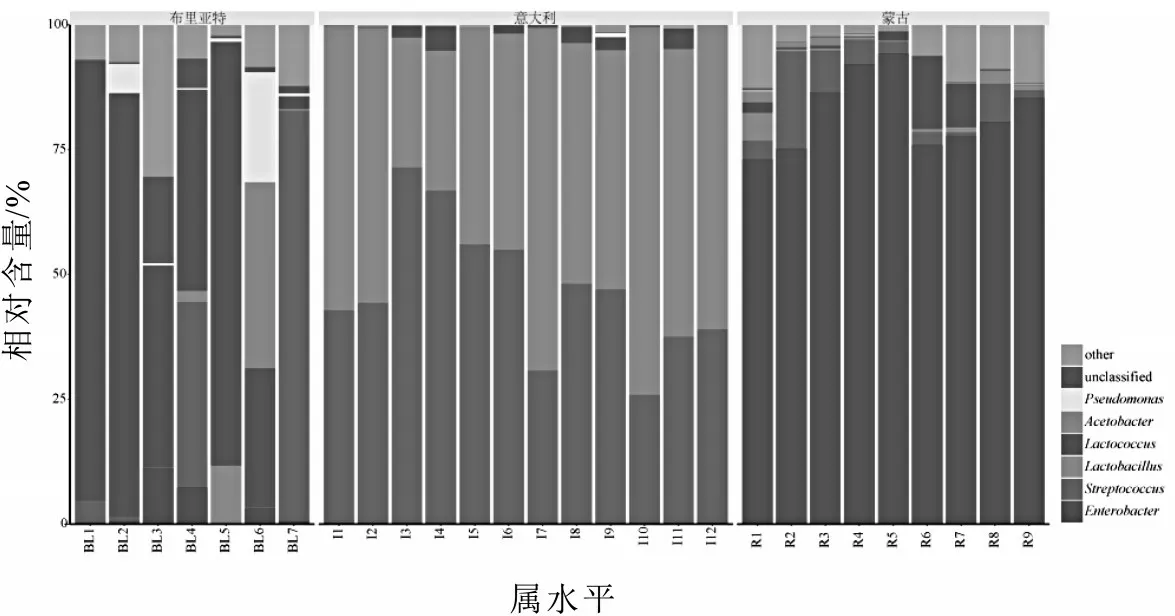
圖2 在屬水平奶酪樣品的細菌組成
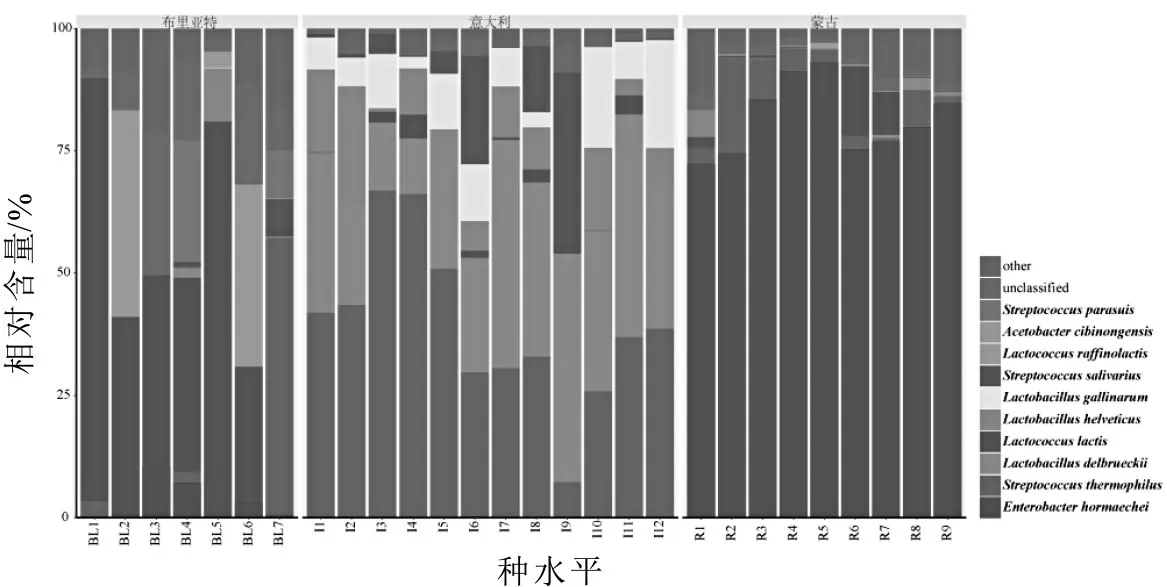
圖3 在種水平奶酪樣品的細菌組成
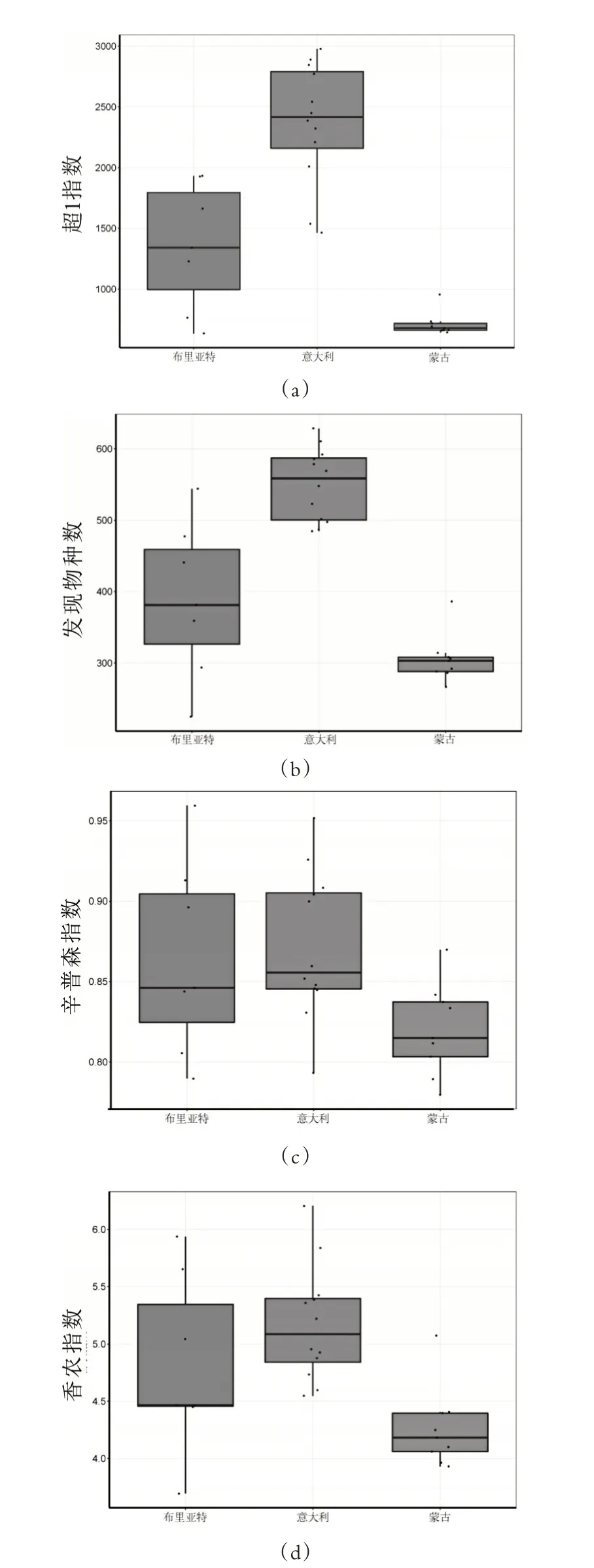
圖4 不同地區樣品測序量及α多樣性比較
然后,利用加權和非加權的UniFrac 距離進行了PcoA 主坐標分析來研究三個地區奶酪樣品的細菌菌群結構差異,在加權和非加權評分圖上(圖5),代表三個地區的點都明顯的區分開,說明三個地區的奶酪樣品間存在著顯著的差異(P<0.005)。
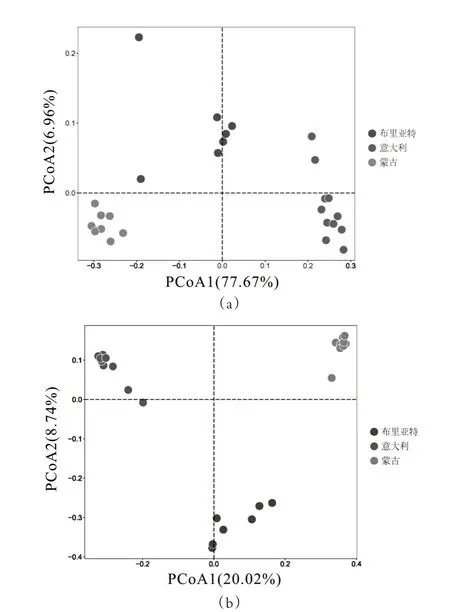
圖5 基于加權和非加權的UniFrac距離主坐標分析
為了進一步確定三個地區奶酪樣品的菌群結構差異體現在哪些菌株上,本研究采用Wilcoxon 秩和檢驗和Kruskal-Wallis 檢驗對三個地區奶酪樣品中菌種進行了比較分析,差異顯著且相對含量大于0.5%的菌種如圖所6 示。發現與其它兩個地區奶酪樣品相比較蒙古奶酪樣品差異顯著的菌種為霍氏腸桿菌和巴氏醋桿菌(P<0.05),布里亞特奶酪樣品與其它兩個地區奶酪樣品相比較差異顯著的菌種為乳酸乳球菌、棉子糖乳球菌和副豬鏈球菌(P<0.01),而意大利奶酪樣品與其他兩個地區奶酪樣品相比較差異顯著的菌種為嗜熱鏈球菌、德氏乳桿菌、瑞士乳桿菌和雞乳桿菌(P<0.005)。
3 討 論
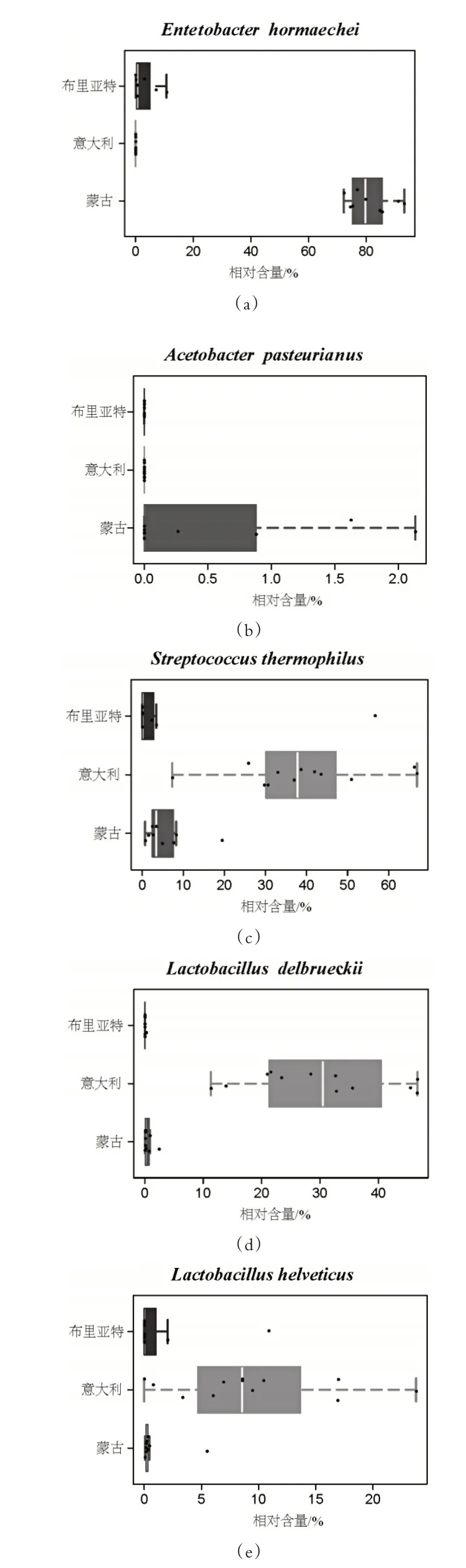
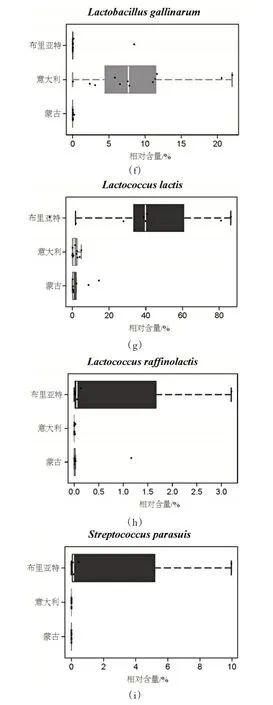
圖6 不同地區奶酪樣品中差異顯著的細菌比較分析
本研究采用SMRT 測序技術來描述蒙古奶酪的細菌多樣性,共鑒定到隸屬于4 個細菌菌門、60 個細菌菌屬的76 個細菌菌種。其中霍氏腸桿菌為蒙古傳統奶酪樣品的絕對優勢菌種,在每個樣品中都以較高的含量存在。除了霍氏腸桿菌之外,本研究還檢測到較高含量的嗜熱鏈球菌、乳酸乳球菌和瑞士乳桿菌等乳酸菌的存在。其中,嗜熱鏈球菌在樣品R2,R3,R4,R5 和R8 中相對含量較高,乳酸乳球菌在樣品R6和R7 中相對含量較高,瑞士乳桿菌在樣品R1 中相對含量較高。由此可見,本研究9 份蒙古奶酪的細菌群落結構既有共性也有一定的差異。可能是由于牧民制作奶酪的方法、時間以及環境的不同而導致的。可以影響奶酪的感官品質[10]。Tornadijo 等人發現在奶酪成熟初期腸桿菌活菌數可達106~107cfu/g,之后隨著奶酪的成熟逐漸減少[11]。霍氏腸桿菌的存在反映了蒙古傳統奶酪制作過程中不良的衛生條件。蒙古傳統奶酪主要是當地牧民以傳統的手工方法進行制作,沒有添加任何的發酵劑,這就意味著其質量和特性主要取決于內源微生物群,同時,奶酪加工過程發酵所處的開放自然環境以及所用到的器具都會對奶酪中微生物群落結構造成影響。在牛奶預處理、制作及儲存過程中,環境、擠奶人員和器具等衛生條件不足可能導致最終奶酪制品受到污染[12-13]。然而,利用純培養技術對本試驗所用樣品中的細菌進行分離鑒定,卻并未分離出霍氏腸桿菌,說明該菌在奶酪發酵初期確實存在過,而隨著加熱等工藝的應用,該菌已經不是活著的狀態。本研究中蒙古傳統奶酪的乳酸菌主要有嗜熱鏈球菌、乳酸乳球菌和瑞士乳桿菌。乳酸菌在奶酪生產過程主要通過發酵乳糖和水解蛋白來提高奶酪的感官特性。嗜熱鏈球菌是乳品工業中最重要的乳酸菌,它可以改善乳酸發酵的生態環境,并且通過抑制膽固醇合成酶的活性而降低膽固醇水平[14]。乳酸乳球菌是工業化生產酸奶及奶酪中常用到的發酵劑,具有快速酸化和凝乳能力,還可以通過促進乳酸形成來增強奶酪風味和質地[15],對奶酪的感官特性做出了顯著貢獻。瑞士乳桿菌也是一種常用的奶酪發酵劑,瑞士乳桿菌具有較高的蛋白水解活性,這一特性在減少奶酪苦味方面發揮了重要作用[16]。此外,一些瑞士乳桿菌的蛋白水解活性被認為在產生抗高血壓肽中具有重要作用[17]。
為了進一步了解蒙古奶酪的細菌群落結構特征,從公共數據庫中下載了布里亞特和意大利的傳統奶酪的16S rRNA 基因序列,并與本研究采集的蒙古奶酪進行了比較分析,PCoA 和MANOVA 分析表明不同地理位置的奶酪菌群結構存在差異。Zhong 等人對中國不同省份的自然發酵乳中細菌菌群進行了分析,跟本研究結果相似,發現乳制品中的細菌群落結構隨著地理位置的不同而表現出了差異性[18]。Xue 等人研究了中國不同地區的乳扇細菌群落結構,發現不同產地的乳扇有不同的細菌群落特征。對傳統發酵蔬菜食品[19]和自制發酵牛奶[20-22]進行細菌多樣性研究也可以得到相似的結論。地理因素主要是指海拔、氣候、溫度、大氣壓、陽光等各種環境因素的差異[23]。這些因素共同塑造了傳統乳制品的細菌群落。除了地理位置之外,奶酪的制作工藝和環境條件等因素也影響發酵后產品的微生物群落結構[24]。這些因素共同促使奶酪產品形成其獨特的細菌菌群結構。
4 結 論
本研究采用SMRT 測序技術對9 份蒙古奶酪樣品進行了細菌群落分析。共鑒定出4 個門,60 個屬,76 個種,其中霍氏腸桿菌(81.46%)、嗜熱鏈球菌(5.65%)和乳酸乳球菌(2.98%)為優勢菌種。通過將本研究產生的16S rRNA 基因序列與從公共數據庫中下載的序列進行比較分析,發現不同地區的奶酪在細菌群落組成確實存在差異。本研究中蒙古傳統奶酪除了檢測到嗜熱鏈球菌、乳酸乳球菌外,還檢測到大量霍氏腸桿菌存在的痕跡,反映了蒙古傳統奶酪在制作過程中不良的衛生條件,在傳統奶酪制作過程中應采取適當的措施預防污染,從而保證奶酪最終質量和產品感官特性。

