丙酮酸鐵氧化還原蛋白酶對藍藻光合生長速率影響的仿真分析
孟雅菲,申 鐵*,彭 棋,陳 震
(1.貴州師范大學貴州省信息與計算科學重點實驗室,貴陽 550000;2.貴州師范大學數學科學院,貴陽 550000)
清潔能源是一種不產生污染物的綠色能源,主要包括核能和可再生能源,可用在生活中的各個領域且對環境沒有影響.可再生能源是一類能在短時期內,經過自然界的不斷循環而不斷得到補充的能源,主要有太陽能、生物質能、風能等[1].隨著世界石油能源越來越匱乏和全球氣候的劇烈演變,清潔能源的開發和使用顯得尤為重要.因此,利用高科技手段獲取更多的清潔能源,已經成為世界上越來越多的國家能源戰略的重要內容.
生物質能作為自然界中一種可再生的清潔能源,具有可獲取性強、污染極小、資源總量大的特點,使它成為無可替代的可再生能源之一.生物質能是以生物質為載體,通過光合生物的光合作用將光能轉化為儲存在生物體內部的化學能,并可轉化成各種形態的燃料,用于日常生產生活.生物質能是唯一可以儲存和運輸的可再生能源.生物質能的有效開發、利用,對世界能源體系的建立和發展有著十分重要的意義.
光合作用是指光合生物利用可見光和光合色素所進行的一系列生化反應[2].這些光合生物通過光合作用將光能轉化為儲存在生物體內的生物質能,生物質能的產生速率受光合生長速率的影響.研究影響光合速率的因素,可以找到提高生物質能生成速率的方法,以便為實驗研究提供可靠依據.生物系統的各組分關系可以用網絡來表示,而基于蛋白質層次的代謝網絡成為比較熱門的研究領域,通過對代謝網絡的研究分析,可以推測影響光合生物生長速率的重要基因.
藍藻作為最早光合放氧的原核微生物,具有遺傳背景簡單且高效固碳的特點.藍藻中的丙酮酸鐵氧化還原蛋白酶(PFOR)是丙酮酸代謝過程中的關鍵酶,丙酮酸在PFOR的催化作用下生成乙酰輔酶A和CO2,其中乙酰輔酶A是脂肪酸、氨基酸和糖類等多種有機物代謝過程的中間產物,在許多的代謝過程中都起著至關重要的作用.乙酰輔酶A在生物醫學的研究之中已有廣泛應用,用于代謝疾病藥物以及抗癌藥物的研發;在三羧酸循環中也有重要作用,并且可作為合成聚酮化合物類藥物的前體分子.由于乙酰輔酶A的藥物研究價值,使得促進其合成的酶PFOR成為重要研究對象.有研究報道稱,酶PFOR廣泛存在于古細菌、真核生物和厭氧原生動物中,由pforA基因編碼,并在丙酮酸異化中起到關鍵的作用.酶PFOR中含有的Fe-S簇蛋白,影響并參與了生物的部分分解代謝途徑和合成代謝途徑,是重要的參與酶之一.在藍藻的光自養性生長過程中,是一種氧敏感酶;敲除酶PFOR會更利于藍藻的生長,使得光合生長速率提高[3].因此,對酶PFOR的功能研究有待進一步證實與探究.
實驗研究是單點式,逐步研究是一個假設驅動的過程,周期較長.利用計算、仿真等方法,則可以提供初步的猜想、解釋,也在研究基因對藍藻代謝的作用中有重要價值.本實驗采用通量平衡分析方法(FBA)、通量可變性分析方法(FVA)和多目標優化方法(MOO)來研究藍藻代謝網絡(iJB785模型)中酶與光合生長速率的關系,推測出影響光合生物生長速率的重要基因,從而對后續的實驗研究起指導性作用.
1 材料與方法
1.1 實驗環境及數據
主機配置:CPU為Intel Core i5-3230M,主頻2.60 G,內存12.0 GB.
實驗平臺:Matlab R2017a,Cobra Toolbox 2.0,libSBML 5.16.0,SBML Toolbox 4.1.0.
實驗數據:本實驗數據采用BIGG數據庫提供的代謝模型iJB785為實驗研究對象,包括藍藻中心代謝過程的全部信息[4].
1.2 方法介紹
1.2.1 通量平衡分析方法 通量平衡分析方法(Flux Balance Analysis,FBA)是一種基于約束的模擬方法,在假設代謝系統處于一個穩定狀態及目標函數達到最優的前提下,加入化學計量系數等約束條件,將代謝網絡封閉在一個可行區域,使用線性規劃(LP)方法得到最優解,并計算出該代謝網絡的通量分布[5].FBA方法可以用于計算、預測代謝流分布,并通過這些信息反映生物代謝系統的生理特性和評估影響生物生長的重要因素.
FBA方法是基于靜態的代謝網絡分析方法,廣泛用于生物代謝網絡中代謝物通量的研究,通過通量值可預測生物的生長速率和代謝物生成速率.FBA方法應用于研究的時間較早,后續的通量分析方法也是以其為基礎而進行更深入的實驗探究.目前,該方法已經在大腸桿菌、幽門螺桿菌等生物上成功應用,所得的實驗結果對生物代謝網絡的下一步研究提供更為科學的實驗依據.
1.2.2 通量可變性分析方法 通量可變性分析(Flux Variability Analysis,FVA)是基于靜態的、在 FBA方法基礎上發展而來的通量分析方法,在保證目標反應能夠達到最優的前提下,求得代謝網絡中每個反應的最小和最大通量值,并得到各個反應的通量可變范圍.
必需反應是指在代謝網絡達到最優的前提下,通量可變范圍總是大于0或總是小于0的反應,由于一個反應通常對應一個或多個同工酶基因,因此與這些反應相對應的基因并非反應所必需的全部基因.除了必需反應外的反應為非必需反應[6].通過FVA方法,可以刪除代謝系統中功能相同的基因,來縮小生物基因組的規模,并可以將代謝系統中的所有反應分為必需反應和非必需反應.然后利用單基因敲除方法,去除代謝系統中的某個基因,使該基因所催化的反應通量值變成0.如果計算出來的目標函數值維持原狀態不變,則該基因就是非必需基因;反之,如果發生了改變,則為必需基因[7].
1.2.3 多目標優化方法 多目標優化(Multi-objective Optimization,MOO)是同時考慮兩個或兩個以上的目標函數,并根據目標函數在給定的區域上獲得一組最優解的方法,得到的解代表不同目標函數之間的最佳權衡[8].
在自然界和細胞中,不存在單一的一個目標函數,而是不同的細胞目標是相互獨立且存在于不同的代謝反應中,通常需要在多個目標函數之中做權衡取舍,并在決策空間中獲得一個最優化目標.多目標優化的解決方案是一組無限點集,稱為帕累托最優集,因此可將MOO問題的結果表示為帕累托最優集[9].由于線性目標和凸解空間的存在,則可以利用ε約束法來獲得帕累托最優集,把多目標優化轉化為在目標最大化前提下的一個單目標問題,而另一個目標則合并為不等式約束ε,以此來獲得最終的最優解集,并根據所得出的最優解集進行結論性的分析和總結.
2 結果和討論
2.1 通量平衡分析方法
在如下的圖形處理中,由于藍藻的代謝網絡較為龐大,只能選取對比結果中的部分數據作圖分析.橫坐標代表iJB785模型代謝系統中849個反應的部分反應(90個反應),其中,第90個反應為目標函數(BIOMASS_1);縱坐標表示敲除酶PFOR前、后各個反應所對應的通量值(單位:mmol·h-1).
圖1是野生型代謝系統中部分反應FBA通量值和敲除酶PFOR后突變型代謝系統中部分反應FBA通量值的對比圖,綜合橫向和縱向分析,在敲除酶PFOR后的突變型代謝系統中,有大多數反應的通量受到了影響.由此可知,酶PFOR敲除后的代謝網絡,通過代謝物與輔酶耦連,對離其較遠的反應也會產生影響.
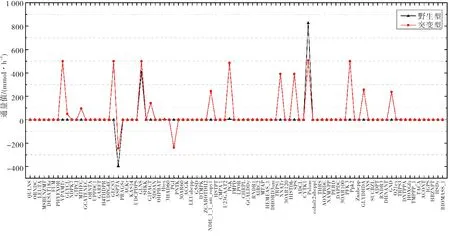
圖1 敲除酶PFOR后的突變型和野生型部分反應通量值的比較Fig.1 Comparison of partial reaction flux values with mutant and wild-type after knocking out the enzyme PFOR
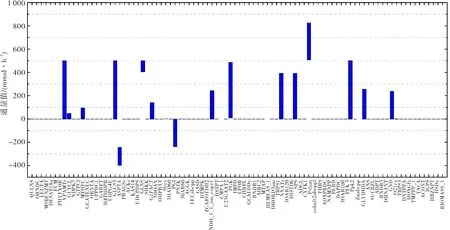
圖2 敲除酶PFOR后突變型和野生型部分反應通量差值Fig.2 Difference between partial reaction flux values with mutant and wild-type after knocking out the enzyme PFOR
圖2是敲除酶PFOR后突變型代謝系統與野生型代謝系統中部分反應FBA的差值,第90個反應為目標函數,可依據目標函數值的前后變化來推測敲除某種酶后,對代謝系統是否有影響.由圖1、圖2可知,野生型目標函數通量值為0.053 9 mmol·h-1,敲除酶PFOR后為0.053 9 mmol·h-1,且敲除酶PFOR前后,FBA通量差值為0 mmol·h-1,即目標函數值保持不變.因此,該代謝系統敲除酶PFOR后,不會對藍藻的光合生長速率產生影響[10].
綜上所述,藍藻代謝系統中敲除酶PFOR后,有多數反應的通量受到了影響,說明酶敲除后,通過代謝物與輔酶的耦連,對離其較遠的反應也會產生影響[11].酶PFOR敲除前后,目標函數通量值保持不變,說明酶PFOR不會對藍藻的光合生長速率產生影響,即在藍藻的光自養性生長過程中,酶PFOR不起作用,是一種氧敏感酶.
2.2 通量可變性分析方法
在如下的圖形處理中,由于藍藻的代謝網絡較為龐大,只能選取對比結果中的部分數據作圖分析.橫坐標代表iJB785模型中849個反應中的部分反應,其中,反應BIOMASS_1為目標函數;縱坐標代表各個反應所對應的FVA通量值(單位:mmol·h-1).
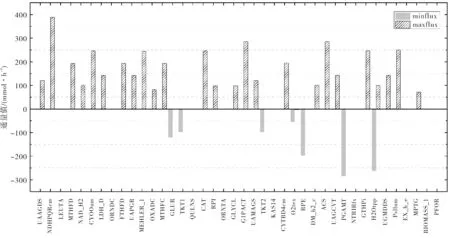
圖3 野生型代謝系統中部分反應FVA通量值Fig.3 Partially reacted FVA flux values in the wild-type metabolic system
圖3是野生型代謝系統中部分反應FVA通量值,即最小通量值(minflux)和最大通量值(maxflux).由圖可知,野生型代謝系統中酶PFOR的最小通量值為0 mmol·h-1,最大通量值為0 mmol·h-1.因此,可推測,酶PFOR的FVA通量值應保持在很小范圍內,數值大了不利于藍藻的生長.
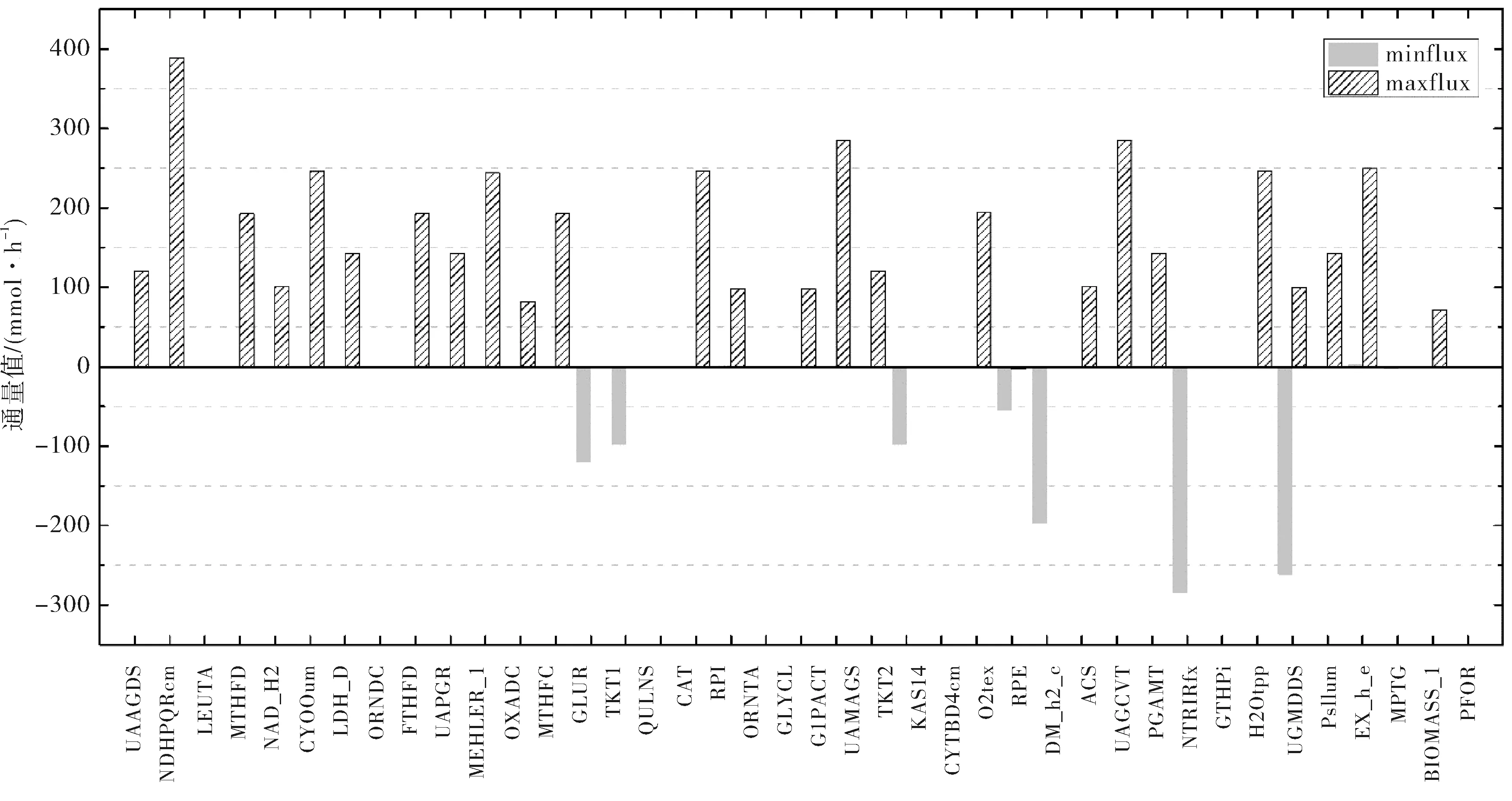
圖4 敲除酶PFOR后代謝系統中部分反應FVA通量值Fig.4 Partial reaction FVA flux value in the metabolic system after knocking out the enzyme PFOR
圖4是敲除酶PFOR后代謝系統中部分反應的最小通量值(minflux)和最大通量值(maxflux),其中BIOMASS_1是目標函數.綜合圖3和圖4可知,野生型和突變型代謝系統中目標函數minflux值均為0.053 9 mmol·h-1,maxflux均為0.053 9 mmol·h-1.由此可知,敲除代謝系統中通量值為0 mmol·h-1的酶PFOR后,目標函數值保持不變.因此,可推測酶PFOR是代謝系統中的非必需基因[12].
綜上所述,FVA方法能夠在已經得到FBA方法預測的最優解的前提下,得到代謝系統的通量變化范圍,即最小通量值和最大通量值.通過FVA方法的分析,可以將代謝系統中的所有反應分為必需反應和非必需反應,運用單基因敲除方法來篩選出非必需基因[13].依據本實驗所得到的數據,野生型代謝系統中酶PFOR的最小通量值為0 mmol·h-1,最大通量值為0 mmol·h-1;敲除通量值為0 mmol·h-1的酶PFOR后,目標函數值保持不變(0.053 9 mmol·h-1),那么可以推測,酶PFOR的通量值應保持在很小范圍內,數值大了不利于藍藻的生長;酶PFOR為該代謝系統的一個非必需基因.
2.3 多目標優化方法
在如下的圖形中,橫坐標表示酶PFOR不同的代謝速率(通量值,單位:mmol·h-1),縱坐標表示目標函數(BIOMASS_1)的生物量(單位:mmol·h-1).
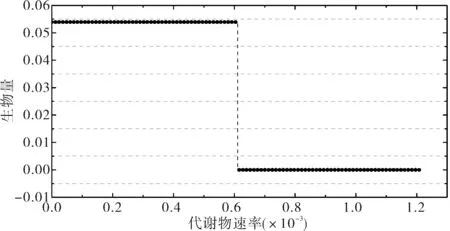
圖5 酶PFOR的代謝物速率與生物量產量的關系Fig.5 Relationship between metabolite rate and biomass yield of the enzyme PFOR
圖5是利用ε約束法[14]得到的酶PFOR代謝速率和目標函數(BIOMASS_1)生物質產量之間的關系.據圖分析,當酶PFOR的通量值在0~0.000 6 mmol·h-1的范圍變化時,目標函數的通量值保持在0.053 9 mmol·h-1不變,即改變酶PFOR的通量值界限,不會對藍藻的光合生長速率產生影響;當酶PFOR通量值在0.000 6~0.001 2 mmol·h-1的范圍變化時,目標函數的通量值保持在0 mmol·h-1不變.由此可知,當酶PFOR的通量值在一定范圍內變化時,對藍藻的光合生長速率沒影響(正常生長),但當酶PFOR的通量超過某一個值后,將會對藍藻的生長起抑制作用.
綜上所述,基因敲除仿真實驗是一種實驗結果全和無比較強烈的方法,這種方法比較容易使仿真結果產生失真,而多目標優化方法能更好的描述酶PFOR和光合生長速率的關系,使得仿真結果更加符合實際[15].由結果可知,酶PFOR的通量值應在很小范圍內變化,這樣才更有利于藍藻的生長,數值大了會對藍藻的生長起抑制作用.
3 結論
BiGG Models是由美國基于約束的代謝流分析方面的創立者University of California,San Diego的帕爾遜教授研究組開發的專業代謝網絡數據庫,是相關代謝網絡的主要來源.該數據庫的最大特點是含有各類模式生物的代謝譜圖模型,用戶可以直觀的調取各種生物的整體代謝通路,也可以查看某個具體的生化反應.目前,BIGG數據庫含有2 766個代謝產物和3 311條代謝生化反應,可以對代謝產物進行搜索.所以,BIGG數據庫在數據量和可靠性方面都是具有科學性和可使用性的.
藍藻的代謝系統中敲除酶PFOR后,有多數反應的通量受到了影響[16],說明酶敲除后,通過代謝物與輔酶的耦連,對離其較遠的反應產生了影響.敲除代謝系統中通量值為0 mmol·h-1的酶PFOR后,目標函數通量值保持不變,說明酶PFOR是藍藻代謝系統中的非必需基因.在FBA方法預測得到的最優解的前提下,通過FVA方法得到酶PFOR的最小通量和最大通量值均為0 mmol·h-1,說明應將代謝系統中酶PFOR的通量值保持在很小范圍內,數值大了不利于藍藻的光合生長.基因敲除仿真是一種全和無的實驗方法,通過多目標優化方法能更好地描述酶濃度和生物量之間的關系,在模擬實驗中,藍藻的光合生長速率受酶PFOR濃度的影響,當增加酶的濃度,會不利于藍藻生長;當酶的濃度降低,光合生長率會得到提高.通過仿真計算的結果與參考文獻三中的實驗結果比較一致,說明通過仿真能更好的預測和了解代謝系統.
本文利用藍藻的代謝網絡為實驗模型,模擬敲除其中的酶PFOR,并利用通量平衡分析、通量可變性分析和多目標優化方法研究基因敲除后的突變型代謝網絡.通過仿真實驗研究,一方面推測出了酶PFOR對藍藻光合生長速率的影響,有利于更好地了解酶PFOR的作用;另一方面,這種仿真方法具有一定的效果,對預測和了解代謝系統提供了一個可行方法.對于代謝系統,酶PFOR可能還具有其他未知功能,有待進一步實驗去探究.

