阿拉伯巖芥AP2基因家族的生物信息學分析
付春 劉曉偉 王玲

摘要 AP2基因作為一種調控植物花發育的功能基因,參與花分生特性建立。利用生物信息學方法對阿拉伯巖芥AP2蛋白的理化性質、高級結構和系統進化關系等進行了詳細的預測和分析。結果表明,除了成員AA40G00375、AA54G00162為穩定蛋白,其余AP2蛋白均為不穩定蛋白,且大多數蛋白定位在細胞核。只有成員AA8G00350屬于分泌蛋白,其余均為非分泌蛋白,且均不屬于跨膜蛋白。磷酸化位點分析表明AP2蛋白的生物功能主要是通過在絲氨酸(Ser)位點進行磷酸化來實現的。AP2蛋白的二級結構以無規則卷曲為主,各個成員的二級結構元件組成含量大小排列順序為無規則卷曲>α-螺旋>延伸鏈>β-轉角。其中14個AP2蛋白成員有2個保守結構域,其余均有1個保守結構域。系統進化樹表明阿拉伯巖芥AP2基因家族與擬南芥、小麥有較近的親緣關系。這些結果可為進一步研究其生物學功能提供一定的參考依據。
關鍵詞 阿拉伯巖芥;AP2基因;生物信息學
Abstract AP2 gene,as a functional gene regulating plant flower development,is involved in the establishment of floral meristems.The physicochemical properties,advanced structure and phylogenetic relationship of AP2 proteins from Aethionema arabicum were predicted and analyzed in detail by bioinformatics.The study shows that apart from the stable proteins AA40G00375 and AA54G00162,the other AP2 proteins are unstable,and most of them were located in the nuclear.Only member AA8G00350 belongs to secretory proteins,the rest are nonsecretory proteins,and they are not transmembrane proteins.Analysis of phosphorylation sites shows that the biological function of AP2 protein is mainly achieved by phosphorylation of serine (Ser) site.The secondary structure of AP2 protein is dominated by random coil,and the content of secondary structural elements of each member is presented in the order of random coil > alpha helix > extended strand > beta turn.Fourteen members of AP2 protein have two conserved domains,and the rest have one conserved domain.Phylogenetic tree shows that AP2 gene family of A.arabicum is closely related to that of Arabidopsis thaliana,Triticum urartu.These results provides some references for further study of its biological function.
Key words Aethionema arabicum;AP2 gene;Bioinformatics
阿拉伯巖芥(Aethionema arabicum)是十字花科巖芥菜屬半灌木植物,短周期,快速開花的一年生植物,較適合生長在砂質土壤中,喜熱植物[1-2]。阿拉伯巖芥是在4—6月完成其生命周期,在陡峭的上坡上缺少植被覆蓋,阿拉伯巖芥可以承受強紫外線的照射,在炎熱的夏季,寒冷的冬季它都可以比其他植物表現更好,具有耐旱和耐寒的特點[3-4]。果實異形性被定義為同一花序上產生的果實和種子的大小、顏色、形狀、休眠與萌發都不同[5]。短周期和異態表型可能是其適應不可測的本地環境的增長趨勢[5]。GLS是十字花科植物中防御病原體和食草動物的一套新的代謝產物,除了它們的防御功能,GLS也可以作為引誘劑,通常用于芥末味和抗癌基因活性研究,對于增加十字花科植物的多樣性有一定的價值[6]。
在植物的體內存在大量的轉錄因子[7],轉錄因子(transcription factor)指能夠與基因啟動子區的順式作用元件(Cis-acting element)相互作用,進而激活或抑制目標基因轉錄的一類蛋白,也被稱為反式作用因子[8]。轉錄因子不僅在植物的生長發育、形態建成等方面發揮著重要作用,而且在植物抗逆性及次生代謝等方面也起著重要的調控作用。轉錄因子根據其DNA結構域的不同,被劃分為bZIP、MYB、AP2/EREBP、WRKY和NAC等幾個大家族[9-10]。作為植物最大轉錄因子家族之一,AP2/EREBP轉錄因子廣泛存在于植物中。AP2/EREBP家族又被分為5個亞族:AP2、ERF、RAV、CBF/DREB和Soloist [11-12]。AP2轉錄因子家族成員至少含有一段60個左右氨基酸構成的AP2保守結構域[13-14],除了在擬南芥、水稻、玉米和番茄等植物中,還在藍藻、線蟲和病毒中也發現了具有AP2結構域和位點特異核酸內切酶的蛋白。所以有人認為當今植物中的AP2基因起源于細菌或者病毒基因的橫向轉移,AP2結構域可能來自后來進化為葉綠體的原始藍細菌的內共生[15]。AP2轉錄因子可通過響應乙烯、赤霉素、脫落酸、細胞分裂素和生長素的調節[16-24],直接或間接參與種子發育過程、花和果實等器官的形態建成等植物發育的多個進程[25-26];除了初生代謝,AP2轉錄因子還在植物次生代謝尤其是在調控藥用植物主要藥用活性成分(如青蒿素、紫杉醇和木質素)合成方面效果顯著[27]。同時,有報道稱擬南芥AP2基因具有正向調節抗灰霉病的功能,AP2基因在植物應對高鹽、干旱、缺氧、低溫等非生物脅迫方面具重要功能,為植物的耐受性研究提供新契機[28-37]。
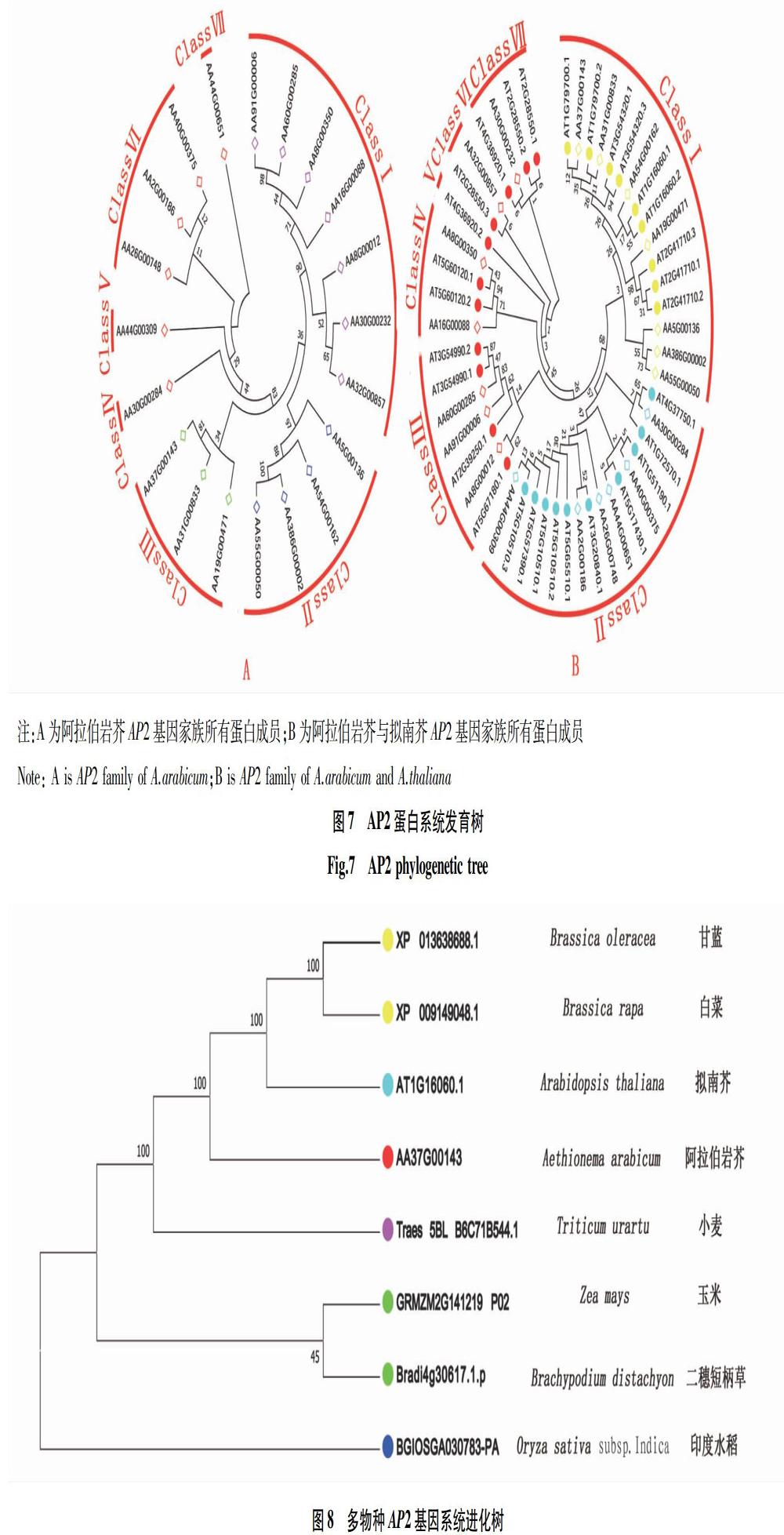
2.8 阿拉伯巖芥AP2基因系統進化樹構建
對阿拉伯芥AP2基因家族的所有蛋白成員系統發育進化關系分析表明,阿拉伯巖芥AP2蛋白家族分為7組,組 Ⅰ 有7個成員,成員數最多,組 Ⅱ 有4個成員,組Ⅲ和組Ⅵ有3個成員,其余組都只有1個成員。第Ⅶ組成員AA44G00651是最原始的一支,第 Ⅰ 組成員AA91G00006和AA60G00285是距離原始親緣最遠的一支,其進化速度最快(圖7A)。
阿拉伯巖芥與擬南芥AP2蛋白的系統進化分析表明,阿拉伯巖芥與擬南芥親緣關系很近。整體共分為7個組,組 Ⅰ 共16個基因成員,阿拉伯巖芥成員有7個,擬南芥成員有9個,其中擬南芥成員AT1G79700.1是進化速度最快的1個;阿拉伯巖芥成員AA37G00143與其處在同一地位,親緣關系最近。組 Ⅱ 共有16個成員,阿拉伯巖芥成員有6個,擬南芥成員有10個,其中成員AT4G37750.1與成員AA30G00284、成員AA40G00375與成員AT1G51190.1、成員AA44G00651和AT5G17430.1、成員AA2G00186和AT3G20840.1、成員AA44G00309和AT5G10510.3有較近的親緣關系,同為姊妹關系。組Ⅲ共有7個成員,阿拉伯巖芥成員有3個,擬南芥成員有4個,其中只有成員AA8G00012和AT5G67180.1的親緣關系最近,可能為親緣關系較近的一類蛋白。組Ⅳ共有4個成員,阿拉伯巖芥成員數量和擬南芥成員數量相等,都是2個;組Ⅴ只有1個擬南芥成員;組Ⅵ共有2個成員,阿拉伯巖芥和擬南芥成員各1個;組Ⅶ共有4個成員,阿拉伯巖芥成員只有1個,其余均為擬南芥成員,其中擬南芥成員AT2G28550.1是最原始的一支(圖7B)。
阿拉伯巖芥AP2基因家族成員AA37G00143與擬南芥等多個物種AP2成員分子進化關系分析表明,阿拉伯巖芥與其他成員都有較高的親緣關系。其中阿拉伯巖芥成員AA37G00143與擬南芥成員AT1G16060.1和小麥成員Traes-5BL-B6C71B544.1的親緣關系最近,與印度水稻的親緣關系最遠。從十字花科分類的角度分析,阿拉伯巖芥與擬南芥親緣關系最近,可能與其同屬于十字花科有關,但是與甘藍的親緣關系最遠;從雙子葉植物與單子葉植物分類分析,阿拉伯巖芥亦是與擬南芥和小麥親緣關系最近,然而與印度水稻的親緣關系最遠,可能與它們分別屬于單雙子葉植物有關(圖8)。
3 討論與結論
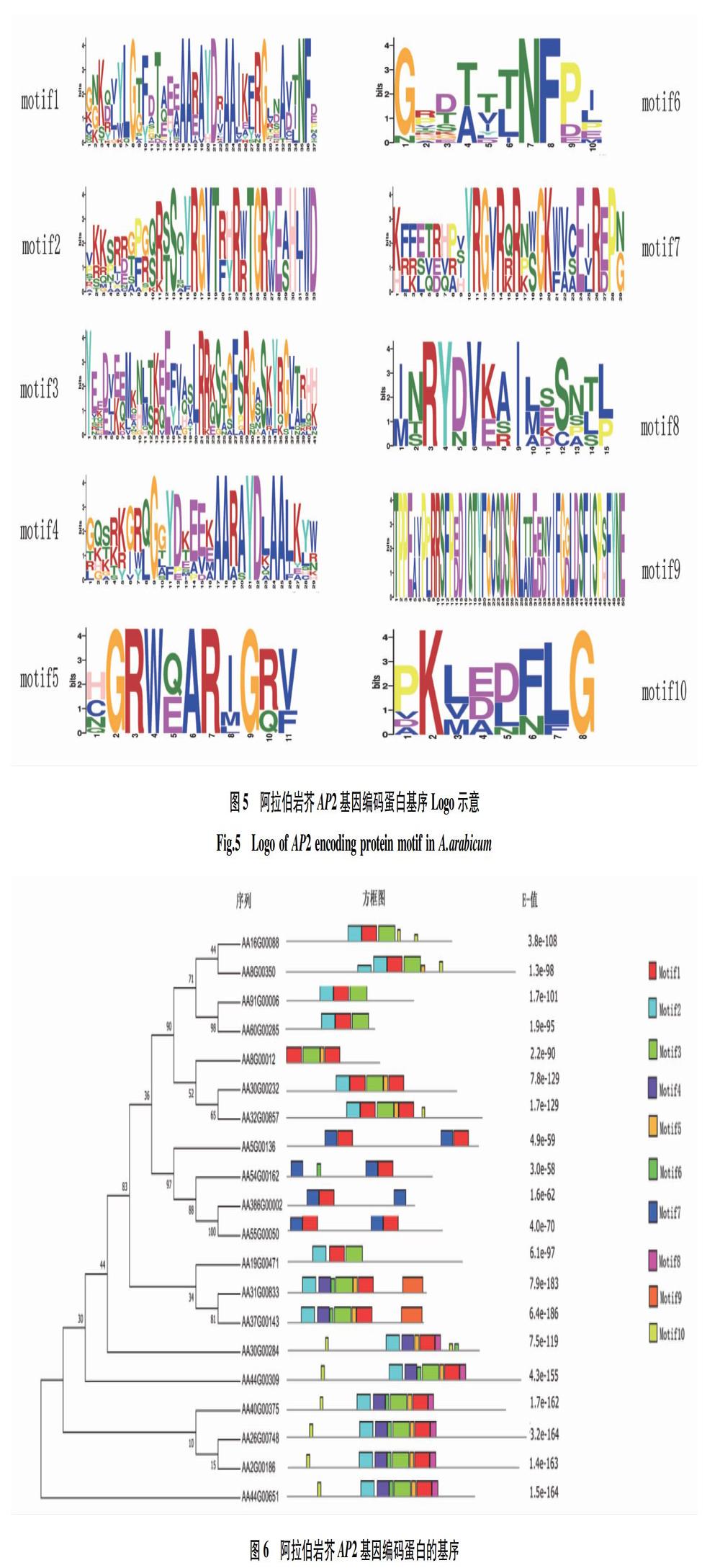
郭慧等[8]對甘藍AP2/ERF轉錄因子的研究分析表明甘藍AP2蛋白質結構為不穩定性蛋白。該研究中阿拉伯巖芥AP2基因編碼蛋白之間存在差異性,且為不穩定蛋白,這與郭慧等[8]對甘藍AP2蛋白結構研究結果相同。謝藤等[72]對新疆紫草AP2/ERF轉錄因子進行生物信息學分析表明其亞細胞定位存在于細胞核,不存在跨膜域,不屬于分泌蛋白。該研究表明阿拉伯巖芥AP2蛋白亞細胞主要存在于細胞核,跨膜域分析顯示所有編碼蛋白都不屬于跨膜蛋白,成員AA8G00350屬于分泌蛋白,跨膜域分析與謝藤等[72]的研究結果相同,但亞細胞定位分析和信號肽分析結果與其不同,不同的原因可能與不同物種蛋白的結構組成存在差異性有關。趙金玲等[73]對楊樹AP2/ERF基因家族性質的研究表明楊樹AP2/ERF家族蛋白二級結構都以隨機卷曲為主,其二級結構特點與阿拉伯巖芥AP2基因編碼蛋白的二級結構特點相似,都以無規則卷曲結構為主,且阿拉伯巖芥AP2蛋白各個成員的二級結構元件組成含量大小排列順序都表現為無規則卷曲>α-螺旋>延伸鏈>β-轉角,模擬生成三級結構的圖像與二級結構預測的結果相一致。不同物種之間其AP2家族成員數量各有不同,例如擬南芥為30個,玉米為54個,甘藍為43個。但是不同物種之間的AP2基因具有一定的保守性,都含有特定的AP2結構域[30,71]。該研究顯示阿拉伯巖芥AP2基因編碼蛋白20個成員都含有AP2保守結構域,其中14個成員有2個保守結構域,其余均只有1個保守結構域。植物為了克服生長發育中不利影響,自身會在體內形成各種應答機制,有些蛋白會在非生物脅迫下作為保護因子調控植物細胞的穩定,進而提升其抗逆性[71,74]。該研究中阿拉伯巖芥AP2蛋白整體成員表現為親水性蛋白,這可能是利于阿拉伯巖芥增強抗逆性的因素。YRG和RAYD元件都已被證實參與了DNA或其他轉錄因子的結合。研究證實,AP2中的一個轉錄因子WXP1與苜蓿和擬南芥的抗旱性都有關[71,75-77]。基因序列對比表明阿拉伯巖芥AP2基因序列含有YRG元件和RAYD元件,推測阿拉伯巖芥抗干旱功能也與其有關。莊靜[78]對白菜與擬南芥進行研究分析建構進化樹表明,白菜與擬南芥有較近的親緣關系。該研究中阿拉伯巖芥與多個物種建構系統進化樹表明阿拉伯巖芥AP2基因家族與擬南芥、小麥等都有較近的親緣關系,完善了莊靜[78]的系統進化分析結果。
該研究結果表明,阿拉伯巖芥AP2基因家族編碼蛋白不同成員之間存在差異,其中只有成員AA40G00375、AA54G00162為穩定蛋白,其余均為不穩定蛋白,其大多數蛋白的亞細胞定位以細胞核為主,只有成員AA8G00350屬于分泌蛋白,其余為非分泌蛋白,且所有蛋白為親水性蛋白,不屬于跨膜蛋白,都含有至少1個AP2結構域,通過絲氨酸位點處進行磷酸化來實現功能。其二級結構較整齊,都以無規則卷曲為主,各個成員的二級結構元件組成含量大小排列順序都表現為無規則卷曲>α-螺旋>延伸鏈>β-轉角,三級結構的圖像與二級結構預測的結果相一致。阿拉伯巖芥AP2基因家族與擬南芥、白菜、玉米、小麥等都有較近的親緣關系,其中阿拉伯巖芥成員AA37G00143與擬南芥成員AT1G16060.1和小麥成員Traes_5BL_B6C71B544.1的親緣關系最近,與印度水稻的親緣關系最遠。阿拉伯巖芥與擬南芥的親緣關系最近,可能與其同屬于十字花科有關,而與印度水稻的親緣關系最遠,可能與它們分別屬于單雙子葉植物有關。該研究結果為進一步研究阿拉伯巖芥AP2基因家族在其生長發育過程中的生物學功能提供了一定的參考依據。
[21] KONG X P,TIAN H Y,YU Q Q,et al.PHB3 maintains root stem cell niche identity through ROSresponsive AP2/ERF transcription factors in Arabidopsis[J].Cell reports,2018,22(5):1350-1363.
[22] GU C,GUO Z H,HAO P P,et al.Multiple regulatory roles of AP2/ERF transcription factor in angiosperm[J].Botanical studies,2017,58(1):1-8.
[23] JISHA V,DAMPANABOINA L,VADASSERY J,et al.Overexpression of an AP2/ERF type transcription factor OsEREBP1 confers biotic and abiotic stress tolerance in rice[J].PLoS One,2015,10(6):1-24.
[24] LIU Z N,KONG L J,ZHANG M,et al.Genome-wide identification,phylogeny,evolution and expression patterns of AP2/ERF genes and cytokinin response factors in Brassica rapa ssp.pekinensis[J].PLoS One,2013,8:1-15.
[25] 徐倩,殷學仁,陳昆松.基于乙烯受體下游轉錄因子的果實品質調控機制研究進展[J].園藝學報,2014,41(9):1913-1923.
[26] YU Y,DUAN X B,DING X D,et al.A novel AP2/ERF family transcription factor from Glycine soja,GsERF71,is a DNA binding protein that positively regulates alkaline stress tolerance in Arabidopsis[J].Plant molecular biology,2017,94(1):509-530.
[27] WUDDINEH W A,MAZAREI M,TURNER G B,et al.Identification and molecular characterization of the switchgrass AP2/ERF transcription factor superfamily,and overexpression of PvERF001 for improvement of biomass characteristics for biofuel[J].Frontiers in bioengineering and biotechnology,2015,3:1-21.
[28] KITOMI Y,ITO H,HOBO T,et al.The auxin responsive AP2/ERF transcription factor CROWN ROOTLESS5 is involved in crown root initiation in rice through the induction of OsRR1, a typeA response regulator of cytokinin signaling[J].Plant journal,2011,67(3):472-484.
[29] 聞可心,劉雪梅.AP2功能基因在植物花發育中的重要作用[J].生物技術通報,2010(2):1-7.
[30] 張計育,王慶菊,郭忠仁.植物AP2/ERF類轉錄因子研究進展[J].遺傳,2012,34(7):835-847.
[31] 季愛加,羅紅梅,徐志超,等.藥用植物轉錄因子AP2/ERF研究與展望[J].科學通報,2015,60(14):1272-1284.
[32] DOSSA K,WEI X,LI D H,et al.Insight into the AP2/ERF transcription factor superfamily in sesame and expression profiling of DREB subfamily under drought stress[J].BMC Plant Biology,2016,16(1):1-16.
[33] LIU Q,KASUGA M,SAKUMA Y,et al.Two transcription factors,DREB1 and DREB2,with an EREBP/AP2 DNA binding domain separate two cellular signal transduction pathways in droughtand lowemperatureresponsive gene expression,respectively,in Arabidopsis[J].The plant cell,1998,10(8):1391-1406.
[34] XU Z S,ChEN M,LI L C,et al.Functions and application of the AP2/ERF transcription factor family in crop improvement[J].Journal of integrative plant biology,2011,53(7):570-585.
[35] SAKUMA Y,LIU Q,DUBOUZET J G,et al.DNAbinding specificity of the ERF/AP2 domain of Arabidopsis DREBs,transcription factors involved in dehydrationand coldinducible gene expression[J].Biochemical and biophysical research communications,2002,290(3):998-1009.
[36] LI H,WANG Y,WU M,et al.Genomewide identification of AP2/ERF transcription factors in cauliflower and expression profiling of the ERF family under salt and drought stresses[J].Frontiers in plant science,2017,8:946.
[37] MAWLONG I,ALI K,SRINIVASAN R,et al.Functional validation of a droughtresponsive AP2/ERF family transcription factorencoding gene from rice in Arabidopsis[J].Molecular breeding,2015,35(8):1-14.
[38] PARK C S,GO Y S,SUH M C.Cuticular wax biosynthesis is positively regulated by WRINKLED4,an AP2/ERF-type transcription factor,in Arabidopsis stems[J].The plant journal,2016,88(2):257-270.
[39] THAMILARASAN S K,PARK J I,JUNG H J,et al.Genomewide analysis of the distribution of AP2/ERF transcription factors reveals duplication and CBFs genes elucidate their potential function in Brassica oleracea[J].BMC Genomics,2014,15(1):1-13.
[40] WANG X H,HAN H Y,YAN J,et al.A new AP2/ERF transcription factor from the oil plant Jatropha curcas confers salt and drought tolerance to transgenic tobacco[J].Applied biochemistry and biotechnology,2015,176(2):582-597.
[41] YANG Y Q,DONG C,LI X,et al.A novel AP2/ERF transcription factor from Stipa purpurea leads to enhanced drought tolerance in Arabidopsis thaliana[J].Plant cell reports,2016,35(11):2227-2239.
[42] 周媛媛,董曉靜,呂婭,等.擬南芥分子生物學研究進展[J].中國農學通報,2018,34(30):56-62.
[43] 綦洋,王柬鈞,桑園園,等.大白菜YUCCA基因家族的鑒定與生物信息學分析[J].江蘇農業科學,2019,47(3):49-54.
[44] 袁明月,樓軼,張競,等.禾谷鐮刀菌AP蛋白家族的生物信息學分析[J/OL].基因組學與應用生物學,2019-03-01[2019-08-16].http:kns.cnki.net/kcms/detail/45.1369.Q.20190301.0942.002.html.
[45] 張書芹,樂愉,武斐.陸地棉GRF基因家族的鑒定和生物信息學分析[J].分子植物育種,2019,17(12):3817-3824.
[46] 白羽聰,李翔宇,程占超,等.毛竹PIN基因家族的鑒定與生物信息學分析[J].分子植物育種,2019,17(16):5238-5247.
[47] 逄洪波,潘英杰,李嘉琦,等.向日葵Hsp70基因家族成員的全基因組鑒定與生物信息學分析[J].分子植物育種,2019,17(18):5918-5929.
[48] 郭棟,杜媚,周寶元,等.玉米CCT基因家族的鑒定與生物信息學分析[J].植物遺傳資源學報,2019,20(4):1001-1010.
[49] 狄建軍,孫佳欣,楊智慧,等.蓖麻PLA2α基因啟動子的克隆及生物信息學分析[J].分子植物育種,2019,17(15):4961-4966.
[50] 何焱,李忠玥,李卿,等.藏藥翼首草角鯊烯合成酶的生物信息學分析[J/OL].基因組學與應用生物學,2019-01-14[2019-08-16].http:∥kns.cnki.net/kcms/detail/45.1369.q.20190111.1501.002.html.
[51] 趙思華,尹晗,呂昊哲,等.大豆GmTAA1-1基因的生物信息及時空表達分析[J].分子植物育種,2019,17(21):6946-6952.
[52] 吳桂梅,趙小峰,汪瑤,等.大鼠單核細胞趨化蛋白-1基因的生物信息學分析[J].安徽農業科學,2019,47(1):93-95.
[53] 張西燕,付玉榮,伊正君.結核分枝桿菌PE_PGRS47蛋白的生物信息學分析[J].中國病原生物學雜志,2019,14(1):44-49.
[54] 徐小欽,杜雪,李國勤,等.白羽王鴿卵清蛋白關聯蛋白Y基因(OVALY)克隆與生物信息學分析[J].浙江農業學報,2019,31(5):704-708.
[55] 馮立娟,尹燕雷,楊雪梅,等石榴查爾酮合成酶蛋白的生物信息學分析[J].山東農業科學,2019,51(2):7-12.
[56] 宋雪,秦彩艷,霍金龍,等.版納微型豬近交系CPN10克隆、亞細胞定位、組織表達和生物信息學分析[J].云南農業大學學報,2019,34(2):255-262.
[57] 姜錚,王芳,何湘,等.蛋白質磷酸化修飾的研究進展[J].生物技術通訊,2009,20(2):233-237.
[58] 李杰,杭柏林,寧春妹,等.牛源cathelicidin類抗菌肽的生物信息學分析[J].河南科技學院學報(自然科學版),2019,47(1):40-45.
[59] 龍芳,李紹鵬,李茂富.7種植物ALAD基因的生物信息學分析[J].基因組學與應用生物學,2013,32(6):802-814.
[60] 韓長志.禾谷炭疽菌RGS蛋白生物信息學分析[J].微生物學通報,2014,41(8):1582-1594.
[61] 宋志忠,楊順瑛,金曼,等.楊樹基因組AMT轉運蛋白的生物信息學特性(英文)[J].基因組學與應用生物學,2011,30(6):641-648.
[62] 郭溆,羅紅梅,陳士林.靈芝GLUGT1基因的全長cDNA克隆和生物信息學分析[C]∥中藥與天然藥高峰論壇暨全國中藥和天然藥物學術研討會論文集.北京:中國藥學會,2012.
[63] 劉金鴿.擬南芥AtDPBF4基因克隆及生物信息學分析與表達研究[D].蘭州:蘭州理工大學,2013.
[64] 張加強,劉慧春,周江華,等.植物赤霉素氧化酶GA20ox基因的生物信息學分析[J].分子植物育種,2019,17(15):4986-5002.
[65] 皮博藝,阮穎,黃勇.植物串聯CCCH 鋅指蛋白RR-TZF家族研究進展及生物信息學分析[J].分子植物育種,2019,17(7):2171-2177.
[66] 唐躍輝,包欣欣,王健,等.小桐子Dof基因家族生物信息學與表達分析[J].江蘇農業學報,2019,35(1):15-25.
[67] 王雁,王小貝,陳海江,等.桃GLV多肽受體PpRGI基因家族的生物信息學及表達分析[J].園藝學報,2019,46(5):855-863.
[68] 袁夢如,楊楊,張磊,等.沙柳SpsTAC2基因克隆、生物信息學及組織特異表達分析[J].分子植物育種,2019,17(7):2144-2151
[69] 王麗珊.擬南芥和水稻Cel基因家族的生物信息學分析[J].閩西職業技術學院學報,2019,21(1):101-106.
[70] 郭娟,彭秋菊,佘明明,等.辣椒生育酚環化酶(TC)基因克隆和生物信息學分析[J].分子植物育種,2019,17(10):3171-3179.
[71] 阮先樂,張杰,張福麗,等.甘薯AP2基因家族的生物信息學分析[J].分子植物育種,2017,15(6):2066-2072.
[72] 謝騰,王升,周良云,等.新疆紫草AP2/ERF轉錄因子的電子克隆和生物信息學分析[J].中國中藥雜志,2014,39(12):2251-2257.
[73] 趙金玲,姚文靜,王升級,等.楊樹AP2/ERF轉錄因子家族生物信息學分析[J].東北林業大學學報,2015,43(10):21-29.
[74] 張麗麗,李博,譚燕華,等.玉米轉錄因子基因ZmASR家族的生物信息學分析[J].分子植物育種,2016,14(4):803-811.
[75] OKAMURO J K,CASTER B,VILLARROEL R,et al.The AP2 domain of APETALA2 defines a large new family of DNA binding proteins in Arabidopsis[J].Proceedings of the national academy of sciences,1997,94(13):7076-7081.
[76] ZHANG J Y,BROECKLING C D,BLANCAFLOR E B,et al.Overexpression of WXP1,a putative Medicago truncatula AP2 domaincontaining transcription factor gene,increases cuticular wax accumulation and enhances drought tolerance in transgenic alfalfa (Medicago sativa)[J].Plant journal,2005,42(5):689-707.
[77] LIN R C,PARK H J,WANG H Y.Role of Arabidopsis RAP2.4 in regulating lightand ethylenemediated developmental processes and drought stress tolerance[J].Molecular plant,2008,1(1):42-57.
[78] 莊靜.大白菜和甘藍型油菜AP2/ERF家族轉錄因子的克隆與分析[D].南京:南京農業大學,2009.

