基于復(fù)雜網(wǎng)絡(luò)理論的肺癌特異性基因識別研究
于曉


摘? 要:基于復(fù)雜網(wǎng)絡(luò)理論分別構(gòu)建以肺癌組織和健康肺組織基因為節(jié)點,基因間調(diào)控關(guān)系為邊的基因調(diào)控網(wǎng)絡(luò),并從拓撲結(jié)構(gòu)、分布特征、節(jié)點中心性三方面分析網(wǎng)絡(luò)特性,挖掘網(wǎng)絡(luò)核心節(jié)點生物功能差異性以識別出肺癌特異性基因。結(jié)果發(fā)現(xiàn),肺癌組織和健康肺組織基因調(diào)控網(wǎng)絡(luò)拓撲參數(shù)極其相似且兩者都為無標度網(wǎng)絡(luò),兩網(wǎng)絡(luò)核心節(jié)點集高度重疊,但非重疊部分核心節(jié)點的生物功能十分特殊,并據(jù)此識別出肺癌特異性基因。該方法識別出的肺癌特異性基因,能夠成為潛在肺癌生物標記物,為肺癌的早期診斷提供幫助,同時該方法能夠適用于其他疾病特異性基因的識別。
關(guān)鍵詞:復(fù)雜網(wǎng)絡(luò);肺癌;中心性;HUB節(jié)點
中圖分類號:TP319? ? ?文獻標識碼:A
Abstract: Based on complex network theory, the study constructs lung cancer tissue and healthy lung tissue gene regulatory networks with genes as nodes and inter-gene regulatory relationships as edges. The paper analyzes network characteristics from three aspects: topological structure, distribution characteristics, and node centrality. By comparing the biological function of the key nodes of networks to identify lung cancer-specific genes, it is found that the topological parameters of the two gene regulatory networks are extremely similar and they are both scale-free networks. The key nodes set of the two networks are highly overlapping, but the non-overlapping key nodes' biological functions are very special, and then the lung cancer-specific genes are identified base on this. The lung cancer-specific genes identified by this method may be potential biomarkers for lung cancer, which promotes the early diagnosis of lung cancer. The method can also be applied in the identification of other disease-specific genes.
Keywords: complex network; lung cancer; centrality; HUB node
1? ?引言(Introduction)
肺癌是最常見和嚴重的癌癥之一,其發(fā)病率和死亡率在世界大多數(shù)國家都處于首位,且增長率驚人[1]。在中國,肺癌已成為致死率最高的癌癥[2],根據(jù)中國衛(wèi)生部于2006年進行的第三次全國性死因抽樣調(diào)查,肺癌造成的死亡率自1990年以來增加了75.77%,并且在剔除年齡結(jié)構(gòu)變化因素后再次增加了33.25%。盡管吸煙這一肺癌的最相關(guān)危險因素已被廣泛認識,但是對肺癌發(fā)病機制和診斷治療等一系列問題的相關(guān)研究依舊進展緩慢。其次,由于肺癌通常在早期不會引發(fā)任何癥狀,許多患者的相關(guān)體征和癥狀僅在疾病發(fā)展時發(fā)生,這讓肺癌的確診存在相當大的難度,若在肺癌早期通過檢測特異性基因使其確診并得到充分的治療,其死亡率將會在很大程度上降低。此外,大多數(shù)肺癌患者仍在接受化學(xué)療法,但這些患者中的大多數(shù)經(jīng)過化學(xué)療法后并未好轉(zhuǎn),這是因為化學(xué)療法并沒有解決患者潛在遺傳缺陷這一導(dǎo)致肺癌癥發(fā)生的根本病因。因此,現(xiàn)在面臨的巨大挑戰(zhàn)是如何準確地識別這些潛在遺傳缺陷,以便使治療與患者相匹配,獲得更高的治愈率。這一系列的問題讓識別有診斷用途的肺癌生物標記成了攻克肺癌這一高致死率疾病的關(guān)鍵。本文以復(fù)雜網(wǎng)絡(luò)理論為工具,提出了系統(tǒng)性識別肺癌特異性基因的方法:根據(jù)肺癌組織中基因關(guān)系構(gòu)建肺癌組織基因調(diào)控網(wǎng)絡(luò),分析網(wǎng)絡(luò)拓撲結(jié)構(gòu)和分布規(guī)律,結(jié)合中心性算法挖掘網(wǎng)絡(luò)中的關(guān)鍵節(jié)點,根據(jù)其生物功能推測肺癌特異性基因。該方法為醫(yī)學(xué)工作者們揭示肺癌發(fā)生機制和針對性治療肺等方面提供了依據(jù)。
2? ?文獻綜述(Literature review)
網(wǎng)絡(luò)生物學(xué)是生物醫(yī)學(xué)研究中一個快速發(fā)展的領(lǐng)域,它提供了一個總體的觀點,即遺傳疾病的發(fā)生不是單個基因突變的結(jié)果,而是由基因網(wǎng)絡(luò)環(huán)境的擾動引起。因此,認識這些基因相互作用網(wǎng)絡(luò)的拓撲結(jié)構(gòu),以及確定在其結(jié)構(gòu)和調(diào)控中起關(guān)鍵作用的基因是理解復(fù)雜遺傳性疾病的關(guān)鍵。
現(xiàn)代生物網(wǎng)絡(luò)的研究著重分析網(wǎng)絡(luò)的拓撲結(jié)構(gòu)[3],基于對大規(guī)模生物網(wǎng)絡(luò)的研究通常比臨床實驗室更有優(yōu)勢[4]。此外研究表明生物網(wǎng)絡(luò)中關(guān)鍵基因的發(fā)現(xiàn)對解決生物問題有重要意義,因為疾病生物網(wǎng)絡(luò)中的關(guān)鍵基因往往與致病基因有著高度的相關(guān)性[5]。在對蛋白質(zhì)相互作用網(wǎng)絡(luò)的研究中驗證了這一觀點,外國研究者們發(fā)現(xiàn)癌癥致病基因或蛋白質(zhì)在網(wǎng)絡(luò)中存在非常高的中心性[6],同時癌癥相關(guān)致病基因或蛋白質(zhì)之間更傾向于密切聯(lián)系使其成為一個社區(qū)[7],在社區(qū)之內(nèi)它們存在更廣泛的相互作用關(guān)系。
在眾多識別生物網(wǎng)絡(luò)關(guān)鍵節(jié)點的方法中,節(jié)點中心性得分是一種快速且可重現(xiàn)的方法,其根本思想是根據(jù)節(jié)點在網(wǎng)絡(luò)中所處位置的關(guān)鍵性對其進行排名。這些網(wǎng)絡(luò)節(jié)點中心性度量通常基于路徑長度和隨機游走,例如接近度中心、介數(shù)中心、聚類系數(shù)。同時,針對不同類型生物網(wǎng)絡(luò)不同中心性算法的表現(xiàn)也不盡相同,例如,Kim等人[8]發(fā)現(xiàn),基于最短路徑的介數(shù)中心性度量可以作為檢測關(guān)鍵性基因的一種可靠標準。此外,He等人[9]表明,在蛋白質(zhì)相互作用網(wǎng)絡(luò)中接近度中心性并不能準確預(yù)測出關(guān)鍵蛋白質(zhì)。此外,子圖中心性在高密度網(wǎng)絡(luò)中的效果拔群,但在酵母菌蛋白質(zhì)相互作用網(wǎng)絡(luò)這種較稀疏網(wǎng)絡(luò)中預(yù)測關(guān)鍵蛋白質(zhì)的能力尚未超過度中心性。
國內(nèi)針對肺癌特異性基因的研究工作主要集中在單個基因的突變檢測上[10,11],很少有從系統(tǒng)或者網(wǎng)絡(luò)角度出發(fā)的相關(guān)研究,對此我們利用復(fù)雜網(wǎng)絡(luò)理論進行了系統(tǒng)性對比實驗,通過對比肺癌與健康肺組織基因調(diào)控網(wǎng)絡(luò)的拓撲參數(shù)以分析兩個網(wǎng)絡(luò)的差異性。其次針對上述文獻中單一拓撲中心性算法難以適配的問題,我們提出全局與局部中心性相結(jié)合觀點,利用四種局部和七種全局中心性算法綜合確定關(guān)鍵基因。
3? ?研究設(shè)計(Research design)
3.1? ?基因調(diào)控網(wǎng)絡(luò)構(gòu)建
我們所用到的數(shù)據(jù)來自文獻[12],研究者們提供來自32個人體不同細胞、組織、器官的基因調(diào)控數(shù)據(jù),以邊表的形式存儲。我們選定其中肺癌組織和健康肺組織的基因調(diào)控數(shù)據(jù)作為我們的實驗數(shù)據(jù),然后對其進行預(yù)處理,根據(jù)權(quán)重分布規(guī)律剔除了一部分噪聲,將剩下的核心數(shù)據(jù)構(gòu)建成了肺癌和健康肺組織基因調(diào)控網(wǎng)絡(luò)。其中健康肺組織基因調(diào)控網(wǎng)絡(luò)如圖1(a)所示,肺癌基因調(diào)控網(wǎng)絡(luò)如圖1(b)所示。圖中的節(jié)點分別代表參與肺癌組織和健康肺組織細胞調(diào)控的基因,網(wǎng)絡(luò)中的邊代表這些基因之間的相互作用關(guān)系。
(a)健康肺組織基因調(diào)控網(wǎng)絡(luò)
(b)肺癌組織基因調(diào)控網(wǎng)絡(luò)
3.2? ?網(wǎng)絡(luò)拓撲參數(shù)與分布特征
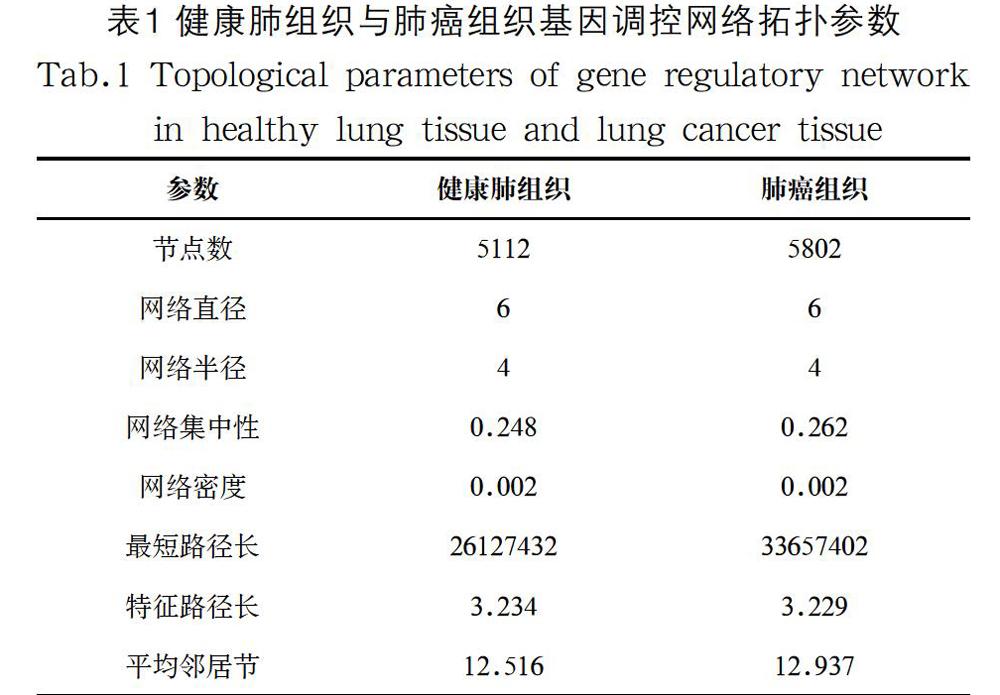
本文計算了肺癌組織基因調(diào)控網(wǎng)絡(luò)和健康肺組織基因調(diào)控網(wǎng)絡(luò)的拓撲參數(shù),通過對比發(fā)現(xiàn)肺癌組織和健康肺組織基因調(diào)控網(wǎng)絡(luò)拓撲參數(shù)極其相似,如表1所示,這就解釋了肺癌細胞和健康肺細胞在基因調(diào)控關(guān)系結(jié)構(gòu)上的同源性。其中,網(wǎng)絡(luò)直徑被定義為兩個節(jié)點對間的最大距離,而節(jié)點間距離是指連接兩個節(jié)點所需要的最小邊數(shù);網(wǎng)絡(luò)半徑與網(wǎng)絡(luò)直徑相似,被定義為兩個節(jié)點對間的最小距離;網(wǎng)絡(luò)集中性用來描述網(wǎng)絡(luò)拓撲結(jié)構(gòu)與星形網(wǎng)結(jié)構(gòu)的相似程度,相似性越高該數(shù)值越接近于1;網(wǎng)絡(luò)密度被定義為網(wǎng)絡(luò)中實際存在邊的數(shù)量與網(wǎng)絡(luò)最大可容納邊數(shù)量的比值;最短路徑長度表示從原始節(jié)點到目標節(jié)點所需經(jīng)過的最小邊數(shù);特征路徑長度被定義為存在于網(wǎng)絡(luò)中所有最短路徑的均值。
分別統(tǒng)計肺癌組織和健康肺組織基因調(diào)控網(wǎng)絡(luò)的度分布,其中健康肺組織基因調(diào)控網(wǎng)絡(luò)的度分布如圖2(a)所示,肺癌組織基因調(diào)控網(wǎng)絡(luò)的度分布如圖2(b)所示。圖中橫坐標代表節(jié)點度,縱坐標代表網(wǎng)絡(luò)中對應(yīng)橫坐標節(jié)點度下的節(jié)點數(shù),由圖可知網(wǎng)絡(luò)度分布均存在明顯下降趨勢,說明網(wǎng)絡(luò)屬于無標度網(wǎng)絡(luò),即網(wǎng)絡(luò)中的大多數(shù)節(jié)點只存在很少的連邊與此同時網(wǎng)絡(luò)中存在為數(shù)不多連邊數(shù)巨大的HUB節(jié)點,低連通節(jié)點與高連通節(jié)點之間的邊在網(wǎng)絡(luò)中占優(yōu)勢[13]。無標度網(wǎng)絡(luò)的特征是它們具有許多外圍節(jié)點和幾個高度連接的節(jié)點,這些網(wǎng)絡(luò)具有通用的組織原則:“小世界”屬性(即,它們通過節(jié)點之間相對較短的路徑高度連接)。擁有這種屬性網(wǎng)絡(luò)具有魯棒性,可以淘汰外圍節(jié)點并保留核心網(wǎng)絡(luò)功能。
3.3? ?網(wǎng)絡(luò)節(jié)點中心性分析
網(wǎng)絡(luò)中心性算法是度量節(jié)點在網(wǎng)絡(luò)中重要性的度量方法,這里分別用到四種局部性中心性和七種全局性中心性算法綜合挖掘網(wǎng)絡(luò)中的HUB節(jié)點。我們先通過計算得到兩個網(wǎng)絡(luò)中所有基因節(jié)點的11種中心性得分,再對11種中心性得分累加歸一化作為最終的中心性得分。最終中心性得分排在前面的基因在對應(yīng)組織基因調(diào)控網(wǎng)絡(luò)中有強中性心,據(jù)此挑選得分排名前10的節(jié)點作為對應(yīng)組織基因調(diào)控網(wǎng)絡(luò)的HUB做后續(xù)分析,見表2。
其中,四種局部性算法的度量標準為:節(jié)點度(Degree),最大鄰居連通分量(Maximum Neighborhood Component)[14],最大鄰居連通分量密度(Density of Maximum Neighborhood Component)[14],最大集團中心性(Maximal Clique Centrality)[15]。其中,節(jié)點度被定義為與該節(jié)點直接相連其他網(wǎng)絡(luò)中節(jié)點的數(shù)量;最大鄰居連通分量定義為節(jié)點鄰居網(wǎng)絡(luò)中最大連通分量包含的節(jié)點數(shù),這里的鄰居網(wǎng)絡(luò)是節(jié)點與它的鄰居所構(gòu)成的網(wǎng)絡(luò);最大鄰居連通分量密度則為節(jié)點鄰居網(wǎng)絡(luò)實際邊數(shù)與所能容納最大邊數(shù)的比值;最大集團中心性則與節(jié)點所在的最大集團中連通分量個數(shù)相關(guān)。
七種全局性算法的度量標準為:接近度(Closeness)[16]、偏心率(Eccentricity)[17]、輻射率(Radiality)[18]、障礙率(Bottleneck)[19]、壓力值(Stress)[20]、介數(shù)(Betweenness)[21]、邊擴散度(Edge Percolated Component)[22]。其中,接近度定義為節(jié)點到其他節(jié)點距離的倒數(shù)和,該值越高節(jié)點越容易到達其他節(jié)點;偏心率定義為節(jié)點到距離它最遠的節(jié)點距離的倒數(shù);輻射率以網(wǎng)絡(luò)直徑為標準,比較節(jié)點到其他節(jié)點的距離遠近;障礙率則是在網(wǎng)絡(luò)中構(gòu)造以節(jié)點為根的最小生成樹,通過統(tǒng)計遍歷該最小生成樹過程中所經(jīng)過網(wǎng)絡(luò)中的其他節(jié)點來定量根節(jié)點的中心性;壓力值定義為通過該節(jié)點最短路徑的數(shù)量,壓力值越高,該節(jié)點更傾向于位于其他節(jié)點對的最短路徑上;介數(shù)被定義為通過該節(jié)點最短路徑數(shù)與網(wǎng)絡(luò)中最短路徑總數(shù)的比值,介數(shù)值越大說明該節(jié)點越傾向于處于網(wǎng)絡(luò)中樞軸位置;邊擴散度則按照相等概率連續(xù)除去節(jié)點所在網(wǎng)絡(luò)中的邊,執(zhí)行多次,并記錄每次操作后當前網(wǎng)絡(luò)中該節(jié)點的鄰居節(jié)點數(shù),算法的結(jié)果取決于這些記錄。
3.4? ?特異性基因的確定
為了識別肺癌組織中的特異性基因,我們重點關(guān)注僅存在于肺癌組織卻不存在于健康肺組織中的HUB基因,這些HUB基因如圖3所示,由圖可知肺癌組織和健康肺組織HUB基因大部分重合,僅存在三個肺癌獨有的HUB基因。通過在 Genecards[23]中匹配這些肺癌獨有HUB基因的功能,能夠進一步得到它們的生物功能信息。這里Genecards是人類基因的綜合數(shù)據(jù)庫,提供以基因數(shù)據(jù)為中心的基因組、轉(zhuǎn)錄組、蛋白質(zhì)組、遺傳和臨床相關(guān)的生物功能信息。
根據(jù)Genecards的匹配結(jié)果發(fā)現(xiàn)肺癌組織獨有的三個HUB基因(POU3F2、KLF7、SHOX2)確實在生物功能上與重合基因存在差異,我們因此將其作為肺癌特異性基因。具體來說,多于一半的HUB基因同時在于肺癌組織網(wǎng)絡(luò)和健康肺組織網(wǎng)絡(luò)中重復(fù)出現(xiàn),它們?yōu)镾P1、SP4、KLF4、RFX2、CTCF、KLF16、RFX3,且它們大多為不同類型的轉(zhuǎn)錄因子,主要參與調(diào)控那些不論癌細胞還是正常細胞都要經(jīng)歷的細胞過程,包括細胞分化、細胞生長、細胞凋亡。而我們重點關(guān)注的肺癌組織基因調(diào)控網(wǎng)絡(luò)獨有HUB基因為:POU3F2、KLF7、SHOX2,其中POU3F2編碼的蛋白質(zhì)參與神經(jīng)元分化,并增強促腎上腺皮質(zhì)激素釋放激素調(diào)節(jié)基因的激活,它的過表達與黑色素瘤細胞增殖有關(guān)。KLF7編碼的蛋白通過抑制胰島β細胞中胰島素的表達和分泌,以及通過調(diào)節(jié)脂肪細胞中脂細胞因子的分泌來促進2型糖尿病的發(fā)展。SHOX2也是一種蛋白質(zhì)編碼基因,與它相關(guān)的疾病包括Turner綜合征和DeLange綜合征,其患者先天身形矮小。從簡單功能上已經(jīng)能看出這三個肺癌獨有HUB基因與其他基因的差異。
接下來,為了驗證所挖掘出肺癌特異性基因(POU3F2、KLF7、SHOX2)的有效性,我們查閱相關(guān)肺癌臨床實驗文獻試圖從生物實驗角度使其特異性再次得到驗證。結(jié)果是,對于SHOX2基因,多個研究已經(jīng)證明SHOX2基因DNA甲基化是檢測肺癌的強有力的生物標記物[24,25]。對于KLF7基因也有研究證明與鄰近的正常組織相比,患者肺腺癌(Lung Adenocarcinoma,LAC)組織中的KLF7表達升高,且KLF7的高蛋白水平與腫瘤大小相關(guān),此外,高的KLF7表達水平與肺腺癌患者較差的臨床結(jié)局顯著相關(guān),表明了KLF7作為新型預(yù)后生物標志物和治療靶標的潛在作用[26]。以上結(jié)果均表明本文利用復(fù)雜網(wǎng)絡(luò)節(jié)點中心性方法所識別出的肺癌特異性基因與臨床生物實驗所得出的結(jié)論是一致的,這證明了本文所提出方法的有效性。
4? ?結(jié)論(Conclusion)
隨著基因組學(xué)、蛋白質(zhì)組學(xué)、轉(zhuǎn)錄組學(xué)的發(fā)展,生物大數(shù)據(jù)得以涌現(xiàn),針對癌癥這一復(fù)雜人類疾病的相關(guān)研究也逐漸從臨床實驗轉(zhuǎn)移到數(shù)據(jù)分析上來。本文正是在這一背景下,借助復(fù)雜網(wǎng)絡(luò)理論這一系統(tǒng)性工具對肺癌基因數(shù)據(jù)進行建模分析,借助多種拓撲中心性算法,以及人類基因綜合數(shù)據(jù)庫提出在生物大數(shù)據(jù)中挖掘癌癥特異性基因的系統(tǒng)性方法:(1)構(gòu)建癌癥與對照組基因調(diào)控網(wǎng)絡(luò)。(2)分析網(wǎng)絡(luò)拓撲結(jié)構(gòu)和分布規(guī)律。(3)計算網(wǎng)絡(luò)節(jié)點中心性得分。(4)比對并匹配HUB節(jié)點生物功能。
通過分析發(fā)現(xiàn)基因調(diào)控網(wǎng)絡(luò)存在廣泛的無標度特性,即人類器官組織對應(yīng)的基因調(diào)控系統(tǒng)由少數(shù)重要基因來主導(dǎo)控制,它們調(diào)控著整個系統(tǒng)的行為,識別這些基因?qū)φJ識人類細胞的生物進程有重要意義。此外,健康肺組織細胞和肺癌組織細胞對應(yīng)的基因調(diào)控網(wǎng)絡(luò)從拓撲結(jié)構(gòu)到分布特征上都極其相似,這也證明了同處病變與健康組織的同源性。最后,利用本文所提出系統(tǒng)性方法識別出的肺癌特異性基因,其肺癌相關(guān)性也在本文中得到了證實,該基因?qū)⒆鳛橐粋€重要的肺癌生物標記物利用在臨床檢測上。總的來說,本文方法對從大規(guī)模癌癥基因數(shù)據(jù)中識別特異性基因提供了可靠思路,而癌癥特異性基因的存在將使癌癥在早期更容易被識別,進而提升治愈率。
參考文獻(References)
[1] Alzahouri K,Martinet Y,Briancon S,et al.Staging practices of primary non-small-cell lung cancer:a literature review[J].European Journal of Cancer Care,2006,15(4):348-354.
[2] Chen W,Zhang S,Zou X.Estimation and Projection of Lung Cancer Incidence and Mortality in China[J].Chinese journal of lung cancer,2010,13(5):488-493.
[3] Carlsson G.Topology and data[J].Bulletin of the American Mathematical Society,2009,46(2):255-308.
[4] Furney SJ,Alba MM,Lopezbigas N.Differences in the evolutionary history of disease genes affected by dominant or recessive mutations[J].BMC Genomics,2006,7(1):165-175.
[5] Park D,Park J,Park SG,et al.Analysis of human disease genes in the context of gene essentiality[J].Genomics,2008,92(6):414-418.
[6] Sun J,Zhao Z.A comparative study of cancer proteins in the human protein-protein interaction network[J].BMC Genomics,2010,11(S3):1471-1481.
[7] Gandhi TKB,Zhong J,Mathivanan S,et al.Analysis of the human protein interactome and comparison with yeast,worm and fly interaction datasets[J].Nature Genetics,2006,38(3):285-293.
[8] Kim J,Kim I,Han SK,et al.Network rewiring is an important mechanism of gene essentiality change[J].Scientific Reports,2012(2):900-907.
[9] He X,Zhang J.Why do hubs tend to be essential in protein networks?[J].PLoS genetics,2006,2(6):826-834.
[10] 周建平,梁立軒,李志芳.非小細胞肺癌細胞系中關(guān)鍵致癌相關(guān)基因突變檢測[J].攀枝花學(xué)院學(xué)報,2016,33(05):71-74.
[11] 俞訓(xùn)彬,陳小巖,陳靈鋒.采用Illumina測序技術(shù)檢測非小細胞肺癌驅(qū)動基因關(guān)鍵位點突變[J].臨床與實驗病理學(xué)雜志,2019,35(7):861-862.
[12] Marbach D,Lamparter D,Quon G,et al.Tissue-specific regulatory circuits reveal variable modular perturbations across complex diseases[J].Nature Methods,2016,13(4):366-370.
[13] Maslov S,Sneooen K.Specificity and Stability in Topology of Protein Networks[J].Science,2002,296(5569):910-913.
[14] Lin CY,Chin CH,Wu HH,et al.Hubba: hub objects analyzer—a framework of interactome hubs identification for network biology[J].Nucleic Acids Research,2008,36(S2):438-443.
[15] Chin CH,Chen SH,Wu HH,et al.cytoHubba: identifying hub objects and sub-networks from complex interactome[J].BMC Systems Biology,2014,8(S4):1752-1761.
[16] Sabidussi G.The centrality index of a graph[J].Psychometrika,1966,31(4):581-603.
[17] Dankelmann P,Goddard W,Swart C S.The Average Eccentricity of a Graph and its Subgraphs[J].Utilitas Mathematica,2004,65(2):41-51.
[18] Thomas WV,Robert KF.Integration and radiality:Measuring the extent of an individual's connectedness and reachability in a network[J].Social Networks,1998,20(1):89-105.
[19] Chin CS,Samanta MP.Global snapshot of a protein interaction network—a percolation based approach[J].Bioinformatics,2003,19(18):2413-2419.
[20] Alfonso S.Structural parameters of communication networks[J].Bulletin of Mathematical Biophysics,1953,15(4):501-507.
[21] Barthélemy M.Betweenness centrality in large complex networks[J].European Physical Journal B,2004,38(2):163-168.
[22] Przuij N,Wigle DA,Jurisica I.Functional topology in a network of protein interactions[J].Bioinformatics,2004,20(3):340-348.
[23] Rebhan M,Chalifa-Caspi V,Prilusky J,et al.GeneCards:a novel functional genomics compendium with automated data mining and query reformulation support[J].Bioinformatics,1998,14(8):656-664.
[24] Ilse P,Biesterfeld S,Pomjanski N,et al.Analysis of SHOX2 Methylation as an Aid to Cytology in Lung Cancer Diagnosis[J].Cancer genomics & proteomics,2014,11(5):251-258.
[25] Llse P,Biesterfeld S,Pomjanski N,et al.SHOX2 DNA Methylation Is a Tumour Marker in Pleural Effusions[J].Cancer genomic & proteomics,2013,10(5):217-223.
[26] Cai XD,Zhou YB,Huang LX,et al.Reduced expression of Krüppel-like factor 17 is related to tumor growth and poor prognosis in lung adenocarcinoma[J].Biochemical and Biophysical Research Communications,2012,418(1):67-73.
作者簡介:
于? ?曉(1991-),男,碩士生.研究領(lǐng)域:復(fù)雜網(wǎng)絡(luò).

