霜霉菌侵染后葡萄葉片酵母雙雜交cDNA文庫(kù)構(gòu)建
劉露露 曲俊杰 郭澤西 孫大運(yùn) 潘鳳英 尹玲
摘要:【目的】構(gòu)建霜霉菌侵染后葡萄葉片的酵母雙雜交cDNA文庫(kù),篩選致病因子在寄主葡萄體內(nèi)的互作靶標(biāo),為葡萄霜霉病菌致病的分子機(jī)理研究提供材料基礎(chǔ)。【方法】以一年生歐亞種葡萄西拉盆栽苗為試驗(yàn)材料,提取并等量混合霜霉菌侵染后0、12、18、24和36 h的葡萄葉片總RNA,利用基于Gateway技術(shù)的CloneMiner II cDNA文庫(kù)構(gòu)建試劑盒:首先將cDNA與pDONR222載體進(jìn)行BP重組反應(yīng)連接,連接產(chǎn)物轉(zhuǎn)化大腸桿菌DH10B感受態(tài)細(xì)胞構(gòu)建初級(jí)文庫(kù);然后初級(jí)文庫(kù)質(zhì)粒與pGADT7-DEST載體通過(guò)LR重組反應(yīng),重組反應(yīng)產(chǎn)物轉(zhuǎn)化大腸桿菌DH10B感受態(tài)細(xì)胞構(gòu)建次級(jí)文庫(kù);再隨機(jī)挑取次級(jí)文庫(kù)轉(zhuǎn)化Y187酵母菌株構(gòu)建酵母雙雜交cDNA文庫(kù);最后利用ImageJ軟件和PCR反應(yīng)分別統(tǒng)計(jì)庫(kù)容量和鑒定重組率。【結(jié)果】構(gòu)建的初級(jí)文庫(kù)庫(kù)容量為4.6×107 CFU,次級(jí)文庫(kù)庫(kù)容量為2.7×107 CFU,均達(dá)到構(gòu)建cDNA文庫(kù)的標(biāo)準(zhǔn)。酵母雙雜交cDNA文庫(kù)的插入片段主要集中在1000~2000 bp,且具有很好的多態(tài)性,文庫(kù)質(zhì)量較高。【結(jié)論】構(gòu)建的霜霉菌誘導(dǎo)葡萄葉片酵母雙雜交cDNA文庫(kù)符合酵母雙雜交篩選要求,可用于篩選霜霉菌致病效應(yīng)蛋白在寄主體內(nèi)的靶標(biāo)及霜霉菌侵染寄主葡萄早期抑制寄主免疫過(guò)程中的致病機(jī)理研究。
關(guān)鍵詞: 霜霉菌;葡萄;酵母雙雜交;cDNA文庫(kù)
Abstract:【Objective】To construct yeast two-hybrid cDNA library of grape leaves after downy mildew infection, screen the target of pathogenic factors in host grapes and provide reference for molecular mechanism of pathogenic factors of Plasmopara viticola. 【Method】Total RNA was extracted and mixed fromgrape leaves of Vitis vinifera Shiraz one-year potted seedlings at 0,12,18,24 and 36 h after downy mildew infection. The library was constructed using the CloneMiner II cDNA library construction kit based on Gateway technology. The cDNA was ligated into the pDONR222 vector by BP recombination reaction, and the ligation product was transformed into Escherichia coli DH10B competent cell to construct a primary library. Then the primary library plasmid was introduced into the pGADT7-DEST vector by LR recombination reaction, recombinant reaction products transformed E. coli DH10B competent cell to construct primary library, and secondary libraries were randomly selected to transform Y187 yeast strain to construct yeast two-hybrid cDNA library. The library capacity was counted by ImageJ software and the recombination rate was identified by PCR. 【Result】The tests showed that the capacity of the primary library obtained by the experiment was 4.6×107 CFU, the capacity of secondary library was? 2.7×107 CFU, reaching the standard to construct cDNA library. The inserts of the yeast twohybrid cDNA library were mainly concentrated at 1000-2000 bp and had good polymorphism. The library quality was high. 【Conclusion】A high-quality yeast two-hybrid cDNA library from grape leaves infected with downy mildew is obtained in this experiment. It meets the requirements of yeast two-hybrid screening, is a target that can be used to screen downy mildew pathogenic effect proteins in the host, and study the pathogenic mechanism during the early inhibition on host immunity of downy mildew infected host grapes.
0 引言
【研究意義】葡萄是經(jīng)濟(jì)價(jià)值較高的果樹(shù)之一,2018年國(guó)際葡萄及葡萄酒組織(OIV)發(fā)布的全球葡萄種植及葡萄酒生產(chǎn)、消費(fèi)和貿(mào)易統(tǒng)計(jì)數(shù)據(jù)顯示,我國(guó)已成為全球第二大葡萄種植國(guó),但生產(chǎn)上葡萄病害的流行限制了我國(guó)葡萄與葡萄酒產(chǎn)業(yè)的可持續(xù)發(fā)展。葡萄霜霉病是葡萄產(chǎn)業(yè)上危害嚴(yán)重的一大卵菌病害,其病原菌是葡萄霜霉菌(Plasmopara viticola),在植物十大病原卵菌排名中位居第六(Kamoun et al.,2015)。因此,構(gòu)建霜霉菌侵染后葡萄葉片酵母雙雜交cDNA文庫(kù),用于篩選霜霉菌效應(yīng)蛋白在寄主中的互作靶標(biāo),以深入研究葡萄霜霉菌效應(yīng)蛋白早期抑制葡萄免疫反應(yīng)過(guò)程的機(jī)理,可為今后基于靶標(biāo)研發(fā)新型殺菌劑提供理論依據(jù)。【前人研究進(jìn)展】過(guò)去的十多年,對(duì)葡萄霜霉菌的研究主要集中在種群遺傳多樣性、遺傳結(jié)構(gòu)及不同種群對(duì)殺菌劑的敏感性或抗藥性等方面。迄今為止,尚未明確我國(guó)葡萄霜霉菌種群的遺傳多樣性和起源,但有學(xué)者認(rèn)為我國(guó)葡萄霜霉菌是地方性和引進(jìn)菌株的混合體(Zhang et al.,2017a);在基因流動(dòng)性大且重組更頻繁的地區(qū),葡萄霜霉菌的遺傳多樣性更廣泛(Boso et al.,2019)。生產(chǎn)上對(duì)霜霉病的防治一直以化學(xué)防治為主,經(jīng)常使用的兩類殺菌劑是保護(hù)性殺菌劑和內(nèi)吸性殺菌劑,但目前在國(guó)內(nèi)已發(fā)現(xiàn)對(duì)烯酰嗎啉和嘧菌酯具有抗性的霜霉菌菌株(王喜娜,2017;Zhang et al.,2017b)。近年來(lái),隨著高通量測(cè)序技術(shù)的發(fā)展,對(duì)病原菌致病分子機(jī)理的研究得到迅猛發(fā)展,尤其是對(duì)病原菌效應(yīng)蛋白互作靶標(biāo)的研究取得了很多突破性進(jìn)展。研究表明,疫霉菌通過(guò)產(chǎn)生糖基水解酶(Ma et al.,2015)和果膠裂解酶(Fu et al.,2015)等類型的質(zhì)外體效應(yīng)蛋白破壞宿主細(xì)胞壁的物理屏障以獲得營(yíng)養(yǎng),發(fā)揮毒力;通過(guò)蛋白酶、蛋白酶抑制劑等其他幾種類型的質(zhì)外體效應(yīng)蛋白作用于寄主植物的β-1,3-葡聚糖酶(Bishop et al.,2005)、絲氨酸蛋白酶(Tian et al.,2004,2005)和半胱氨酸蛋白酶(Song et al.,2009;Kaschani et al.,2010)等PTI(PAMP-triggered immunity,病原相關(guān)分子模式觸發(fā)的免疫反應(yīng))過(guò)程相關(guān)的不同靶標(biāo)來(lái)干擾寄主的免疫反應(yīng),促進(jìn)侵染。胞內(nèi)效應(yīng)蛋白則靶向多種防衛(wèi)相關(guān)途徑促進(jìn)其侵染,如26S蛋白酶體降解(Bos et al.,2010;Yang et al.,2016)、激素抗病信號(hào)(Evangelisti et al.,2013)、絲裂原活化蛋白激酶抗病信號(hào)(King et al.,2014)、活性氧代謝(Zhang et al.,2015)及內(nèi)質(zhì)網(wǎng)脅迫(Jing et al.,2016)等。在葡萄霜霉菌中,目前僅發(fā)現(xiàn)PvRxLR131能靶向富含亮氨酸重復(fù)序列的類受體蛋白激酶(Leucine-rich repeat receptor-like protein kinase,LRR-RLK)相關(guān)的抑制子BKI1來(lái)干擾寄主的防御促進(jìn)侵染(Lan et al.,2019)。本課題組相繼完成了葡萄霜霉菌的轉(zhuǎn)錄組和全基因組測(cè)序,并通過(guò)生物信息學(xué)方法挖掘出大量的致病基因,共鑒定出1301個(gè)假定的分泌蛋白,包括100個(gè)RXLR(R: 精氨酸;X: 任意氨基酸:L: 亮氨酸)類型的效應(yīng)蛋白和90個(gè)CRN(Crinkling and necrosis protein,皺縮壞死蛋白)類型的效應(yīng)蛋白(Yin et al.,2015,2017)。【本研究切入點(diǎn)】本課題組前期已從葡萄霜霉菌基因組和轉(zhuǎn)錄組中克隆出82個(gè)候選RXLR效應(yīng)蛋白基因,并通過(guò)煙草葉片瞬時(shí)表達(dá)系統(tǒng)明確其對(duì)植物免疫的抑制作用,以及這些效應(yīng)蛋白進(jìn)入植物體后發(fā)揮作用的位置(Xiang et al.,2017;Liu et al.,2018),但其作用靶標(biāo)及具體的致病機(jī)理尚不明確。【擬解決的關(guān)鍵問(wèn)題】利用Gateway體系,采用Gateway位點(diǎn)特異性重組技術(shù)(Cloneminer cDNA library construction kit)構(gòu)建西拉葡萄葉片在霜霉菌侵染下的酵母雙雜交文庫(kù),以期用于篩選霜霉菌效應(yīng)蛋白在葡萄葉片中的互作靶標(biāo),為研究葡萄霜霉病菌致病的分子機(jī)理提供材料基礎(chǔ)。
1 材料與方法
1. 1 試驗(yàn)材料
供試葡萄品種為一年生歐亞種葡萄西拉盆栽苗,種植在廣西農(nóng)業(yè)科學(xué)院科研核心區(qū)玻璃溫室。試驗(yàn)所用葡萄霜霉菌菌株由廣西作物遺傳改良生物技術(shù)重點(diǎn)開(kāi)放實(shí)驗(yàn)室分離保存。
1. 2 試驗(yàn)方法
1. 2. 1 霜霉菌侵染 采用離體葉盤接種法,用蒸餾水將葡萄枝條頂端完全伸展的第3~5片葉清洗干凈,用吸水紙擦干備用。將葡萄葉片用打孔鉗打成直徑為1 cm的葉盤(避開(kāi)主葉脈和較大側(cè)葉脈),葉盤背面朝上置于鋪有完全濕潤(rùn)濾紙的培養(yǎng)皿上。吸取35 μL霜霉菌孢子囊懸浮液(濃度5×105個(gè)孢子囊/mL)滴于每個(gè)葉盤背面,23 ℃、16 h/8 h光周期培養(yǎng)12 h后,用濾紙吸取多余的菌液,葉盤繼續(xù)培養(yǎng),分別于接種0、12、18、24和36 h后取樣,每個(gè)時(shí)間點(diǎn)采集12個(gè)葉盤,液氮速凍后-80 ℃保存。
1. 2. 2 RNA提取、反轉(zhuǎn)錄 采用Spectrum Plant Total RNA Kit (Sigma)提取各時(shí)間點(diǎn)葉片的總RNA,1%瓊脂糖凝膠檢測(cè)RNA質(zhì)量和濃度;將每個(gè)時(shí)間點(diǎn)的RNA等量混合成一個(gè)樣品,利用NanoDrop和Agilent 2100檢測(cè)混合后樣品的質(zhì)量和濃度;使用Oligotex mRNA Midi Kit進(jìn)行mRNA分離純化,并以1%瓊脂糖凝膠檢測(cè)其質(zhì)量,用NanoDrop 2000分光光度計(jì)檢測(cè)其濃度。
1. 2. 3 初級(jí)文庫(kù)(Uncut型)構(gòu)建及保存 參照CloneMiner II cDNA文庫(kù)構(gòu)建試劑盒說(shuō)明,在上一步得到的mRNA中加入Biotin-aatB2-Oligo(dT)Primer和SuperScript Ⅲ RT酶進(jìn)行反轉(zhuǎn)錄,得到cDNA的第一鏈;再以得到的cDNA第一鏈為PCR模板,加入大腸桿菌DNA Ligase、大腸桿菌DNA Polymerase I、大腸桿菌RNase H及T4聚合酶合成cDNA的第二鏈;cDNA與三框attB1重組接頭連接(3種接頭分別各連接1份),目的是在鏈末端連接attB1序列。然后利用cDNA Size Fraction Columns對(duì)cDNA進(jìn)行分級(jí)分離和收集,收集500~4000 bp的雙鏈cDNA。將收集到的雙鏈cDNA與pDONR222載體混合,通過(guò)BP重組反應(yīng)連入載體。重組產(chǎn)物電轉(zhuǎn)化導(dǎo)入大腸桿菌DH10B感受態(tài)細(xì)胞中,搖菌培養(yǎng)(37 ℃,220 r/min,1 h)獲得cDNA文庫(kù)。培養(yǎng)1 h后,取轉(zhuǎn)化后大腸桿菌原液10 ?L,將其稀釋100倍后,吸取50 ?L稀釋液涂布LB培養(yǎng)基(含氨芐青霉素),從而進(jìn)行庫(kù)容量鑒定;從培養(yǎng)基上長(zhǎng)出的菌落中隨機(jī)挑取24個(gè)單克隆,進(jìn)行菌落PCR鑒定,對(duì)其插入片段長(zhǎng)度和重組率進(jìn)行分析;在剩余菌液中加入滅菌甘油至終濃度為20%,液氮速凍存于-80 ℃,此菌液為初級(jí)文庫(kù)菌液。
庫(kù)容量鑒定:CFU/mL=培養(yǎng)基上的克隆數(shù)/50 ?L×稀釋倍數(shù)×1×103 ?L。
文庫(kù)總CFU= CFU/mL×文庫(kù)菌液總體積(mL)。
1. 2. 4 次級(jí)文庫(kù)構(gòu)建及保存 將驗(yàn)證成功的初級(jí)文庫(kù)加入相應(yīng)抗性的LB培養(yǎng)基,30 ℃搖床過(guò)夜培養(yǎng),培養(yǎng)結(jié)束后抽提質(zhì)粒,測(cè)OD并進(jìn)行電泳檢測(cè)。將抽提得到的質(zhì)粒稀釋至300 ng/?L,與pGADT7-DEST載體混合進(jìn)行LR重組反應(yīng),反應(yīng)產(chǎn)物電轉(zhuǎn)化大腸桿菌DH10B感受態(tài)細(xì)胞,將轉(zhuǎn)化的產(chǎn)物接種到新的15 mL離心管,37 ℃、250 r/min搖床中培養(yǎng)1 h;將培養(yǎng)物涂布培養(yǎng)基,從培養(yǎng)基上長(zhǎng)出的菌落中隨機(jī)挑取24個(gè)單克隆,進(jìn)行菌落PCR鑒定,對(duì)其插入片段長(zhǎng)度和重組率進(jìn)行分析;在剩余菌液中加入滅菌甘油至終濃度為20%,液氮速凍存于-80 ℃,此菌液為次級(jí)文庫(kù)菌液。
1. 2. 5 酵母雙雜交cDNA文庫(kù)質(zhì)量鑒定 用5 ?g次級(jí)文庫(kù)質(zhì)粒轉(zhuǎn)化Y187酵母菌株,在150-mm SD/–Leu培養(yǎng)基上鋪100 ?L稀釋比例為1∶10、1∶100、1∶1000和1∶10000的稀釋液,30 ℃倒置溫育培養(yǎng)基直到克隆出現(xiàn)(3~6 d);收集轉(zhuǎn)化子,確定文庫(kù)的滴度,并挑取24個(gè)克隆,利用T7SP Primer和3' AD Primer進(jìn)行PCR擴(kuò)增,電泳檢測(cè)插入cDNA片段的大小。
1. 2. 6 菌落計(jì)數(shù)方法 在ImageJ軟件中打開(kāi)圖片,轉(zhuǎn)換RGB圖像為灰度圖片:Image–>Type–>8-bit。閾值選定:Image–>Adjust–>Threshold–>閾值選定->紅色代表選中。培養(yǎng)皿邊緣高亮部分影響菌落的識(shí)別,使用橢圓選框大致選中菌落所在區(qū)域。Analyze–>Analyze particles,點(diǎn)擊OK,得到菌落計(jì)數(shù)結(jié)果。
2 結(jié)果與分析
2. 1 總RNA提取及mRNA分離純化
為研究葡萄霜霉菌效應(yīng)蛋白早期抑制葡萄免疫反應(yīng)過(guò)程的機(jī)理,利用Spectrum Plant Total RNA Kit分別提取霜霉菌侵染西拉葡萄0、12、18、24和36 h后的葉片總RNA,核酸電泳檢測(cè)結(jié)果(圖1)顯示,從5個(gè)時(shí)間點(diǎn)樣品提取的總RNA的28S和18S條帶均清晰明亮,且完整性較好。將5個(gè)樣品等量混合后,用NanoDrop 2000和Agilent 2100進(jìn)行測(cè)定,結(jié)果顯示,混合后的RNA樣品濃度為596 ng/?L,A260/A280=2.14,A260/A230=2.13,28S/18S=2.0,RIN=8.4,質(zhì)量滿足試驗(yàn)要求(RIN≥7且28S/18S≥0.7),可進(jìn)行后續(xù)文庫(kù)建庫(kù)試驗(yàn)。瓊脂糖凝膠電泳檢測(cè)結(jié)果(圖2)顯示,經(jīng)Oligotex mRNA Midi Kit分離純化后的mRNA在≥0.5 kb較大范圍內(nèi)呈彌散狀,無(wú)明顯28S和18S rRNA區(qū)帶,大部分的mRNA集中在0.5~2.0 kb范圍內(nèi)呈彌散狀,mRNA無(wú)DNA污染且完整性較好,適用于后續(xù)構(gòu)建文庫(kù)。
2. 2 初級(jí)文庫(kù)的構(gòu)建及鑒定
將pDONR222載體與反轉(zhuǎn)錄得到的雙鏈cDNA按比例混合進(jìn)行BP重組反應(yīng),轉(zhuǎn)化大腸桿菌DH10B感受態(tài)細(xì)胞后,得到初級(jí)文庫(kù)。吸取初級(jí)文庫(kù)原液10 ?L稀釋100倍,將50 ?L稀釋液涂布于LB+Amp的培養(yǎng)基上,ImageJ軟件讀出的克隆總數(shù)為5719個(gè),菌液量為4 mL,根據(jù)庫(kù)容量的計(jì)算方法,文庫(kù)總CFU=5719/50 ?L×100倍×1×103 ?L×4 mL≈4.6×107 CFU(圖3),說(shuō)明本研究構(gòu)建的庫(kù)容量較高。在庫(kù)中隨機(jī)挑取24個(gè)克隆進(jìn)行菌落PCR鑒定,瓊脂糖凝膠電泳檢測(cè)結(jié)果(圖4)顯示,陽(yáng)性率為96%,擴(kuò)增出的插入片段大小不一,分布在750~2000 bp,僅有一個(gè)菌落未擴(kuò)增出條帶(白色箭頭所示),大多數(shù)條帶大小集中于1000~2000 bp。綜上所述,該cDNA文庫(kù)的質(zhì)量和庫(kù)容量較好,可用于篩選后續(xù)效應(yīng)因子的靶標(biāo)蛋白。
2. 3 次級(jí)文庫(kù)的構(gòu)建及質(zhì)量鑒定
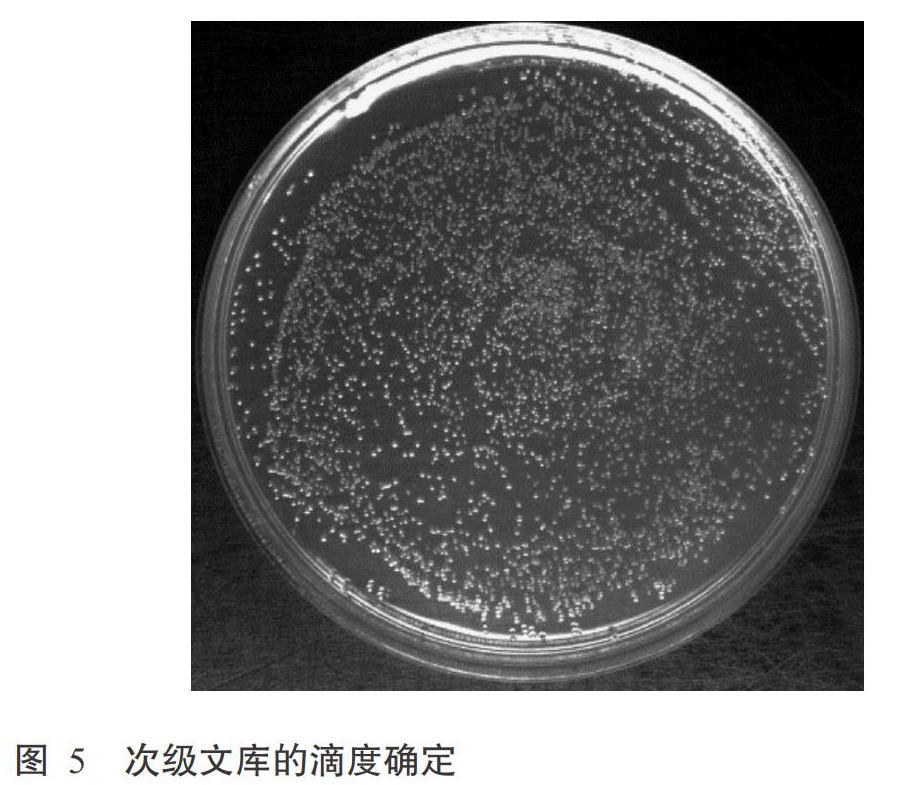
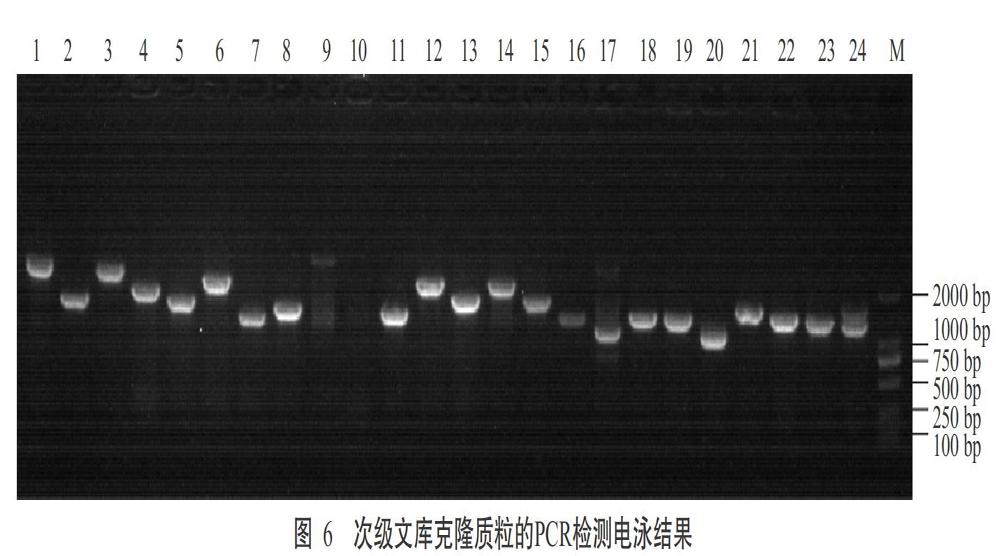
將pGADT7-DEST載體與構(gòu)建合格的初級(jí)文庫(kù)質(zhì)粒按比例混合后進(jìn)行LR反應(yīng),重組產(chǎn)物轉(zhuǎn)化大腸桿菌DH10B感受態(tài)細(xì)胞后得到次級(jí)文庫(kù)菌液。吸取10 ?L細(xì)菌原液稀釋100倍后,取50 ?L涂布于LB+Amp的培養(yǎng)基上,ImageJ軟件讀出的克隆數(shù)為3352個(gè),根據(jù)公式計(jì)算次級(jí)文庫(kù)庫(kù)容量總CFU=3352/50 ?L×100倍× 1×103 ?L×4 mL≈2.7×107 CFU(圖5)。在次級(jí)文庫(kù)中隨機(jī)挑取24個(gè)克隆進(jìn)行PCR檢測(cè),瓊脂糖凝膠電泳檢測(cè)結(jié)果(圖6)顯示,陽(yáng)性重組率為96%,擴(kuò)增出的插入片段分布在750~2000 bp,主要集中在1000~2000 bp,說(shuō)明本研究構(gòu)建的庫(kù)容量較高。
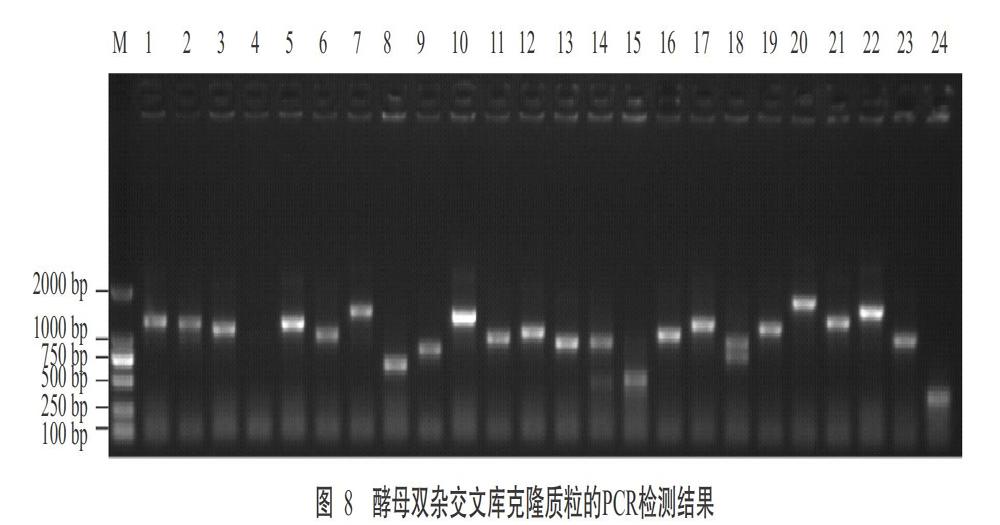
用5 ?g文庫(kù)質(zhì)粒轉(zhuǎn)化Y187酵母菌株,在150-mm SD/–Leu培養(yǎng)基上鋪100 ?L稀釋比例為1∶10、1∶100、1∶1000和1∶10000的稀釋液,30 ℃倒置溫育直到克隆出現(xiàn)(3~6 d)(圖7);收集轉(zhuǎn)化子,確定文庫(kù)的滴度,并挑取24個(gè)克隆,利用T7SP Primer和3' AD Primer進(jìn)行PCR擴(kuò)增,電泳檢測(cè)插入cDNA片段的大小(圖8),擴(kuò)增出的插入片段分布在250~2000 bp,主要集中在1000~2000 bp,說(shuō)明本文庫(kù)重組的cDNA序列的完整性高,符合酵母雙雜交篩選要求。
3 討論
目前,用于酵母雙雜交cDNA文庫(kù)構(gòu)建的主流方法有Clonetech的SMART方法和Invitrogen公司基于Gateway技術(shù)的方法,近年來(lái)上海海科公司還推出了All-Direct方法。Clonetech的SMART方法主要優(yōu)點(diǎn)在于建庫(kù)所需的RNA起始量很小,只要幾微克即可,成本相對(duì)較低。Invitrogen的方法對(duì)起始RNA的要求較高,建議起始用量大于200 ?g,不需要PCR擴(kuò)增,保證了文庫(kù)基因的覆蓋度和真實(shí)性;SMART方法不分離mRNA,而Invitrogen的方法在建庫(kù)過(guò)程中會(huì)進(jìn)行mRNA的分級(jí)分離,去除文庫(kù)內(nèi)核糖體RNA及基因組DNA等對(duì)反轉(zhuǎn)錄的影響;SMART方法使用酶切連接或酵母細(xì)胞內(nèi)重組的方式,進(jìn)行片段與載體連接,而Invitrogen使用高效率的Gateway體外重組方式,文庫(kù)的重組率及原始庫(kù)容均較高,且得到的文庫(kù)質(zhì)粒還可轉(zhuǎn)移到其他任意載體上形成其他用途的文庫(kù),既提高了文庫(kù)的使用率,又?jǐn)U大了文庫(kù)的使用范圍;SMART方法構(gòu)建的文庫(kù)只能采用Mating的方法進(jìn)行篩庫(kù),而Invitrogen的文庫(kù)既可用共轉(zhuǎn)也可采用Mating的方法。All-Direct方法是在Invitrogen方法的基礎(chǔ)上進(jìn)行進(jìn)一步優(yōu)化,只需經(jīng)過(guò)一次重組直接連接到目的載體上,從而極大提高了文庫(kù)的質(zhì)量。
為高效率和高質(zhì)量地篩選互作靶蛋白,構(gòu)建高質(zhì)量的酵母雙雜交cDNA文庫(kù)具有必要性。評(píng)價(jià)一個(gè)酵母雙雜交cDNA文庫(kù)是否為高質(zhì)量主要有兩個(gè)方面:(1)庫(kù)容量,是指構(gòu)建的cDNA文庫(kù)中所包含的重組子克隆數(shù),其數(shù)量反映了一個(gè)cDNA文庫(kù)的覆蓋度,理論上一個(gè)覆蓋度完整的cDNA文庫(kù)庫(kù)容量至少有1×106 CFU(李珣等,2016)。本研究構(gòu)建的初級(jí)和次級(jí)cDNA文庫(kù)的庫(kù)容量分別為4.6×107 CFU和2.7×107 CFU,均達(dá)到建庫(kù)的標(biāo)準(zhǔn),說(shuō)明本文庫(kù)覆蓋度完整。(2)基因的重組率和插入片段長(zhǎng)度,即重組cDNA序列的完整性(王淑紅等,2008)。本研究構(gòu)建的初級(jí)和次級(jí)cDNA文庫(kù)基因重組率均為96%,且插入片段主要集中在1000~2000 bp,說(shuō)明本文庫(kù)重組的cDNA序列完整性高。
目前已報(bào)道的2個(gè)葡萄霜霉菌侵染葡萄葉片cDNA文庫(kù)均運(yùn)用SMART方法構(gòu)建,利用的材料是霜霉菌侵染抗病品種葡萄葉片不同時(shí)間點(diǎn)的RNA,主要用于抗病基因的抗病分子機(jī)理研究(王沛雅,2008;王沛雅等,2009)。本研究利用Gateway技術(shù)構(gòu)建的酵母雙雜交cDNA文庫(kù),為篩選霜霉菌致病效應(yīng)蛋白在寄主體內(nèi)的靶標(biāo)打下基礎(chǔ),為今后針對(duì)性地設(shè)計(jì)殺菌藥物的靶標(biāo)提供可行的思路和策略。
4 結(jié)論
本研究通過(guò)Gateway技術(shù)構(gòu)建了高質(zhì)量的霜霉菌侵染后葡萄葉片酵母雙雜交cDNA文庫(kù),在庫(kù)容量、基因重組率和外源插入片段長(zhǎng)度方面均符合cDNA文庫(kù)構(gòu)建要求,且該方法得到的文庫(kù)質(zhì)粒還可轉(zhuǎn)移到其他任意載體上形成其他用途的文庫(kù),簡(jiǎn)單易行,為篩選霜霉菌致病效應(yīng)蛋白在寄主體內(nèi)的靶標(biāo)提供了材料基礎(chǔ),可用于下一步深入研究葡萄霜霉菌侵染葡萄的具體致病機(jī)理。
參考文獻(xiàn):
李珣,陳思宇,胡傳活,王曉曄. 2016. PK-15細(xì)胞酵母雙雜交三框cDNA文庫(kù)構(gòu)建及鑒定[J]. 南方農(nóng)業(yè)學(xué)報(bào),47(5):726-730. [Li X,Chen S Y,Hu C H,Wang X Y. 2016. Construction and identification of yeast two-hybrid three-frame cDNA library of PK-15 cells[J]. Journal of Sou-thern Agriculture,47(5):726-730.]
王沛雅. 2008. 葡萄霜霉菌誘導(dǎo)華東葡萄抗霜霉病基因cDNA文庫(kù)構(gòu)建及EST分析[D]. 楊凌: 西北農(nóng)林科技大學(xué). [Wang P Y. 2008. Construction of cDNA library and analysis of expressed sequence tags of Chinese wild Vitis pseudoreticulata induced by Plasmopara Viticola[D]. Yangling: Northwest A & F University.]
王沛雅,王躍進(jìn),張建文,朱自果,張朝紅. 2009. 華東葡萄白河-35-1株系抗霜霉病基因cDNA文庫(kù)構(gòu)建及EST分析[J]. 農(nóng)業(yè)生物技術(shù)學(xué)報(bào),17(2): 294-300. [Wang P Y,Wang Y J,Zhang J W,Zhu Z G,Zhang C H. 2009. Construction of cDNA library and analysis of expressed sequence tags of Chinese wild Vitis pseudoreticulata clone baihe-35-1 resistance to downy mildew[J]. Journal of Agri-cultural Biotechnology,17(2): 294-300.]
王淑紅,鄒志華,張子平,施永春,王藝?yán)? 2008. 副溶血弧菌感染的九孔鮑血細(xì)胞均一化全長(zhǎng)cDNA文庫(kù)的構(gòu)建[J]. 臺(tái)灣海峽,27(3): 278-285. [Wang S H,Zou Z H,Zhang Z P,Shi Y C,Wang Y L. 2008. Construction a normali-zed full-length cDNA library of haemocytes from Halio-tis diversicolor supertexta infected with Vibrio parahaemolyticus[J]. Journal of Oceanography in Taiwan Strait,27(3): 278-285.]
王喜娜. 2017. 我國(guó)主要葡萄產(chǎn)區(qū)霜霉菌對(duì)烯酰嗎啉和嘧菌酯的抗藥性分析[D]. 北京: 中國(guó)農(nóng)業(yè)科學(xué)院. [Wang X N. 2017. Detection of dimethomorph and asoxystrobin resistant isolates of Plasmopara viticola from main grapevine areas in China[D]. Beijing: Chinese Academy of Agricultural Sciences.]
Bishop J G,Ripoll D R,Bashir S,Damasceno C M B,Seeds J D,Rose J K C. 2005. Selection on glycine beta-1,3-endoglucanase genes differentially inhibited by a Phytophthora glucanase inhibitor protein[J]. Genetics,169: 1009-1019.
Bos J I B,Armstrong M R,Gilroy E M,Boevink P C,Hein I,Taylor R M,Tian Z,Engelhardt S,Vetukuri R R,Harrower B,Dixelius C,Bryan G,Sadanandom A,Whisson S C,Kamoun S,Birch P R J. 2010. Phytophthora infestans effector AVR3a is essential for virulence and manipulates plant immunity by stabilizing host E3 ligase CMPG1[J]. Proceedings of the National Academy of Sciences of the United States of America,107(21): 9909-9914.
Boso S,Gago P,Santiago J L,F(xiàn)uente M,Martínez M C. 2019. Factors affecting the vineyard populational diver-sity of Plasmopara viticola[J]. The Plant Pathology Journal,35(2):125-136.
Evangelisti E,Govetto B,Minet-Kebdani N,Kuhn M L,Attard A,Ponchet M,Panabieres F,Gourgues M. 2013. The Phytophthora parasitica RXLR effector penetration-specific effector 1 favours Arabidopsis thaliana infection by interfering with auxin physiology[J]. New Phytologist,199(2): 476-489.
Fu L,Zhu C Y,Ding X M,Yang X Y,Morris P F,Tyler B M,Zhang X. 2015. Characterization of cell-death-indu-cing members of the pectate lyase gene family in Phytophthora capsici and their contributions to infection of pe-pper[J]. Molecular Plant-Microbe Interactions,28(7): 766-775.
Jing M,Guo B,Li H,Yang B,Wang H,Kong G,Zhao Y,Xu H,Wang Y,Ye W,Dong S,Qiao Y,Tyler B M,Ma W,Wang Y. 2016. A Phytophthora sojae effector suppresses endoplasmic reticulum stress-mediated immunity by stabilizing plant binding immunoglobulin proteins[J]. Nature Communications,7(1): 1-17.
Kamoun S,F(xiàn)urzer O,Jones J D G,Judelson H S,Ali G S,Dalio R J D,Roy S G,Schena L,Zambounis A,Panabieres F,Cahill D,Ruocco M,F(xiàn)igueiredo A,Chen X R,Hulvey J,Stam R,Lamour K,Gijzen M,Tyler B M,Grunwald N J,Mukhtar M S,Tome D F A,Tor M,Ackerveken G V D,Mcdowell J,Daayf F,F(xiàn)ry W E,Kreuze H L,Meijer H J G,Petre B,Ristaino J,Yoshida K,Birch P R J,Govers F. 2015. The Top 10 oomycete pathogens in molecular plant pathology[J]. Molecular Plant Pathology,16(4): 413-434.
Kaschani F,Shabab M,Bozkurt T,Shindo T,Schornack S,Gu C,IIyas M,Win J,Kamoun S,Hoorn R A L. 2010. An effector-targeted protease contributes to defense against Phytophthora infestans and is under diversifying selection in natural hosts[J]. Plant Physiology,154(4): 1794-1804.
King S R F,McLellan H,Boevink P C,Armstrong M R,Bukharova T,Sukarta O,Win J,Kamoun S,Birch P R J,Banfield M J. 2014. Phytophthora infestans RXLR effector PexRD2 interacts with host MAPKKK to suppress plant immune signal[J]. The Plant Cell,26(3): 1345-1359.
Lan X,Liu Y X,Song S R,Yin L,Xiang J,Qu J J,Lu J. 2019. Plasmopara viticola effector PvRXLR131 suppresses plant immunity by targeting plant receptor-like kinase inhibitor BKI1[J]. Molecular Plant Pathology,20(6): 765-783.
Liu Y X,Lan X,Song S R,Yin L,Dry I,Qu J J,Xiang J,Lu J. 2018. In planta functional analysis and subcellular localization of the oomycete pathogen Plasmopara viticola candidate RXLR effector repertoire[J]. Frontiers in Plant Science,9: 286.
Ma Z C,Song T Q,Zhu L,Ye W W,Wang Y,Shao Y Y,Dong S M,Zhang Z G,Dou D L,Zheng X B,Tyler B M,Wang Y C. 2015. A Phytophthora sojae glycoside hydrolase 12 protein is a major virulence factor during soybean infection and is recognized as a PAMP[J]. The Plant Cell,27(7): 2057-2072.
Song J,Win J,Tian M Y,Schornack S,Kaschani F,IIyas M,Hoorn R A L,Kamoun S. 2009. Apoplastic effectors secreted by two unrelated eukaryotic plant pathogens target the tomato defense protease Rcr3[J]. Proceedings of the National Academy of Sciences of the United States of America,106: 1654-1659.
Tian M Y,Benedetti B,Kamoun S. 2005. A second Kazal-like protease inhibitor from Phytophthora infestans inhi-bits and interacts with the apoplastic pathogenesis-related protease P69B of tomato[J]. Plant Physiology,138: 1785-1793.
Tian M Y,Huitema E,Cunha L,Torto-Alalibo T,Kamoun S. 2004. A Kazal-like extracellular serineprotease inhibitor from Phytophthora infestans targets the tomato pathogene-sis-related protease P69B[J]. The Journal of Biological Chemistry,279: 26370-26377.
Xiang J,Li X L,Yin L,Liu Y X,Zhang Y L,Qu J J,Lu J. 2017. A candidate RxLR effector from Plasmopara viticola can elicit immune responses in Nicotiana benthamiana[J]. BMC Plant Biology,17(1): 75.
Yang L N,McLellan H,Naqvi S,He Q,Boevink P C,Armstrong M,Giuliani L m,Zhang W,Tian Z,Zhan J,Gilroy E M,Birch P R J. 2016. Potato NPH3/RPT2-like protein StNRL1,targeted by a Phytophthora infestans RXLR effector,is a susceptibility factor[J]. Plant Physiology,171(1): 645-657.
Yin L,An Y H,Qu J J,Li X L,Zhang Y L,Dry I,Wu H J,Lu J. 2017. Genome sequence of Plasmopara viticola and insight into the pathogenic mechanism[J]. Scientific Reports,7: 46553.
Yin L,Li X L,Xiang J,Qu J J,Zhang Y L,Dry I,Lu J. 2015. Characterization of the secretome of Plasmopara viticola by de novo transcriptome analysis[J]. Physiological and Molecular Plant Pathology,91: 1-10.
Zhang H,Kong F F,Wang X N,Liang L S,Schoen C D,Wang Z Y. 2017b. Tetra-primer ARMS PCR for rapid detection and characterization of Plasmoparav iticola phenotypes resistant to carboxylic acid amide(CAA) fungicides[J]. Pest Management Science,73(5): 557-563.
Zhang M X,Li Q,Liu T L,Liu L,Shen D Y,Zhu Y,Liu P H,Zhou J M,Dou D L. 2015. Two cytoplasmic effectors of Phytophthora sojae regulate plant cell death via intera-ctions with plant catalases[J]. Plant Physiology,167(1): 164-175.
Zhang W,Manawasinghe I S,Zhao W S,Xu J P,Brooks S,Zhao X Y,Hyde K,Chethana K,Liu J H,Li X H,Yan J Y. 2017a. Multiple gene genealogy reveals high genetic diversity and evidence for multiple origins of Chinese Plasmopara viticola population[J]. Scientific Reports,7(1): 17304.
(責(zé)任編輯 麻小燕)

