華山松SSR-PCR反應體系優化
高麗云 徐劍 李偉 陳喬穩 王凡 董章宏 瞿紹宏 常曉勇 辛培堯

摘要:【目的】建立華山松SSR-PCR最佳反應體系,為華山松種質資源的分子標記輔助育種、遺傳多樣性分析及遺傳圖譜構建研究提供理論參考。【方法】以華山松幼嫩針葉為材料,分別采用試劑盒法、SDS法和改良CTAB法提取華山松基因組DNA,確定華山松DNA最佳的提取方法。通過L16(44)正交試驗設計對華山松SSR-PCR反應體系中引物用量(A)、dNTP用量(B)、Taq DNA聚合酶用量(C)和DNA模板用量(D)進行優化,獲得華山松SSR-PCR最佳反應體系。【結果】改良CTAB法提取的基因組DNA濃度為92.1~1786.3 ng/μL,OD260/OD280為1.80~2.07,OD260/OD230比值約2.0,條帶明亮且清晰,無彌散現象,表明該方法提取效果最佳。正交試驗極差分析結果顯示,4個因素影響華山松SSR-PCR反應體系擴增效果的主次順序為A>D>B>C,最優水平組合為A4B2C2D2。正交試驗方差分析結果顯示,4個因素影響華山松SSR-PCR反應體系擴增效果的主次順序為A>D>C>B,最優水平組合為A4D2C2B2。結合正交試驗16個組合的評分結果,最終確定華山松SSR-PCR最佳反應體系(20.00 ?L):10 ?mol/L引物0.35 ?L,50 ng/?L DNA模板1.00 ?L,10 mmol/L dNTP 2.00 ?L,5 U/?L Taq DNA聚合酶1.20 ?L,10×PCR Buffer(含Mg2+ 15 mmol/L)2.00 ?L,用ddH2O補足至20.00 ?L。【結論】優化獲得的華山松SSR-PCR最佳反應體系可應用于華山松種質資源評價及分子標記輔助育種等研究。
關鍵詞: 華山松;SSR-PCR反應體系;優化;DNA提取
Abstract:【Objective】To establish the optimal SSR-PCR reaction system for Pinus armandii Franch.,and provide the theoretical and practical guidance for the germplasm resources evaluation and molecular marker-assisted breeding of P.armandii. 【Method】The young planting needles of P.armandii were as materials. P.armandii genome DNA were extracted by DNA extraction kit,SDS method and modified CTAB method to find the best DNA extraction method of P.armandii? by comparing the three? results. Through the L16(44) orthogonal design, four factors of Taq DNA polymerase amount,dNTPs amount,primer amount and template DNA amount in the SSR-PCR reaction system of P.armandii were optimized to obtain the best P.armandii SSR-PCR reaction system. 【Result】The concentration of genomic DNA, which extracted by the improved CTAB method, was 92.1-1786.3 ng/μL, and OD260/OD230 was 1.80-2.07, the ratio of OD260/OD230 was about 2.0, and the bands were bright and clear, without dispersion phenomenon, indicating that this method had the best extraction effect. The range analysis results of orthogonal test showed that the four factors which affected the amplification effect of P.armandii SSR-PCR reaction system were: A>D>B>C, and the optimal level combination was: A4B2C2D2. Orthogonal test analysis of variance showed that the four factors affecting the amplification effect of P.armandii SSR-PCR reaction system were: A>D>C>B, and the optimal level combination was: A4D2C2B2. Combining the scoring results of 16 combinations of orthogonal experiments, the best reaction system for SSR-PCR of P.armandii(20.00 ?L) was as follows: 0.35 μL primer(10 μmol/L);1.00 μL template DNA (50 ng/μL);2.00 μL dNTP (10 mmol/L),1.20 μL(5 U/μL) Taq DNA polymerase and 2.00 μL of 10×PCR Buffer(containing Mg2+ 15 mmol/L),then added to a volume of 20.00 μL with ddH2O. 【Conclusion】The optimal reaction system of P.armandii SSR-PCR? can be applied to the evaluation of germplasm resources and molecular marker-assisted breeding of P.armandii.
0 引言
【研究意義】華山松(Pinus armandii Franch.)又名白松,隸屬于松科(Pinaceae)松屬(Pinus)單維管束亞屬的高大常綠喬木,是我國特有的五針松樹種(辛培堯等,2010)。華山松生長較快,材質優良,其木材可作為建筑、家具原料,樹干可割取松脂,樹皮可提煉拷膠,針葉可提煉芳香油,種子還可食用。因此,華山松被我國多個省(區)引種造林。但由于人們沿襲“見樹采種,見種造林”的習慣,導致人工林分質量不高、病蟲害控制能力較差,致使其生長量持續減少,出現嚴重的衰退現象(孫海燕等,2007;辛培堯等,2010)。目前,運用遺傳改良手段選育良種是華山松種質創新的有效途徑之一。其中,以特異引物PCR為基礎的分子標記輔助育種技術廣受人們青睞,可在育種早期較準確地檢測目標性狀,從而達到苗期高效選擇的目的。SSR分子標記已廣泛應用于植物遺傳多樣性分析、遺傳圖譜構建、基因定位等研究(孫文婷等,2016;馮飛等,2018;邱芬等,2018;龐新華等,2019)。因此,優化華山松SSR-PCR反應體系對研究華山松的輔助育種及遺傳多樣性分析具有重要意義。【前人研究進展】目前,已有大量采用正交設計或單因素試驗等方法對思茅松、火炬松和云南松等樹種SSR-PCR反應體系進行優化的研究報道。張冬梅等(2007)分析了Taq DNA聚合酶、Mg2+、dNTPs、引物及DNA模板等因素對PCR擴增產物結果的影響,建立了一套穩定可靠的油松SSR-PCR反應體系。馮富娟等(2010)采用單因素試驗與正交試驗相結合的方法對影響紅松SSR-PCR擴增效果的DNA模板、Mg2+、dNTPs、引物、Taq DNA聚合酶、延伸時間和循環次數等進行優化,建立了穩定的紅松SSR-PCR最佳反應體系和擴增程序。邵俊培等(2012)采用單因素試驗對馬尾松EST-SSR PCR反應體系中各組分及退火溫度對擴增效果的影響進行研究,并對優化后的體系和擴增程序進行驗證。張瑞麗等(2012)、蔡年輝等(2013)分別采用正交試驗和單因素試驗分析云南松SSR-PCR反應體系中各組分對擴增效果的影響,篩選出各因素的最佳水平。何衛龍等(2013)利用正交試驗,以馬尾松胚乳DNA為模板進行SSR-PCR反應體系優化,建立了最佳SSR-PCR反應體系。張蝶等(2016)采用單因素試驗和正交試驗相結合的方法對SSR-PCR反應體系的DNA模板、引物、dNTPs、Mg2+和Taq DNA聚合酶進行優化,從而獲得火炬松SSR-PCR最佳反應體系。王大瑋等(2018)以思茅松基因組DNA為模板,對SSR-PCR反應體系中的DNA模板、Mg2+、引物、Taq DNA聚合酶和dNTPs進行正交試驗,建立了一套穩定可靠的SSR-PCR反應體系,可用于思茅松遺傳多樣性研究。【本研究切入點】雖然Dong等(2016)、祝娟(2016)利用SSR分子標記對華山松及其近緣種的群體遺傳學和物種形成進行研究,但未對華山松SSR-PCR反應體系進行優化。目前,鮮見運用正交試驗和SSR分子標記對華山松SSR-PCR反應體系各組分進行優化的研究報道。【擬解決的關鍵問題】以幼嫩無病蟲害的華山松針葉為材料,采用改良CTAB法提取華山松基因組DNA,通過L16(44)正交試驗對華山松SSR-PCR反應體系中的Taq DNA聚合酶、dNTPs、引物和DNA模板等4個組分進行優化,以獲得最佳的SSR-PCR反應體系,為華山松的分子標記輔助育種、遺傳多樣性分析及遺傳圖譜構建等研究提供理論參考。
1 材料與方法
1. 1 試驗材料
供試華山松種源為云南會澤、巍山、騰沖、楚雄、南華和宜良,種植于云南省楚雄市紫溪山紫金山林場華山松無性系種子園內(表1)。Taq DNA聚合酶、10×PCR Buffer(含15 mmol/L Mg2+)和dNTPs均購自北京博邁德基因技術有限公司;DNA Marker為1500 bp廣譜高密度分子量標準,購自北京天恩澤基因科技有限公司;Ezup柱式植物基因組DNA抽提試劑盒購自生工生物工程(上海)股份有限公司。主要儀器設備:低溫高速離心機、微量迷你離心機FC5306[奧豪斯國際貿易(上海)有限公司]、PCR擴增儀(S1000 Thermal Cycler,美國Bio-Rad)、瓊脂糖凝膠電泳儀(JY100型,北京君意東方電泳設備有限公司)、超微量紫外分光光度計(NanoDrop 2000,美國Thermo Scientific)、凝膠成像系統(SYNGENEG:BOX)等。
1. 2 試驗方法
1. 2. 1 樣品采集 分別采集華山松當年生幼嫩無病蟲害的針葉,按單株編號并裝入自封袋內,置于-80 ℃冰箱保存。
1. 2. 2 基因組DNA提取 隨機選取10個華山松無性系針葉樣品,分別采用試劑盒法(Ezup柱式植物基因組DNA抽提試劑盒)、SDS法和改良CTAB法提取華山松基因組DNA,利用超微量紫外分光光度計檢測其純度和濃度,并用0.8%瓊脂糖凝膠電泳進行檢測并拍照(謝冬梅等,2014),比較3種方法的提取效果,篩選出最佳的華山松基因組DNA提取方法。
1. 2. 3 引物初篩 利用會澤、巍山、騰沖和楚雄4個種源的華山松無性系基因組DNA進行PCR擴增。所用SSR引物序列(祝娟,2016)見表2,委托生工生物工程(上海)股份有限公司合成。擴增產物用1.0%瓊脂糖凝膠電泳進行檢測,篩選出擴增效果較好的SSR引物用于SSR-PCR體系優化。
1. 2. 4 正交試驗 從6個華山松種源的樣品中各選一個無性系,以其基因組DNA為模板,利用初篩出的SSR引物進行SSR-PCR反應體系優化。以引物(10 ?mol/L)、dNTPs(10 mmol/L)、Taq DNA聚合酶(5 U/μL)和DNA模板(50 ng/mL)的用量為4個因素進行L16(44)正交試驗(李春喜等,2008)。每個因素設4個水平(表3),2次重復,每重復16個組合。按照表4配制SSR-PCR反應體系,各體系均加入2.00 μL 10×PCR Buffer緩沖液,用ddH2O補足至20.00 μL。擴增程序:94 ℃預變性5 min;94 ℃ 50 s,52 ℃ 90 s,72 ℃ 1 min,進行30個循環;72 ℃延伸10 min,擴增產物4 ℃保存。
1. 3 統計分析
根據擴增產物條帶清晰明亮度、彌散程度及特異性等對各組合的電泳圖譜進行直觀評分(1~16分)(何正文等,1998)。其中,條帶清晰、特異性高、無彌散的處理計為16分,條帶淺、彌散嚴重、無特異性的處理計為1分。對2次重復打分的結果進行極差分析,計算每因素在同一水平下的試驗值之和(Ki)、平均值(Xi)及各因素不同水平下的極差(R),并利用SPSS 19.0進行方差分析。
2 結果與分析
2. 1 華山松基因組DNA提取結果
用0.8%瓊脂糖凝膠電泳檢測3種方法提取的華山松基因組DNA,結果如圖1所示。試劑盒法提取的基因組DNA條帶不清晰,彌散明顯;SDS法提取的基因組DNA條帶整齊但顏色較淺,且操作用時較長;改良CTAB法提取的基因組DNA條帶明亮且清晰,無彌散現象,表明改良CTAB法提取效果最佳。
改良CTAB法提取的華山松DNA濃度和純度經超微量紫外分光光度計檢測,結果顯示DNA濃度為92.1~1786.3 ng/μL,OD260/OD280為1.80~2.07,OD260/OD230約2.0,DNA目的條帶清晰完整、無彌散現象。
2. 2 引物篩選結果
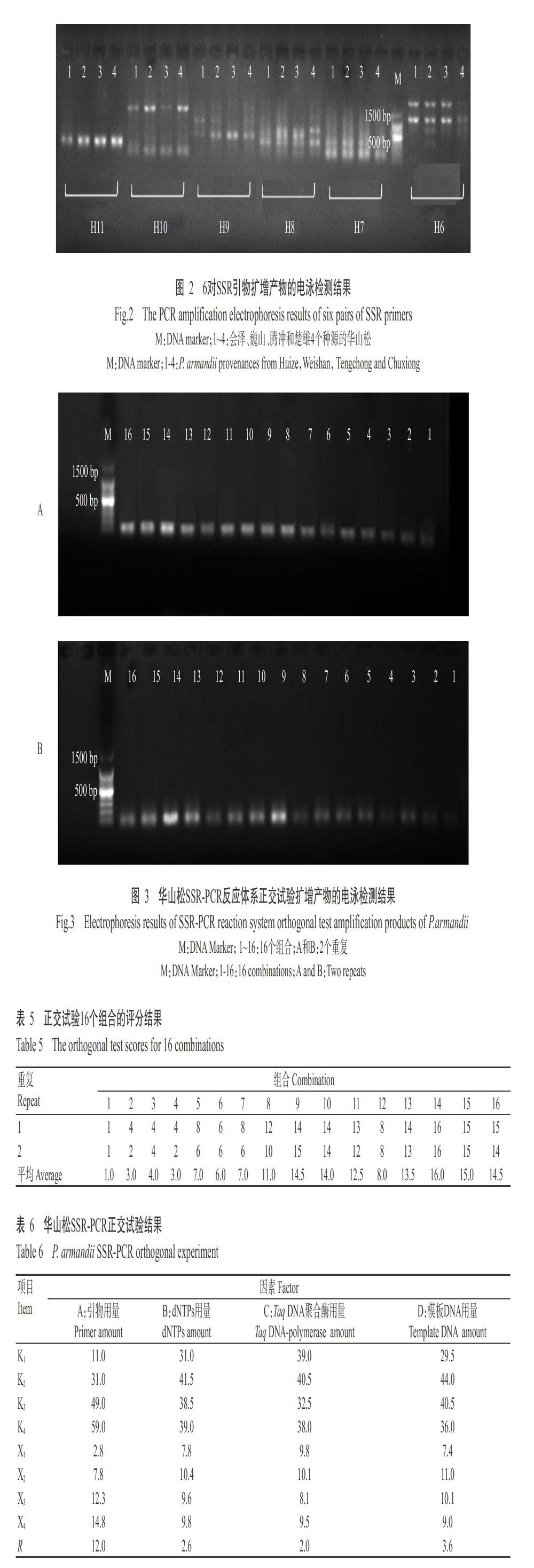
如圖2所示,6對SSR引物均能擴增出明顯的目的條帶,其中,引物H11的擴增效果最佳。因此,利用該引物進行華山松SSR-PCR反應體系優化。
2. 3 正交試驗結果
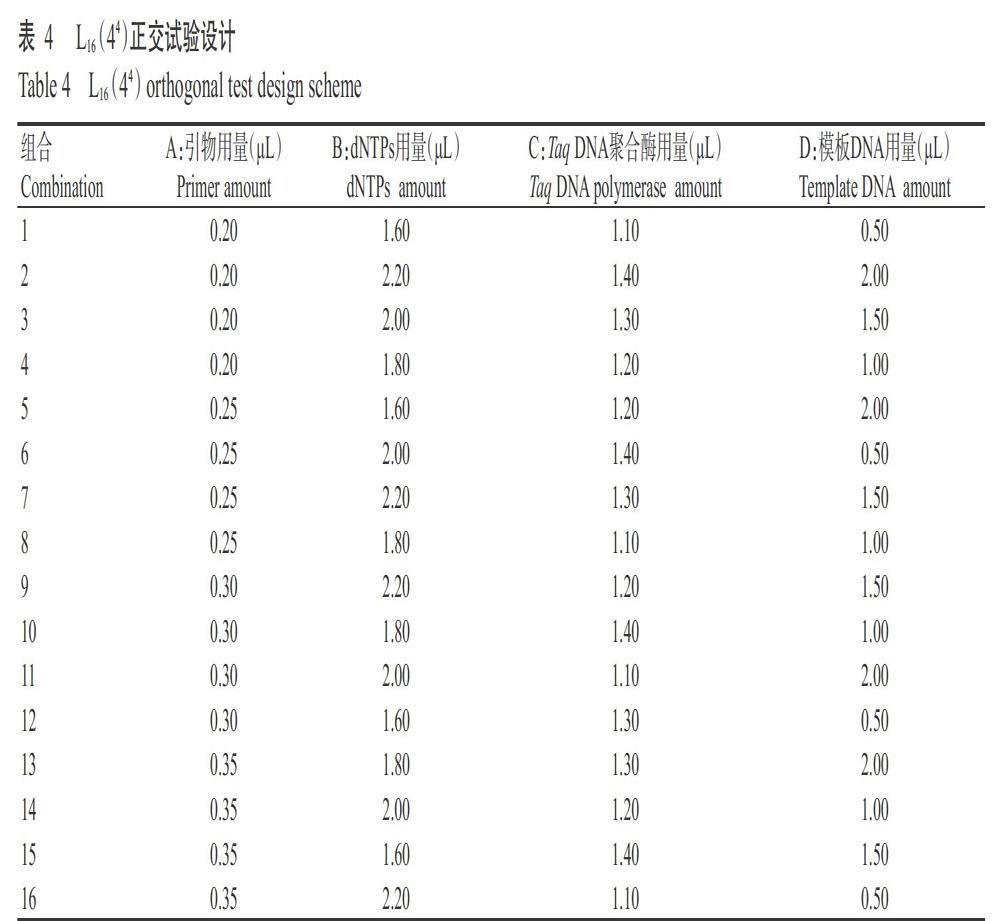
2. 3. 1 評分及極差分析結果 由圖3和表5可知,16個處理中,處理1~8和處理12的條帶較弱,評分較低,尤其是處理1幾乎無條帶,處理9、14、15和16的條帶較亮,評分較高,其中又以處理14的評分最高。R反映各因素對華山松SSR-PCR反應體系的影響程度,R越大,說明該因素對反應體系的影響越顯著。正交試驗結果(表6)顯示,RA>RD>RB>RC,因此,4個因素影響華山松SSR-PCR反應體系擴增效果的主次順序為:A>D>B>C,即引物用量>DNA模板用量>dNTPs用量>Taq DNA聚合酶用量,說明引物用量對華山松SSR-PCR反應體系擴增效果的影響最大,Taq DNA聚合酶用量的影響最小。A因素4個水平的擴增效果排序為:A4>A3>A2>A1,即引物用量為0.35 ?L時擴增效果最佳;B因素4個水平的擴增效果排序為:B2>B4>B3>B1,即dNTPs用量為1.80 ?L時擴增效果最佳;C因素4個水平的擴增效果排序為:C2>C1>C4>C3,即Taq DNA聚合酶用量為1.20 ?L時擴增效果最佳;D因素4個水平的擴增效果排序為:D2>D3>D4>D1,即DNA模板用量為1.00 ?L時擴增效果最佳。綜上所述,最優水平組合為A4B2C2D2,即引物用量0.35 ?L、dNTPs用量1.80 ?L、Taq DNA聚合酶用量1.20 ?L、DNA模板用量1.00 ?L。
2. 3. 2 方差分析結果 由表7可知,引物用量對華山松SSR-PCR反應體系擴增效果的影響達極顯著水平(P<0.01,下同),需要進行多重比較分析;Taq DNA聚合酶用量、DNA模板用量和dNTPs用量對華山松SSR-PCR反應體系的影響不顯著(P>0.05,下同),無需進行多重比較分析。由表7還可知:FA>FD>FC>FB,即影響華山松SSR-PCR反應體系擴增效果的主次順序為:引物用量>DNA模板用量>Taq DNA聚合酶用量>dNTPs用量,與極差分析結果略有差異。根據正交試驗方差評分的估算邊際均值(圖4)可知,A因素4個水平的擴增效果排序為:A4>A3>A2>A1,即引物用量為0.35 ?L時擴增效果最好,與極差分析結果一致;B因素4個水平的擴增效果排序為:B2>B4>B3>B1,即dNTPs用量為1.80 ?L時擴增效果最好,與極差分析結果一致;C因素4個水平的擴增效果排序為:C1>C2>C4>C3,即Taq DNA聚合酶用量為1.10 ?L時擴增效果最佳,與極差分析結果存在差異;D因素4個水平的擴增效果排序為:D3>D2>D4>D1,即模板DNA用量為1.5 ?L時擴增效果最好,與極差分析結果存在差異。綜上所述,最優水平組合為:A4D2C2B2,即引物用量0.35 ?L、DNA模板用量1.50 ?L、Taq DNA聚合酶用量1.10 ?L、dNTPs用量1.80 ?L。
2. 3. 3 引物用量的多重比較分析結果 對引物用量進行多重比較分析,結果如表8所示。引物用量在水平3與水平4間差異不顯著(P>0.05),二者均極顯著大于水平2和水平1;而水平2極顯著大于水平1。因此,引物用量0.30和0.35 ?L時擴增效果較好。
3 討論
提取高質量的基因組DNA是分子試驗的重要環節。但松屬植物的針葉內含較多次生代謝產物和多糖,多糖會抑制聚合酶、連接酶等分子生物學酶類的活性,因此從中提取質量好、純度高的DNA較困難(謝冬梅等,2014)。謝冬梅等(2014)以華山松針葉為材料,分別采用改良CTAB法、Zigenhagen法等4種方法提取華山松基因組DNA,結果發現改良CTAB法提取的基因組DNA穩定性最好,純度最高。本研究分別采用試劑盒法、SDS法和改良CTAB法3種方法提取華山松基因組DNA,結果發現,改良CTAB法提取華山松DNA的效果最佳。與謝冬梅等(2014)的研究結果基本一致。究其原因是改良CTAB法可有效地去除樣品中含有的多糖、色素、黃酮等雜質,因此也可用于提取其他松科植物的DNA。
本研究采用L16(44)正交試驗設計對影響華山松SSR-PCR體系擴增效果的4個因素進行優化,參考何正文等(1998)的直觀評分法對擴增產物的電泳圖譜進行評分,并通過極差分析和方差分析獲得最佳的SSR反應體系。董勝君等(2019)、王開芳等(2019)也使用直觀評分法分別對毛梾和野杏SSR-PCR反應體系進行優化。可見,直觀評分法是一種便捷、通用的方法。本研究的極差分析結果表明,華山松SSR-PCR反應體系中的引物、DNA模板、dNTPs、Taq DNA聚合酶的最佳用量分別為0.35、1.00、1.80和1.20 ?L;但方差分析結果表明,華山松SSR-PCR反應體系中的引物、模板DNA、dNTPs、Taq DNA聚合酶的最佳用量分別為0.35、1.50、1.80和1.10 ?L,且引物用量對華山松SSR-PCR反應體系擴增效果的影響達極顯著水平;多重比較分析結果顯示,引物用量為0.30和0.35 ?L時擴增效果較好。當極差分析和方差分析得出的最佳組合存在極小差異時,應考慮交互作用(辛益軍,2001)。結合本研究正交試驗16個組合的評分結果(表5)可知,組合14的擴增效果最佳,引物的最佳用量為0.35 ?L;DNA模板的最佳用量為1.00 ?L,其原因是DNA模板量過多時,不僅會增加非特異性產物,還會抑制Taq DNA聚合酶的活性;dNTPs和Taq DNA聚合酶的最佳用量為2.00和1.20 ?L,表明dNTPs和Taq DNA聚合酶用量過低或過高時擴增出的條帶不清晰。因此,最終確定的華山松SSR-PCR最佳反應體系(20.00 ?L):10 ?mol/L引物0.35 ?L,50 ng/?L DNA模板1.00 ?L,10 mmol/L dNTPs 2.00 ?L,5 U/?L Taq DNA聚合酶1.20 ?L,10×PCR Buffer(含15 mmol/L Mg2+)2.00 ?L,用ddH2O補足至20.00 ?L。
目前,大量研究人員利用SSR分子標記對華山松進行遺傳分析時均使用Taq PCR Mix(Dong et al.,2016;祝娟,2016)。雖然Taq PCR Mix在操作時相對方便,但不同品牌不同型號的Taq PCR Mix的擴增效果存在明顯差異。本研究分別對華山松SSR-PCR反應體系中的dNTPs、Taq DNA聚合酶、DNA模板和引物的用量進行具體分析及篩選,能較精準地確定各因素對PCR擴增的影響大小,進而獲得最佳的反應體系,促使實驗結果更加穩定可靠。
4 結論
華山松SSR-PCR最佳反應體系(20.00 ?L)為:10 ?mol/L引物0.35 ?L,50 ng/?L DNA模板1.00 ?L,10 mmol/L dNTPs 2.00 ?L,5 U/?L Taq DNA聚合酶1.20 ?L,10×PCR Buffer(含Mg2+ 15 mmol/L)2.00 ?L,用ddH2O補足至20.00 ?L。該體系可應用于華山松種質資源評價及分子標記輔助育種等研究。
參考文獻:
蔡年輝,呂學輝,賀斌,陳詩,李根前. 2013. 云南松SSR-PCR反應體系的優化研究[J]. 西部林業科學,42(6):57-61. [Cai N H,Lü X H,He B,Chen S,Li G Q. 2013. Study on optimization of SSR reaction system of Pinus yunnanensis[J]. Journal of West China Forestry Science,42(6):57-61.]
董勝君,夏澤臻,盧彩云,劉明國,吳月亮,陳建華. 2019. 野杏SSR-PCR反應體系優化[J]. 經濟林研究,37(1):25-31. [Dong S J,Xia Z Z,Lu C Y,Liu M G,Wu Y L,Chen J H. 2019. Optimization of SSR-PCR reaction system for Armeniaca vulgaris Lam. var. ansu[J]. Non-wood Forest Research,37(1):25-31.]
馮飛,汪磊,傅春玲,譚美蓮,汪魏,嚴興初,王力軍. 2018. 基于SSR標記的向日葵DNA指紋圖譜構建[J]. 中國油料作物學報,40(4):508-515. [Feng F,Wang L,Fu C L,Tan M L,Wang W,Yan X C,Wang L J. 2018. Establishment of DNA fingerprint for sunflower by SSR markers[J]. Chinese Journal of Oil Crop Sciences,40(4):508-515.]
馮富娟,趙丹,孫曉艷,韓士杰,許小妍. 2010. 紅松SSR-PCR反應體系的建立與優化[J]. 經濟林研究,28(1):35-40. [Feng F J,Zhao D,Sun X Y,Han S J,Xu X Y. 2010. Establishment and optimization of the SSR-PCR reaction system in Pinus koraiensis Sieb. et Zucc[J]. Non-wood Forest Research,28(1):35-40.]
何衛龍,楊立恒,王曉鋒,林能慶,季孔庶. 2013. 馬尾松胚乳DNA高效提取及SSR-PCR體系優化[J]. 林業科技開發,27(1):15-18. [He W L,Yang L H,Wang X F,Lin N Q,Ji K S. 2013. Efficient DNA extraction from endosperm of Pinus massoniana and SSR-PCR system optimization[J]. Journal of Forestry Engineering,27(1):15-18.]
何正文,劉運生,陳立華,曹美鴻. 1998. 正交設計直觀分析法優化PCR條件[J]. 湖南醫科大學學報,23(4):403. [He Z W,Liu Y S,Chen L H,Cao M H. 1998. Orthogonal design-direct analysis for PCR optimization[J]. Bulletin of Hunan Medical University,23(4):403.]
李春喜,邵云,姜麗娜. 2008. 生物統計學[M]. 第4版. 北京:科學出版社. [Li C X,Shao Y,Jiang L N. 2008. Biostatistics[M]. The 4th Edition. Beijing:Science Press.]
龐新華,檀小輝,韋麗君,梁芳,張繼,呂平,程琴,黃秋偉,周全光. 2019. 35份甘蔗種質的遺傳多樣性分析及DNA指紋圖譜構建[J]. 南方農業學報,50(6):1157-1164. [Pang X H,Tan X H,Wei L J,Liang F,Zhang J,Lü P,Cheng Q,Huang Q W,Zhou Q G. 2019. Genetic diversity analysis for 35 sugarcane germplasm resources and construction of DNA fingerprint[J]. Journal of Southern Agriculture,50(6):1157-1164.]
邱芬,雷瀚,陳杰,楊生超,白斌,劉成,葉鵬,高麗云,辛培堯. 2018. 云貴地區金鐵鎖EST-SSR遺傳多樣性分析[J]. 中草藥,49(16):3895-3906. [Qiu F,Lei H,Chen J,Yang S C,Bai B,Liu C,Ye P,Gao L Y,Xin P Y. 2018. EST-SSR analysis of genetic diversity of Psammosilene tunicoides in Yunnan-Guizhou provincial region[J]. Chinese Traditional and Herbal Drugs,49(16):3895-3906.]
邵俊培,李志輝,楊模華,黃麗群,洪永輝,黃以法. 2012. 馬尾松EST-SSR PCR體系的優化[J]. 中南林業科技大學學報,32(4):159-163. [Shao J P,Li Z H,Yang M H,Huang L Q,Hong Y H,Huang Y F. 2012. Optimization of PCR system in EST-SSR analysis of Pinus massonia-na Lamb[J]. Journal of Central South University of Fo-restry & Technology,32(4):159-163.]
孫海燕,李桐森,周長富. 2007,華山松高結實無性系子代苗期測定與家系選擇[J]. 西南林學院學報(自然科學版),27(1):37-40. [Sun H Y,Li T S,Zhou C F. 2007. Sapling test of filial generation and family selection of high seed yielding clones of Pinus armandii[J]. Journal of Southwest Forestry University(Natural Sciences),27(1):37-40.]
孫文婷,于大德,董明亮,趙健,王小平,張鴻景,張金鳳. 2016. 華北落葉松種子園控制授粉子代遺傳多樣性分析[J]. 西北植物學報,36(8):1662-1670. [Sun W T,Yu D D,Dong M L,Zhao J,Wang X P,Zhang H J,Zhang J F. 2016. Genetic diversity of control-pollinated progenies in seed orchard of Larix gmelinii var. principis-rupprechtii[J]. Acta Botanica Boreali-Occidentalia Sinica,36(8):1662-1670.]
王大瑋,保云瑩,唐紅燕,段安安,蔡年輝,許玉蘭,周軍,李思廣. 2018. 思茅松SSR-PCR反應體系優化研究[J]. 西南林業大學學報(自然科學),38(5):34-37. [Wang D W,Bao Y Y,Tang H Y,Duan A A,Cai N H,Xu Y L,Zhou J,Li S G. 2018. Optimization of SSR-PCR reaction system in Pinus kesiya var. langbianensis[J]. Journal of Southwest Forestry University(Natural Sciences),38(5):34-37.]
王開芳,燕麗萍,任飛,趙登超,孫超,劉翠蘭,吳德軍. 2019. 毛梾SSR-PCR反應體系優化及引物篩選[J]. 分子植物育種,17(23):7834-7839. [Wang K F,Yan L P,Ren F,Zhao D C,Sun C,Liu C L,Wu D J. 2019. Optimization and primers screening of SSR-PCR reaction system for Cornus walteri[J]. Molecular Plant Breeding,17(23):7834-7839.]
謝冬梅,陳國明,何德,李翠新. 2014. 華山松針葉基因組DNA不同提取方法比較[J]. 廣西林業科學,43(4):378-384. [Xie D M,Chen G M,He D,Li C X. 2014. Comparison on the different genomic DNA extraction methods of leaves in Pinus armandi[J]. Guangxi Forestry Science,43(4):378-384.]
辛培堯,周軍,段安安,孫正海,何承忠. 2010. 我國華山松遺傳改良研究進展[J]. 北方園藝,(19):210-214. [Xin P Y,Zhou J,Duan A A,Sun Z H,He C Z. 2010. Research advance of genetic improvement on Pinus armandii in China[J]. Northern Horticulture,(19):210-214.]
辛益軍. 2001. 方差分析與試驗設計[M]. 北京:中國財政經濟出版社. [Xin Y J. 2001. Variance analysis and test design[M]. Beijing:China Financial Economics Press.]
張蝶,郭小丹,鄧亞婷,徐剛標. 2016. 火炬松SSR-PCR反應體系的建立及引物篩選[J]. 廣西林業科學,45(3):253-258. [Zhang D,Guo X D,Deng Y T,Xu G B. 2016. Establishment and primer screening of the SSR-PCR reaction system for Pinus taeda[J]. Guangxi Forestry Scien-ce,45(3):253-258.]
張冬梅,楊婭,沈熙環,茹廣欣. 2017. 油松SSR-PCR引物篩選及反應體系的建立[J]. 北京林業大學學報,(2):13-17. [Zhang D M,Yang Y,Shen X H,Ru G X. 2017. Selection of primers and establishment of SSR-PCR reaction system on Pinus tabulaeformis Carr[J]. Journal of Beijing Forestry University,(2):13-17.]
張瑞麗,許玉蘭,王大瑋,呂學輝,何承忠,段安安. 2012. 云南松SSR-PCR反應體系的建立與優化[J]. 生物技術通報,(4):93-97. [Zhang R L,Xu Y L,Wang D W,Lü X H,He C Z,Duan A A. 2012. Establishment and optimization of the SSR reaction system for Pinus yunnanensis using orthogonal design[J]. Biotechnology Bulletin,(4):93-97.]
祝娟. 2016. 華山松及其近緣種的群體遺傳學和物種形成研究[D]. 西安:西北大學. [Zhu J. 2016. Population gene-tics and speciation study of Pinus armandii and its three affinities[D]. Xian:Northwest University.]
Dong W L,Wang R N,Yan X H,Niu C,Gong L L,Li H Z. 2016.Characterization of polymorphic microsatellite mar-kers in Pinus armandii(Pinaceae),an endemic conifer species to China[J]. Application in Plant Science,4(10):1-5.
(責任編輯 陳 燕)

