鰲肢動物Wnt基因家族的全基因組與進化分析
梁園,周雁,2*
鰲肢動物基因家族的全基因組與進化分析
梁園1,周雁1,2*
1. 復旦大學生命科學學院遺傳工程國家重點實驗室, 上海 200438 2. 中國國家人類基因組中心上海市疾病與健康基因組學重點實驗室, 上海 201203
Wnt蛋白是由基因家族編碼的一類富含半胱氨酸的分泌性蛋白,在多種生物過程中扮演了至關重要的角色,如胚胎發育過程中細胞形態的分化,組織的再生,體細胞穩態維持以及免疫應答的調節。基于其功能的多樣性和重要性,基因家族在多種后生動物中已經被廣泛研究。但是,在節肢動物門中的螯肢動物中,仍然缺乏關于基因及蛋白的系統研究。在本研究中,從8種已有基因組或轉錄組資源的螯肢動物對基因家族進行了生物信息學鑒定和分析,共鑒定出87個基因家族成員。對蛋白序列結構的分析結果顯示螯肢動物的Wnt蛋白包含高度保守的Wnt結構域、信號肽、跨膜區域或低復雜度區域;對基因在基因組上的位置分析表明螯肢動物的Wnt基因在基因組上的分布情況主要有兩種模式,包括基因簇和基因簇,此外還有一些在基因的方向和數量上有差異的物種特異性的分布;系統發育樹的構建結果顯示螯肢動物中的,和亞家族有擴增現象,而和亞家族發生了丟失。本研究作為螯肢動物中第一個基因家族的全基因組研究,有望豐富螯肢動物基因組學的研究內容,為基因家族在節肢動物乃至后生動物中的和進化和功能的進一步研究提供有力的基礎。
螯肢動物;基因家族; 全基因組; 進化
基因編碼了一類分泌性的糖蛋白配體,長度通常在350到400個氨基酸之間,以具有23~24個保守的半胱氨酸為特征[1-4]。分子激發的信號通路參與了多種生物過程的調節,在生物體的發育過程中起著至關重要的作用,包括細胞形態的分化、體細胞形態和功能穩態的維持、胚軸形成,組織的再生和免疫應答調節[5,6]。
基因家族的起源非常古老,且具有多樣性。利用已有的基因組信息對基因家族的系統發育分析結果顯示,在刺胞動物和兩側對稱動物的共同祖先中可能存在13個亞家族[7,8]。其中,在腔腸動物門的海葵()中發現了12個Wnt亞家族[9]。此外,在哺乳動物中還發現了基因的擴增現象,例如在人類的基因組中共鑒定出了19個基因,包括了12個亞家族[10]。然而,在節肢動物中,基因的數量存在著物種間的差異。在昆蟲類的果蠅和蜜蜂的基因組中分別發現了7個基因亞家族[11],然而在甲殼綱動物蚤狀溞()中則鑒定出了12個基因亞家族[12]。另外,根據基因在基因組上分布的位置關系,研究者發現基因在基因組上存在一定程度的連鎖分布現象,形成了具有共線性的基因簇。在刺胞動物和兩側對稱動物的共同祖先中可能有2個具有共線性的基因簇,其中一個基因簇由,-1,-6和-10連鎖分布形成,另一個由和組成的另一個基因簇,但目前尚不清楚在節肢動物中是否仍然存在這一基因簇[7,11,13]。
迄今為止,在節肢動物門的螯肢動物中,仍然缺乏關于基因的全基因組的分析研究。不過,近期發表的拼接質量較高的螯肢動物的基因組數據為全面研究螯肢動物中的基因的特征提供了資源。在本研究中,我們采用生物信息學方法對螯肢動物的基因序列進行了鑒定和分析,并進一步比較了其數量、結構、基因組分布和系統發育關系,以期為基因家族在節肢動物乃至后生動物中的和進化過程和功能的進一步研究提供有力的基礎。
1 材料與方法
1.1 螯肢動物基因組序列獲取
我們收集了公開數據庫中的7種螯肢動物的基因組和1種的轉錄組,包括蝎子類(),蜘蛛類(),蜱螨類(,,),鱟類(,)和海蜘蛛類(sp.)。所收集的7種螯肢動物的基因組都拼接到了scaffold或染色體水平。海蜘蛛sp.的原始RNA測序數據從NCBI SRA數據庫下載得到,檢索號為SRR8745912。
1.2 螯肢動物Wnt基因家族的核酸序列和氨基酸序列的獲取
利用NCBI數據庫中代表性的節肢動物的基因的氨基酸序列為搜索序列,來搜索螯肢動物基因組和轉錄組中的的基因家族成員[11,14,15],首先使用BlastP和TBlastN算法[16]搜索螯肢動物預測基因的蛋白序列和基因組序列,e值閾值選擇10-5。然后將獲得的序列與Pfam數據庫進行比較,檢測是否存在Wnt保守結構域。接下來使用PF00110這一Wnt保守結構域的隱馬爾可夫文件,利用HMMER程序包[17]的HMMsearch程序在螯肢動物的預測基因中搜索是否存在其它基因。第三,使用ClustalW[18]對每個物種已搜索到的基因進行聚類,使用HMMbuild程序構建物種特異的Wnt隱馬爾可夫文件,然后再一次用HMMsearch對螯肢動物的預測基因進行搜索,以獲得物種特異的基因家族候選序列。鑒定得到的Wnt蛋白序列在SMART中進行比對,以鑒定信號肽和其它保守結構。鑒定出的Wnt蛋白結構域使用IBS 1.0.3軟件繪制[19]。
1.3 Wnt蛋白多序列比對和系統發育分析
選擇鑒定得到的2種鱟、1種蝎子、1種蜘蛛、1種螨蟲、1種蜱蟲、1種海蜘蛛,以及已知的1種蜜蜂、1種甲蟲、1種蚜蟲、1種蜈蚣、1種水蚤和1種家鼠的基因用于進行系統發育分析。首先用Clustal W[18]的BLOSUM打分矩陣進行多序列比對,然后使用PhyML 3.0[20]采用最大似然法構建系統發育樹,使用SH-aLRT方法對分支的支持度進行評估。
2 結果與分析
2.1 螯肢動物Wnt基因家族的鑒定
我們在8種螯肢動物中共鑒定得到87個基因家族成員。其中,在中華鱟和美洲鱟中分別發現了15個和18個基因,在蝎子中發現了13個基因,在蜘蛛中發現了12個基因,在二斑葉螨、肩突硬蜱和天鵝絨螨蟲中分別鑒定到了8、7、6個基因,在海蜘蛛中發現了8個基因。每個基因都在之后的系統發育分析中進一步進行了亞家族分類。在蝎子,蜘蛛和海蜘蛛中,鑒定到的基因的氨基酸序列均由300至400個氨基酸構成。在二斑葉螨、肩突硬蜱、和天鵝絨螨蟲中,大多數鑒定得到的基因的氨基酸序列長度在300至400之間,除了在每個物種中分別鑒定出了各2個相對較短的Wnt氨基酸序列以及一個較長的Wnt16序列。在中華鱟和美洲鱟的基因組中,Wnt氨基酸序列的長度出現了更高的變異度,包含了兩條較長的TtWnt1和TtWnt5c以及若干較短的Wnt蛋白序列。
2.2 螯肢動物的Wnt基因氨基酸序列的保守結構域分析
進一步對螯肢動物的基因的氨基酸序列特征和結構域進行分析,結果顯示,在包括鱟、蝎子、蜘蛛和海蜘蛛在內的物種中的Wnt氨基酸序列都含有高度保守的Wnt結構域(見圖1a),位于序列的C末端附近,在這些Wnt氨基酸序列的N末端還發現了多種結構序列,包括長度為20~30個氨基酸的信號肽序列、跨膜區序列或低復雜度區域。然而在3個蜱螨類物種中鑒定到的一部分基因的氨基酸序列的N末端則缺少了這些結構域(見圖1b)。此外,我們對在蝎子、蜘蛛和海蜘蛛中鑒定到的長度在300至400 aa之間的基因的氨基酸序列做了多序列比對,結果顯示這些序列中存在21個保守的半胱氨酸殘基,呈分散分布于序列當中,這與在其它后生生物中鑒定到的基因家族的氨基酸序列的特征是一致的,這些半胱氨酸殘基可能有助于這些Wnt蛋白質的正確折疊。

圖 1 螯肢動物Wnt基因結構
2.3 螯肢動物的Wnt基因在基因組上的連鎖分布
為了進一步探索螯肢動物基因的進化現象,我們比較了其中6個物種的基因在基因組上的分布(見圖2)。結果發現除了美洲鱟以外,其它螯肢動物都保留了進化上保守的基因簇中的一部分,各自伴隨著譜系特異性重排或丟失現象。中華鱟基因組中的和基因位于其3號染色體上,蝎子和蜘蛛基因組中的和基因分別位于各一條scaffold上。此外,在蝎子的另一條scaffold上還發現了的連鎖分布,在肩突硬蜱的基因組中的一條scaffold上發現了相鄰的基因簇。有趣的是,我們在中華鱟、美洲鱟、蝎子和蜘蛛的基因組中還發現了和基因簇,分別位于中華鱟的2條染色體上和美洲鱟的3條scaffolds上。除此之外,我們還發現了除了這兩個基因簇以外的新的基因簇。在美洲鱟基因組中,有一串相鄰分布的基因簇,位于1條scaffold上。在蝎子中,發現了相鄰分布的的和基因簇。在蜘蛛基因組中,和被發現相鄰分布于1條scaffold上。
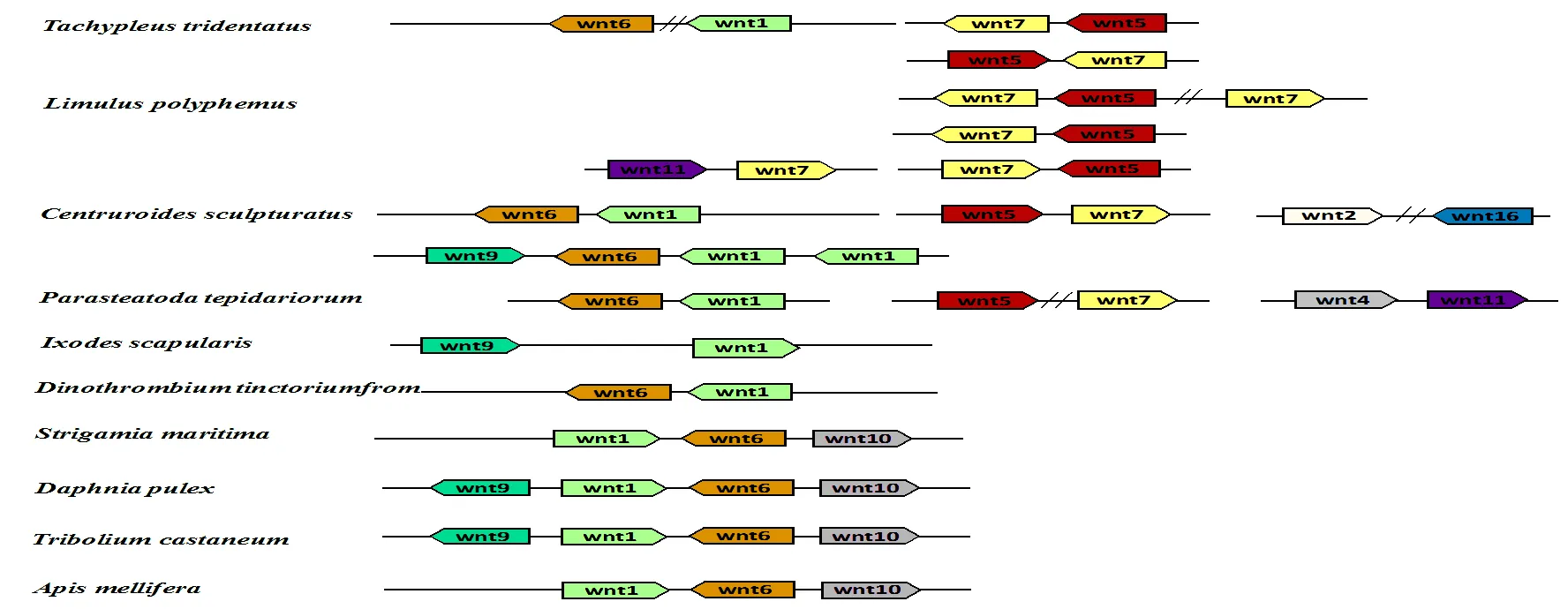
圖 2 螯肢動物Wnt基因的連鎖分布
2.4 螯肢動物Wnt基因的系統發育樹構建及分析
為了進一步闡明螯肢動物的基因的系統發育關系,我們利用7個螯肢動物以及其它節肢動物(包括蜈蚣、水蚤、蜜蜂、蚜蟲、以及甲蟲)的基因的氨基酸序列,采用最大似然法構建了基因家族的系統發生樹,采用小鼠的基因序列作為外類群。系統發育樹顯示,螯肢動物的家族可分為11個不同的亞家族(見圖3)。如在其它節肢動物中所發現的,這些螯肢動物的基因組發生了亞家族的缺失。有趣的是,螯肢動物的基因組中還另外缺失了亞家族。同時,我們僅在蝎子和肩突硬蜱中鑒定到了亞家族序列。與其它類節肢動物基因組大多擁有單拷貝的基因所不同,螯肢動物的,和亞家族基因表現出具有多拷貝的擴增現象(見圖3),這可能暗示了在螯肢動物進化過程的早期出現了基因組的擴增現象。對于每個物種而言,在中華鱟、美洲鱟,蝎子和蜘蛛中分別鑒定出9、10、9、10個亞家族。在海蜘蛛基因組中,鑒定到了8個亞家族。值得注意的是,在螨蟲和蜱蟲中共鑒定出6個和7個亞家族,與其它螯肢動物相比,表現出了和的丟失現象,這可能與其進化速率是有關的。此外,從系統發育樹上,我們發現和亞家族作為姐妹類群聚成一支,且具有分數較高的的支持度,這表明這兩個亞家族可能是由于串聯重復進化而來的。我們還發現,,和亞家族的基因都分為了支持度較高的兩個分支(螯肢動物分支和非螯肢動物分支),這表明這些基因亞家族在螯肢動物和其它節肢動物之間的進化距離更遠。
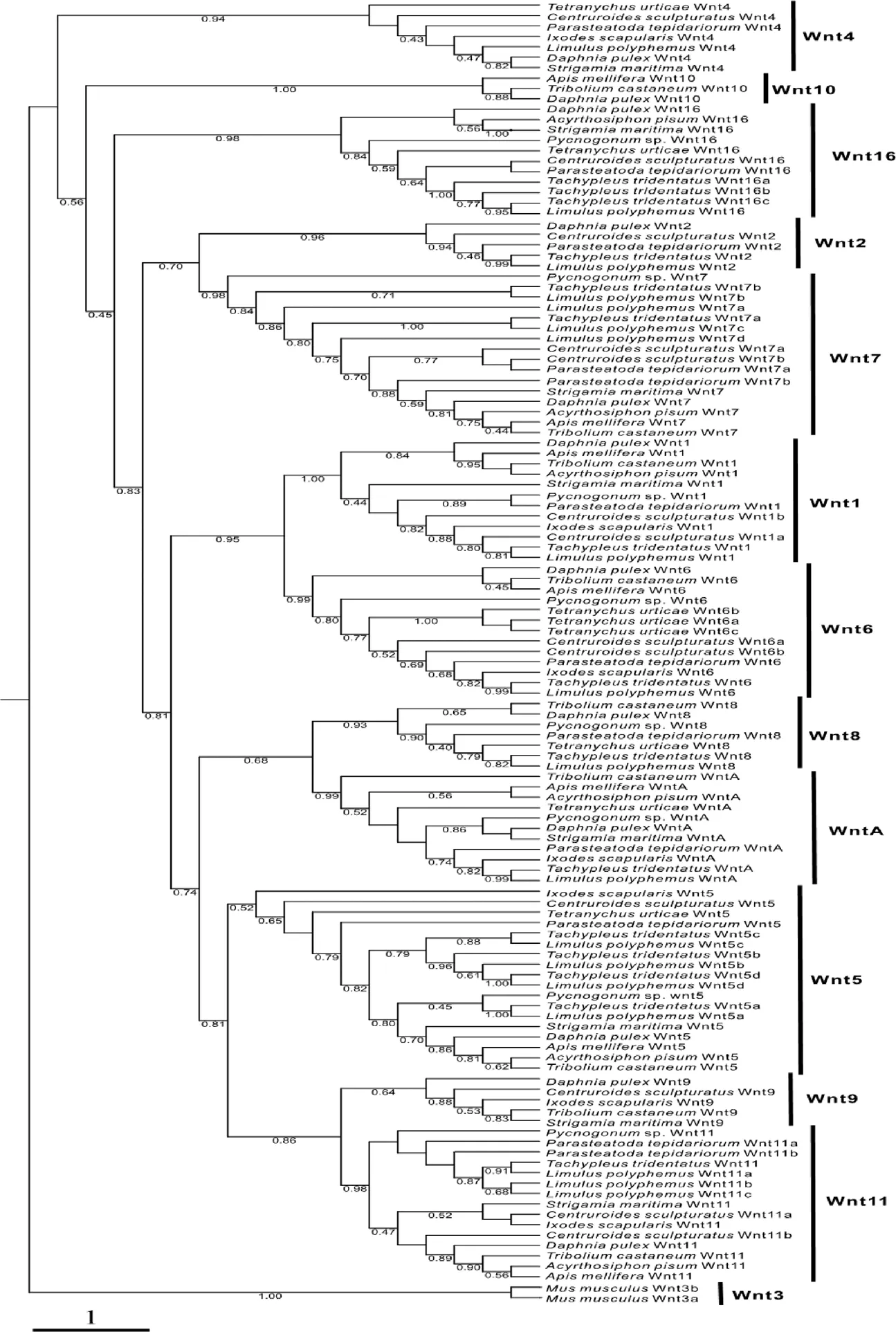
圖 3 Wnt基因家族系統發生樹
3 討 論
在本研究中,我們用生物信息學方法全面鑒定了8種螯肢動物的基因家族,這可以為進一步理解基因在后生動物進化過程中的進化模式提供基礎。盡管在基因組的測序和拼接過程中會有一定的錯誤,但是這8個高深度、高覆蓋度、高拼接水平的基因組和轉錄組資源為我們提供了從全基因組和轉錄組水平鑒定基因的條件。從結構分析結果來看,在蝎子、蜘蛛和海蜘蛛中,大多數Wnt氨基酸序列都包含可識別的信號肽區域,而其它物種中的一些序列則顯然缺乏該基序。考慮到信號肽的生物信息學預測算法仍是有限的,這些序列仍然有包含現有預測軟件無法識別的信號肽的可能性,需要進一步的細胞生物學實驗來確定這些缺乏信號肽的序列是否為非分泌型Wnt蛋白。從基因組的分布情況來看,在蝎子中我們發現了2個位于不同scaffold上的-簇,同時分別在兩種鱟的不同scaffold和染色體上的發現了2個和3個-簇,這可能暗示了在鱟的祖先中曾發生了全基因組復制事件。從系統發育分析結果來看,螯肢動物的基因家族包括了11個亞家族,丟失了和亞家族,這表明螯肢動物的共同祖先可能已缺失了這兩個亞家族。其它亞家族的丟失是在螯肢動物的進化過程中發生的。此外,在螨蟲和蜱蟲中鑒定出了數量較少的6個和7個亞家族,這可能是因為它們在整個進化過程中具有更快的進化速度從而導致了大量的基因丟失。相反,,和亞家族在鱟和蝎子中具有多個基因的拷貝,表現出了較大程度的擴張,而在其它節肢動物(如昆蟲和甲殼類動物)中則表現為單拷貝。對這些螯肢動物中擴張的亞家族的具體功能研究正在進行當中。
4 結 論
在本研究中,我們對螯肢動物中的基因家族做了全基因組的生物信息學鑒定和分析,從螯肢動物的8個物種中共鑒定出了87個基因。我們進一步分析了這些基因的基因結構,發現這些基因包含高度保守的結構域、信號肽、跨膜區域或低復雜度區域。對這些基因在基因組上的分布研究發現,在螯肢動物中的基因簇主要存在兩種連鎖分布模式(--或-W),并且還有一些譜系特異性的基因簇。螯肢動物中的基因家族的系統發育分析結果顯示了、和的擴張以及和的缺失,這些發現為進一步研究螯肢動物中的基因家族的功能提供了基礎。
[1] Cadigan KM, Nusse R. Wnt signaling: a common theme in animal development [J]. Genes & development, 1997,11(24):3286-305
[2] Miller JR. The Wnts [J]. Genome Biology, 2001,3(1):1-15
[3] Clevers H, Nusse R. Wnt/β-Catenin Signaling and Disease [J]. Cell, 2012,149(6):1192-205
[4] Amerongen VR. Alternative Wnt Pathways and Receptors [J]. Cold Spring Harbor Perspectives in Biology, 2012,4(10):715-22
[5] Karner C, Wharton KA, Carroll TJ. Apical–basal polarity, Wnt signaling and vertebrate organogenesis [J]. Seminars in cell & developmental biology, 2006,17(2):214-22
[6] Gordon MD, Dionne MS, Schneider DS,WntD is a feedback inhibitor of Dorsal/NF-κB in Drosophila development and immunity [J]. Nature, 2005,437(7059):746
[7] Cho SJ, Vallès Y, Giani Jr VC,. Evolutionary dynamics of the wnt gene family: a lophotrochozoan perspective [J]. Molecular biology and evolution, 2010,27(7):1645-1658
[8] Bolognesi R, Farzana L, Fischer TD,Multiple Wnt genes are required for segmentation in the short-germ embryo of Tribolium castaneum [J]. Current Biology, 2008,18(20):1624-1629
[9] Kusserow A, Pang K, Sturm C,. Unexpected complexity of the Wnt gene family in a sea anemone [J]. Nature, 2005,433(7022):156
[10] Garriock RJ, Warkman AS, Meadows SM,Census of vertebrate Wnt genes: isolation and developmental expression of Xenopus Wnt2, Wnt3, Wnt9a, Wnt9b, Wnt10a, and Wnt16 [J]. Developmental dynamics: an official publication of the American Association of Anatomists, 2007,236(5):1249-1258
[11] Bolognesi R, Beermann A, Farzana L,. Tribolium Wnts: evidence for a larger repertoire in insects with overlapping expression patterns that suggest multiple redundant functions in embryogenesis [J]. Development genes and evolution, 2008,218(3-4):193-202
[12] Janssen R, Le Gouar M, Pechmann M,. Conservation, loss, and redeployment of Wnt ligands in protostomes: implications for understanding the evolution of segment formation [J]. BMC evolutionary biology, 2010,10(1):374
[13] Sullivan JC, Ryan JF, Mullikin JC,. Conserved and novel Wnt clusters in the basal eumetazoan[J]. Development genes and evolution, 2007,217(3):235-239
[14] Damen WG. Parasegmental organization of the spider embryo implies that the parasegment is an evolutionary conserved entity in arthropod embryogenesis [J]. Development, 2002,129(5):1239-1250
[15] Hayden L, Arthur W. The centipede Strigamia maritima possesses a large complement of Wnt genes with diverse expression patterns [J]. Evolution & development, 2014,16(3):127-138
[16] Altschul SF, Gish W, Miller W,Basic local alignment search tool [J]. Journal of molecular biology, 1990,215(3):403-410
[17] Eddy SR. Accelerated profile HMM searches [J]. PLoS computational biology, 2011,7(10):e1002195
[18] Larkin MA, Blackshields G, Brown N,. Clustal W and Clustal X version 2.0 [J]. bioinformatics, 2007,23(21):2947-2948
[19] Liu W, Xie Y, Ma J,. IBS: an illustrator for the presentation and visualization of biological sequences [J]. Bioinformatics, 2015,31(20):3359-3361
[20] Guindon S, Dufayard JF, Lefort V,. New algorithms and methods to estimate maximum-likelihood phylogenies: assessing the performance of PhyML 3.0 [J]. Systematic biology, 2010,59(3):307-321
Genome-wide and Evolution Analysis ofGene Family in Chelicerata
LIANG Yuan1, ZHOU Yan1,2*
1.200438,2. Shanghai-MOST Key Laboratory of Health and Disease Genomics, Chinese National Human Genome Center at Shanghai, Shanghai 201203, China
The secreted, cysteine-rich Wnt proteins plays an essential role in a diversity of biological processes, encompassing embryo development, the differentiation and maintenance of cell morphology, tissue regeneration and regulation in immune response. On the basis of its functional diversity and importance, the Wnt gene family has been extensively investigated in a variety of metazoans. However, for Chelicerata in arthropods, limited information is available about the systematic identification and analysis of the Wnt gene family. In the present study, the Wnt gene family were characterized in eight Chelicerata, with extensive genomic or transcriptomic resources. Totally 87 Wnt members were identified in Chelicerata. Investigation of functional domains showed that Wnt proteins contain a highly conserved Wnt domain, a signal peptide, a transmembrane region, or a low complexity region. The genomic organization analysis found that there are two major patterns (Wnt9-Wnt6-Wnt1 or Wn5-Wnt7) existing in Chelicerata species, with lineage specific clusters including variation of the orientation and number of the genes. The phylogenetic reconstruction suggested expansion of Wnt5, Wnt7 and Wnt11 and loss of Wnt3 and Wnt10 in Chelicerates. To our best knowledge, this study would provide an insight into the first genome-wide study of the Wnt gene family in Chelicerata, and furthermore, could provide preliminary implications for further understanding of Wnts in Chelicerata.
Chelicerata;gene family; genome-wide; evolution
Q1
A
1000-2324(2020)03-0432-06
10.3969/j.issn.1000-2324.2020.03.008
2019-03-05
2019-05-12
國家重點研發項目(2017YFC0907503);上海市科學技術研究委員會(17DZ2270800);中國科學院(KFJ-STS-QYZD-126)
梁園(1994-),女,研究生,主要從事螯肢動物基因組研究工作. E-mail:yliang17@fudan.edu.cn
Author for correspondence. E-mail:Yan Zhou, zhouy@fudan.edu.cn

