山東主要刺參養殖區幼參腸道抗生素耐藥菌及耐藥基因分布特征*
閆倩倩 李 彬 廖梅杰 王印庚 張 正 于永翔 榮小軍 李德軍
山東主要刺參養殖區幼參腸道抗生素耐藥菌及耐藥基因分布特征*
閆倩倩1,2李 彬2,3廖梅杰2,3①王印庚2,3張 正2,3于永翔2,3榮小軍2,3李德軍4
(1. 上海海洋大學水產與生命學院 上海 201306;2. 中國水產科學研究院黃海水產研究所 青島 266071; 3. 青島海洋科學與技術試點國家實驗室海洋漁業科學與食物產出過程功能實驗室 青島 266071; 4. 青島市西海岸新區漁業技術推廣站 青島 266400)
為解析目前刺參()苗種攜帶耐藥菌及耐藥基因現狀,本研究從煙臺、威海、青島3個刺參主要養殖區的6家養殖場采集了幼參,對其腸道內容物中常用抗生素的耐藥菌數量、占比和種類進行檢測。利用熒光定量PCR技術,對樣本中4類抗生素的7種耐藥基因分布情況進行分析。結果顯示,所檢測的6個采樣點中均有6種抗生素耐藥菌的檢出,從耐藥菌占比來看,耐藥菌占比最高的種類是乙酰甲喹、萘啶酸和四環素耐藥菌,占比分別為0.05%~40.06%、2.16%~39.94%和0.06%~23.15%,氟苯尼考、慶大霉素和鏈霉素耐藥菌的占比均不高,占比范圍為0.01%~4.15%。由所分離到的98株耐藥菌的鑒定結果可以看出,可培養的抗生素耐藥菌分為4門5綱30屬,主要集中在弧菌屬、芽孢桿菌屬和嗜冷桿菌屬,占檢出率的15.30%、13.27%和12.25%。基于屬水平的不同抗生素耐藥菌種類的分布統計結果顯示,各采樣點耐藥菌種類差異較大,而且弧菌屬、芽孢桿菌屬和嗜冷桿菌屬中均存在同一菌屬中有耐多種抗生素的情況。對6個樣本7種抗生素耐藥基因的豐度檢測結果顯示,同一類抗生素的不同耐藥基因的含量差異顯著,除氨基糖苷類抗生素耐藥基因的相對拷貝數比例和鏈霉素以及慶大霉素耐藥菌占比之間存在顯著相關性(<0.05)外,其他抗生素耐藥基因的豐度與耐藥菌占比之間相關性不顯著(>0.05)。研究表明,苗期刺參中存在一定的攜帶耐藥菌和耐藥基因的風險。
刺參;腸道;耐藥菌;耐藥基因;熒光定量PCR
隨著環保風暴和民眾食品質量安全意識的提高,對耐藥菌及耐藥基因的研究逐步成為熱點。目前,國內外耐藥菌和耐藥基因的相關研究主要集中在畜禽類糞便(Mu,2015; 張昊等,2018; Brooks,2014)和環境介質等方面(Lin, 2011; Ji, 2012),針對海水養殖環境下,養殖動物體內的耐藥菌及耐藥基因(Antimicrobial resistant genes, ARGs)方面的研究較少,而在刺參()方面的相關研究還屬于空白。近年來,刺參養殖在我國北方沿海地區發展迅速,并成為沿海地區重要的支柱性產業,為沿海漁業經濟發展提供了重要途徑。但是隨著產業的快速發展,由于養殖過程中的不規范操作以及近岸環境污染等原因,導致疾病頻發,病害成為海水養殖產業可持續發展的主要瓶頸。使用抗生素成為養殖者進行病害防控的重要手段,而抗生素的大量使用不僅嚴重影響養殖生物的質量和安全,也極大地破壞了鄰近海區的生態環境,導致水質環境惡化、藥物殘留、細菌耐藥以及微生態結構失衡(黨宏月等, 2006; 趙小慧等, 2019; Niu, 2016; Zhang, 2012)。據統計,2012年我國抗生素年產量約21萬t,其中,有46.1%的抗生素用于禽畜及水產養殖業;2013年這一比例上升至52%,且由于吸收和代謝不完全等釋放到河流水體中的抗生素達到了5.38萬t (Zhang, 2015)。此外,食品源動物體內殘留的抗生素經食物鏈的富集作用也嚴重威脅人類的健康與安全。2016年刺參抗生素事件曾引發廣泛的社會關注。除了對于耐藥菌株的關注外,由于耐藥基因具有持久性存在和可轉移性的特性(Tamminen, 2011),耐藥基因可通過質粒水平轉移到其他非致病菌或條件致病菌中(張騫月等, 2015),對水產養殖業和人類健康也存在潛在的威脅(羅義等, 2008; 王瑞旋等, 2010),因此,亟需對養殖刺參環境及腸道的耐藥菌現狀進行監測。
刺參的養殖主要分為保苗期和池塘養成期2個時期,保苗期具有生長環境可控的特點,是刺參整個生長周期中的用藥高峰期。目前,刺參苗種培育區主要包括遼寧和山東2個省區,其中,山東省苗種產量占全國苗種供應量的60%以上(中國漁業統計年鑒, 2018)。研究調查發現,目前刺參養殖常用抗生素包括四環素、萘啶酸、氟苯尼考、乙酰甲喹、慶大霉素和鏈霉素。鑒于此,本研究選取山東省3個主要的刺參主養區的6家育苗場,開展6個采樣點刺參苗種腸道中耐藥菌數量、種類及耐藥基因豐度的檢測,以期為解析刺參苗期抗生素耐藥菌及耐藥基因的現狀和防控刺參養殖過程中耐藥細菌的產生提供科學依據。
1 材料與方法
1.1 樣品采集
2018年1~4月,在山東省沿海3個刺參主養區 (青島、煙臺和威海)選取6個育苗場采集10月齡的幼參,采樣位點信息見圖1。每個采樣點各隨機采集 50頭刺參帶回實驗室,參苗規格為(12.35±5.60) g/頭,活體解剖后,獲取其腸道內容物,混勻后立即進行細菌培養,其余樣品于–20℃保存。

圖1 采樣點的地理位置
1.2 總可培養細菌和抗生素耐藥菌的培養及所占比例測定
取1 g腸道內容物樣品加入1.5%的滅菌NaCl溶液制成10 ml懸液,然后10倍梯度稀釋后分別涂布于滅菌的不含抗生素的2216E平板和分別含單種抗生素的2216E平板上,各種抗生素的濃度參考CLSI (2016)標準(Patel, 2016)和畜禽及水產動物耐藥菌篩選的終濃度進行設定(李壹等, 2016; 譚愛萍等,2019),分別為四環素(50 μg/ml)、萘啶酸(50 μg/ml)、乙酰甲喹(50 μg/ml)、氟苯尼考(50 μg/ml)、慶大霉素(32 μg/ml)和鏈霉素(50 μg/ml)。28℃培養24 h后,根據相應平板上的菌落數計算可培養細菌總量及耐藥菌數量,按照以下公式計算相應抗生素耐藥菌所占比例。
耐藥細菌比例(%)=耐藥細菌數/可培養細菌總數× 100%
1.3 抗生素耐藥菌分離純化及鑒定
根據相應抗生素平板上菌落形態(菌落大小、邊緣形狀、顏色和透明度等)對所篩選得到的耐藥菌進行分類和優勢度統計后,用接種環挑取優勢菌的單菌落,在2216E固體培養基進行2次純化培養和保種。用細菌DNA提取試盒(TIANamp Bacteria DNA Kit)提取所分離耐藥菌的細菌DNA,利用細菌16S rDNA通用引物(27F: 5′-AGAGTTTGATC(C/A)TGGCTCAG-3′, 1492R: 5′-GGTTACCTTGTTACGACTT-3′)擴增16S rDNA序列。PCR擴增反應體系為50 μl,擴增程序:94℃預變性5 min;30個循環:94℃變性60 s,55℃復性60 s,72℃延伸80 s;最后,72℃溫育10 min。PCR產物用1%瓊脂糖凝膠電泳檢測合格后送交生工生物工程(上海)股份有限公司進行純化、雙向測序,測序結果在EzBioCloud數據庫(http://www.ezbiocloud.net)中比對,確定相應的菌株分類地位。
1.4 腸道中耐藥基因豐度檢測
取0.5 g腸道內容物,用E.Z.N.A.TMSoil DNA Kit (OMEGA, 美國)提取總DNA,操作步驟參照試劑盒說明書,DNA的完整性和純度用1.0%瓊脂糖凝膠電泳檢測,利用Thermo Nanodrop 2000微量分光光度計(Thermo Scientific, 美國)測定濃度,檢測合格的DNA用于熒光定量PCR檢測。
針對四環素類、喹諾酮類、氯霉素類和氨基糖苷類這4類抗生素選取7種常見耐藥基因進行定量分析,引物信息見表1。利用PCR對目的片段進行擴增、純化后,連接到pMD18-T vector (TaKaRa),之后轉化到大腸桿菌DH5α感受態細胞進行克隆。利用Plasmid Mini Kit I Protocol (OMEGA, 美國)進行重組質粒的提取,以所提取的質粒為模板進行實時熒光定量PCR (qRT-PCR)擴增,構建相應基因擴增的標準曲線,利用Eppendrof實時熒光定量PCR儀自帶的軟件程序計算各基因擴增的t值、擴增效率()及其相關系數(2)。采用qRT-PCR測定6個采樣點樣品中7種耐藥基因和內參基因含量,每個樣品設置3個平行。qRT-PCR反應體系如下:10 μl SYBR Premix ExTMⅡ(2×) (TaKaRa),上、下游引物(20 μmol/L)各0.8 μl,5 ng DNA模板(約5 ng),總體積為20 μl。qRT-PCR反應程序:95℃ 1min;95℃10 s,m30 s,72℃ 30 s, 40個循環。反應結束后,測定熔解曲線。根據標準曲線計算各基因拷貝數的絕對含量,并以各耐藥基因的絕對拷貝數和16S rDNA絕對拷貝數的比值來計算各耐藥基因的相對豐度。
1.5 數據統計方法
利用SPSS 19.0軟件對四環素、萘啶酸、氟苯尼考、慶大霉素和鏈霉素5種抗生素耐藥菌所占比例與7種耐藥基因的相對拷貝數進行線性回歸分析,以< 0.05作為差異顯著水平,確定耐藥菌所占比例和相關耐藥基因相對拷貝數的相關性。
2 結果
2.1 可培養抗生素耐藥菌的數量分析
山東地區的3個刺參主養區6個采樣點刺參苗種腸道中可培養細菌與耐藥菌培養結果見表2。由結果可知,6個采樣點刺參苗種腸道中可培養細菌總數為1.37×105~6.21×106CFU/g。6種抗生素耐藥菌的數量分別為:四環素耐藥菌的數量為(3.00±0.50)×103~(1.15±0.30)×105CFU/g;萘啶酸耐藥菌的數量為(7.77±0.61)× 103~(1.04±0.21)×106CFU/g;氟苯尼考耐藥菌的數量為(2.33±0.59)×102~(5.17± 0.33)×104CFU/g;乙酰甲喹耐藥菌的數量為(1.63±0.29)×103~(4.31±0.23)×105CFU/g;慶大霉素藥菌的數量為(4.00±0.09)×102~(5.90±0.71)× 104CFU/g;鏈霉素耐藥菌的數量為(1.17±0.62)×103~ (1.13±0.20)×105CFU/g。根據相應結果計算耐藥菌在總可培養細菌中的占比(圖2)。由圖2可以看出,同一個采樣點刺參腸道內容物中的不同抗生素耐藥菌占比差異顯著。Y1、Y2、Q2單個采樣點均表現出單一種類抗生素耐藥菌豐度高的特點,相應耐藥菌種類分別為四環素耐藥菌(23.15%)、萘啶酸耐藥菌(37.04%)和萘啶酸耐藥菌(16.76%)。W2采樣點中萘啶酸耐藥菌和乙酰甲喹耐藥菌的占比均很高,分別達到39.94%和40.06%。W1和Q1采樣點雖然有多種抗生素耐藥菌的檢出,但是占比均較低,檢出率最高的萘啶酸耐藥菌的比例也僅為7.32%和6.95%。所有采樣點中氟苯尼考、慶大霉素和鏈霉素耐藥菌的占比均不高,占比范圍為0.01%~4.15%。
2.2 幼參腸道中可培養耐藥菌的分離及鑒定結果
根據含抗生素2216E培養基上形成的菌落形態、顏色、形狀、透明度等表型形態指標,對篩選得到的耐藥菌進行排重,選取目標菌落分離純化,用于菌株保藏及分類地位鑒定,結果見表3。由表3可知,共獲得98株耐藥菌株,其中,四環素、萘啶酸、氟苯尼考、乙酰甲喹、慶大霉素和鏈霉素耐藥菌株分別為18、22、10、14、11和23株。依據16S rDNA序列的EzBioCloud數據庫比對結果,可以判斷耐藥菌分別分布在30個屬。屬水平統計結果顯示,6個采樣點所獲的耐藥菌株分別分布到6、7、13、6、14和8個屬中;所分離到的98株耐藥菌中,檢出率最高的3個屬為弧菌屬()、芽孢桿菌屬()和嗜冷桿菌屬(),分別檢出15株、 13株和12株,占耐藥菌株總數的15.30%、13.27%和12.25%。
鑒于16S rDNA鑒定結果在屬水平上的準確性高,根據相應菌株在屬水平上耐藥菌的分類地位和在抗生素平板上的優勢度,統計屬水平上各采樣點不同抗生素耐藥菌種類的豐度分布(圖3)。各采樣點耐藥菌種類差異較大,其中,Y1采樣點的耐藥菌種類豐度較高的菌為嗜冷桿菌屬和假交替單胞菌屬(),Y2采樣點耐藥菌種類主要為芽孢桿菌屬和嗜冷桿菌屬,W2采樣點耐藥菌種類主要為弧菌屬和動性球菌屬(),Q2采樣點耐藥菌種類主要為弧菌屬,以上4個采樣點都存在同一個屬的不同菌株耐多種抗生素的情況。W1和Q1這 2個采樣點耐藥菌種類較多;在W1采樣點同一種抗生素耐藥菌株的多樣性較高;在Q1采樣點耐氟苯尼考、乙酰甲喹和慶大霉素3種抗生素的耐藥菌株各集中于單一屬的細菌中,而耐四環素、萘啶酸和鏈霉素的耐藥菌株的多樣性較高,分布在4~5個屬中。

表2 不同采樣點可培養細菌及耐藥細菌數量(CFU/g)
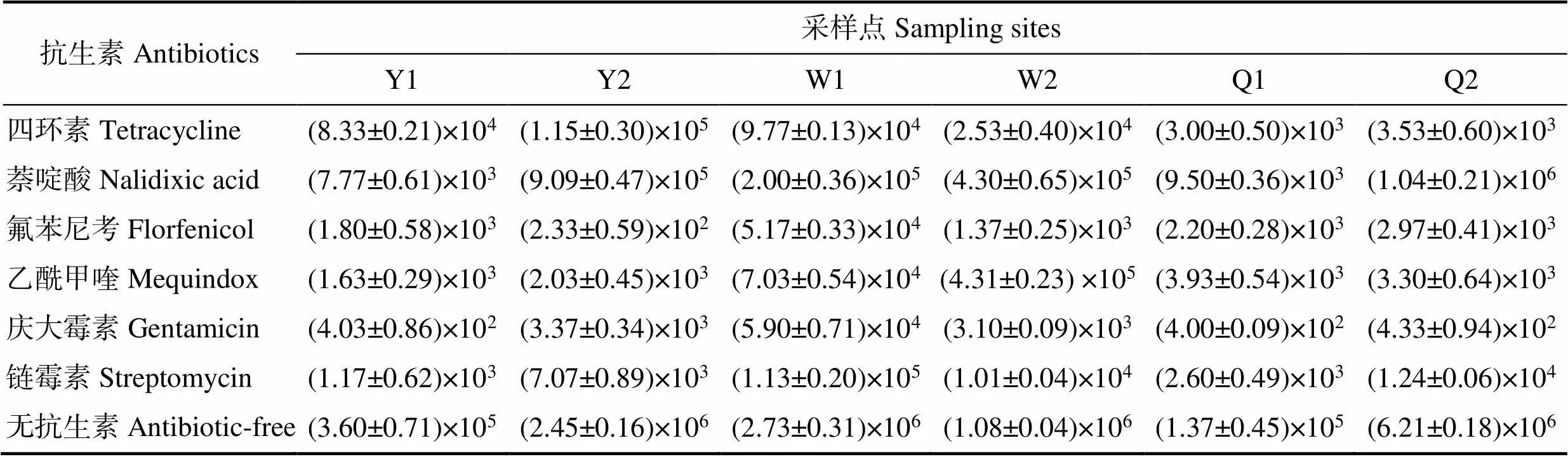
Tab.2 The number of total cultured bacteria and drug-resistant bacteria at different sampling sites (CFU/g)

圖2 各采樣點刺參腸道中不同抗生素耐藥菌占總可培養細菌比例
2.3 各樣品中耐藥基因豐度分析
對刺參腸道中的四環素類、喹諾酮類、氯霉素類和氨基糖苷類共7種抗生素耐藥基因的標準曲線公式見表1,對7個基因的絕對含量和相對于16S rDNA的相對含量進行測算,結果見圖4和圖5。所檢測的2個四環素耐藥基因和拷貝數絕對含量分別為7.63×104~5.24×105和1.95×106~1.91×107copies/g,相對16S rDNA的含量分別為1.69×10–2~1.01×10–1,同一采樣點拷貝數是拷貝數的12~94倍。所檢測的2個喹諾酮類耐藥基因和的拷貝數絕對含量分別為1.71×103~1.51×104和8.57×104~2.21×108copies/g,相對16S rDNA的含量分別為4.09×10–4~7.87×10–3和3.53× 10–2~4.18×101,同一采樣點拷貝數是拷貝數的9~1.02×105倍。所檢測的2個氯霉素類耐藥基因和的拷貝數絕對含量分別為1.29×105~ 8.66×107和7.25× 104~1.64×107copies/g,相對16S rDNA的含量分別為0.17~16.4和0.28~4.56,同一采樣點拷貝數是拷貝數的1.77~78.7倍。氨基糖苷類耐藥基因的拷貝數絕對含量為1.18×104~1.20×106copies/g,相對16S rDNA的含量為1.56×10–2~ 8.73×10–1。
表3 不同采樣點抗生素培養基篩選到的耐藥菌株數及屬水平上的種類數

Tab.3 Number of strains and genera of drug-resistant bacteria screened by antibiotic culture medium at different sampling sites
注:括號外數字為所分離到的耐藥菌株數,括號內的數字為根據16S rDNA鑒定結果所匹配到的屬水平的種類數
Note: Data outside the brackets is the number of drug-resistant strains isolated from the antibiotic plates, data inside the brackets is the genus number classified by 16S rDNA sequences
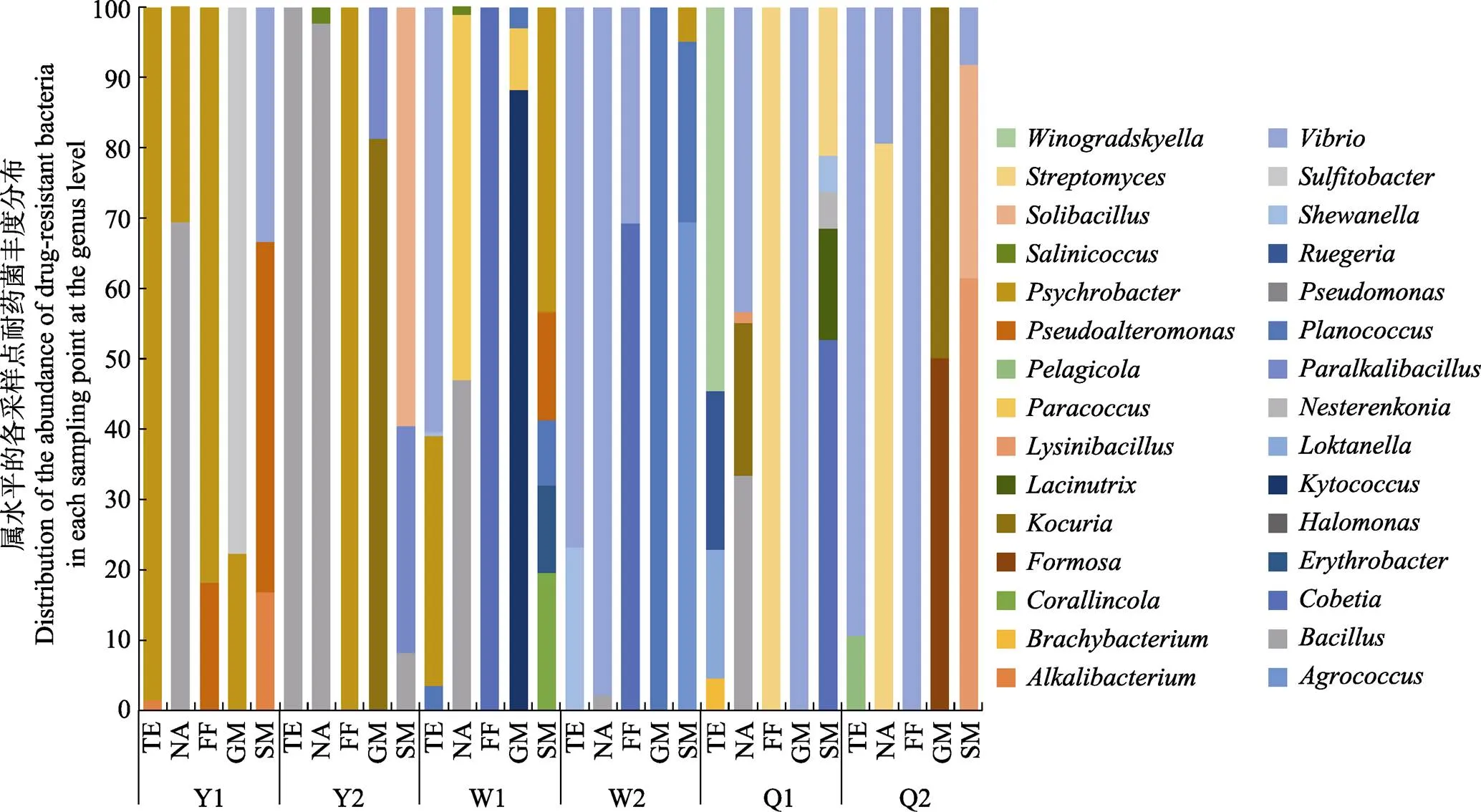
圖3 基于屬水平的各采樣點耐藥菌豐度分布
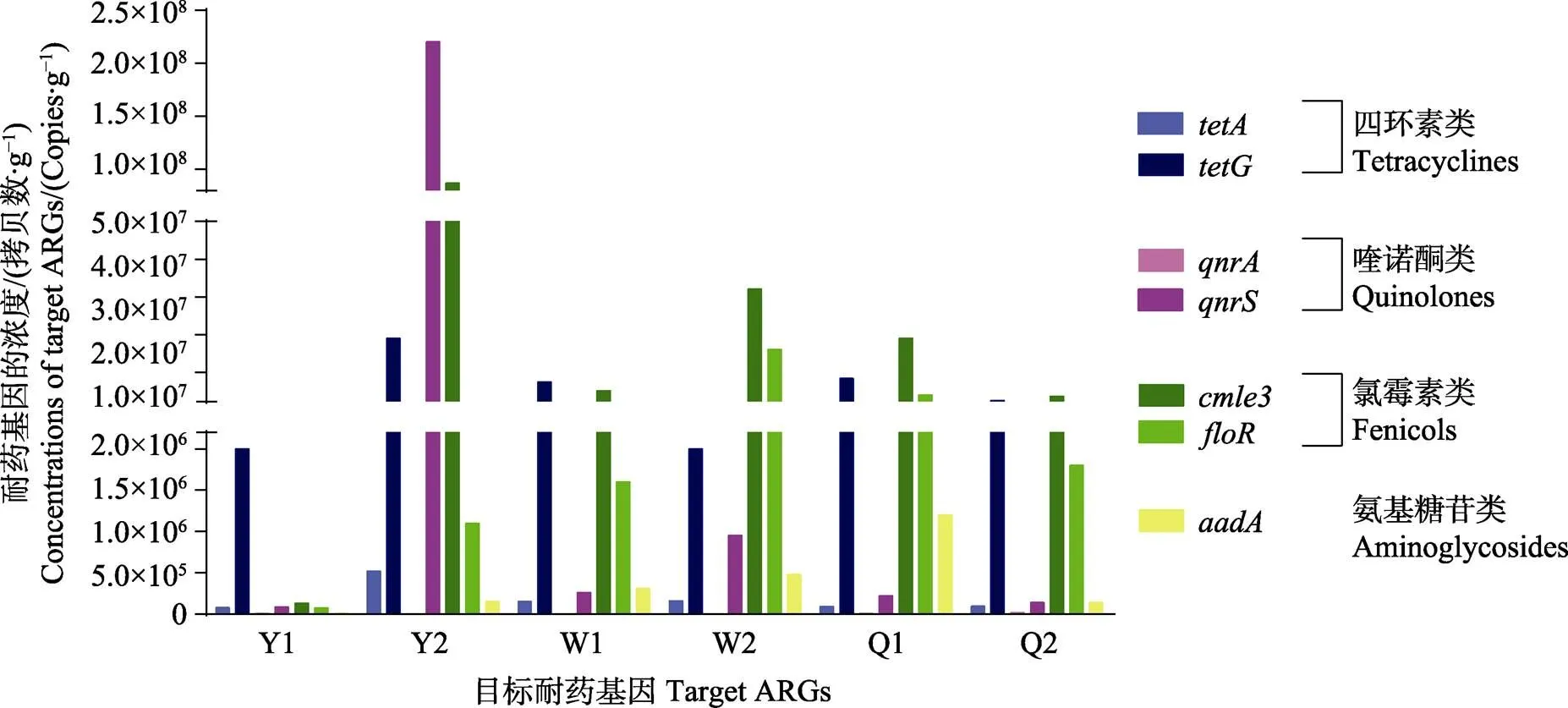
圖4 刺參腸道中目標耐藥基因的濃度
2.4 耐藥菌總數和比例與耐藥基因豐度的相關性分析
針對四環素、萘啶酸、氟苯尼考、慶大霉素和鏈霉素5種藥物作用下耐藥菌所占比例與對應的7種耐藥基因的相對拷貝數(基因絕對拷貝數與16S rDNA基因絕對拷貝數的比值)分別做線性回歸分析(表4)。結果顯示,基因相對拷貝數比例和鏈霉素以及慶大霉素耐藥菌占比之間存在顯著相關性(<0.05)。除此之外,和與四環素耐藥菌占比、和與萘啶酸耐藥菌占比、和與氟苯尼考耐藥菌占比之間相關性不顯著(>0.05)。

圖5 刺參腸道中目標耐藥基因的相對拷貝數
表4 耐藥菌占比與耐藥基因相對含量的相關性分析

Tab.4 Analysis of correlation between the percentage of antibiotic-resistant bacteria and the relative abundance of antibiotic resistance genes
3 討論
3.1 幼參腸道中耐藥細菌的數量特征
水產養殖已經成為我國北方沿海重要產業,養殖過程的疾病頻發導致了抗生素的廣泛使用。養殖動物腸道微生物在抗生素的選擇壓力下,易產生耐藥性,多項研究證實,抗生素耐藥菌已普遍存在養殖環境中(張昊等2018; 王嵐等, 2017; 左志晗等,2018)。本研究對山東地區3個刺參主產區的刺參苗種腸道中6種抗生素耐藥菌進行了調查,不同采樣點均獲得了6種抗生素耐藥菌,說明耐藥現象在刺參苗期養殖中普遍存在。6種抗生素中,乙酰甲喹、萘啶酸和四環素耐藥菌的數量較高,最高值分別達到了4.31×105、1.04×106和1.15×105CFU/g,占可培養細菌總數的40.06%、39.94%和23.15%,而氟苯尼考、慶大霉素和鏈霉素耐藥菌的占比相對較低。李壹等(2016)對山東地區海水養殖環境中的耐藥菌進行了調查,發現在山東海水養殖區存在一定程度的抗生素耐藥性菌,四環素耐藥菌的比例為22.8%~49.0%,而氟苯尼考占比為0.1%~ 7.1%,說明養殖刺參腸道中的耐藥菌與沿海海水環境中的耐藥菌相似性較高,可能是由于養殖生物排泄物排入外海以及外海海水引入養殖系統再次使用的交互作用引起的。從3個地區6個采樣點的耐藥菌占比分布特點還可以看出,同一個地區不同采樣點各抗生素耐藥菌占比存在很大差異,如Y2中萘啶酸耐藥菌豐度顯著高于Y1,W2中萘啶酸和乙酰甲喹耐藥菌的占比顯著高于W1,相對于Q1耐藥菌種類較多,Q2中僅有萘啶酸耐藥菌是優勢耐藥菌,這些情況說明,刺參腸道中耐藥菌的組成和分布除了受外海海水環境影響外,與不同養殖業者各自的養殖管理和用藥習慣存在很大關系。
3.2 幼參腸道中耐藥菌的分類特征
據98株抗生素耐藥菌的16S rDNA鑒定分類結果可以看出,耐藥菌檢出率排序:弧菌屬>芽孢桿菌屬>嗜冷桿菌屬>假交替單胞菌屬。這一結果與Dang等(2007)研究發現的弧菌是海水養殖環境最具優勢的耐藥菌屬結論一致。弧菌和假交替單胞菌均是刺參主要的致病菌(王印庚等, 2006; 逄慧娟等, 2017),刺參養殖生產中大量使用的抗生素主要針對病原性弧菌和假交替單胞菌,藥物的長期使用易導致其在抗生素選擇的壓力下耐藥性的產生。芽孢桿菌屬細菌作為水產養殖中重要的益生菌,被廣泛應用于水環境和養殖動物腸道菌群調控(Pandiyan, 2013),本研究中,芽孢桿菌屬細菌在耐藥菌中占較高的優勢,可能與養殖環境中大量使用芽孢桿菌屬的益生菌,再利用抗生素進行疾病防控,導致芽孢桿菌屬細菌產生耐藥性有關。李研東(2004)利用體外耐藥性誘導實驗誘導出對氟喹諾酮類藥物耐藥的蠟樣芽孢桿菌(),說明芽孢桿菌的耐藥性與抗生素的誘導作用息息相關。因此,在養殖生產中益生菌和抗生素的使用應設置相應的時間差,以降低因抗生素使用帶來的耐藥益生菌產生風險。
3.3 幼參腸道耐藥基因的分布特征
鑒于耐藥基因具有存在的持久性和可轉移性,處于可移動元件上的耐藥基因可轉移到其他非致病菌或條件致病菌中,較多學者通過檢測ARGs評估耐藥風險。在智利三文魚養殖場(Miranda, 2003)、泰國綜合養魚場(Agers?, 2007)、澳大利亞水產養殖場(Hoa, 2008)和日本金槍魚養殖場(Furushita, 2003)均已經檢測到多種ARGs。我國對水產養殖環境、養殖廢水和養殖生物(Dang, 2006、2007; Su, 2017; Gao, 2012; Wen, 2016; Huang, 2017)中的耐藥基因檢測結果表明,水產養殖環境及養殖動物體內也存在多種ARGs。本研究所檢測的7種抗生素耐藥基因在刺參腸道中均被檢出,進一步證明了相應耐藥菌株的存在。但同一個采樣點同一類抗生素的不同耐藥基因的絕對含量卻存在很大差異,如四環素耐藥基因的含量遠高于,喹諾酮類耐藥基因的含量高于,這與李壹等(2016)對海水的檢測結果相似,本研究對氯霉素類 2個耐藥基因和基因表達量檢測也出現了類似的趨勢,即的含量高于,這可能是由于不同基因在環境中的代謝速率差異以及基因在傳遞過程中的質粒或宿主局限造成的。從不同耐藥基因相對含量與相應耐藥菌占比的相關性分析可以看出,除基因相對豐度與鏈霉素和慶大霉素耐藥菌占比極顯著相關(<0.01)外,其他耐藥基因相對豐度與相應抗生素耐藥菌的占比均不存在顯著相關性。李壹等(2016)對海水中耐藥菌占比和相關耐藥基因相對豐度的相關性分析也得出類似結論,即只有氯霉素相關耐藥基因和相對拷貝數比例和氯霉素耐藥菌占比存在顯著相關性,其他所有基因的相對豐度均與相應耐藥菌占比不存在顯著相關性。這可能是由兩方面原因造成的:一是可培養細菌占所有細菌的比例很低,目前培養的耐藥菌不能全部反映出總細菌中耐藥菌的占比;二是因為不同耐藥菌基因在不同細菌中的相對拷貝數和降解速率不同。
綜合以上結果可以看出,目前,苗期刺參中存在一定的攜帶耐藥菌和耐藥基因的風險,其耐藥菌的分布不僅受外海海水環境影響,也與不同養殖業者各自的養殖管理和用藥習慣密切相關;弧菌屬病原菌和芽孢桿菌屬益生菌的耐藥菌檢出比例較高,提示了抗生素的使用已對致病性耐藥菌和益生耐藥菌的產生造成了一定的威脅;耐藥基因豐度與耐藥菌豐度相關性不顯著,因此,后期應同時密切關注二者的分布特征,為評價耐藥風險提供科學依據。此外,細菌耐藥性在“腸道–環境–腸道”的循環中存在傳遞和放大的效應,隨腸道排泄進入環境的耐藥菌和耐藥基因將極大的增加管控的難度和成本,因此,養殖過程中應密切關注腸道耐藥細菌和腸道耐藥基因的檢測,建立抗生素使用的監測和預警機制,為科學選藥提供依據,也為研究和推廣降低耐藥菌數量及耐藥基因豐度的無害化處理過程提供科學參考。
Agers? Y, Petersen A. The tetracycline resistance determinant Tet 39 and the sulphonamide resistance geneare common among resistantsppisolated from integrated fish farms in Thailand. Journal of Antimicrobial Chemotherapy, 2007, 59(1): 23–27
Brooks JP, Adeli A, McLaughlin MR. Microbial ecology, bacterial pathogens, and antibiotic resistant genes in swine manure wastewater as influenced by three swine management systems. Water Research, 2014, 57: 96–103
Dang HY, Song LS, Chen MN,. Concurrence ofandgenes in multiple antibiotic-resistant bacteria isolated from a sea cucumber and sea urchin mariculture farm in China. Microbial Ecology, 2006, 52(4): 634–643
Dang HY, Song LS, Zhang ZN. Harmfulness of bacterial antibiotic resistance from maricultural environments. Studia Marina Sinica, 2006, 47(1): 29–40 [黨宏月, 宋林生, 張志南. 海水養殖環境細菌耐藥性的危害. 海洋科學集刊, 2006, 47(1): 29–40]
Dang H, Zhang X, Song L,. Molecular determination of oxytetracycline-resistant bacteria and their resistance genes from mariculture environments of China. Journal of Applied Microbiology, 2007, 103(6): 2580–2592
Furushita M, Shiba T, Maeda T,. Similarity of tetracycline resistance genes isolated from fish farm bacteria to those from clinical isolates. Applied and Environmental Microbiology, 2003, 69(9): 5336–5342
Gao PP, Mao DQ, Luo Y,. Occurrence of sulfonamide and tetracycline-resistant bacteria and resistance genes in aquaculture environment. Water Research, 2012, 46(7): 2355–2364
Hoa PTP, Nonaka L, Viet PH,. Detection of the sul1, sul2, and sul3 genes in sulfonamide—Resistant bacteria from wastewater and shrimp ponds of north Vietnam. Science of the Total Environment, 2008, 405(3): 337–384
Huang L, Xu YB, Xu JX,. Antibiotic resistance genes (ARGs) in duck and fish production ponds with integrated or non-integrated mode. Chemosphere, 2017, 168: 1107–1114
Ji XL, Shen QL, Liu F,. Antibiotic resistance gene abundances associated with antibiotics and heavy metals in animal manures and agricultural soils adjacent to feedlots in Shanghai, China. Journal of Hazardous Materials, 2012, 235–236: 178–185
Li YD. Study on characteristics of drug susceptibility and mechanisms of quinolones resistance in. Master′s Thesis of Jilin University, 2004 [李研東. 蠟樣芽孢桿菌的藥敏特性及對氟喹諾酮類藥物耐藥機制的初步研究. 吉林大學碩士研究生學位論文, 2004]
Li Y, Qu LY, Zhu PF,. Distribution characteristics of antibiotic resistance bacteria and related resistance genes in mariculture area of Shandong. Marine Environmental Science, 2016, 35(1): 55–62 [李壹, 曲凌云, 朱鵬飛, 等. 山東地區海水養殖區常見抗生素耐藥菌及耐藥基因分布特征. 海洋環境科學, 2016, 35(1): 55–62]
Lin T, Hu LM, Guo ZG,. Sources of polycyclic aromatic hydrocarbons to sediments of the Bohai and Yellow Seas in East Asia. Journal of Geophysical Research, 2011, 116: D23305
Luo Y, Zhou QX. Antibiotic resistance genes (ARGs) as emerging pollutants. Acta Scientiae Circumstantiae, 2008, 28(8): 1499–1505 [羅義, 周啟星. 抗生素抗性基因(ARGs)—一種新型環境污染物. 環境科學學報, 2008, 28(8): 1499– 1505]
Miranda CD, Kehrenberg C, Ulep C,. Diversity of tetracycline resistance genes in bacteria from Chilean salmon farms. Antimicrobial Agents and Chemotherapy, 2003, 47(3): 883–888
Mu QH, Li J, Sun YX,. Occurrence of sulfonamide-, tetracycline-, plasmid-mediated quinolone- and macrolide- resistance genes in livestock feedlots in Northern China. Environmental Science and Pollution Research, 2015, 22(9): 6932–6940
Niu ZG, Zhang K, Zhang Y. Occurrence and distribution of antibiotic resistance genes in the coastal area of the Bohai Bay, China. Marine Pollution Bulletin, 2016, 107(1): 245– 250
Pang HJ, Liao MJ, Li B,. Etiology and treatment of enteritis disease in cultured junviles of. Progress in Fishery Sciences, 2017, 38(3): 188–197 [逄慧娟,廖梅杰, 李彬, 等. 刺參()保苗期“腸炎病”及其治療方法. 漁業科學進展, 2017, 38(3): 188– 197]
Patel JB, Franklin R, George M,. Performance standards for antimicrobial susceptibility testing: Twenty-six informational supplement. Wayne: Clinical and Laboratory Standards Institute, 2016
Pandiyan P, Balaraman D, Thirunavukkarasu R,Probiotics in aquaculture. Drug Invention Today, 2013, 5(1): 55–59
Su HC, Liu S, Hu XJ,. Occurrence and temporal variation of antibiotic resistance genes (ARGs) in shrimp aquaculture: ARGs dissemination from farming source to reared organisms. Science of the Total Environment, 2017, 607– 608: 357–366
Tamminen M, Karkman A, L?hmus A,. Tetracycline resistance genes persist at aquaculture farms in the absence of selection pressure. Environmental Science and Technology, 2011, 45(2): 386–391
Tan AP, Deng YT, Jiang L,. Correlation between PMQR genes/QRDR mutations and quinolones resistance infrom aquatic animals.Journal of China Agricultural University, 2019, 24(4): 102–111 [譚愛萍, 鄧玉婷, 姜蘭, 等. 水產動物源氣單胞菌喹諾酮類耐藥與PMQR基因、QRDR突變相關性分析. 中國農業大學學報, 2019, 24(4): 102–111]
Van TTH, Chin J, Chapman T,. Safety of raw meat and shellfish in Vietnam: An analysis ofisolations for antibiotic resistance and virulence genes. International Journal of Food Microbiology, 2008, 124(3): 217–223
Wang HP, Yan H, Zhao JR,. Quantitative detection of six classes of antibiotic resistance and classⅠintegron genes in aquatic products. Modern Food Science and Technology, 2017, 33(5): 270–276 [王慧平, 閆鶴, 趙俊仁, 等. 水產品中6類抗生素抗性基因和Ⅰ類整合子的定量檢測. 現代食品科技, 2017, 33(5): 270–276]
Wang L, Wang YG, Zhang Z,. Diversity and drug resistance of bacterial pathogens isolated from bacterial ascetic disease in culturedturbot.Progress in Fishery Sciences 2017, 38(4): 17–24 [王嵐, 王印庚, 張正, 等. 養殖大菱鲆()腹水病的病原多樣性及其耐藥性分析. 漁業科學進展, 2017, 38(4): 17–24]
Wang RX, Feng J, Geng YJ,. Studies on the drug resistance of aquatic bacteria. Marine Environmental Science, 2010, 29(5): 770–777 [王瑞旋, 馮娟, 耿玉靜, 等. 水產細菌耐藥性的最新研究概況. 海洋環境科學, 2010, 29(5): 770– 777]
Wang YG, Fang B, Zhang CY,. Etiology of skin ulcer syndrome in cultured juveniles ofand analysis of reservoir of the pathogens. Journal of Fishery Sciences of China, 2006, 13(4): 610–616 [王印庚, 方波, 張春云, 等. 養殖刺參保苗期重大疾病“腐皮綜合征”病原及其感染源分析. 中國水產科學, 2006, 13(4): 610–616]
Wen YP, Pu XY, Zheng W,. High prevalence of plasmid-mediated quinolone resistance and incQ plasmids carryinggene in bacteria from rivers near hospitals and aquaculture in China. PLoS One, 2016, 11(7): pone0159418
Zhang H, Wang PL, Yang QX,. Distribution of multidrug- resistant bacteria and antibiotic-resistant genes in livestock manures. Environmental Science, 2018, 39(1): 460–466 [張昊, 王盼亮, 楊清香, 等. 畜禽糞便中多重耐藥細菌及耐藥基因的分布特征. 環境科學, 2018, 39(1): 460–466]
Zhang RJ, Zhang G, Zheng Q,. Occurrence and risks of antibiotics in the Laizhou Bay, China: Impacts of river discharge. Ecotoxicology and Environmental Safety, 2012, 80(1): 208–215
Zhang QY, Zhao WW, Wu W. Antibiotics resistance gene pollution and its research progress acheived in aquaculture environment. Journal of Agricultural Science and Technology, 2015, 17(6): 125–134 [張騫月, 趙婉婉, 吳偉. 水產養殖環境中抗生素抗性基因污染及其研究進展. 中國農業科技導報, 2015, 17(6): 125–134]
Zhang QQ, Ying GG, Pan CG,. Comprehensive evaluation of antibiotics emission and fate in the river basins of China: Source analysis, multimedia modeling, and linkage to bacterial resistance. Environmental Science and Technology, 2015, 49(11): 6772–6782
Zhao XH, Su J, Fan JF,. Research progress on bacterial resistance in marine environment. Chinese Journal of Antibiotics, 2019, 44(4): 406–412 [趙小慧, 蘇潔, 樊景鳳, 等. 海洋環境中細菌耐藥性研究進展. 中國抗生素雜志, 2019, 44(4): 406–412]
Zuo ZH, Li YH, Shao YC,. Isolation, identification and antibiotic resistance analysis of intestinal strains of aquaculturein surrounding areas of Tianjin. Journal of Fisheries of China, 2018, 42(5): 797–807 [左志晗, 李艷紅, 邵迎春, 等. 天津市周邊地區養殖凡納濱對蝦腸道菌株的分離鑒定及耐藥性分析. 水產學報, 2018, 42(5): 797–807]
Distribution Characteristics of Antibiotic Resistant Bacteria and Antimicrobial Resistant Genes in the Intestine of Cultured Sea Cucumber () Seedlings in Shandong Province
YAN Qianqian1,2, LI Bin2,3, LIAO Meijie2,3①, WANGYingeng2,3, ZHANG Zheng2,3, YU Yongxiang2,3, RONG Xiaojun2,3, LI Dejun4
(1. College of Fisheries and Life Science, Shanghai Ocean University, Shanghai 201306; 2. Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao 266071; 3. Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology(Qingdao), Qingdao 266071; 4. Aquatic Technology Extension Station of Qingdao West Coast New Economic District, Qingdao 266400)
In order to understand the current status of drug-resistant bacteria and drug-resistant genes in the sea cucumber, seedlings were collected from 6 aquaculture farms in Shandong Province. The number, proportion and species of antibiotic-resistant bacteria in the intestinal were tested. Meanwhile, the distribution of seven drug-resistant genes of four classes of antibiotics was analyzed by quantitive real-time PCR (qRT-PCR). The results were listed as follows: All of the six kinds of antibiotic-resistant bacteria are detected at all six sampling sites. According to the proportion of drug-resistant bacteria to total cultured bacteria, the top three highest proportions are mequindox, nalidixic acid and tetracycline-resistant bacteria, with the proportions are 0.05%~40.06%, 2.16%~39.94% and 0.06%~23.15%, respectively. The proportion of florfenicol, gentamicin and streptomycin-resistant bacteria range from 0.01% to 4.15%. The 98 antibiotic-resistant bacteria strains are identified as 4 phyla, 5 classes and 30 genera based on 16S rDNA sequence. The top 3 genera are,andwith the detection rates of 15.30%, 13.27% and 12.25%, respectively. The components of drug-resistant bacteria species varied at different sampling sites, and there are compound antibiotic-resistant strains in,andgenera. The abundances of seven antibiotic resistance genes in six samples show that the contents of different antibiotic resistance genes against the same antibiotic are significantly different. There is significant correlation between the relative copy number ratio of aminoglycoside resistance gene () and the proportion of streptomycin and gentamicin-resistant bacteria. The correlation between the abundance of the other six genes and the proportion of drug-resistant bacteria is not significant. The results indicate that there is a certain risk of drug-resistant bacteria and drug-resistant genes in the seedling stage of sea cumber.
;Intestine; Antibiotic resistant bacteria; Antimicrobial resistant genes; Quantitive real-time PCR
LIAO Meijie, E-mail: liaomj@ysfri.ac.cn
S966.9
A
2095-9869(2020)04-0134-10
10.19663/j.issn2095-9869.20190424001
http://www.yykxjz.cn/
閆倩倩, 李彬, 廖梅杰, 王印庚, 張正, 于永翔, 榮小軍, 李德軍. 山東主要刺參養殖區幼參腸道抗生素耐藥菌及耐藥基因分布特征. 漁業科學進展, 2020, 41(4): 134–143
Yan QQ, Li B, Liao MJ, Wang YG, Zhang Z, Yu YX, Rong XJ, Li DJ. Distribution characteristics of antibiotic resistant bacteria and antimicrobial resistant genes in the intestine of cultured sea cucumber () seedlings in Shandong Province. Progress in Fishery Sciences, 2020, 41(4): 134–143
* 國家重點研發計劃(2018YFD0901603)、中國水產科學研究院中央級公益性科研院所基本科研業務費專項資金(2018GH10)、中國水產科學研究院黃海水產研究所級基本科研業務費(20603022016008)和山東省農業良種工程重大課題(2017LZGC010)共同資助 [This work was supported by National Key R&D Program of China (2018YFD0901603), Central Public-Interest Scientific Institution Basal Research Fund, CAFS (2018GH10), Special Scientific Research Funds for Central Non-Profit Institutes, Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences (20603022016008), and Agriculture Seed Improvement Project of Shandong Province (2017LZGC010)]. 閆倩倩,E-mail: qian_qian_1314@126.com
廖梅杰,副研究員,E-mail: liaomj@ysfri.ac.cn
2019-04-24,
2019-05-03
(編輯 馬璀艷)

