泥東風螺EST-SSR開發及其群體遺傳多樣性分析*
熊 鋼 王曉清 王 佩 陳貞年 周先文,3 康 驪 曾志南
泥東風螺EST-SSR開發及其群體遺傳多樣性分析*
熊 鋼1,2王曉清2①王 佩2陳貞年2周先文2,3康 驪1曾志南4
(1. 湖南生物機電職業技術學院動物科技系 長沙 410127;2. 湖南農業大學動物科技學院 長沙 410128;3. 湘西州水產工作站 吉首 416000;4. 福建省水產研究所 廈門 361013)
本研究采用MISA軟件分析泥東風螺()轉錄組中微衛星信息。結果顯示,從轉錄組中共獲得16324個SSR,共有181種重復基元;泥東風螺轉錄組中不同類型微衛星的重復基元具有不同的分布特征,其中,二核苷酸重復基元中AC/GT(70.58%)重復基元以重復6次出現頻率占優;長度為12~20 bp的SSR占63.95%,長度為21~25 bp的SSR占9.14%,總體的平均長度為18.4 bp。隨機選取其中50條序列設計引物,通過對福建野生泥東風螺群體(WP)DNA樣本進行PCR擴增和分型,結果獲得23個多態性位點,等位基因數目為2~7個不等,期望雜合度(e)為0.190~0.937,觀察雜合度(o)為0.065~0.936,多態性信息含量(PIC)為0.061~0.777,有4個位點顯著偏離哈迪溫伯格平衡(Hardy-Weinberg equilibrium, HWE) (<0.05)。對野生群體和養殖群體(BP)遺傳多樣性分析顯示,野生群體和養殖群體的平均e分別為0.491和0.544,平均o分別為0.477和0.564,平均PIC分別為0.541和0.407。is結果顯示,野生群體和養殖群體分別有13個和9個位點雜合子過剩。群體間遺傳分化指數(st)為0.001~0.655,平均值為0.053 (0.05 泥東風螺;轉錄組;EST-SSR;遺傳多樣性 東風螺(sp)隸屬于軟體動物的腹足綱、新腹足目、蛾螺科、東風螺屬(王如才等, 1988),俗稱花螺、泥螺、南風螺等,分布于熱帶和亞熱帶海域,是我國沿海重要的經濟軟體動物。我國現有方斑東風螺()、泥東風螺()和臺灣東風螺 () 3種(張漢華等, 2004; 陳利雄等, 2004)。泥東風螺分布于我國福建至廣西沿海地區。隨著人類活動對海洋的影響,海洋中野生泥風螺銳減,我國研究人員已開展了泥東風螺的人工選育(葉泉土等, 2015a; 林國清等, 2015)、人工養殖(葉泉土等, 2015b)、人工增殖放流(葉泉土等, 2015b)和群體遺傳多樣性AFLP分析(秦溱等, 2014)的相關研究。 簡單重復序列(Simple sequence repeats, SSR)又稱微衛星DNA、短串聯重復序列,一般以1~6個堿基為核心序列,廣泛存在真核生物的基因組中,具有數量豐富、多態性高、共顯性等特點(Powell, 1996)。Morgante等(1993)將微衛星用于遺傳和物理圖譜的構建、品種鑒定、基因定位、遺傳多樣性、分類和進化及比較基因組等方面的研究。目前,SSR標記主要分為基因組SSR(Genomic SSR, gSSR)和表達序列標簽SSR(Expressed sequence tag SSR, EST-SSR)兩種。EST-SSR反映的是基因的編碼部分,可為功能基因提供更可靠的功能性標記,所以它在物種起源與進化、資源多樣性、遺傳作圖、功能基因的發現與定位和比較基因組學研究等方面都有重要的利用價值。EST-SSR多態性可能與基因功能直接相關,因此,比gSSR標記具有更高通用性(Eujayl, 2002)。新一代測序技術可以對全基因組范圍內的轉錄本進行大規模的高通量測序,并能產生海量的轉錄組數據(Simon, 2009),這為功能基因組SSR標記的開發提供了更豐富和極有價值的可利用資源(Graham, 2010)。我國研究工作者采用新一代測序技術已從曼氏無針烏賊()(管奧等, 2018)、長江刀鱭()(于愛清等, 2018)、櫛江珧()(李東明等, 2017)、凡納濱對蝦()(李東宇等, 2017)、扇貝() (張廣明等, 2018; 倪守勝等, 2018)、黃口荔枝螺()(李威等, 2015)和泥蚶() (史松富等, 2013)等水生動物中成功開發并應用于物種群體遺傳多樣性分析的SSR標記。本課題組利用軟件分析泥東風螺轉錄組數據中微衛星分布特點和規律,利用篩選SSR標記對野生群體和養殖群體的遺傳多樣性進行分析,可為泥東風螺微衛星標記的開發及應用研究奠定基礎。 野生泥東風螺采自福建連江和長樂,共62只。養殖群體來源于福建長樂泥東風螺育種場,共206只。 1.2.1 泥東風螺轉錄組數據來源 泥東風螺轉錄組數據是由腹足和肝胰腺組織的mRNA建立混合池,經Illumina HiSeqTM2000高通量測序平臺獲得的轉錄組數據文庫。 1.2.2 泥東風螺轉錄組SSR的篩選及引物設計 利用軟件MISA(Kanehisa, 2008) (http://pgrc. ipk-gatersleben.de/misa/)對泥東風螺轉錄組中Unigene的cDNA序列數據進行SSR搜索,設置參數:單堿基最少重復10次以上,雙堿基最少重復6次以上,3~6堿基最少重復5次以上;混合重復的2個重復之間的距離不能大于100 bp。隨機挑選EST-SSR序列,參照引物設計原則(張新宇等, 2004),利用軟件Primer premier 5.0在重復序列兩側保守區設計微衛星擴增引物。 1.2.3 DNA提取 取泥東風螺腹足,采用天澤基因柱式動物DNA提取試劑盒提取泥東風螺總DNA。總DNA用1%瓊脂糖凝膠電泳檢測,?20℃保存備用。SSR分析采用20 μl PCR擴增反應體系,擴增條件為:94℃預變性5 min;94℃變性30 s,退火(退火溫度見表3) 30 s,72℃延伸30 s,30個循環;72℃延伸5 min,在4℃條件下保存。擴增產物采用8%變性聚丙烯酰胺凝膠電泳,銀染顯色,人工讀帶后記錄帶型。 1.2.4 EST-SSR分析 用PopGene 1.3.1(Yeh, 1997)軟件計算有效等位基因數(Effective numbers of allele,e)、觀測雜合度(Observed heterozygosity,o)、(Expected heterozygosity,e)、群體間基因流(m)、固定系數(st)和群體近交系數(is)。位點多態性(Polymorphis information content, PCI)采用軟件包計算,Cervus 3.03 (Kalinowsk, 2007)軟件計算哈迪溫伯格平衡(Hardy-Weinberg equilibrium, HWE)。 利用MISA軟件對Unigenes的cDNA序列數據中篩選1 kb以上的Unigenes進行SSR分析,共篩選到16324個SSR符合設置條件(表1),泥東風螺轉錄組中SSR種類豐富,共有181種重復類型;各種類型的出現頻率差異較大,主要集中在單核苷酸重復、二核苷酸重復和三核苷酸重復,分別占SSR總數量的40.17%、29.75%和15.27%,四核苷酸重復和五核苷酸重復數量少,分別占0.76%和0.04%,未發現多于5個核苷酸重復。SSR在整個泥東風螺轉錄組中的出現頻率和發生頻率分別為13.62%和6.86%,平均每350.78 kb出現1個SSR。各種重復基元出現頻率差異大(表2),如:AC/GT重復基元占二核苷酸重復的70.58%,AGG/CCT和ACC/GGT重復基元分別占三核苷酸重復的22.60%和20.50%。從整體分布分析中發現多核苷酸重復序列以重復6次出現頻率占優勢。長度為12~20 bp的SSR占63.95%,長度為21~25 bp的SSR占9.14%,總體的平均長度為18.4 bp。 表1 泥東風螺轉錄組中EST-SSRs重復次數分布 Tab.1 Repeat number of EST-SSRs in B. lutosa transcriptome 表2 二核苷酸和三核苷酸EST-SSRs不同重復基元分布比例 Tab.2 Percentage of different motifs dinucleotide and trinucleotide EST-SSRs 隨機選取其中50條序列設計引物,通過對福建野生泥東風螺62只個體DNA樣本進行PCR擴增和分型,獲得23個多態性位點(表3)。23個位點的等位基因數為2~7個不等,平均a為3.5,平均e為0.491,平均o為0.477,平均PIC為0.451。HWE平衡檢驗顯示,4個位點顯著偏離平衡(<0.05)(表4)。 表3 泥東風螺EST-SSR引物信息 Tab.3 Information of EST-SSR primer in the B. lutosa 利用23個多態性位點分析泥東風螺野生群體和養殖群體遺傳多樣性(表4),結果顯示,福建野生群體e值為0.190~0.937,o為0.065~0.936,PIC為0.061~ 0.777,其中,9個微衛星位點表現為高度多態性(PIC>0.5),11個位點表現為中度多態性(0.5>PIC>0.25),3個位點表現為低度多態性(PIC<0.25)。養殖群體e(0.183~0.979)和o(0.130~0.980)的平均值均高于野生群體,但PIC(0.020~0.787)的平均值低于野生群體。SPSS分析群體間e、o和is差異顯著(<0.05),PIC差異極顯著((<0.01)。養殖群體高度多態性位點和低度多態性位點分別為7個和5個。在養殖群體樣本中檢測到B05、B08、B12、B15、B16和B20位點中出現等位基因缺失現象。野生群體和養殖群is分別為–0.214~0.377和–0.130~0.129,群體的is平均值都大于0,在野生群生群體和養殖群體中,位點is<0的分別有13個和9個。HWE平衡檢驗顯示,野生群體和養殖群體分別有4個和9個位點偏離平衡。 Popgene分析野生群體與養殖體間的位點基因流值(m)為0.132~543.787(表4),平均m為4.450,說明這2個群體間的遺傳分化小。群體間位點遺傳分化指數(st)為0.001~0.655(表4),平均st為0.053,屬于遺傳分化中等(0.05 不同物種轉錄組數據庫中微衛星分布的頻率存在差異。本研究分析了泥東風螺轉錄組中SSR的分布頻率和重復基元的特點。從泥東風螺SSR重復基元來看,二核苷酸重基元中AC/GT占70.58.%,這與其他水產動物中AC/GT重復最多一致(曾聰等, 2013)。三核苷酸重基元占總數15.27%,其中,CCG/CGG重復基元數量最少,這與一些真核生物中發現一定比例的CCG/CGG重復相似(Toth, 2000)。Schlottere等(1992)推測微衛星的長度因在復制過程中的滑動而反映了微衛星位點獲得(或失去)重復基元的活躍程度。Schlotterer(2000)認為,SSR位點的變異頻率與基元重復數存在一定正相關,即重復次數越多,SSR產生變異的可能性越大。Temnykh等(2001)進一步研究表明,SSR的長度是影響其多態性高低的重要因素,據此分析,泥東風螺轉錄組基因所含的微衛星長度絕大部分分布在12~25 bp之間,推測是受到強烈趨同選擇的壓力影響。 表4 泥東風螺23個EST-SSR位點的野生群體和養殖群體遺傳多樣性分析 Tab.4 Genetic diversity of wild and breeding populations in B. lutosa at 23 EST-SSR loci 對福建野生群體和養殖群體的位點各項參數進行分析,結果顯示,野生群體雜合子過剩(is<0,表明存在雜合子過剩)現象比養殖群體多4個位點。養殖群體有6個位點檢測到等位基因缺失,這可能與本研究選取樣本數量較少及養殖群體選擇的親本來自不同地理群體有關。o和e是判定群體遺傳多樣性水平的重要指標,養殖群體中平均o和e顯著高于野生群體,推測是因目前養殖群體選育采用的技術策略導致養殖群體的遺傳多樣性發生變化。 野生群體和養殖群體間st為0.053 (0.05 本研究中,泥東風螺養殖群體的遺傳多樣性顯著低于野生群體,這與中國明對蝦()(王軍等, 2018)、草魚()(王沈同等, 2018)、大黃魚() (趙廣泰等, 2010)、鱖()(鄭荷子等, 2013)水產動物養殖群體比野生群體遺傳多性低的研究結果一致,其原因可能為目前養殖泥東風螺繁育親本群體來源和個體數量問題。研究表明,本研究所開發的SSR具有較高的多態性,研究的群體具有較高的遺傳多樣性,具有進一步選育的價值。 SSR標記在水生生物之間通用性較好(張瓊等, 2010; 劉必謙等, 2007),SSR遺傳作圖將使物種之間連鎖信息的轉換更快,實現多個圖譜整合,從而更有利于比較基因組學的研究。本研究開發的泥東風螺EST-SSR標記對泥東風螺的親子譜系分析、群體遺傳結構分析、圖譜構建、增殖放流效果評估和分子輔助育種等研究方面具有重要的意義。 Chen LX, Wu JF. Culture technique and industrial prospect of ivory shell. Shandong Fisheries, 2004, 21(10): 9–11 [陳利雄, 吳進鋒. 東風螺的增養殖技術及產業化前景. 齊魯漁業, 2004, 21(10): 9–11] Eujayl I, Sorrells ME, Baum M,. Isolation of EST-derived microsatellite markers for genotyping the A and B genomes of wheat. Theoretical and Applied Genetics, 2002, 104(2–3): 399–407 Graham IA, Besser K, Blumer S,. The genetic map ofL. identifies loci affecting yield of the antimalarial drug artemisinin. Science, 2010, 327(5963): 328–331 Guan A, Wu YT, Chen Y,. Deep sequence-based transcriptome analysis of microsatellites in the cuttlefish (). Progress in Fishery Sciences, 2018, 39(3): 144–151 [管奧, 毋玉婷, 陳宇, 等. 曼氏無針烏賊轉錄組微衛星特征分析. 漁業科學進展, 2018, 39(3): 144–151] Kalinowski ST, Taper ML, Marshall TC. Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment. Molecular Ecology, 2007, 16(5): 1099–1106 Kanehisa M, Araki M, Goto S,. KEGG for linking genomes to life and the environment. Nucleic Acids Research, 2008, 36: D480–D484 Li DM, Yang AG, Wu B,. Development and application of the EST-SSR markers in. Progress in Fishery sciences, 2017, 38(2): 137–142 [李東明, 楊愛國, 吳彪, 等. 櫛江珧() EST-SSR標記的開發與應用. 漁業科學進展, 2017, 38(2): 137–142] Li DY, Meng XH, Kong J,. The difference of genetic diversity and the comparison of growth performance between selected population and hybridized population of pacific white shrimp () under low temperature conditions. Progress in Fishery sciences, 2017, 38(4): 69–77 [李東宇, 孟憲紅, 孔杰, 等. 凡納濱對蝦()選育群體與雜交群體遺傳多樣性差異及其在低溫條件下生長性能的比較. 漁業科學進展, 2017, 38(4): 69–77] Li W, Zhao S, Jiao HF,. Characterization and analysis of microsatellite markers inusing next generation sequencing. Marine Sciences, 2015, 39(11): 61–67 [李威, 趙姍, 焦海峰, 等. 黃口荔枝螺轉錄組數據的微衛星標記開發與分析. 海洋科學, 2015, 39(11): 61–67] Lin GQ, Lin D, Chen XF,. Research on the scale artificial breeding technique and the morphological observation of the early developmental stages of. Journal of Fujian Fisheries, 2015, 37(1): 20–28 [林國清, 林丹, 陳曦飛, 等. 泥東風螺()規模化人工育苗技術和早期發育觀察. 福建水產, 2015, 37(1): 20–28] Liu BQ, Zeng QG, Wang YJ,. The cross-species amplification and validation of EST-SSR loci in. Acta Hydrobiolonica Sinica, 2007, 31(2): 149–154 [劉必謙, 曾慶國, 王亞軍, 等. 條斑紫菜EST-SSR引物種間轉移擴增真實性研究. 水生生物學報, 2007, 31(2): 149–154] Morgante M, Olivieri AM. PCR-amplified microsatellites as markers in plant genetics. Plant Journal, 1993, 3(1): 175–182 Ni SS, Yang Y, Liu SF,. Microsatellite analysis ofusing next-generation sequencing method. Progress in Fishery sciences, 2018, 39(1): 107–113 [倪守勝, 楊鈺, 柳淑芳, 等. 基于高通量測序的蝦夷扇貝基因組微衛星特征分析. 漁業科學進展, 2018, 39(1): 107–113] Powell WMGCP. Polymorphism revealed by simple sequence repeats. Trends in Plant Science, 1996, 7(1): 215–222 Qin Z, Wang XQ, Zeng ZN,. Genetic distance in four populations of(Lamer) assessed by AFLP makers. Journal of Hunan Agricultural University (Natural Sciences), 2014, 40(3): 299–304 [秦溱, 王曉清, 曾志南, 等. 泥東風螺4個群體遺傳多樣性的AFLP分析. 湖南農業大學學報(自然科學版), 2014, 40(3): 299–304] Schlotterer C, Tautz D. Slippage synthesis of simple sequence DNA. Nucleic Acids Research, 1992, 20(2): 211–215. Schlotterer C. Evolutionary dynamics of microsatellite DNA. Chromosoma, 2000, 109(6): 365–371 Shi SF, Yao HH, Lin ZH,. Characterization and analysis of 24 polymorphic EST-SSR loci in. Marine Sciences, 2013, 37(8): 42–46 [史松富, 姚韓韓, 林志華, 等. 24個泥蚶EST-SSR標記的開發與分析. 海洋科學, 2013, 37(8): 42–46] Simon SA, Zhai J, Nandety RS,. Short-read sequencing technologies for transcriptional analyses. Annual Review of Plant Biology, 2009, 60: 305–333 Temnykh S, DeClerck G, Lukashova A,. Computational and experimental analysis of microsatellites in rice (L.): Frequency, length variation, transposon associations, and genetic marker potential. Genome Research, 2001, 11(8): 1441–1452 Toth G, Gaspari Z, Jurka J. Microsatellites in different eukaryotic genomes: Survey and analysis. Genome Research, 2000, 10(7): 967–981 Wang J, Wang QY, Kong J,. SSR analysis on genetic diversity in breeding and wild populations of. Progress in Fishery sciences, 2018, 39(2): 104–111 [王軍, 王清印, 孔杰, 等. 中國明對蝦人工選育群體與野生群體遺傳多樣性的SSR分析. 漁業科學進展, 2018, 39(2): 104–111] Wang RC. China aquatic shellfish primary color guide. Hangzhou: Zhejiang Science and Technology Press, 1988 [王如才. 中國水生貝類原色圖鑒. 杭州: 浙江科學技術出版社, 1988] Wang ST, Shen YB, Meng XZ,. Genetic variability in wild and selected populations ofusing microsatellite markers. Journal of Fisheries of China, 2018(8): 1273–1284 [王沈同, 沈玉幫, 孟新展, 等. 草魚野生與選育群體遺傳變異微衛星分析. 水產學報, 2018(8): 1273–1284] Ye QT, Liu Y, Zeng ZN,. Tracking survey after enhancement and releasing ofand its effects analysis. Journal of Fujian Fisheries, 2015a, 37(2): 140–147 [葉泉土, 劉勇, 曾志南, 等. 泥東風螺增殖放流跟蹤調查及效果分析. 福建水產, 2015a, 37(2): 140–147] Ye QT, Wu QS, Zeng ZN,. Effect of different bottom sowing density on growth and survival ofjuveniles. Journal of Fujian Fisheries, 2015b, 37(1): 36–42 [葉泉土, 巫旗生, 曾志南, 等. 不同底播密度對泥東風螺()幼螺生長和存活的影響. 福建水產, 2015b, 37(1): 36–42] Yeh FC,Boyle TJB. Population genetic analysis of codominant and dominant markers and quantitative traits. Belgian Journal of Botany, 1997, 129–157 Yu AQ, Shi YH, Xu JB,. Characteristic analysis of microsatellites in the selectedbased on transcriptome dataset. Progress in Fishery Sciences, 2019, 40(5): 101–109 [于愛清, 施永海, 徐嘉波, 等. 長江刀鱭選育群體轉錄組EST-SSR的分布特征分析. 漁業科學進展, 2019, 40(5): 101–109] Zeng C, Gao ZX, Luo W,. Characteristics of microsatllites in blunt snout bream () EST sequences using 545 FLX. Acta Hydrobiolonica Sinica, 2013, 37(5): 982–988 [曾聰, 高澤霞, 羅偉, 等. 基于454GS FLX高通量測序的團頭魴ESTs中微衛星特征分析. 水生生物學報, 2013, 37(5): 982–988] Zhang GM, Sun XJ, Wu B,. Transferability of EST-SSR frominto. Progress in Fishery Sciences, 2018, 39(4): 1–8 [張廣明, 孫秀俊, 吳彪, 等. 蝦夷扇貝EST-SSR標記在櫛孔扇貝中的通用性研究. 漁業科學進展, 2018, 39(4): 1–8] Zhang HH, Wu JF, Chen LX,. Prospect of artificial breeding, breeding and industrialization of. Southern Aquaculture, 2004(11): 2–5 [張漢華, 吳進鋒, 陳利雄, 等. 東風螺人工育苗、養殖及產業化發展前景. 南方水產, 2004(11): 2–5] Zhang Q, Liu XL, Li XL,. Application of simple sequence repeats derived from expression sequence tags (EST-SSRs) in aquatic animal genomics. Fisheries Science, 2010, 29(5): 302–306 [張瓊, 劉小林, 李喜蓮, 等. EST-SSR分子標記在水生動物遺傳研究中的應用. 水產科學, 2010, 29(5): 302–306] Zhang XY, Gao YN. To design PCR primers with Oligo 6 and Primer premier 5. China Journal of Bioinformatics, 2004, 2(4): 15–18 [張新宇, 高燕寧. PCR引物設計及軟件使用技巧. 生物信息學, 2004, 2(4): 15–18] Zhao GT, Liu XD, Wang ZY,. Genetic structure and genetic diversity analysis of four consecutive breeding generations of large yellow croaker () using microsatellite markers. Journal of Fisheries of China, 2010, 34(4): 500–507 [趙廣泰, 劉賢德, 王志勇, 等. 大黃魚連續4代選育群體遺傳多樣性與遺傳結構的微衛星分析. 水產學報, 2010, 34(4): 500–507] Zheng HZ, Yi TL, Liang XF,. Genetic structure and genetic diversity analysis of four consecutive breeding generations of. Freshwater Fisheries, 2013, 43(6): 8–12 [鄭荷子, 易提林, 梁旭方, 等. 翹嘴鱖連續4代選育群體遺傳多樣性及遺傳結構分析. 淡水漁業, 2013, 43(6): 8–12] Development and Genetic Diversity Analysis ofwith EST-SSR Markers XIONG Gang1,2, WANG Xiaoqing2①, WANG Pei2, CHEN Zhennian2, ZHOU Xianwen2,3, KANG Li1, ZENG Zhinan4 (1. Department of Animal Science and Technology, Hunan Biological and Electromechanical Polytechnic, Changsha 410127;2. College of Animal Science and Technology, Hunan Agriculture University, Changsha 410128;3. Station of Aquaculture in Xiang xi, Jishou 416000;4. Fisheries Research Institute of Fujian, Xiamen 361013) is a marine shellfish that has high economic values. In recent decades, the natural resource ofhas declined due to the environment destruction and overfishing. To further understand the level of genetic diversity and population genetic structure ofwe have evaluated the information characteristics of’s microsatellites, after obtaining transcriptome sequences using MISA software. The results show that a total of 16342 microsatellites and 181 nucleotide repeat motifs were identified. Different types of repeat microsatellites had considerably different distribution characteristics. Mononucleotide and dinucleotide microsatellite repeating units were the most abundant in thetranscriptome, in which 6 repeats of AC/GT (70.58%) were the dominant repeating dinucleotide units. The length of the dominant repeating units was 12~20 bp (63.95%) and 21~25 bp (9.14%), respectively, and the average length was 18.4 bp. Among the 50 designed primer pairs, 23 proved to be polymorphic microsatellite markers in thewild populations (WP). The results showed that the allele number of these microsatellites ranged from 2 to 7. The expected heterozygosity (e) and observed heterozygosity (o) ranged from 0.190 to 0.937, and 0.065 to 0.936, respectively. The polymorphism information content (PIC) ranged from 0.061 to 0.777. Theevalues of the WP and breeding population (BP) were 0.491 and 0.544, respectively. Theovalues of the WP and BP populations were 0.477 and 0.564, respectively. The PIC values for WP and BP were 0.541 and 0.407, respectively. There were 13 population loci that were heterozygote excesses in WP, and 13 population loci in BP. The genetic differentiation index (st) ranged from 0.001 to 0.655, with an average value of 0.053 (0.05 ; Transcriptome; EST-SSR; Polymorphism WANG Xiaoqing, E-mail: wangxiao8258@126.com S917.4 A 2095-9869(2020)04-0117-08 10.19663/j.issn2095-9869.20190417002 http://www.yykxjz.cn/ 熊鋼, 王曉清, 王佩, 陳貞年, 周先文, 康驪, 曾志南. 泥東風螺EST-SSR開發及其群體遺傳多樣性分析. 漁業科學進展, 2020, 41(4): 117–124 Xiong G, Wang XQ,Wang P, Chen ZN, Zhou XW, Kang L, Zeng ZN. Development and genetic diversity analysis ofwith EST-SSR markers. Progress in Fishery Sciences, 2020, 41(4): 117–124 * 海洋公益性行業科研專項經費項目(201205021)、國家自然科學基金(31672640)、湖南省自然科學基金(2017JJ3134; 2016NK2115)和湖南省教育廳基金項目(17C0935)共同資助[This work was supported by Public Science and Technology Research Funds Projects of Ocean (201205021), National Natural Science Foundation of China (31672640), Natural Science Foundation of Hunan Province(2017JJ3134; 2016NK2115), and Scientific Research Fund of Hunan Province Education Department (17C0935)]. 熊 鋼,E-mail: xionggang709@126.com 王曉清,教授,E-mail: wangxiao8258@126.com 2019-04-17, 2019-05-12 (編輯 馮小花)1 材料與方法
1.1 材料
1.2 方法
2 結果
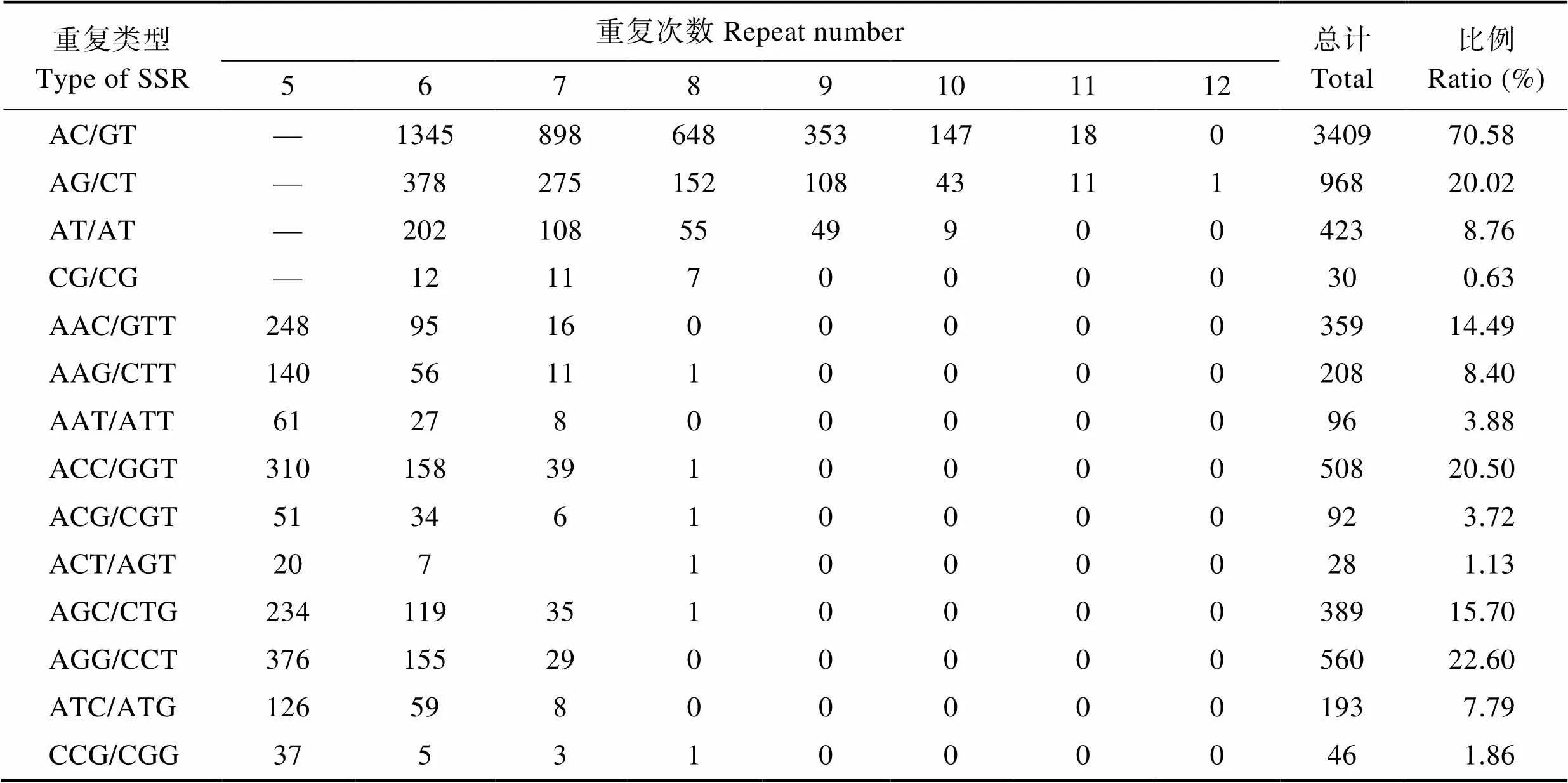
2.1 轉錄組中EST-SSR特性

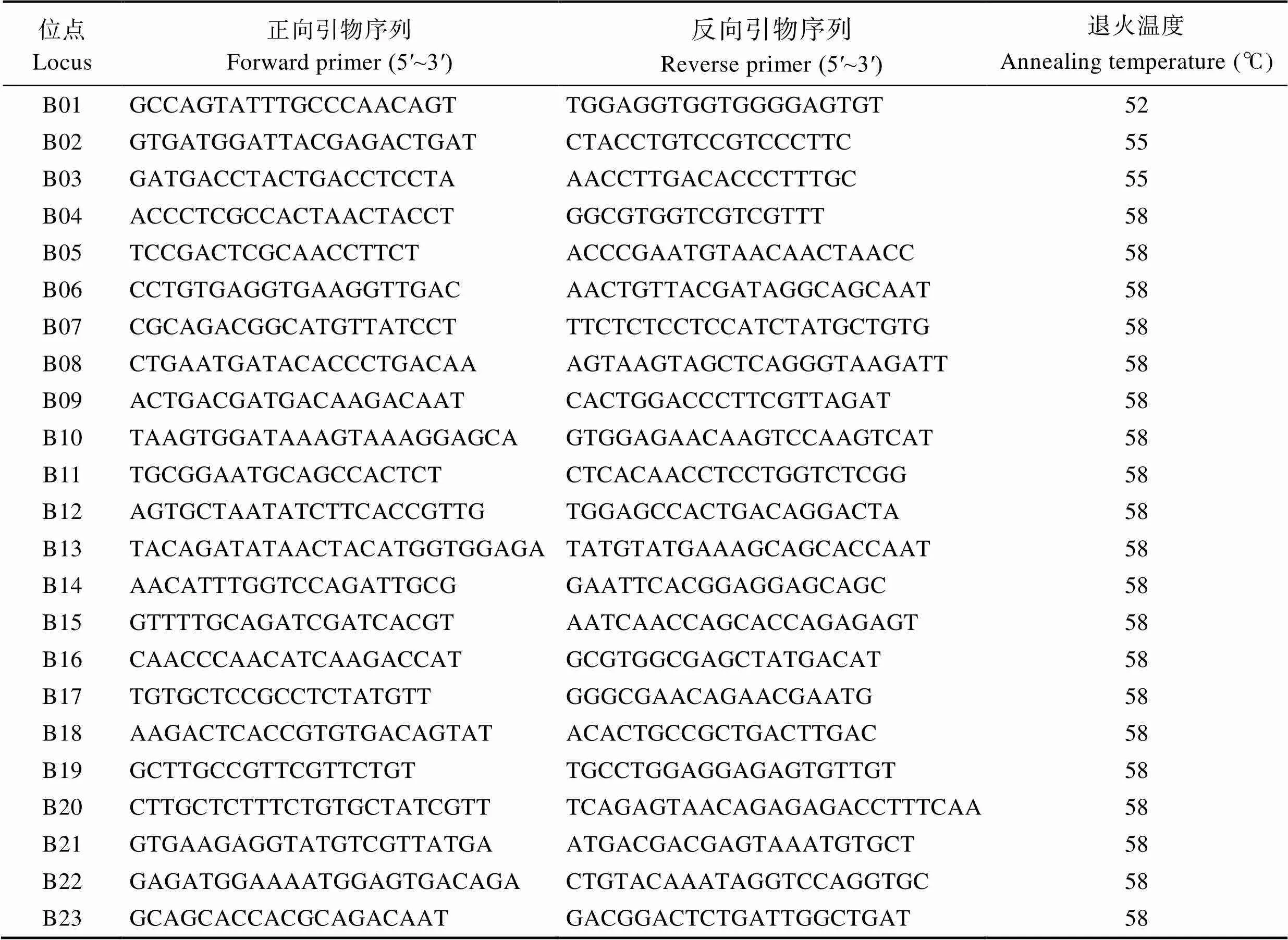
2.2 擴增位3點的多態性
2.3 野生群體和養殖群體遺傳多樣性
2.4 野生群體和養殖群體遺傳分化
3 討論
3.1 轉錄組中EST-SSR分布

3.2 EST-SSR在泥東風螺野生和養殖群體中的多態性變化
3.3 泥東風螺野生群體和養殖群體遺傳多樣性與遺傳分化

