基于Cytb和D-Loop的日本囊對蝦遺傳多樣性分析
杜景豪,王偉峰,陳秀荔,侯春秀,王煥嶺
( 1.華中農業大學 水產學院,湖北 武漢 430070; 2.廣西壯族自治區水產科學研究院,廣西水產遺傳育種與健康養殖重點實驗室,廣西 南寧 530021 )
日本囊對蝦(Marsupenaeusjaponicus)屬甲殼綱、十足目、對蝦科、囊對蝦屬。日本囊對蝦的分布極其廣泛,在我國主要分布在長江口以南沿海,其具有耐低溫、生長快、營養高等特點,在我國對蝦養殖中具有較高的經濟地位[1]。近年來,捕撈量的增加、養殖規模的擴大以及水域生態環境的惡化等,導致其自然資源不斷衰退。而遺傳變異水平與生物的生長速度、抗病能力等生產性狀密切相關[2]。因此開展日本囊對蝦的遺傳多樣性分析對其種質資源保護與合理開發利用具有重要的意義。
線粒體DNA(mtDNA)是一種核外遺傳物質,由于具有分子量小、結構簡單、進化速度快、母系遺傳及無組織特異性等特點[3],而成為評估群體遺傳和生物進化的有效標記[4]。Tsoi等[5-6]曾利用mtDNA和微衛星標記研究西太平洋日本囊對蝦,發現其可以分成2個變種,并且都具有很高的遺傳多樣性。郭慧等[7-8]利用微衛星標記對我國沿海3個日本囊對蝦群體遺傳多樣性進行了研究,發現不同地理群體間具有不同程度的分化。
而動物線粒體基因組中含細胞色素氧化酶3個亞基基因 (COⅠ、COⅡ、COⅢ)、細胞色素b(Cytb)、2個ATP酶亞基因(ATPase6、ATPase8)和7個NADH還原酶復合體亞基因共13個結構蛋白基因[9]。其中由于Cytb基因的進化速度適中,諸多學者選擇其作為遺傳多樣性分析的標記,用于分析種群母系起源、系統進化、遺傳結構和遺傳多樣性等[10-13]。另外線粒體DNA控制區(D-Loop)處于非編碼區,富含A-T堿基,進化速度較快,是揭示種群遺傳結構常用的分子標記[14-15]。近年來,研究發現利用D-Loop序列進行遺傳多樣性和系統進化研究信息更加全面,結果更為準確[16-17]。因此,筆者基于Cytb和D-Loop序列,對我國日本囊對蝦遺傳多樣性展開研究,為日本囊對蝦種質資源保護及合理利用和雜交育種的群體選擇提供科學依據。
1 材料與方法
1.1 試驗材料
試驗所用日本囊對蝦于2018年8月—2019年3月分別采自浙江舟山、福建福州、福建廈門、廣西北海、廣東湛江5個近海水域,每個群體約20尾。所有樣品均取對蝦腹部第六腹節肌肉,于-20 ℃無水乙醇中保存。
1.2 基因組DNA的提取
取肌肉100~150 mg,采取SDS法[18]提取基因組DNA,使用超微量紫外分光光度計測定DNA質量濃度并稀釋到150 ng/μL,1%瓊脂糖凝膠電泳檢測DNA,于4 ℃保存。
1.3 PCR擴增
根據美國國立生物技術信息中心中日本囊對蝦線粒體的基因組序列(序列號:AP006346)設計引物用于擴增Cytb基因和D-Loop區。其中擴增Cytb的引物序列為:Cb-L 5′-GGTCCTTTACG CTTATCATC-3′和Cb-R 5′-GGGTGAGTGGGTTAATAATG-3′;擴增D-Loop的引物序列為:Dp-L 5′-ATTAGCACTAGGTACTGAGA-3′和Dp-R 5′-GGTAAGGCTTGACTTACATA-3′;由武漢天一輝遠生物科技有限公司合成引物。PCR反應體系為20 μL,其中雙蒸水15.2 μL,10×PCR Buffer 2.0 μL,DNA模板1.0 μL,左右引物各0.6 μL,dNTPs 0.3 μL,TaqDNA聚合酶0.3 μL。PCR擴增反應程序為:94 ℃預變性5 min,94 ℃變性30 s,53 ℃退火30 s,72 ℃延伸75 s,共32個循環,最后72 ℃終延伸7 min。1%瓊脂糖凝膠電泳檢測PCR產物,送至武漢天一輝遠生物科技有限公司進行雙向測序。
1.4 數據分析
使用Seqman軟件對比測序結果,并輔以人工校正;DnaSP[19]軟件統計多態位點數、單倍型數、單倍型多態性、核苷酸多態性、多態簡約信息位點數、Fu和Li′sD檢驗參數、Tajima′sD檢驗參數;MEGA[20]軟件用于分析序列的平均堿基組成、轉換/顛換比率R、群體內和群體間的遺傳距離以及系統發育樹[21]的構建;Arlequin 3.1[22]軟件分析群體間及群體內的遺傳變異和核苷酸錯配分布情況。
2 結 果
2.1 堿基組成
測序結果經過對比和校對后,分別選取長度1000 bp和900 bp的Cytb和D-Loop區用于后續分析。分析結果顯示,5個群體中Cytb和D-Loop序列的堿基顛換比值R(TS/TV)分別為6.267和2.694,突變以堿基的顛換為主。4種堿基在所得序列中平均含量A為24.77%和37.22%、T為35.88%和45.32%、C為24.07%和8.68%、G為15.28%和8.78%,其中A+T含量為60.65%和82.54%,顯著高于C+G含量(表1),D-Loop序列較Cytb序列堿基組成更具偏倚性。
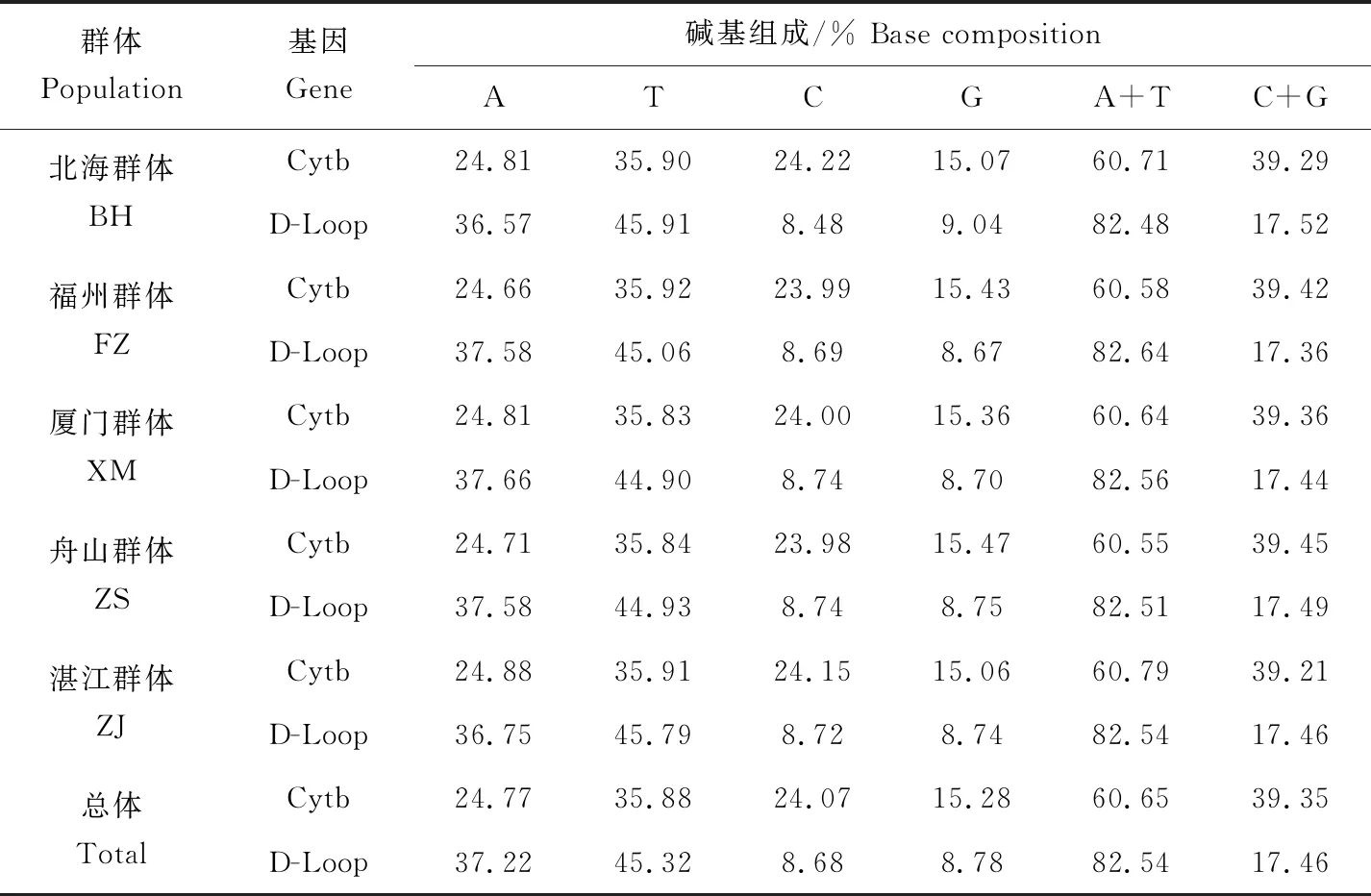
表1 日本囊對蝦5個群體Cytb和D-Loop序列堿基組成
2.2 遺傳多樣性和遺傳結構
基于Cytb序列在5個群體中檢測到76個簡約信息位點,共定義了55種單倍型,其中Hap-30、Hap-36數量最多,均為10個,占樣本總數的10.1%;Hap-6數量為9個,占樣本總數9.1%;Hap-50、Hap-53數量均為4個,占樣本總數的4.1%;Hap-21、Hap-54數量均為3個,占樣本總數的3.1%;Hap-2、Hap-9、Hap-19、Hap-29、Hap-35、Hap-47、Hap-49、Hap-51數量均為2個,占樣本總數的2.1%;其他單倍型均只有1個(表2)。單倍型多態性為0.969,核苷酸多態性為0.033。群體內遺傳距離為0.004~0.022,群體間遺傳距離為0.007~0.068,其中遺傳距離最大是舟山群體與北海群體,為0.068(表3)。基于D-Loop序列分析,在5個群體中發現了75個簡約信息位點,共定義了78種單倍型,其中Hap-22數量為4個,占樣本總數的4%;Hap-7、Hap-65數量均為3個,占樣本總數的3%;Hap-2、Hap-6、Hap-19、Hap-26、Hap-30、Hap-38、Hap-42和Hap-49數量均為2個,占樣本總數的2%;其余單倍型均為1個。單倍型多態性為0.995,核苷酸多態性為0.036。群體內遺傳距離為0.005~0.017,群體間遺傳距離為0.012~0.075,其中遺傳距離最大是舟山群體與北海群體(0.075)(表4)。D-Loop序列中單倍型數、單倍型多態性及核苷酸多態性均高于Cytb序列,這表明D-Loop序列具有更高的遺傳多樣性,這可能與其進化速度較快有關。
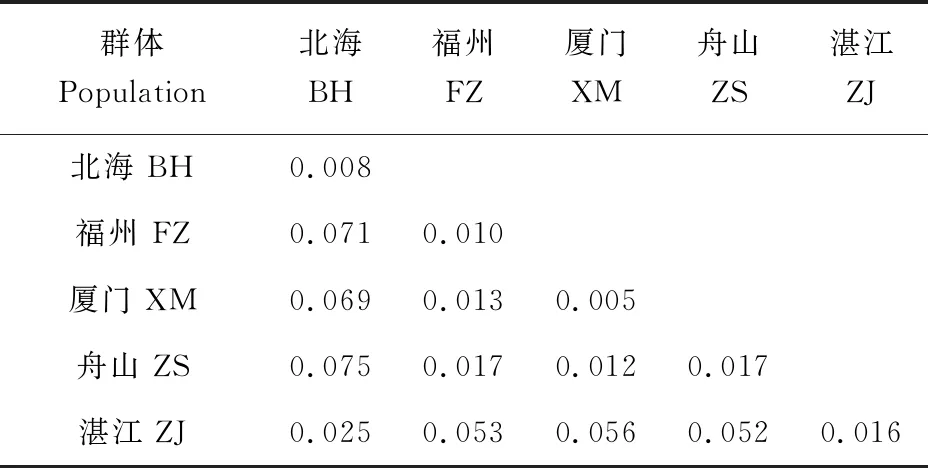
表4 基于D-Loop序列的群體內及群體間遺傳距離
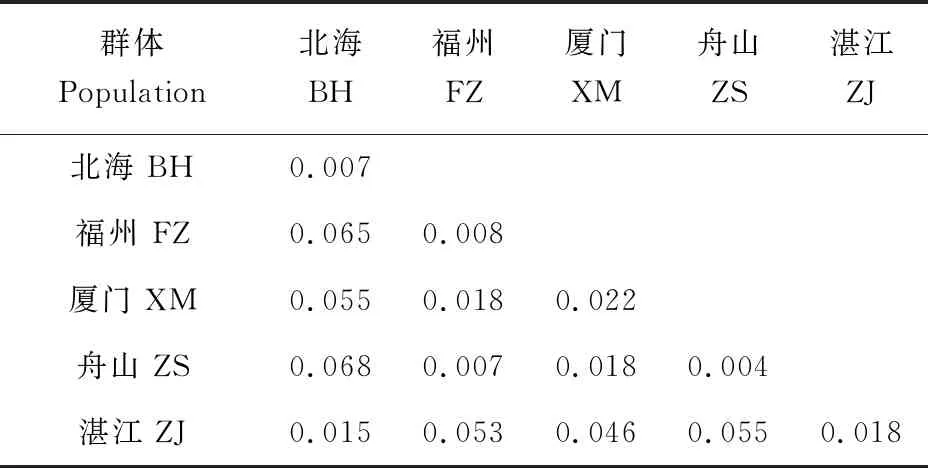
表3 基于Cytb序列的群體內及群體間遺傳距離
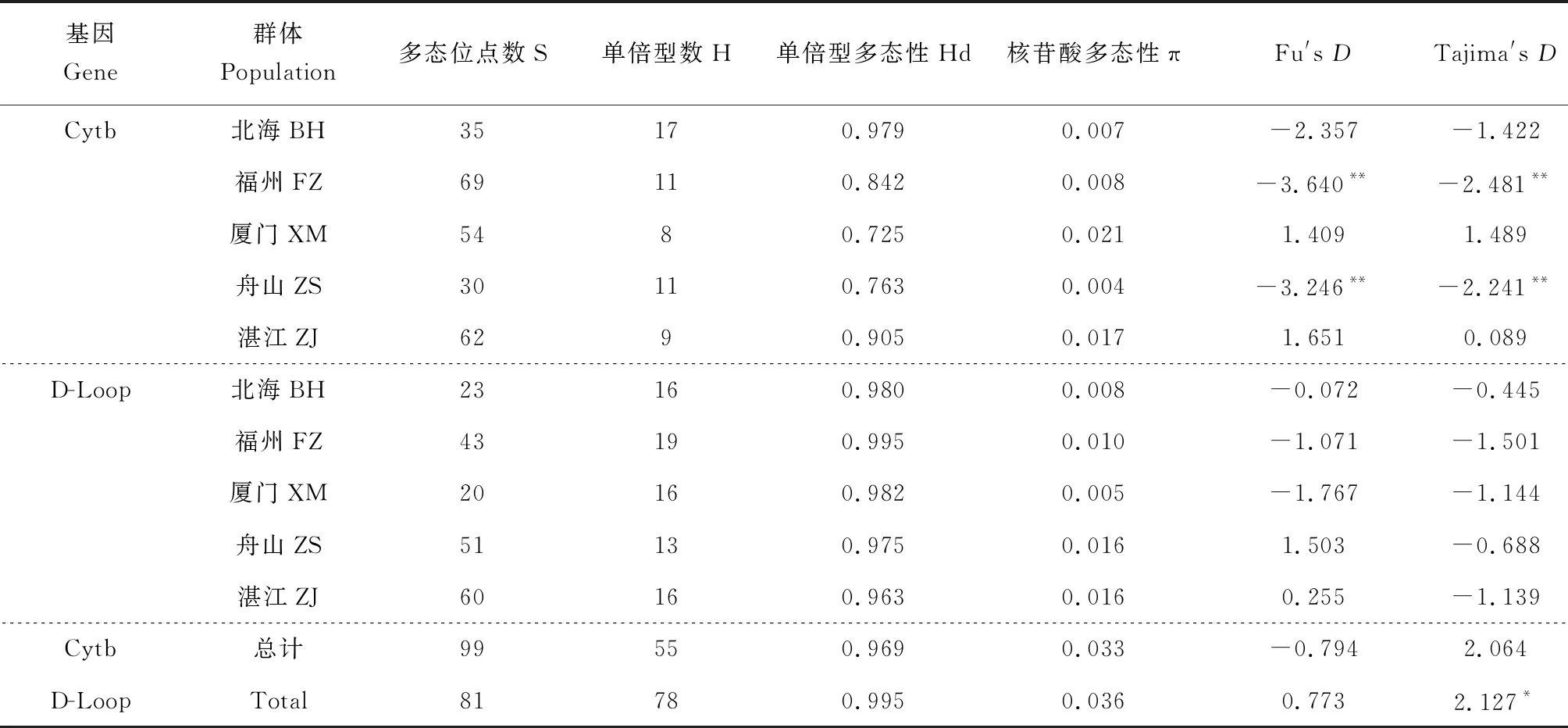
表2 群體遺傳多樣性參數
分子方差分析結果顯示,基于Cytb和D-Loop序列獲得的群體內變異貢獻率分別為29.99%和25.61%,群體間變異貢獻率達到70.01%和74.39%,可見群體間變異是變異的主要來源(表5)。遺傳分化指數(FST)常用于表示群體間的遺傳關系及分化程度,值越大表示群體分化程度越大[23]。兩者分化指數分別為0.7001和0.7439,表明群體間遺傳分化程度明顯。
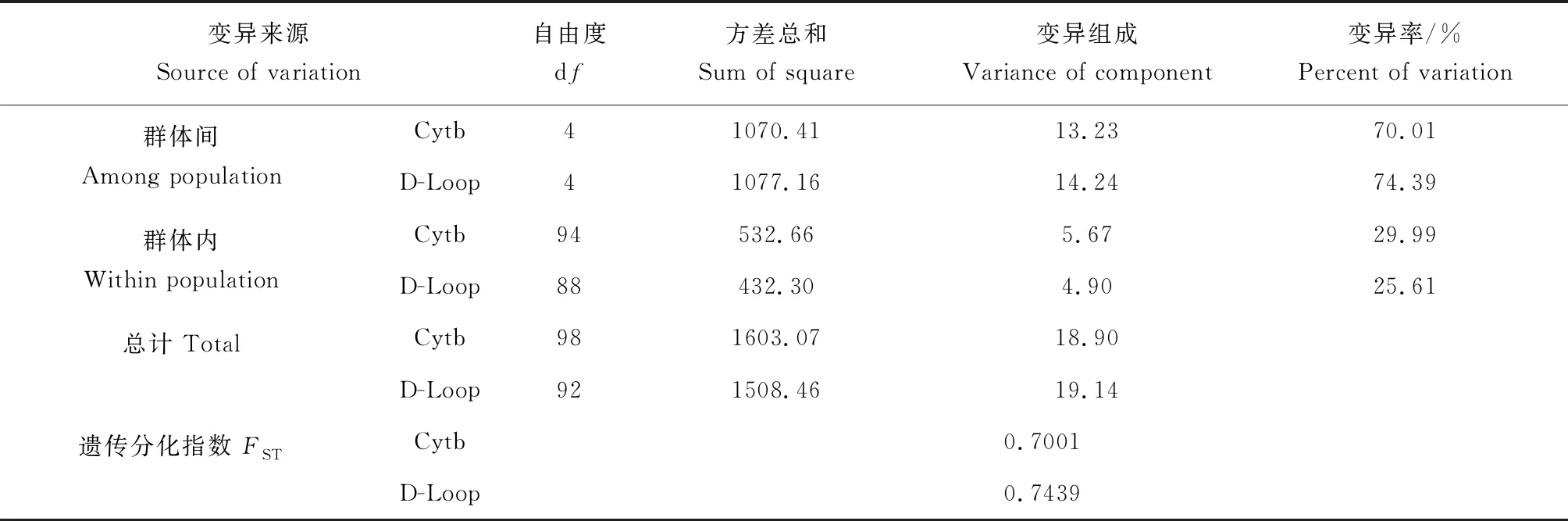
表5 群體分子方差分析
2.3 單倍型系統發育樹
基于單倍型分別構建系統發育進化樹,結果顯示(圖1),不同群體間單倍型分化比較明顯,主要分化成兩個單系,即北海群體和湛江群體聚成一支,其他3個群體聚成另一支,同一單系間又有不同程度的分化,說明日本囊對蝦5個群體間存在比較明顯的地理譜系,種群間分化具有明顯的地域性。
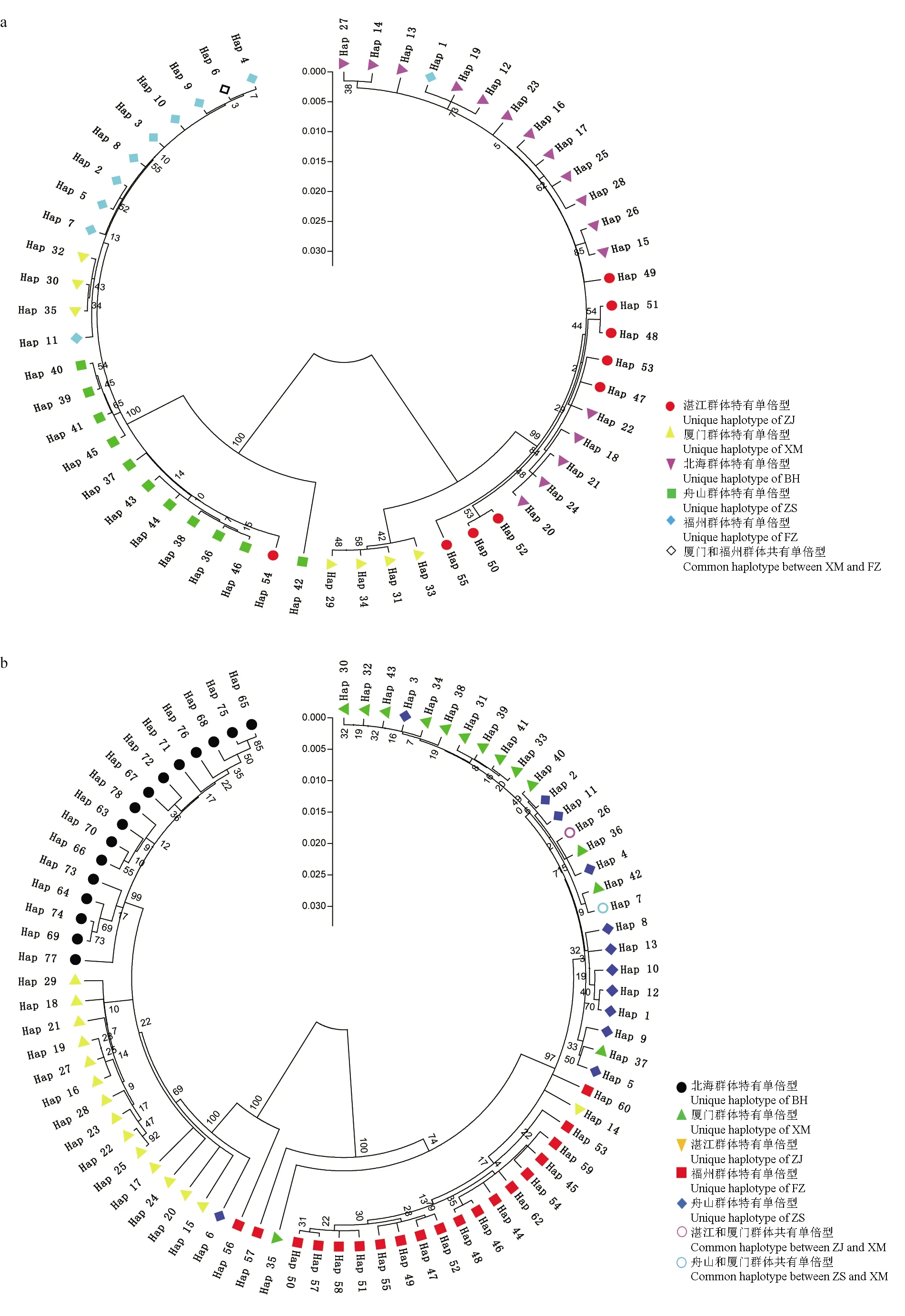
圖1 基于Cytb(a)和D-Loop(b)序列單倍型系統發育樹
2.4 種群歷史動態
中性檢驗結果見表2,基于Cytb序列發現,北海、福州和舟山3個群體Fu′D值和Tajima′D值為負值,而其他2個群體Fu′D值和Tajima′D值為正值,表明北海、福州和舟山3個群體近期發生了種群擴張,但總體未明顯偏離中性突變,種群存在平衡選擇,近期未出現明顯擴張。基于D-Loop序列研究發現,5個群體Tajima′D值均為負值,總體 Tajima′D值為2.127,具有顯著性差異,表明各群體之間存在定向選擇,總體存在平衡選擇。而基于Cytb和D-Loop序列中性檢驗值在5個群體內具有差異性,可能是由于不同群體所處環境不同且D-Loop區更容易發生堿基突變。日本囊對蝦5個群體Cytb和D-Loop區核苷酸錯配分布見圖2,兩者成多峰分布,表明整個種群保持相對穩定,近期未曾經歷過瓶頸效應和明顯擴張。
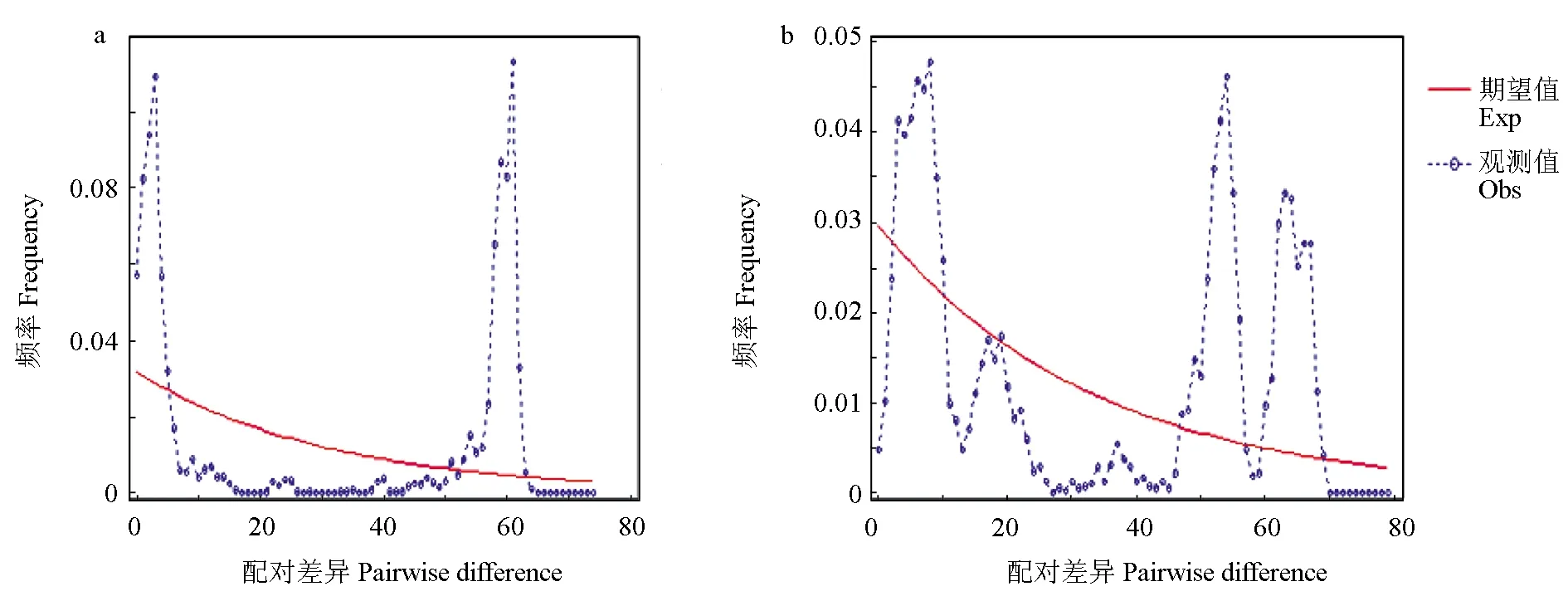
圖2 群體Cytb(a)和D-Loop(b)核苷酸錯配分布
3 討 論
3.1 序列堿基組成分析
本研究基于線粒體Cytb和D-Loop序列對我國沿海5個日本囊對蝦群體的遺傳多樣性進行了分析,發現4種堿基出現明顯的偏隨機分布,即A+T含量顯著高于C+G含量,符合節肢動物mtDNA堿基組成中G+C含量相對缺乏,A+T含量高的普遍現象[24]。曾凡榮等[25]基于日本囊對蝦線粒體Cytb的研究發現,A+T含量高達59.4%,與本研究相關結果基本相符。利用顛換比值R,可以估計序列的飽和度,R>5表示序列未達到飽和,一般來說分歧時間越長、親緣關系越遠的群體間堿基發生顛換的頻率越高,即R值越小,由此可初步判斷群體間的進化關系[26]。Cytb和D-Loop區堿基顛換比值R分別為5.970和2.694,說明Cytb突變未達到飽和而D-Loop區突變可能已達到飽和,兩者均產生了一定程度的進化。
3.2 群體遺傳多樣性與遺傳分化分析
遺傳多樣性即基因多樣性,存在于生物個體內、單個物種內以及物種間,是生物適應和進化的基礎,種內的遺傳多樣性與該物種對環境的適應能力呈正向線性關系[27]。物種遺傳多樣性的分布不僅受到現階段演化力量(如遷移)的影響, 還受到歷史的影響,生活環境的不連續性和種群數量變動的不穩定,均會造成種群間的分化[28]。劉若余等[29]研究表明,線粒體DNA的單倍型多態性和核苷酸多態性是衡量群體遺傳多樣性和遺傳分化的重要指標,兩者數值越大,群體遺傳多樣性越高。本研究從Cytb和D-Loop區兩個角度分析發現,兩者的單倍型多態性分別為0.969和0.995,核苷酸多態性為0.033和0.036,這說明我國沿海5個日本囊對蝦群體遺傳多樣性處于較高的水平。Tzeng等[30]研究日本囊對蝦線粒體控制區發現,東亞地區日本囊對蝦具有很高的遺傳多樣性,與本研究的結果基本相符。
從遺傳距離來看,物種內的遺傳距離通常小于10%[31]。大于6%的群體間會有明顯的亞種或者種的分化[32-33],不論是從Cytb或D-Loop來衡量,北海群體和湛江群體與其他3個群體遺傳距離均較遠,接近亞種的分化水平,可能由于不同群體間地理位置相距較遠,阻礙了群體間基因交流。基于Cytb和D-Loop序列分子方差分析顯示,群體間變異是總變異的主要來源。而曾凡榮等[25]基于Cytb基因的研究也發現,日本囊對蝦群體間遺傳變異占69.9%。據溯祖理論[34],Cytb中單倍型Hap-30和Hap-36數量最多可能為祖先類型,而D-Loop區單倍型Hap-22可能為祖先類型。D-Loop區單倍型分析發現,5個群體間共享單倍型較少,群體分化十分明顯,這與Cytb基因分析結果有所不同,可能是由于D-Loop區受到的選擇壓力小,進化速度較快,而Cytb是編碼蛋白質的功能基因,受到的選擇壓力較大,進而導致Cytb和D-Loop的變異存在不均一性[35]。基于Cytb和D-Loop序列單倍型進化樹發現,各群體特有單倍型基本聚在一起,部分存在交叉的現象,可能是群體間的部分個體在自然生長過程中遺傳背景存在交叉[36-37]。基于Cytb和D-Loop序列分析發現,D-Loop進化速度要高于Cytb,遺傳多樣性更加豐富,相對更加接近分子鐘假說。因此,D-Loop比Cytb更加適合于研究日本囊對蝦群體遺傳多樣性的分子標記。
由種群的錯配分布圖可見,種群呈多峰分布,表明種群近期未曾經歷過瓶頸效應和明顯擴張[38],保持相對穩定的狀態,自然選擇將突變保持在平衡水平。此外,日本囊對蝦產卵洄游并不是特別明顯,僅隨個體生長由近岸向遠岸遷移,這樣地理距離相距較近的群體更容易發生基因交流。氣候急劇變化會引起群體數量突然減少,可能導致種群遺傳多樣性差異,而洋流、遷徙和人類活動會促進不同群體間的基因交流而減少分化[39-40]。
本研究通過日本囊對蝦不同地理種群的遺傳多樣性分析發現,我國日本囊對蝦群體具有較高的遺傳多樣性,野生資源狀況良好,為日本囊對蝦的種質資源保護工作提供科學依據,也為雜交育種的群體選擇提供參考資料。

