肉雞CMTR2基因分子特征及其表達譜分析
李修嶺 徐香慧 邢晉祎


摘要:【目的】分析肉雞加帽甲基轉移酶基因(CMTR2)分子特征及其表達譜,為后續開展雞CMTR2基因結構及生物學功能研究打下基礎。【方法】以艾拔益加(AA)肉雞為研究對象,利用RT-PCR克隆CMTR2基因,使用DNAMAN、ProtParam、SOPMA、SWISS-MODEL、TMHMM 2.0和SignalP 3.0等在線軟件進行生物信息學分析,并以實時熒光定量PCR檢測分析其表達特征。【結果】肉雞CMTR2基因編碼區(CDS)序列全長為2350 bp(登錄號MN296490),其開放閱讀框(ORF)為2292 bp,編碼763個氨基酸殘基;肉雞CMTR2氨基酸序列與原雞CMTR2氨基酸序列的相似性為99.74%,只有2個氨基酸殘基差異,與火雞CMTR2氨基酸序列的親緣關系較近(相似性為95.94%)。肉雞CMTR2蛋白分子量為86855.48 Da,理論等電點(pI)為6.08,在哺乳動物網織紅細胞體外表達的半衰期為30 h,在酵母體內表達的半衰期大于20 h,在大腸桿菌體內表達的半衰期大于10 h。肉雞CMTR2蛋白為不穩定的親水性蛋白,無信號肽和跨膜區域,包含61個磷酸化位點、15個二硫鍵、1個N-糖基化位點及8個O-糖基化位點;其二級結構由α-螺旋(49.93%)、無規則卷曲(35.65%)、延伸鏈(11.53%)和β-轉角(2.88%)組成,但無法獲得完整的三級結構。CMTR2基因在42日齡AA肉雞8種組織中的相對表達量排序為脾臟>肺臟>胸肌>腹脂>肝臟>腎臟>腿肌>心臟,以在脾臟中的相對表達量最高,顯著高于在其他組織中的相對表達量(P<0.05,下同);肉雞CMTR2基因的時空表達變化趨勢表現為21日齡的相對表達量最高、14日齡的相對表達量最低。【結論】CMTR2基因在不同物種中具有較高的保守性,因其在肉雞脾臟中的表達水平最高,故推測CMTR2基因是參與機體免疫相關的關鍵基因。
關鍵詞: 肉雞;CMTR2基因;生物信息學;表達譜;機體免疫
中圖分類號: S831.92? ? ? ? ? ? ? ? ? ? ? ? ? ?文獻標志碼: A 文章編號:2095-1191(2020)08-1832-08
Molecular characterization and expression pattern of
CMTR2 gene in broiler
LI Xiu-ling, XU Xiang-hui, XING Jin-yi*
(School of Life Sciences, Linyi University, Linyi, Shandong? 276000, China)
Abstract:【Objective】The aim of this paper was to study the molecular characteristics and expression patterns of cap-specific mRNA (nucleoside-2'-O-)- methyltransferase 2(CMTR2) in broilers, so as to lay a foundation for further deve-lopment of CMTR2 gene structure and biological function. 【Method】RT-PCR was used to clone CMTR2 gene in Arbor Acres(AA) broilers. The bioinformatics analysis was carried out using DNAMAN, ProtParam, SOPMA, SWISS-MODEL, TMHMM 2.0 and SignalP 3.0 software, and the expression characteristics were analyzed by real-time fluorescence quantitative PCR. 【Result】The length of coding region(CDS) of CMTR2 gene in broiler was 2350 bp(accession number: MN296490), and its open reading frame(ORF) was 2292 bp, encoding 763 amino acid residues. The similarity of amino acid sequence between broiler CMTR2 and native chicken was 99.74%, only two amino acid residues were different. The phylogenetic relationship between broiler CMTR2 and Turkey CMTR2 amino acid sequence was the closest(95.94% similarity). The results showed that the molecular weight of CMTR2 protein was 86855.48 Da and the theoretical isoelectric point(PI) was 6.08. The half-life of CMTR2 protein expressed in mammalian reticulocytes in vitro was 30 h, that in yeast was more than 20 h, and that in Escherichiacoli was more than 10 h. The CMTR2 protein was an unstable hydrophilic protein with no signal peptide and transmembrane domain. It contained 61 phosphorylation sites, 15 disulfide bonds, 1 N-glycosylation site and 8 O-glycosylation sites. Its secondary structure consisted of α-helix(49.93%), random coil(35.65%), extension chain(11.53%) and β-turn(2.88%), but there was no complete tertiary structure. The relative expression of CMTR2 gene in 42-day-old AA broilers was spleen>lung>chest muscle>abdominal fat>liver>kidney>leg muscle>heart. The relative expression level of CMTR2 in spleen was the highest, which was significantly higher than that in other tissues(P<0.05, the same below); the temporal and spatial expression trend of CMTR2 gene in broilers was the highest at 21-day-old and the lowest at 14-day-old. 【Conclusion】CMTR2 gene is highly conserved in different species, and its expression level is the highest in spleen. Therefore, it is speculated that CMTR2 gene is a key gene involved in immune system.
Key words: broiler; CMTR2 gene; bioinformatics; expression patterns; body immunity
Foundation item: National Natural Science Foundation of China(31372333);? Shandong Natural Science Foundation(ZR2017LC018)
0 引言
【研究意義】加帽甲基轉移酶[Cap-specific mRNA (nucleoside-2'-O-)-methyltransferase 2,CMTR2]也稱FTSJD1,分布在人類的細胞核和細胞質中,可催化重組RNA的核糖甲基化,對基因表達有直接影響,包括增強RNA穩定性、剪接、核質轉運和翻譯起始,以及促進細胞核與細胞質帽結合蛋白的相互作用等(Schroeder et al.,2004;Werner et al.,2011;Trotman and Schoenberg,2019)。雞是重要的模式動物之一,常作為研究哺乳動物胚胎發育機制的理想材料(張月娟,2004),在生物醫學研究領域發揮著重要作用,且易于飼養和便于手術操作及觀察。因此,研究雞CMTR2基因分子特征及其表達譜,可為進一步探究CMTR2基因的生物學功能積累基礎資料。【前人研究進展】甲基轉移酶(Methyltransferase)是動物機體內普遍存在的一種重要轉移酶,能調控DNA甲基化反應,從而調節染色質結構和基因表達(蘇玉等,2009;Du et al.,2015;Jensen et al.,2019)。CMTR2是一類重要的甲基轉移酶,由CMTR2基因編碼,其結構包括具有催化RFM(Rossmann-fold MTase)結構域的氨基端和具有非催化RFM結構域的羧基端。CMTR2的催化結構域能對mRNA帽子結構進行識別,使甲基供體與帽寡核苷酸結合形成配合物,從而促進甲基化反應(Werner et al.,2011;Smietanski et al.,2014)。人類CMTR2基因定位于16號染色體的q22.2位置上(Werner et al.,2011),而小鼠CMTR2基因定位在8號染色體的8D3位置上(Gaudet et al.,2011)。轉錄組測序結果表明,CMTR2基因能在多種組織中表達(Werner et al.,2011;Fagerberg et al.,2014;Yue et al.,2014);近期還在肺腺癌和鱗狀細胞瘤的相關研究中發現CMTR2基因會發生顯著突變,且因移碼突變而富集,故推測CMTR2基因是引起此類疾病的誘因之一(Campbell et al.,2016)。此外,CMTR2基因變異與妊娠糖尿病存在顯著相關性,與2型糖尿病不相關(Huerta-Chagoya et al.,2015),但CMTR2基因的生物學功能及調控機制至今尚未明確。【本研究切入點】目前,有關CMTR2基因的研究報道較少,且主要集中在酵母、病毒、小鼠及人類等方面(Saha et al.,1999;Egloff et al.,2002;Werner et al.,2011;Smietanski et al.,2014;Campbell et al.,2016),而針對雞CMTR2基因的研究尚無報道。【擬解決的關鍵問題】克隆肉雞CMTR2基因及進行生物信息學分析,并以實時熒光定量PCR檢測分析其表達模式,為后續開展雞CMTR2基因結構及生物學功能研究打下基礎。
1 材料與方法
1. 1 試驗材料
從山東新希望六和集團有限公司臨沂種雞場選購100羽健康狀況良好的1日齡艾拔益加(AA)肉雞,按AA肉雞生產性能標準進行正常飼養管理和免疫接種,分別于1、7、14、21、28、35和42日齡時挑選形體大小相似的6羽AA肉雞,禁食12 h后屠宰,采集肝臟、心臟、肺臟、腿肌、腎臟、脾臟、胸肌及腹脂等組織樣品,以PBS進行沖洗,置于無菌冷凍管中,經液氮速凍后轉移至-80 ℃冰箱保存備用。TRIzol(Invitrogen)和DNA凝膠回收試劑盒購自天根生化科技(北京)有限公司;M-MVL反轉錄酶購自Promega公司;質粒提取試劑盒購自天根生化科技(北京)有限公司;pMD19-T載體和LA Taq DNA酶購自TaKaRa公司;Hind III和EcoR I購自賽默飛世爾科技(中國)有限公司;2×SYBR Green Fast qPCR Mix購自寶日醫生物技術(北京)有限公司。主要儀器設備:梯度PCR儀(Eppendorf)、冷凍離心機(Eppendorf)、凝膠成像體系(Bio-Rad)、高低溫振蕩培養箱(JeioTech)及超低溫冰箱(Haier)等。
1. 2 總RNA提取及cDNA合成
采用TRIzol法提取各組織樣品總RNA,以核酸蛋白儀(Bio-Rad)檢測其OD260/OD280比值(1.8~2.1);然后采用M-MVL逆轉錄酶反轉錄合成cDNA。
1. 3 PCR引物設計及PCR擴增
根據GenBank已公布的原雞CMTR2基因序列(XM_004944112),使用Primer Premier 6.0設計CMTR2基因擴增引物(表1)。以42日齡AA肉雞的肝臟組織cDNA為模板,采用引物CMTR2-F和CMTR2-R進行PCR擴增。PCR反應體系25.0 μL:10×LA Buffer 2.5 μL,dNTP 2.0 μL,上、下游引物各0.4 μL,LA Taq DNA酶0.2 μL,cDNA模板1.0 μL,滅菌三蒸水18.5 μL。擴增程序:95 ℃預變性5 min;94 ℃ 30 s,59 ℃ 40 s,72 ℃ 150 s,進行35個循環;72 ℃延伸10 min。以1.2%瓊脂糖凝膠電泳檢測PCR擴增產物。
1. 4 肉雞CMTR2基因測序分析
采用DNA凝膠回收試劑盒對目的條帶進行純化回收,然后連接至pMD18-T載體上,并轉化大腸桿菌DH5α感受態細胞,經37 ℃過夜培養后挑取白色菌落接種至LB培養基上,搖床培養16 h,收集菌液進行PCR檢測。取目標菌株提取重組質粒,以Hind III和EcoR I進行雙酶切鑒定,并將陽性重組質粒送至生工生物工程(上海)股份有限公司測序。
1. 5 肉雞CMTR2基因生物信息學分析
利用NCBI網站中的ORF Finder搜尋肉雞CMTR2基因開放閱讀框(ORF);使用DNAMAN進行肉雞CMTR2基因核苷酸序列及其推導氨基酸序列比對分析,以MEGA 5.0中的鄰接法(Neighbor-joining,NJ)構建系統發育進化樹;運用ProtParam預測肉雞CMTR2蛋白理化性質;采用SOPMA和SWISS-MODEL分別預測肉雞CMTR2蛋白二、三級結構;使用TMHMM 2.0預測CMTR2蛋白跨膜域、PSORT II Prediction預測CMTR2蛋白亞細胞定位、SignalP 3.0預測CMTR2蛋白信號肽;并以NetPhos 3.1預測CMTR2蛋白磷酸化位點、SCRATCH Protein Prediction分析CMTR2蛋白二硫鍵、NetNGlyc 1.0預測CMTR2蛋白N-糖基化位點及YinOYang 1.2預測CMTR2蛋白O-糖基化位點。
1. 6 肉雞CMTR2基因表達分析
對42日齡AA肉雞的肝臟、心臟、肺臟、腿肌、腎臟、脾臟、胸肌及腹脂等組織進行CMTR2基因表達譜分析,以1、7、14、21、28、35和42日齡AA肉雞肝臟組織為對照模板,分析不同發育階段CMTR2基因的表達水平。以CMTR2-exp-F和CMTR2-exp-R為目標基因擴增引物、β-actin為內參基因(表1),采用SYBR Green I染料法進行實時熒光定量PCR檢測。反應體系25.0 μL:10.0 mol/L的上、下游引物各1.0 μL,cDNA模板(10倍稀釋)2.0 μL,2×SYBR Fast qPCR Mix 12.5 μL,以無菌超純水補足至25.0 μL。擴增程序:95 ℃預變性10 min;95 ℃ 20 s,55/58 ℃ 30 s,72 ℃ 20 s,進行40個循環。每個樣品進行3次重復,并通過2-△△Ct法計算CMTR2基因的相對表達量。
2 結果與分析
2. 1 肉雞CMTR2基因克隆及測序分析結果
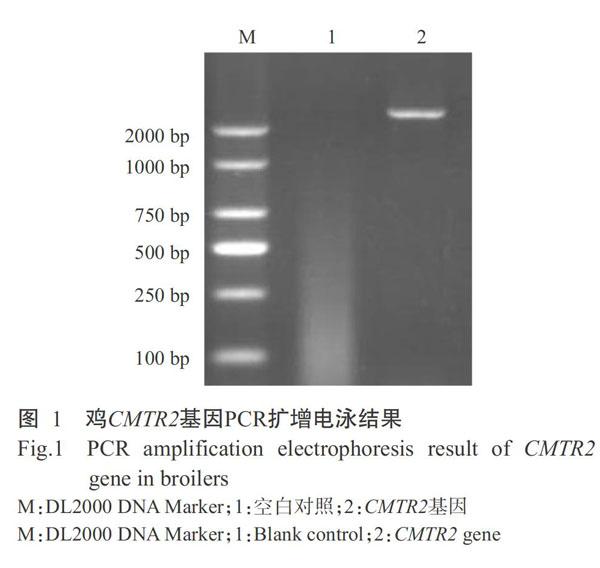
以肝臟cDNA為模板進行PCR擴增,PCR擴增產物經1.2%瓊脂糖凝膠電泳檢測,發現在2350 bp附近出現1條特異性條帶(圖1),與預期結果相符。經測序分析及序列拼接后,確認肉雞CMTR2基因編碼區(CDS)序列全長為2350 bp(登錄號MN296490),其ORF為2292 bp,編碼763個氨基酸殘基。肉雞CMTR2基因核苷酸序列與原雞CMTR2基因核苷酸序列(XM_004944112)的相似性為99.66%,僅有7 bp的差異;肉雞CMTR2氨基酸序列與原雞CMTR2氨基酸序列的相似性為99.74%,只存在2個氨基酸殘基差異,表明已成功克隆獲得肉雞CMTR2基因。
2. 2 肉雞CMTR2氨基酸序列比對及遺傳進化分析結果
由表2可知,肉雞CMTR2氨基酸序列與火雞CMTR2氨基酸序列的相似性最高,與鳥類CMTR2氨基酸序列的相似性均在80.00%以上,而與斑馬魚的相似性最低,為44.21%,說明CMTR2基因在物種間具有一定的保守性。基于CMTR2氨基酸序列相似性,采用鄰接法構建系統發育進化樹,結果顯示肉雞與火雞和日本鵪鶉聚為一類(圖2),即肉雞與火雞和日本鵪鶉的親緣關系較近,與氨基酸序列比對分析結果一致。
2. 3 肉雞CMTR2蛋白理化性質及其結構預測結果
肉雞CMTR2蛋白分子量為86855.48 Da,理論等電點(pI)為6.08;包含82個正電荷氨基酸殘基[天冬氨酸(Asp)+谷氨酸(Glu)]和67個負電荷酸殘基[精氨酸(Arg)+賴氨酸(Lys)];其消光系數為93580。肉雞CMTR2蛋白在哺乳動物網織紅細胞體外表達的半衰期為30 h,在酵母體內表達的半衰期大于20 h,在大腸桿菌體內表達的半衰期大于10 h;其不穩定系數為42.33,屬于不穩定蛋白。肉雞CMTR2蛋白脂肪指數為87.77,總平均親水性為-0.115,屬于親水性蛋白;其亞細胞定位預測結果表明主要分布在細胞質(26.1%)、分泌系統小泡(21.7%)、細胞核(13.0%)、線粒體(13.0%)、質膜(8.7%)、高爾基體(4.3%)、細胞骨架(4.3%)、空泡(4.3%)和內質網(4.3%)。
肉雞CMTR2蛋白信號肽及跨膜結構預測結果(圖3和圖4)表明,不存在信號肽和跨膜區域,不屬于分泌蛋白和跨膜蛋白;蛋白磷酸化預測結果顯示,肉雞CMTR2蛋白包含61個磷酸化位點(圖5),分別為38個絲氨酸(Ser)磷酸化位點、18個蘇氨酸(Thr)磷酸化位點和5個酪氨酸(Tyr)磷酸化位點,均大于閾值0.50,可作為蛋白激酶磷酸化位點。肉雞CMTR2氨基酸序列含有36個半胱氨酸(Cys),即預測CMTR2蛋白中有15個二硫鍵。肉雞CMTR2蛋白存在1個N-糖基化位點,位于第181位氨基酸處(圖6),其潛力值為0.6668(大于閾值0.50);而在第224、305、306、311、532、671、758和760位氨基酸處有8個O-糖基化位點(圖7),其潛力值分別為0.6450、0.5142、0.5131、0.5755、0.5118、0.5442、0.5606和0.5552(大于閾值0.50)。
肉雞CMTR2蛋白二級結構由α-螺旋(49.93%)、無規則卷曲(35.65%)、延伸鏈(11.53%)和β-轉角(2.88%)組成(圖8)。肉雞CMTR2蛋白三級結構預測結果與SWISS-MODEL數據庫模板4n49只有22.48%的氨基酸序列吻合度,故無法獲得其完整的三級結構,僅得到部分三級結構模型(圖9)。
2. 4 肉雞CMTR2基因表達分析結果
實時熒光定量PCR檢測結果顯示,CMTR2基因在42日齡AA肉雞8種組織中的相對表達量排序為脾臟>肺臟>胸肌>腹脂>肝臟>腎臟>腿肌>心臟,以在脾臟中的相對表達量最高,顯著高于在其他組織中的相對表達量(P<0.05,下同),而在心臟中的相對表達量顯著低于在其他組織中的相對表達量(圖10-A)。肉雞CMTR2基因的時空表達變化趨勢顯示,在21日齡時的相對表達量最高,極顯著高于其他日齡;在14日齡的相對表達量最低,而在28~42日齡的相對表達量呈逐漸上升趨勢(圖10-B)。
3 討論
CMTR2能催化多種物質甲基化,在基因表達調控過程中發揮重要作用,進而調節動物的生長發育,但至今其具體作用機制尚不明確。本研究成功克隆獲得肉雞CMTR2基因,其CDS序列全長為2350 bp,編碼763個氨基酸,與GenBank已公布的原雞CMTR2基因核苷酸序列僅有7 bp的差異;肉雞CMTR2氨基酸序列與原雞CMTR2氨基酸序列僅有2個氨基酸殘基差異,其余堿基突變均為同義突變。肉雞CMTR2氨基酸序列與火雞CMTR2氨基酸序列的相似性高達95.94%,且肉雞、火雞、鵪鶉、綠頭鴨和鴯鹋的CMTR2氨基酸序列均為763個氨基酸殘基,表明CMTR2基因在物種間高度保守。肉雞CMTR2氨基酸序列與斑馬魚CMTR2氨基酸序列的相似性最低,說明CMTR2基因在從魚類到鳥類經歷了較強的凈化選擇,與系統發育進化樹分析結果一致。
二硫鍵在蛋白分子中的主要作用是穩定其空間結構(田悅和杜軍保,2007)。肉雞CMTR2蛋白含有36個半胱氨酸(Cys)、15個二硫鍵,說明該蛋白抗外界因素影響的穩定性較高,且CMTR2蛋白穩定性與其半衰期成正比,即半衰期長則蛋白穩定性相應提高(賈浩等,2011)。本研究結果表明,肉雞CMTR2蛋白在哺乳動物網狀紅細胞體外表達的半衰期為30 h,半衰期較長,但屬于不穩定蛋白,可能與不同物種的生理功能有關。信號肽能被內質網膜上的受體識別(鄭斌和詹希美,2005)。本研究的蛋白信號肽預測結果顯示,肉雞CMTR2蛋白無信號肽,說明該蛋白不屬于分泌蛋白,而在胞內發揮作用,且無跨膜區域。因此,確定肉雞CMTR2蛋白是胞內蛋白。
磷酸化修飾和糖基化修飾是機體中最常見、最重要的蛋白翻譯后修飾方式,參與并調控生物機體的許多生命活動(姜錚等,2009;李軍等,2009;楊妍梅等,2019)。其中,蛋白磷酸化在細胞信號轉導過程中發揮重要作用(姜錚等,2009)。肉雞CMTR2蛋白包含61個潛在的磷酸化位點,能活化CMTR2蛋白,說明該蛋白具有較高的生物活性。蛋白糖基化能增強其穩定性(李軍等,2009)。肉雞CMTR2蛋白存在1個N-糖基化位點和8個O-糖基化位點,且其二級結構以α-螺旋和無規則卷曲為主,但這種結構對CMTR2蛋白催化功能的影響還需進一步探究。由于肉雞CMTR2蛋白三級結構僅有部分模型,且與模型中的氨基酸序列吻合度低于30.00%,故無法預測獲得完整的三級結構。CMTR2蛋白三級結構主要是由α-螺旋和無規則卷曲在空間上進一步盤曲、折疊而形成,與二級結構預測結果基本一致。
本研究結果表明,肉雞CMTR2基因在肝臟、心臟、肺臟、腿肌、腎臟、脾臟、胸肌及腹脂等8種組織中均有表達,其中,在脾臟中的相對表達量最高,在肺臟和胸肌中的相對表達量次之,與在豬(Yu et al.,2014)和大鼠(Yue et al.,2014)上的研究結果基本一致。此外,CMTR2基因在人類胸腺和睪丸(Fagerberg et al.,2014)、小鼠中樞神經系統和大腦(Yue et al.,2014)及綿羊子宮(Jiang et al.,2014)中的表達水平較高。可見,CMTR2基因對動物的免疫、繁殖、肌肉形成和生長發育均具有一定影響,但具體調控機制仍需進一步探究。
4 結論
CMTR2基因在不同物種中具有較高的保守性,因其在肉雞脾臟中的表達水平最高,故推測CMTR2基因是參與機體免疫相關的關鍵基因。
參考文獻:
賈浩,張小白,宋曉峰. 2011. 人類胞內蛋白半衰期與其亞細胞定位的相關性研究[J]. 計算機與應用化學,28(4):411-414. [Jia H,Zhang X B,Song X F. 2011. Relationship between intracellular proteins half-life and subcellular localization in human cells[J]. Computer and Applied Chemistry,28(4):411-414.]
姜錚,王芳,何湘,劉大偉,陳宣男,趙紅慶,黃留玉,袁靜. 2009. 蛋白質磷酸化修飾的研究進展[J]. 生物技術通訊,20(2):233-237. [Jiang Z,Wang F,He X,Liu D W,Chen X N,Zhao H Q,Huang L Y,Yuan J. 2009. Progress on protein/peptide phosphorylation[J]. Letters in Biotechnology,20(2):233-237.]
李軍,杜鑫,Hosseini Moghaddam S H,陳玉銀. 2009. 蛋白質糖基化修飾研究進展[J]. 科技通報,25(6):773-778. [Li J,Du X,Hosseini M S H,Chen Y Y. 2009. The research progress in protein glycosylation modification[J]. Bulletin of Science and Technology,25(6):773-778.]
蘇玉,王溪,朱衛國. 2009. DNA甲基轉移酶的表達調控及主要生物學功能[J]. 遺傳,31(11):1087-1093. [Su Y,Wang X,Zhu W G. 2009. DNA methyltransferases:The role in regulation of gene expression and biological processes[J]. Heredity (Beijing),31(11):1087-1093.]
田悅,杜軍保. 2007. 二硫鍵和巰基在蛋白質結構功能中的作用及分析方法[J]. 實用兒科臨床雜志,22(19):1499-1501. [Tian Y,Du J B. 2007. Effect of disulfide bond and mercap to group on structure and function of protein and analytical method[J]. Chinese Journal of Applied Clinical Pediatrics,22(19):1499-1501.]
楊妍梅,李玉,覃圣,蔣劉芽,趙金潭,陸曉東,熊杰,陳偉基,扎西英派. 2019. 靜寧雞PPARα基因克隆與生物信息學分析[J]. 江蘇農業學報,35(2):370-377. [Yang Y M,Li Y,Qin S,Jiang L Y,Zhao J T,Lu X D,Xiong J,Chen W J,Zhaxi Y P. 2019. Cloning and bioinformatic analysis of PPARα gene in Jingning chicken[J]. Jiangsu Journal of Agricultural Sciences,35(2):370-377.]
張月娟. 2004. 模式動物雞和果蠅的心臟發育研究[D]. 長沙:湖南師范大學. [Zhang Y J. 2004. Research of the heart development in chick and Drosophila[D]. Changsha:Hunan Normal University.]
鄭斌,詹希美. 2005. 信號肽序列及其在蛋白質表達中的應用[J]. 生物技術通訊,16(3):296-298. [Zheng B,Zhan X M. 2005. Signal peptide sequence and its application in protein expression[J]. Letters in Biotechnology,16(3):296-298.]
Campbell J D,Alexandrov A,Kim J,Wala J,Berger A H,Peda-mallu C S,Shukla S A,Guo G W,Brooks A N,Murray B A,Imielinski M,Hu X,Ling S Y,Akbani R,Rosenberg M,Cibulskis C,Ramachandran A,Collisson E A,Kwiatkowski D J,Lawrence M S,Weinstein J N,Verhaak R G,Wu C J,Hammerman P S,Cherniack A D,Getz G,Cancer Genome Atlas Research Network,Artyomov M N,Schreiber R,Govindan R,Meyerson M. 2016. Distinct patterns of somatic genome alterations in lung adenocarcinomas and squamous cell carcinomas[J]. Nature Genetics,48(6):607-616.
Du J M,Johnson L M,Jacobsen S E,Patel D J. 2015. DNA methylation pathways and their crosstalk with histone methylation[J]. Nature Reviews. Molecular Cell Biology,16(9):519-532.
Egloff M P,Benarroch D,Selisko B,Romette J L,Canard B. 2002. An RNA cap(nucleoside-2'-O-)-methyltransferase in the flavivirus RNA polymerase NS5:Crystal structure and functional characterization[J]. The EMBO Journal,21(11):2757-2768.
Fagerberg L,Hallstr?m B M,Oksvold P,Kampf C,Djurei-novic D,Odeberg J,Habuka M,Tahmasebpoor S,Da-nielsson A,Edlund K,Asplund A,Sj?stedt E,Lundberg E,Szigyarto C A K,Skogs M,Takanen J O,Berling H,Tegel H,Mulder J,Nilsson P,Schwenk J M,Lindskog C,Danielsson F,Mardinoglu A,Sivertsson A,von Feilitzen K,Forsberg M,Zwahlen M,Olsson I,Navani S,Huss M,Nielsen J,Ponten F,Uhlén M. 2014. Analysis of the human tissue-specific expression by genome-wide integration of transcriptomics and antibody-based proteomics[J]. Molecular & Cellular Proteomics,13(2):397-406.
Gaudet P,Livstone M S,Lewis S E,Thomas P D. 2011. Phylogenetic-based propagation of functional annotations within the gene ontology consortium[J]. Briefings in Bio-informatics,12(5):449-462.
Huerta-Chagoya A,Vázquez-Cárdenas P,Moreno-Macías H,Tapia-Maruri L,Rodríguez-Guillén R,López-Vite E,García-Escalante G,Escobedo-Aguirre F,Parra-Covarrubias A,Cordero-Brie?o R,Manzo-Carrillo L,Zacarías-Castillo R,Vargas-García C,Aguilar-Salinas C,Tusié-Luna T. 2015. Genetic determinants for gestational diabetes mellitus and related metabolic traits in Mexican women[J]. PLoS One,10(5):e0126408.
Jensen T ?,Tellgren-Roth C,Redl S,Maury J,Jacobsen S A B,Pedersen L E,Nielsen A T. 2019. Genome-wide systematic identification of methyltransferase recognition and modification patterns[J]. Nature Communications,10(1):3311.
Jiang Y,Xie M,Chen W B,Talbot R,Maddox J F,Faraut T,Wu C H,Muzny D M,Li Y X,Zhang W G,Stanton J A,Brauning R,Barris W C,Hourlier T,Aken B L,Searle S M J,Adelson D L,Bian C,Cam G R,Chen Y L,Cheng S F,DeSilva U,Dixen K,Dong Y,Fan G Y,Franklin I R,Fu S Y,Guan R,Highland M A,Holder M E,Huang G D,Ingham A B,Jhangiani S N,Kalra D,Kovar C L,Lee S L,Liu W Q,Liu X,Lu C X,Lü T,Mathew T,McWilliam S,Menzies M,Pan S K,Robelin D,Servin B,Townley D,Wang W L,Wei B,White S N,Yang X H,Ye C,Yue Y J,Zeng P,Zhou Q,Hansen J B,Kristensen K,Gibbs R A,Flicek P,Warkup C C,Jones H E,Oddy V H,Nicholas F W,McEwan J C,Kijas J,Wang J,Worley K C,Archibald A L,Cockett N,Xu X,Wang W,Dalrymple B P. 2014. The sheep genome illuminates biology of the rumen and lipid metabolism[J]. Science,344(6188):1168-1173.
Saha N,Schwer B,Shuman S. 1999. Characterization of human,Schizosaccharomyces pombe,and Candida albicans mRNA cap methyltransferases and complete replacement of the yeast capping apparatus by mammalian enzymes[J]. The Journal of Biological Chemistry,274(23):16553-16562.
Schroeder S C,Zorio D A R,Schwer B,Shuman S,Bentley D. 2004. A function of yeast mRNA cap methyltransfera-se,Abd1,in transcription by RNA polymerase II[J]. Molecular Cell,13(3):377-387.
Smietanski M,Werner M,Purta E,Kaminska K H,Stepinski J,Darzynkiewicz E,Nowotny M,Bujnicki J M. 2014. Structural analysis of human 2'-O-ribose methyltransfera-ses involved in mRNA cap structure formation[J]. Nature Communications,5:3004. doi:10.1038/ncomms4004.
Trotman J B,Schoenberg D R. 2019. A recap of RNA recapping[J]. Wiley Interdisciplinary Reviews. RNA,10(1):e1504.
Werner M,Purta E,Kaminska K H,Cymerman I A,Campbell D A,Mittra B,Zamudio J R,Sturm N R,Jaworski J,Bujnicki J M. 2011. 2'-O-ribose methylation of cap2 in human:Function and evolution in a horizontally mobile family[J]. Nucleic Acids Research,39(11):4756-4768.
Yu Y,Fuscoe J C,Zhao C,Guo C,Jia M W,Qing T,Bannon D I,Lancashire L,Bao W J,Du T T,Luo H,Su Z Q,Jones W D,Moland C L,Branham W S,Qian F,Ning B T,Li Y,Hong H X,Guo L,Mei N,Shi T L,Wang K Y,Wolfinger R D,Nikolsky Y,Walker S J,Duerksen-Hughes P,Mason C E,Tong W D,Thierry-Mieg J,Thierry-Mieg D,Shi L M,Wang C. 2014. A rat RNA-Seq transcriptomic BodyMap across 11 organs and 4 developmental stages[J]. Nature Communications,5:3230. doi:10. 1038/ncomms4230.
Yue F,Cheng Y,Breschi A,Vierstra J,Wu W S,Ryba T,Sandstrom R,Ma Z H,Davis C,Pope B D,Shen Y,Pervouchine D D,Djebali S,Thurman R E,Kaul R,Rynes E,Kirilusha A,Marinov G K,Williams B A,Trout D,Amrhein H,Fisher-Aylor K,Antoshechkin I,DeSalvo G,See L H,Fastuca M,Drenkow J,Zaleski C,Dobin A,Prie-to P,Lagarde J,Bussotti G,Tanzer A,Denas O,Li K,Bender M A,Zhang M H,Byron R,Groudine M T,McCleary D,Pham L,Ye Z,Kuan S,Edsall L,Wu Y C,Rasmussen M D,Bansal M S,Kellis M,Keller C A,Morrissey C S,Mishra T,Jain D,Dogan N,Harris R S,Cayting P,Kawli T,Boyle A P,Euskirchen G,Kundaje A,Lin S,Lin Y,Jansen C,Malladi V S,Cline M S,Erickson D T,Kirkup V M,Learned K,Sloan C A,Rosenbloom K R,de Sousa B L,Beal K,Pignatelli M,Flicek P,Lian J,Kahveci T,Lee D,Kent W J,Santos M R,Herrero J,Notredame C,Johnson A,Vong S,Lee K,Bates D,Neri F,Diegel M,Canfield T,Sabo P J,Wilken M S,Reh T A,Giste E,Shafer A,Kutyavin T,Haugen E,Dunn D,Reynolds A P,Neph S,Humbert R,Hansen R S,de Bruijn M,Selleri L,Rudensky A,Josefowicz S,Samstein R,Eichler E E,Orkin S H,Levasseur D,Papayannopoulou T,Chang K H,Skoultchi A,Gosh S,Disteche C,Treuting P,Wang Y L,Weiss M J,Blobel G A,Cao X Y,Zhong S,Wang T,Good P J,Lowdon R F,Adams L B,Zhou X Q,Pazin M J,Feingold E A,Wold B,Taylor J,Mortazavi A,Weissman S M,Stamatoyannopoulos J A,Snyder M P,Guigo R,Gingeras T R,Gilbert D M,Hardison R C,Beer M A,Ren B. 2014. A comparative encyclopedia of DNA elements in the mouse genome[J]. Nature,515(7527):355-364.
(責任編輯 蘭宗寶)
收稿日期: 2019-09-07
基金項目:國家自然科學基金項目(31372333);山東省自然科學基金項目(ZR2017LC018)
作者簡介:*為通訊作者,邢晉祎(1968-),博士,教授,主要從事動物遺傳育種研究工作,E-mail:xingjinyi@lyu.edu.cn。李修嶺(1972-),教授,主要從事動物分子遺傳進化研究工作,E-mail:379124952@qq.com

