CRISPR/Cas9技術介導豬基因組單堿基編輯效率的研究
張婷婷 陳濤 李燕莉 楊漫漫 魏強 王然 李林 李勇
摘要:利用國際上最新的兩種基于CRISPR/Cas9的BE3(Cytosine base editor,CBE)和ABE7.10(Adenine base editor,ABE)單堿基修飾技術對豬基因組靶基因位點進行編輯效率的分析研究。設計、合成并構建4個豬基因組靶基因位點gRNA表達載體,分別與CBE或ABE共轉染PK15細胞,繼續培養48 h,結合二代測序技術測定單堿基替換效率和indels發生率。結果表明,單堿基編輯系統BE3和ABE7.10對豬基因組靶位點堿基修飾的活性編輯窗口主要分別為5個核苷酸和4個核苷酸;兩套單堿基編輯系統主要對編輯窗口內的目標堿基進行單堿基轉換而非indel;兩套單堿基編輯系統對豬基因組堿基置換有一定偏好性,其中CMAH、MC1R(1-2)、MC1R(2-1)基因編輯窗口內C→T的效率分別為2.2%、0.4%和1.3%;豬GGTA、MSTN-2窗口內A→G的效率分別為1.4%、1.4%。表明單堿基編輯系統BE3和ABE7.10均能夠對豬細胞的基因組靶位點序列進行有效的單堿基置換。
關鍵詞:豬;單堿基編輯;CRISPR/Cas9;BE3;ABE7.10
中圖分類號:Q812? ? ? ? ?文獻標識碼:A
文章編號:0439-8114(2020)18-0143-07
DOI:10.14088/j.cnki.issn0439-8114.2020.18.029 開
Programmable base editing efficiency study of CRISPR/Cas9-guided
DNA base editors in pig genome
ZHANG Ting-ting1,2, CHEN Tao2,3, LI Yan-li1,2, YANG Man-man2,3,
WEI Qiang2,3, WANG Ran2,3,LI Lin2,3, LI Yong1,2,3
(1. BGI-Shenzhen Sanshengyuan Technology Co., Ltd., Shenzhen? 518000, Guangdong, China;
2. BGI-Agricultural Application Research Institute, Shenzhen? 518000,Guangdong,China;
3.Shenzhen Engineering Laboratory for Genomics-Assisted Animal Breeding, Shenzhen? 518000, Guangdong,China)
Abstract: The CRISPR/Cas9-based BE3(Cytosine base editor,CBE) and ABE7.10(Adenine base editor,ABE) were used to analyze and study the editing efficiency of target gene loci in pig genome. Four porcine genomic target gene loci gRNA expression vectors were designed, synthesized and constructed, which were co-transfected into PK15 cells with CBE or ABE, respectively. The PK15 cells were further cultured for 48 h, and single-base replacement efficiency and indels incidence were determined by second-generation sequencing technology. The results showed that the active editing Windows of the single base editing system BE3 and ABE7.10 were 5 nucleotides and 4 nucleotides, respectively; Both BE3 and ABE7.10 were mainly contributed to single base conversion but not indel; The two sets of single-base editing systems showed certain preference for base replacement in pig genome. In the CMAH, MC1R(1-2) and MC1R(2-1) gene editing Windows, the efficiency of C→T was 2.2%, 0.4% and 1.3%, respectively; The efficiency of A→G in pig GGTA and MSTN-2 was 1.4% and 1.4%, respectively. It is shown that both BE3 and ABE7.10 can perform effective single base substitutions for genomic target sequences in pig cells.
Key words: pig; single base editing; CRISPR/Cas9; BE3; ABE7.10
CRISPR/Cas9(Clustered regularly interspaced short palindromic repeats (CRISPR)-associated system,Cas9)系統作為第三代基因編輯工具已廣泛應用于各物種的基因修飾,包括小鼠[1]、豬[2]、牛[3]、羊[4]、水稻[5]、擬南芥[6]、煙草[7]、玉米[8]、小麥[9]等。該系統是在sgRNA的引導下Cas9核酸酶造成基因組雙鏈斷裂,斷裂后的基因組進行非同源末端修復(Non-homologous end joining,NHEJ)或同源末端修復(Homology-directed repair,HDR)來實現基因編輯。絕大部分DNA修復是通過NHEJ的方式進行,這種修復通常會產生大量的小片段隨機插入或缺失,抑或造成大片段缺失,進而導致基因的失活[10]。然而,在現實的農業和醫學研究領域中,導致農藝性狀或人類遺傳性疾病的原因則更多的是因點突變引起的功能獲得或缺失,如約2/3的人類遺傳疾病都是由單堿基突變造成,這種單堿基突變卻是常規CRISPR/Cas9系統介導雙鏈斷裂所不能完成的。盡管通過同源重組載體或ssDNA介導的HDR完成,但由于細胞內同源重組的發生概率極低[11],使得這類堿基替換方法的效率極低。
基于CRISPR/Cas9系統發展起來的單堿基替換技術較多。利用Cas9突變體(nCas9)分別與大鼠胞嘧啶脫氨酶(rAPOBEC)融合,開發出在DNA雙鏈不發生斷裂的情況下可以實現更加安全、高效、精準的單堿基編輯器(Cytosine base editor, CBE),而且其效率遠高于DSB引起的HDR修復方式[12]。Gaudelli等[13]把Escherichia coli來源的tRNA腺苷酸脫氨酶(TadA)與nCas9融合,經過多輪選擇和蛋白修飾后,開發出可以將腺嘌呤精準地轉化成鳥嘌呤的新型單堿基轉化系統(Adenine base editor, ABE)。且隨著生物技術的發展,形成了更多高效特異的單堿基替換系統[14,15]。目前,基于CRISPR/Cas9系統開發的單堿基替換系統BE3和ABE7.10已在小鼠[16]、斑馬魚[17]、人類干細胞[18]、水稻[19]、小麥[20]等物種中得到廣泛應用。
豬作為中國最為重要的農業動物,因其與人在解剖、生理、病理以及基因組序列方面高度的相似性,已成為人類相關疾病研究的理想試驗模型和異種器官移植模型。現已通過改造豬基因組開發了一系列的人類疾病模型豬,如內源性轉錄病毒敲除模型[21]、亨廷頓舞蹈癥模型[22]、動脈粥樣硬化模型[23]、杜氏營養不良模型等[24],以及伴隨單堿基替換系統而制備出的模擬人類點突變的疾病模型豬[25,26]。
鑒于單堿基替換系統技術所具備的應用價值,而該技術豬基因組編輯中卻沒有關于活性編輯框、單個堿基替換效率、堿基替換傾向性及脫靶等方面的報道。本研究選取了豬基因組中4個不同的基因作為靶位點,構建其對應的gRNA表達載體,以最新發展起來且應用廣泛的單堿基替換系統 BE3和ABE7.10為介質,并結合靶向基因組測序技術,評價豬基因組特定序列的堿基替換結果。同時,選用4條人源序列開展平行性陽性對照試驗,詳細對比分析了兩項基于CRISPR/Cas9技術的BE3和ABE7.10單堿基編輯技術在豬細胞基因組中的修飾效率和缺失、插入效率。本研究成果將為后續利用單堿基編輯系統在模型豬體內研究、治療人類單點突變疾病提供重要基礎。
1 材料與方法
1.1 材料
1.1.1 細胞及載體 PK15細胞購于美國模式培養物寄存庫(ATCC);293T細胞由深圳國家基因庫母嬰健康研究中心楊熹提供;CRR質粒由深圳華大三生園科技有限公司實驗室設計并合成;pCMV-ABE7.10質粒、pCMV-BE3質粒購于Addgene。
1.1.2 主要試劑 無內毒素質粒提取試劑盒購于Omega公司;Lipofectamine2000轉染試劑盒購于Invitrogen公司;MGIEasy DNA文庫快速制備試劑盒購于深圳華大智造科技有限公司。
1.2 方法
1.2.1 sgRNA設計及活性鑒定 4條陽性對照sgRNA-2TAT、sgRNA-HBG1、sgRNA-EMX1、sgRNA- HEK293T site3序列均來自于文獻[12]和[13]。設計并合成豬GGTA、MSTN、CMAH、MC1R、MYH7等5個基因的sgRNA序列,sgRNA序列信息見表1。sgRNA正向鏈及其互補鏈按照體積比1∶1變性退火(變性退火程序:95 ℃,10 min;25 ℃,30 min),形成帶有黏性末端的雙鏈核苷酸,與經BsaI酶切、膠回收純化后的CRR骨架載體連接。構建的CRR表達載體經過Sanger測序驗證連接正確后,無內毒素試劑盒提取質粒,用于轉染PK15細胞驗證剪切活性。
CRR-sgRNA表達質粒與pCMV-spCas9質粒共轉染,當PK15細胞匯合度50%時進行脂質體轉染,轉染48 h后分別收集細胞并提取基因組DNA(Genomic DNA,gDNA),PCR擴增sgRNA靶點附近序列,產物經T7程序變性退火后用T7核酸內切酶I酶切,sgRNA活性鑒定引物見表2,反應體系如下。
PCR反應體系:gDNA 0.1 μg,premix Ex Taq 10 μL,上下游引物(10 μmol/L)各0.4 μL,ddH2O補足體積至20 μL。PCR反應程序:98 ℃預變性1 min;98 ℃變性10 s,55~62 ℃(Tm)退火30 s,72 ℃延伸10 s,32個循環;再于72 ℃延伸2 min;4 ℃保存。
T7變性程序:95 ℃預變性10 min;以2.0 ℃/s自95 ℃降至85 ℃;以-0.3 ℃/s 自85 ℃降至25 ℃;25 ℃ 1 min。
T7酶切反應體系:PCR產物18 μL,NEB? ? ? Buffer2 2 μL,T7核酸內切酶I 0.2 μL。反應條件37 ℃,45 min。
1.2.2 細胞轉染 PK15細胞、293T細胞復蘇至24孔板,細胞匯合度達50%后進行轉染,轉染策略見表3。DNA轉染總量500 ng,單堿基替換載體和CRR-sgRNA表達載體轉染比例為3∶1。根據脂質體說明書進行轉染,轉染后細胞置于37 ℃、5%CO2培養箱中培養,轉染后24 h更換1次培養基。
1.2.3 靶向文庫構建及測序 轉染48 h后,收集細胞,提取基因組DNA,靶向擴增引物進行擴增。PCR反應體系:gDNA 5 ng,PrimeSTAR 10 μL,上下游引物(10 μmol/L)各0.4 μL,ddH2O補足體積至20 μL。
PCR反應程序:98 ℃預變性1 min;98 ℃變性10 s,55~60 ℃(Tm)退火30 s,72 ℃延伸10 s,30個循環;再于72 ℃延伸2 min;4 ℃保存。靶向擴增引物序列見表4。
取5 μL擴增產物于1.8%瓊脂糖凝膠中檢測,剩余產物純化后進行文庫構建。
使用Qubit? dsDNA HS Assay Kit檢測PCR回收產物濃度,均一化至60 ng/μL,總體積50 μL,使用MGIEasy DNA文庫快速制備試劑盒進行文庫制備。文庫環化產物使用Qsep100檢測片段長度,Qubit? ssDNA Assay Kit測定濃度,使用BGISEQ-500平臺進行PE100測序。
1.2.4 測序數據分析 數據使用SOAPnuke-1.5.0進行過濾,使用BWA-0.7.12進行序列比對,用Samtools1.7的mpileup模塊轉換格式后統計結果。
2 結果與分析
2.1 CRR-sgRNA表達載體構建及活性驗證結果
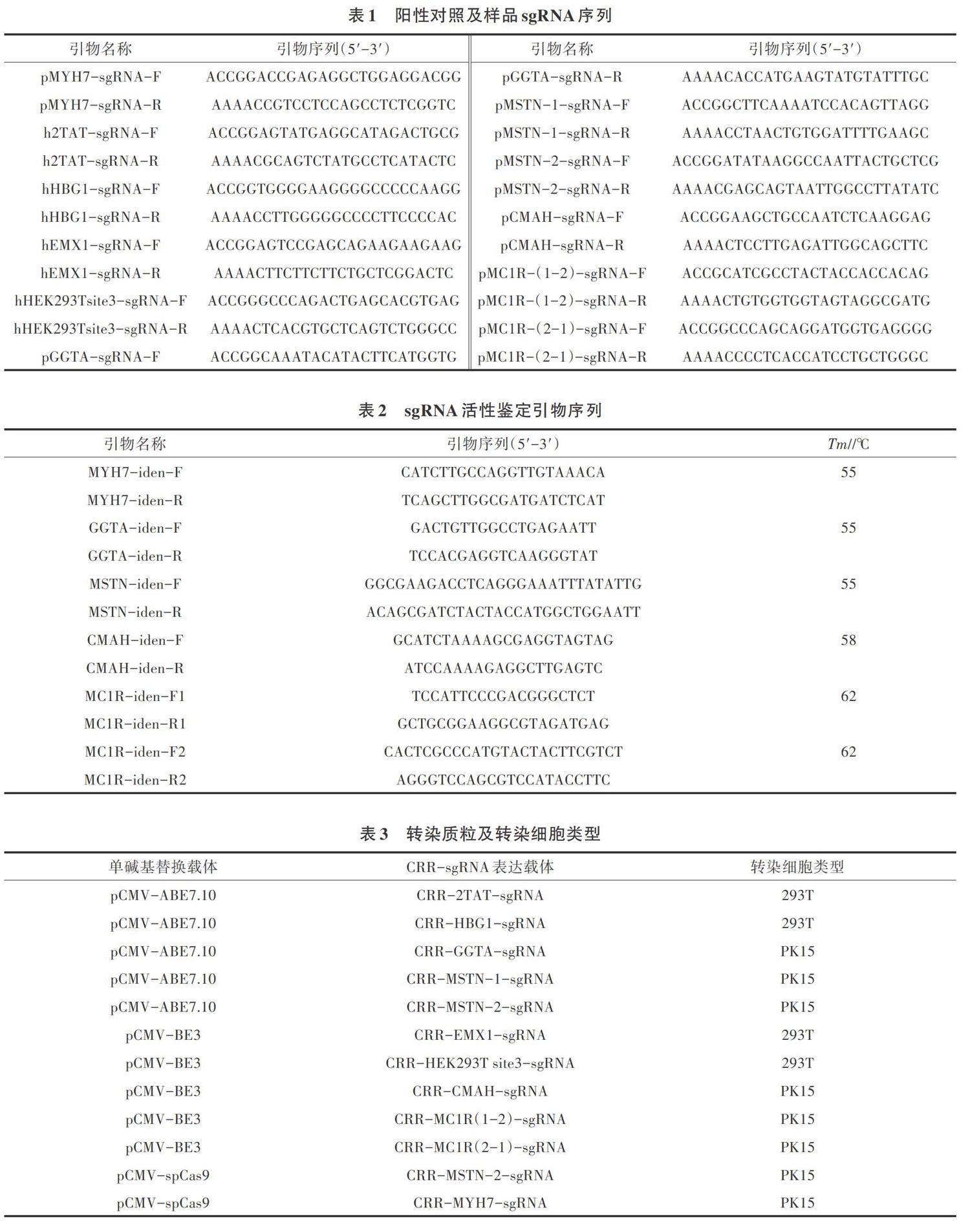
將豬靶基因GGTA、MSTN、CMAH、MC1R、MYH7相關sgRNA,陽性對照2TAT、HBG1、EMX1、HEK293T site3相關sgRNA,分別連入CRR骨架載體中,經Sanger測序驗證,表明sgRNA均成功連接到CRR骨架載體中(圖1)。
將構建成功的豬靶基因相關sgRNA表達載體與pCMV-spCas9表達載體共轉染至PK15細胞中,轉染48 h后提取基因組DNA,用鑒定引物擴增并進行T7酶切鑒定,活性鑒定結果表明,7條豬靶基因相關的sgRNA均有剪切活性,其中MSTN-1活性較弱(圖2)。
2.2 靶向文庫構建
靶向擴增引物分別擴增靶向位點及其附近序列,陽性對照及樣品平行擴增3份,電泳結果(圖3)顯示,目的片段大小正確,樣品目標條帶單一、特異性較好。
2.3 單堿基替換效率分析
BE3單堿基編輯系統中的胞嘧啶脫氨酶的酶活性窗口包含了5個核苷酸,一般是距離PAM序列最遠端的第4至第8個核苷酸[12]。對靶序列測序結果進行分析,結果(圖4A至圖4F)顯示,CMAH、MC1R(1-2)、MC1R(2-1)、陽性對照HEK293Tsite3的堿基替換主要發生在活性編輯窗口內,其中CMAHC5→T5效率為2.2%(圖4A),MC1R(1-2)C7→T7效率為0.4%(圖4B);MC1R(2-1)C7→T7效率為1.3%(圖4C),陽性對照HEK293T site3 C4→T4和C5→T5的效率分別為11%和10%(圖4E)。相比而言,另一個陽性對照EMX1基因窗口區堿基替換效率極低,C6→T6效率僅為0.02%(圖4D)。
ABE7.10單堿基編輯系統中的腺嘌呤脫氨酶的酶活性窗口包含了4個核苷酸,一般是距離PAM序列最遠端的第4至第7個核苷酸[13]。對靶序列測序結果進行分析,結果(圖5A至圖5F)顯示,樣品GGTA和MSTN-2、陽性對照2TAT和HBG1其堿基替換主要發生在活性編輯窗口內,其中GGTA A7→G7的效率為1.4%(圖5A),MSTN-2 A6→G6的效率為1.4%(圖5C),2TAT A5→G5效率為9.6%(圖5D),HBG1A7→G7的效率為1.2%(圖5E)。相比而言,MSTN-1的單堿基修飾效率極低,窗口區A→G效率僅為0.01%(圖5B)。
鑒于單堿基修飾系統極低的indels發生率,同時設計了常規CRISPR/Cas9的對照,并通過高通量測序檢測indels發生率,結果(圖4F、圖5F)顯示,除陽性對照HEK293T site3有2.2%的indels比例外,其他組都遠低于常規CRISPR/Cas9組,這說明單堿基編輯后的樣品主要發生堿基替換。
3 討論
單堿基修飾是近年來發展最為迅速的基因編輯技術之一,它整合了Cas9蛋白的靶向性和脫氨酶堿基替換修復的特點,為農業或生物醫學單點、多點特定堿基定向突變模型制備提供了高效的技術手段[20,27-29]。本研究利用BE3和ABE7.10兩套單堿基編輯系統對4個豬基因組靶位點進行單堿基修飾。結果表明,BE3和ABE7.10兩套單堿基編輯系統都能對豬靶基因進行有效的單堿基修飾,且堿基轉換主要發生在活性編輯窗口內,這與該基因編輯系統在其他物種中的報道類似。只不過在本研究中單堿基替換效率較低,4個豬靶基因區域堿基修飾最高比例僅有2.2%,略低于其他文獻報道中的轉換效率[12,13]。但是在同等試驗條件下,在人293T細胞中對4個基因受BE3和ABE7.10作用而發生單堿基修飾的效率也偏低,整體而言,以此作對照并不影響本研究關于活性編輯框、單個堿基替換效率、堿基替換傾向性在豬和人基因組中進行差異比較與分析的結果。此外,BE3-MC1R(1-2)位點、BE3-EMX1位點以及ABE7.10-MSTN-1未能發生單堿基修飾可能與gRNA活性有關,沒有加入單堿基編輯效率的數據分析之中。本研究發現,BE3系統堿基替換效率整體略高,ABE7.10的要低些。大量研究表明,體外基因編輯效率與細胞轉染效率高度正相關,無論是核轉染、電轉染或者通過病毒載體介導的轉染,都能實現高效的載體導入,從而獲得高效的基因編輯結果[30-32]。
CBE和ABE堿基編輯系統都能夠在不使DNA雙鏈斷裂的情況下,精準、高效地進行目標堿基的替換,精確地生成或者修復點突變。這在本研究中也有體現,以MYH7和MSTN為靶點測定了CRISPR/Cas9基因修飾發生的indel概率為陽性對照,試驗結果表明,經過BE3和ABE7.10單堿基編輯系統編輯后,除HEK293T site3有2.2%的indels比例外,其他組indels都在1.0%以下,編輯產物indels的發生率都比CRISPR/Cas9系統編輯產物indels的發生率低,結果與文獻報道的結果相符合[12,13]。此外,本研究中樣品以及陽性對照都出現了不同程度的編輯窗口以外的堿基轉換,結合在小鼠、豬等多個物種中進行的多項研究[16,25,26,33]顯示,CBE編輯系統用于堿基轉換時,除了目標堿基C-T轉換的同時還會伴隨著少量的C-A或C-G的轉換。鑒于CBE堿基編輯系統中所用的酶是突變體dCas9,一種缺乏雙鏈斷裂能力的核酸酶,可以排除Cas9斷裂雙鏈DNA后引起indels,推測是當編輯窗口內的堿基脫氨基以及堿基切除修復時,把目標堿基做轉換的同時引發了堿基的插入或缺失[27]。
在位點活性評價方面,與常規CRISPR/Cas9基因編輯系統依賴T7酶檢測不同,單堿基編輯系統主要通過高通量測序來檢測堿基替換比率。本研究發現采用靶向PCR建庫的方法可以較為高效地檢測低頻堿基變異,利用該方法最重要的是確保擴增片段的特異性和高保真性。此外,在建庫過程中,接頭連接時容易發生片段自連,從而產生非特異性條帶,但這些條帶在建庫后純化中可去除,不會影響后續的測序。最后,相比高通量測序一個反應上百GB的數據而言,靶向片段的測序數據極少,可以與其他類型的測序樣本進行pooling來降低檢測成本。
總之,本研究利用基于CRISPR/Cas9技術的BE3和ABE7.10兩套單堿基編輯系統對PK15細胞中4種不同基因靶位點進行了研究,探索這兩套堿基修飾系統對豬基因組堿基的編輯情況以及編輯效率。結果表明,BE3和ABE7.10系統都能在體外試驗中對豬基因組中靶基因產生一定的編輯效果,這將為科研工作者研究基因在豬細胞內的生物學功能、為創制人類單堿基突變基因病的小型豬動物模型提供重要生物技術基礎。
參考文獻:
[1] BEIL-WAGNER J,D?SSINGER G,SCHOBER K,et al. T cell-specific inactivation of mouse CD2 by CRISPR/Cas9[J]. Sci Rep,2016,6:21377.
[2] YIN Y J,HAO H Y,XU X B,et al. Generation of an MC3R knock-out pig by CRSPR/Cas9 combined with somatic cell nuclear transfer (SCNT) technology[J]. Lipids Health Dis,2019,18:122.
[3] IKEDA M,MATSUYAMA S,AKAGI S,et al. Correction of a disease mutation using CRISPR/Cas9-assisted genome editing in Japanese black cattle[J]. Sci Rep,2017,7:17827.
[4] VILARINO M,RASHID S T,SUCHY F P,et al. CRISPR/Cas9 microinjection in oocytes disables pancreas development in sheep[J]. Sci Rep,2017,7:17472.
[5] MA K,HAN J L,HAO Y,et al. An effective strategy to establish a male sterility mutant mini-library by CRISPR/Cas9-mediated knockout of anther-specific genes in rice[J]. J Genet Genomics,2019,46(5):273-275.
[6] RYDER P,MCHALE M,FORT A,et al. Generation of stable nulliplex autopolyploid lines of Arabidopsis thaliana using CRISPR/Cas9 genome editing[J]. Plant Cell Rep,2017,36(6):1005-1008.
[7] HIROHATA A,SATO I,KAINO V,et al. CRISPR/Cas9-mediated homologous recombination in tobacco[J]. Plant Cell Rep,2019,38(4):463-473.
[8] FENG C,SU H D,BAI H,et al. High-efficiency genome editing using a dmc1 promoter-controlled CRISPR/Cas9 system in maize[J]. Plant Biotechnol J,2018,16(11):1848-1857.
[9] LIANG Z,CHEN K L,LI T D,et al. Efficient DNA-free genome editing of bread wheat using CRISPR/Cas9 ribonucleoprotein complexes[J]. Nat Commun,2017,8:14261.
[10] OTA S,HISANO Y,IKAWA Y,et al. Multiple genome modifications by the CRISPR/Cas9 system in zebrafish[J]. Genes to cells,2014,19(7):555-564.
[11] LIU M J,REHMAN S,TANG X D,et al. Methodologies for improving HDR efficiency[J]. Front Genet,2019,9:691.
[12] KOMOR A C,KIM Y B,PACKER M S,et al. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage[J]. Nature,2016,533:420-424.
[13] GAUDELLI N M,KOMOR A C,REES H A,et al. Programmable base editing of A·T to G·C in genomic DNA without DNA cleavage[J]. Nature,2017,551:464-471.
[14] JIANG W,FENG S J,HUANG S S,et al. BE-PLUS: A new base editing tool with broadened editing window and enhanced fidelity[J]. Cell Res,2018,28(8):855-861.
[15] FAGAGNA DI F D,WELLER G R,DOHERTY A J,et al. The Gam protein of bacteriophage Mu is an orthologue of eukaryotic Ku[J]. EMBO Rep,2003,4(1):47-52.
[16] LIU Z,LU Z Y,YANG G,et al. Efficient generation of mouse models of human diseases via ABE- and BE-mediated base editing[J]. Nat Commun,2018,9(1):2338.
[17] ZHANG Y H,QIN W,LU X C,et al. Programmable base editing of zebrafish genome using a modified CRISPR-Cas9 system[J]. Nat Commun,2017,8(1):118.
[18] YANG J P,LI J Y,SUZUKI K,et al. Genetic enhancement in cultured human adult stem cells conferred by a single nucleotide recoding[J]. Cell Res,2017,27(9):1178-1181.
[19] YAN F,KUANG Y J,REN B,et al. Highly efficient A·T to G·C base editing by Cas9n-Guided tRNA adenosine deaminase in rice[J]. Mol Plant,2018,11(4):631-634.
[20] ZONG Y,WANG Y P,LI C,et al. Precise base editing in rice, wheat and maize with a Cas9-cytidine deaminase fusion[J]. Nat Biotechnol,2017,35(5):438-440.
[21] NIU D,WEI H J,LIN L,et al. Inactivation of porcine endogenous retrovirus in pigs using CRISPR-Cas9[J]. Science,2017,357(6357):1303-1307.
[22] YAN S,TU Z C,LIU Z M,et al. A huntingtin knockin pig model recapitulates features of selective neurodegeneration in Huntington's disease[J]. Cell,2018,173(4):989-1002.e13.
[23] HUANG L,HUA Z D,XIAO H W,et al. CRISPR/Cas9-mediated ApoE-/- and LDLR-/- double gene knockout in pigs elevates serum LDL-C and TC levels[J]. Oncotarget,2017,8(23):37751-37760.
[24] YU H H,ZHAO H,QING Y B,et al. Porcine zygote injection with Cas9/sgRNA results in DMD-Modified pig with muscle dystrophy[J]. Int J Mol Sci,2016,17(10):1668.
[25] LI Z F,DUAN X Y,AN X M,et al. Efficient RNA-guided base editing for disease modeling in pigs[J]. Cell Discov,2018,4:64.
[26] XIE J K,GE W K,LI N,et al. Efficient base editing for multiple genes and loci in pigs using base editors[J]. Nat Commun,2019,10(1):2852.
[27] NISHIDA K,ARAZOE T,YACHIE N,et al. Targeted nucleotide editing using hybrid prokaryotic and vertebrate adaptive immune systems[J]. Science,2016,353(6305):aaf8729.
[28] LU Y M,ZHU J K. Precise editing of a target base in the rice genome using a modified CRISPR/Cas9 system[J]. Mol Plant,2017,10(3):523-525.
[29] KIM K,RYU S M,KIM S T,et al. Highly efficient RNA-guided base editing in mouse embryos[J]. Nat Biotechnol,2017,35(5):435-437.
[30] CAO F,XIE X Y,GOLLAN T,et al. Comparison of gene-transfer efficiency in human embryonic stem cells[J]. Mol Imaging Biol,2010,12(1):15-24.
[31] ROSS J W,WHYTE J J,ZHAO J G,et al. Optimization of square-wave electroporation for transfection of porcine fetal fibroblasts[J]. Transgenic Res,2010,19(4):611-620.
[32] MAURISSE R,DE SEMIR D,EMAMEKHOO H,et al. Comparative transfection of DNA into primary and transformed mammalian cells from different lineages[J]. BMC Biotechnol,2010,10:9.
[33] LIANG P P,SUN H W,ZHANG X Y,et al. Effective and precise adenine base editing in mouse zygotes[J]. Protein & cell,2018,9(9):808-813.

