甘蔗近緣野生種蔗茅EfGRAS基因克隆及表達(dá)分析
沈先岳 谷書杰 謝林艷 錢禛鋒 曾丹 何麗蓮 李富生


摘? 要:為深入挖掘甘蔗品種的抗旱性能,本研究挑選甘蔗品種中參與調(diào)控抗旱機制的主要轉(zhuǎn)錄因子GRAS的基因進行表達(dá)分析。從轉(zhuǎn)錄組數(shù)據(jù)庫中調(diào)取GRAS轉(zhuǎn)錄因子基因序列信息,采用RACE-PCR和qRT-PCR技術(shù)從‘蔗茅99-2中克隆得到1個GRAS基因,將其命名為EfGRAS(GenBank登錄號MT499789),并對其進行生物信息學(xué)分析與干旱脅迫表達(dá)分析。結(jié)果表明,EfGRAS基因編碼547個氨基酸,CDS全長1644 bp,該蛋白的分子式為C2645H4149N747O816S21,相對分子質(zhì)量為60.14 kDa,不穩(wěn)定系數(shù)為54.85,脂肪系數(shù)為84.37,GRAVY值為?0.293,是一類不穩(wěn)定蛋白和親水蛋白;亞細(xì)胞定位表明其位于細(xì)胞核內(nèi),EfGRAS蛋白主要的二級結(jié)構(gòu)是α-螺旋和無規(guī)則卷曲,與高粱親緣關(guān)系最近;qRT-PCR分析顯示,受到干旱脅迫后,‘蔗茅99-2中基因表達(dá)量相比于對照組上調(diào)約57.6倍,‘滇蔗01-58中基因表達(dá)量相比于對照上調(diào)約27.4倍,‘崖城89-9中基因表達(dá)量相比于對照組上調(diào)倍數(shù)較低。本研究結(jié)果為進一步研究EfGRAS基因在甘蔗中的功能提供理論基礎(chǔ)。
關(guān)鍵詞:蔗茅;干旱脅迫;EfGRAS基因;克隆;表達(dá)分析
中圖分類號:S566.1;Q78? ? ? 文獻(xiàn)標(biāo)識碼:A
Abstract: In order to make deeper research into the drought resistance of sugarcane varieties, the genes of GRAS which playing an important role in the regulation of drought resistance mechanisms in sugarcane varieties for expression analysis were studied. In this experiment, we firstly retrieved the GRAS transcription factor gene sequence information from the transcriptome database, and used RACE-PCR and qRT-PCR techniques to clone a GRAS gene from ‘Sucrose 99-2 and namde it EfGRAS (GenBank registration No. MT499789), then the bioinformatics analysis and expression analysis under drought stress were conducted on the EfGRAS gene. The results showed that the EfGRAS gene could encode 547 amino acids with a CDS 1644 bp in length, the molecular formula of the protein was C2645H4149N747O816S21, the relative molecular mass was 60.14 kDa, the instability coefficient was 54.85, the fat coefficient was 84.37, and the GRAVY value was ?0.293, which was a class unstable and hydrophilic protein. The subcellular location indicated that it was located in the nucleus. The main secondary structure of EfGRAS protein was α-helix and random coils, which were closely related to sorghum; qRT-PCR analysis showed that after drought stress, the gene expression in ‘Sugarcane 99-2 was up-regulated by about 57.6 times compared to the control group, and the gene expression in ‘DZ 01-58 was up-regulated by about 27.4 times compared to the control group. Compared with the control group, the gene expression level in ‘YC 89-9 had a lower fold of up-regulation. The results would provide a theoretical basis for the further study on the function of EfGRAS in sugarcane.
Keywords: Erianthus fluvus; drought stress; EfGRAS gene; clone; expression analysis
DOI: 10.3969/j.issn.1000-2561.2020.10.017
甘蔗(Saccharum spp.)是禾本科多年生熱帶作物,屬于無性繁殖的C4作物,其種植最早起源于中國[1-2]。作為主要的制糖類經(jīng)濟作物,在我國南方地區(qū)甘蔗種植面積較廣[3-4]。中國85%左右的甘蔗種植面積為旱地,干旱成為限制中國甘蔗生產(chǎn)的首要環(huán)境因素[5]。干旱嚴(yán)重影響了甘蔗的產(chǎn)量和品質(zhì),隨著甘蔗品種抗旱性能研究的不斷深入,選育出高產(chǎn)、穩(wěn)產(chǎn)、抗旱能力強的甘蔗品種及挑選最佳的親本材料成為我國目前甘蔗研究的首要任務(wù)[6]。蔗茅(Erianthus fulvus Ness.)是禾本科甘蔗亞族蔗茅屬的一個野生種,具有早熟、含糖量高、抗逆性強等特點[7],其在高海拔寒冷的氣候條件下和干旱嚴(yán)峻的荒坡上都可以生長,甚至在陡峭的石壁巖縫和成土母質(zhì)上也可見到蔗茅,表現(xiàn)出極強的抗寒、抗旱和耐瘠能力,宿根性強、抗銹病等優(yōu)異特性,容易開花且花粉量極多,目前只在中國收集、保存并展開相關(guān)的研究,是一種極其珍貴的種質(zhì)資源[8-10]。
GRAS轉(zhuǎn)錄因子只在植物中存在,GRAS蛋白擁有著獨特的結(jié)構(gòu)域,根據(jù)對模式植物擬南芥和水稻的GRAS基因家族遺傳分析,可將GRAS基因家族劃分為8個亞族(LISCL、PATI、SCL3、DELLA、SCR、SHR、LS和HAM),其中擬南芥、水稻、煙草、胡楊、大白菜、佛手、辣椒和獨行菜的GRAS家族基因響應(yīng)了干旱、低溫和激素等脅迫。馬洪雙等[11]從胡楊中得到抗旱基因PeSCL7,包含可能與干旱脅迫、病害脅迫等應(yīng)答元件。張煥欣等[12]利用生物信息學(xué)方法進行系統(tǒng)鑒定和進化分析表明,辣椒的大部分GRAS家族基因能夠響應(yīng)PEG6000和鹽脅迫。楊輝等[13]從煙草品種NC89克隆的NtGRAS在低溫(4 ℃)、NaCl(200 μmol/L)和干旱下均可誘導(dǎo)NtGRAS基因表達(dá)上調(diào)。殷龍飛等[14]以玉米為材料,發(fā)現(xiàn)ZmGRAS31在玉米中響應(yīng)了抗旱、抗寒及抗鹽等逆境脅迫,推測ZmGRAS31可提高玉米的抗逆性。韓雯毓等[15]利用qRT-PCR技術(shù)檢測干旱和鹽脅迫下蓖麻根、莖和葉不同組織中5個基因的表達(dá),發(fā)現(xiàn)RcGRASs在不同組織中的表達(dá)存在差異性,其中RcGRAS14、RcGRAS21、RcGRAS35的基因表達(dá)量上調(diào),RcGRAS1、RcGRAS10的基因表達(dá)下調(diào)。
研究甘蔗野生種蔗茅的抗旱相關(guān)基因,對未來提高甘蔗產(chǎn)量、品質(zhì)有著重要意義。本研究以‘蔗茅99-2‘滇蔗01-58和‘崖城89-9及其轉(zhuǎn)錄組數(shù)據(jù)為試驗材料和基礎(chǔ),對EfGRAS基因進行克隆以及生物信息學(xué)分析,同時采用qRT-PCR技術(shù)檢測干旱脅迫下EfGRAS基因的表達(dá)情況,以期為甘蔗抗旱機制的后續(xù)研究提供科學(xué)依據(jù)。
1? 材料與方法
1.1? 材料
供試材料為‘蔗茅99-2‘滇蔗01-58和‘崖城89-9,均由云南農(nóng)業(yè)大學(xué)甘蔗研究所提供,在材料苗期分別取其對照組(正常澆水)及處理組(干旱脅迫處理)的+1葉。取樣時間為9:00,剪取后立即放入?80 ℃冰箱保存?zhèn)溆谩?/p>
1.2? 方法
1.2.1? 總RNA提取及cDNA鏈合成? 利用TRNzol Universa試劑盒提取總RNA,cDNA鏈的合成利用FastKing一步法合成反轉(zhuǎn)錄cDNA試劑盒。
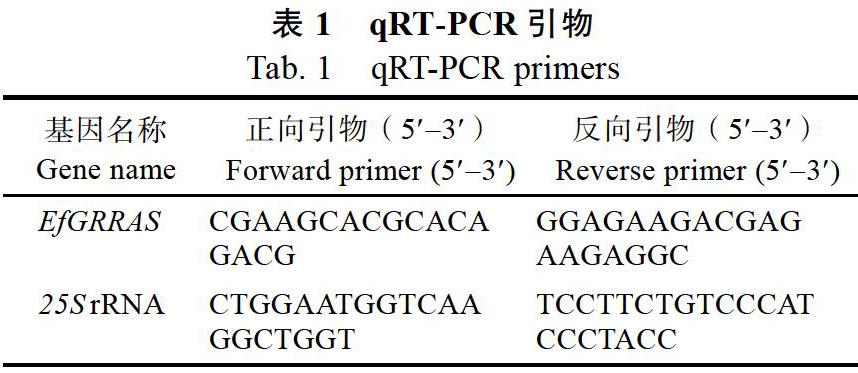
1.2.2? 引物設(shè)計及基因表達(dá)分析? 根據(jù)基因的序列,qRT-PCR使用NCBI Primer Blast以及Primer 5.0設(shè)計其引物,內(nèi)參基因選用闕友雄等[16]設(shè)計的25S引物序列,具體見表1。
PCR程序采用兩步法在ABI-7500儀器上進行qRT-PCR。反應(yīng)程序為:95 ℃預(yù)變性10 min;95 ℃變性15 s,60 ℃退火延伸60 s,采集熒光信號共40個循環(huán),每個樣品設(shè)置3個重復(fù)。反應(yīng)結(jié)束后從qRT-PCR的擴增曲線中得到Ct值,采用2?Ct算法計算差異表達(dá)基因的相對表達(dá)量。
1.2.3? EfGRAS基因克隆? 根據(jù)EfGRAS基因的序列,使用NCBI在線軟件Home-ORF Finder找到基因的開放閱讀框,然后通過NCBI的Primer- BLAST設(shè)計目的基因的擴增引物(表2)。基因擴增的PCR反應(yīng)程序為:98 ℃ 20 s;98 ℃ 10 s,53 ℃ 10 s,72 ℃ 1 min,35個循環(huán);72 ℃ 10 min。
1.2.4? EfGRAS基因生物信息學(xué)分析? EfGRAS蛋白的氨基酸組成、理論分子量和等電點利用Protparam在線軟件分析;EfGRAS蛋白的親疏水性利用ProtScale(https://web.expasy.org/protscale/)在線軟件進行預(yù)測分析;利用ProtComp Version 9.0對EfGRRAS蛋白亞細(xì)胞定位進行分析;通過NCBI的CDD(conserved domain database)進行結(jié)構(gòu)域預(yù)測;利用SOPMA軟件對EfGRAS蛋白的二級結(jié)構(gòu)進行分析;利用Swiss- modle在線軟件對EfGRAS蛋白的三級結(jié)構(gòu)進行預(yù)測;通過NCBI的Blastx進行搜索不同物種間氨基酸序列,再利用DNAMAN 6.0進行同源性分析,系統(tǒng)進化樹由MEGA7.0軟件構(gòu)建。
2? 結(jié)果與分析
2.1? RNA的檢測結(jié)果
提取正常澆水及干旱脅迫處理的‘蔗茅99-2‘滇蔗01-58‘崖城89-93種材料苗期的RNA進行紫外分光光度計檢測,檢測后發(fā)現(xiàn)RNA樣品的OD260/280均在1.8~2.0左右,純度較高,如圖1所示。
2.2? EfGRAS基因克隆
以cDNA為模板,根據(jù)編碼區(qū)起始端、終止端序列設(shè)計的特異引物進行基因編碼區(qū)PCR擴增,將PCR產(chǎn)物經(jīng)1%瓊脂糖TAE凝膠電泳,EfGRAS基因在1769 bp處顯示出1條清晰條帶(圖2),與預(yù)期結(jié)果一致。
2.3? EfGRAS基因生物信息學(xué)分析
2.3.1? EfGRAS轉(zhuǎn)錄因子的氨基酸組成及理化性質(zhì)分析? 將測序結(jié)果在NCBI上進行ORF Finder分析,結(jié)果發(fā)現(xiàn)有12個大小不同的ORF,其中最長的一個ORF有一個起始密碼子ATG,終止密碼子TAG,編碼547個氨基酸,CDS全長1644 bp(圖3)。利用ProParam在線軟件對該基因編碼蛋白質(zhì)的氨基酸序列進行基本性質(zhì)的分析:該蛋白的分子式為C2645H4149N747O816S21,相對分子質(zhì)量為60.14 kDa,由C、H、N、O、S 5種原子組成,共有8378個原子;該蛋白不穩(wěn)定系數(shù)為54.85,脂肪系數(shù)為84.37,GRAVY值為?0.293,是一類不穩(wěn)定蛋白。
2.3.2? EfGRAS蛋白親疏水性及保守結(jié)構(gòu)域? 利用ProScale在線軟件進行親疏水性分析,標(biāo)度值<0的區(qū)域比>0的較為密集(圖4),結(jié)合GRAVY值,預(yù)測該蛋白為親水蛋白;對EfGRAS基因編碼蛋白進行保守結(jié)構(gòu)域預(yù)測分析,該蛋白含有1個典型的GRAS結(jié)構(gòu)域(圖5)。
2.3.3? EfGRAS蛋白亞細(xì)胞定位及序列比對分析? 使用ProtComp Version 9.0對EfGRRAS蛋白進行亞細(xì)胞定位分析,從分析結(jié)果可以看出,該蛋白位于細(xì)胞核的積分值為5.93(表3),從而可以推測該蛋白定位于細(xì)胞核內(nèi)的可能性較大。利用Blastx分析,與EfGRAS蛋白序列相似性高的蛋白序列有高粱(Sorghum bicolor)(96.71%)、糜子(Panicum miliaceum)(93.60%)、狗尾草(Setaria italica)(93.24%)、玉米(Zea mays)(92.78%)、佛肚竹(Bambusa ventricosa)(83.36%)、二穗短柄草(Brachypodium distachyon)(81.67%)、水稻(Oryza sativa Japonica Group)(81.17%)、黃麻(Corchorus capsularis)(65.80%)、油棕(Elaeis guineensis)(65.29%),利用DNAMAN 6.0將EfGRAS基因進行同源蛋白比較,結(jié)果見圖6。
2.3.4? EfGRAS蛋白進化樹分析及結(jié)構(gòu)分析? MEGA 7軟件分析的結(jié)果顯示,EfGRAS蛋白與高粱親緣關(guān)系最近,其次是玉米(圖7)。SOPMA軟件分析結(jié)果表明,該蛋白由α-螺旋(alpha helix) (43.51%)、延伸主鏈(extended strand)(10.97%)、β-轉(zhuǎn)角(beta turn)(4.39%)和無規(guī)則卷曲(random coil)(41.13%)構(gòu)成,總的來看,EfGRAS蛋白主要的二級結(jié)構(gòu)是α-螺旋和無規(guī)則卷曲。EfGRAS蛋白的三級結(jié)構(gòu)預(yù)測如圖8所示。
2.4? EfGRAS基因在干旱脅迫下的定量表達(dá)分析
從圖9可看出,干旱脅迫處理下,EfGRAS基因不同材料中的表達(dá)量有顯著性差異。受到干旱脅迫后,‘蔗茅99-2基因表達(dá)量相比于對照組上調(diào)約57.6倍,‘滇蔗01-58上調(diào)約27.4倍,‘崖城89-9上調(diào)倍數(shù)較低。受到干旱脅迫后經(jīng)過實時熒光定量PCR發(fā)現(xiàn)基因表達(dá)量都呈現(xiàn)不同程度的遞增,其中甘蔗野生種‘蔗茅99-2中的基因表達(dá)量前后增幅最大,其次是雜交種‘滇蔗01-58。
3? 討論
3.1? EfGRAS基因表達(dá)量分析
目前,關(guān)于GRAS蛋白的抗旱研究中,發(fā)現(xiàn)少數(shù)GRAS蛋白參與非生物應(yīng)激反應(yīng)。郭華軍等[17]以擬南芥為試驗材料,并通過生物信息學(xué)技術(shù)和全基因組芯片分析發(fā)現(xiàn)在滲透和干旱脅迫時有10個GRAS家族基因表達(dá)量顯著上調(diào),然后利用功能基因組平臺,初步發(fā)現(xiàn)SCL13基因可能在應(yīng)答過程中參與了ABA依賴型信號轉(zhuǎn)導(dǎo)途徑。李亞飛等[18]通過轉(zhuǎn)錄組數(shù)據(jù)分析了在熱脅迫和干旱脅迫下小麥中GRAS基因的表達(dá)量的差異,發(fā)現(xiàn)該基因在響應(yīng)干旱脅迫的過程中起著關(guān)鍵的作用。Xu等[19]探究了GRAS轉(zhuǎn)錄因子基因OsGRAS23的抗旱能力,結(jié)果表明過表達(dá)OsGRAS23的水稻植株與野生型水稻相比,具有更強的抗旱性和抗氧化能力。Ma等[20]為探究GRAS的抗旱性和耐鹽性,從胡楊中分離出PeSCL7基因,研究發(fā)現(xiàn)PeSCL7基因在擬南芥中的過表達(dá)顯示出提高了耐旱性和耐鹽性。Czikkel等[21]研究發(fā)現(xiàn)NtGRAS1與煙草的抗旱性相關(guān),在干旱脅迫下NtGRAS1表達(dá)被誘導(dǎo)。
研究表明,EfGRAS基因受到干旱脅迫后能被誘導(dǎo),基因表達(dá)量均表現(xiàn)上調(diào),但在‘蔗茅99-2‘崖城89-9‘滇蔗01-58中的上調(diào)倍數(shù)不同,其中在‘蔗茅99-2中EfGRAS基因上調(diào)倍數(shù)最大,本研究與前人研究結(jié)果基本一致,推測EfGRAS在參與蔗茅干旱脅迫響應(yīng)過程中起正調(diào)控的作用。
3.2? EfGRAS轉(zhuǎn)錄因子抗旱性克隆分析
GRAS蛋白是一個多功能蛋白質(zhì),參與許多生物學(xué)過程,除參與植物的生長發(fā)育外,也已經(jīng)鑒定發(fā)現(xiàn)多種GRAS轉(zhuǎn)錄因子具有抵抗不同非生物脅迫的功能。Li等[22]發(fā)現(xiàn)BrLAS編碼一種脅迫反應(yīng)的GRASs轉(zhuǎn)錄因子,該因子可正向調(diào)節(jié)干旱脅迫的耐受性。Liu等[23]以辣椒為試驗材料發(fā)現(xiàn),在冷害、干旱、鹽和赤霉素(GA)處理下,有21種CaGRAS基因差異表達(dá),表明它們可能與植物對非生物脅迫的反應(yīng)有關(guān)。粟莉圓等[24]在熱脅迫下,JrGRAS2基因被顯著誘導(dǎo),發(fā)現(xiàn)轉(zhuǎn)JrGRAS2基因酵母表現(xiàn)出較對照更高的生存活性,表明JrGRAS2基因具有響應(yīng)熱脅迫的能力且能提高酵母的抗性,JrGRAS2基因可作為核桃逆境應(yīng)答的重要候選基因。韓菲等[25]以“富士×特拉蒙”雜交后代柱型株系新梢為試材,用同源克隆方法,得到了柱型蘋果GRAS基因家族的MdSCR基因。李阿英等[26]以擬南芥SCL6和楊樹GRAS cDNA 序列作為模板,對柑橘EST數(shù)據(jù)庫進行同源檢索篩選出柑橘SCL6和GRAS基因的cDNA序列,并以枳花cDNA為模板,利用5 RACE和3 RACE技術(shù)獲得枳的SCL6和GRAS cDNA全長,分別命名為Pt-SCL6和Pt-GRAS。
本研究結(jié)果可以推測出EfGRAS可能是一個抗旱基因,通過對蔗茅EfGRAS基因的克隆、生物信息學(xué)分析,確定該蛋白不穩(wěn)定系數(shù)為54.85,脂肪系數(shù)為84.37,GRAVY值為?0.293,是一類不穩(wěn)定蛋白,EfGRAS蛋白與高粱親緣關(guān)系最近,主要的二級結(jié)構(gòu)是α-螺旋和無規(guī)則卷曲,為進一步深入研究EfGRAS基因的功能奠定了基礎(chǔ)。
參考文獻(xiàn)
方靜平, 闕友雄, 陳如凱. 甘蔗屬起源及其與近緣屬進化關(guān)系研究進展[J]. 熱帶作物學(xué)報, 2014, 35(4): 816-822.
楊翠鳳, 楊麗濤, 李楊瑞. 甘蔗的起源和進化[J]. 南方農(nóng)業(yè)學(xué)報, 2014, 45(10): 1744-1750.
雷敬超, 張保青, 高麗花, 等. 甘蔗常用親本黑穗病抗性初步調(diào)查[J]. 中國糖料, 2018, 40(5): 30-33.
趙建屹. 甘蔗種業(yè)發(fā)展現(xiàn)狀與對策研究[D]. 福州: 福建農(nóng)林大學(xué), 2014.
徐? 榮, 李富生, 何麗蓮. 甘蔗抗旱性遺傳育種的研究進展[J]. 分子植物育種, 2018, 16(6): 1949-1954.
蘭? 靖, 陳? 永, 陸玉朵, 等. 6個甘蔗品種(系)的抗旱性差異評價[J]. 西南農(nóng)業(yè)學(xué)報, 2014, 27(4): 1374-1381.
李嬡甜, 李富生, 李翠英, 等. 含有蔗茅血緣的甘蔗新品系在德宏蔗區(qū)的適應(yīng)性評價[J]. 中國糖料, 2018, 40(5): 1-5, 9.
李富生, 林位夫, 何順長. 開發(fā)利用蔗茅野生種質(zhì)資源的思考[J]. 資源開發(fā)與市場, 2004(4): 266-270.
陸? 鑫, 毛? 鈞, 劉洪博, 等. 甘蔗野生種滇蔗茅種質(zhì)創(chuàng)新利用研究Ⅰ.甘蔗與滇蔗茅遠(yuǎn)緣雜交F1群體構(gòu)建與SSR分子標(biāo)記鑒定[J]. 植物遺傳資源學(xué)報, 2012, 13(2): 321-324.
李文鳳, 蔡? 青, 黃應(yīng)昆, 等. 甘蔗野生資源對蔗茅柄銹菌的抗性鑒定[J]. 植物保護, 2005, 31(2): 51-53.
馬洪雙, 夏新莉, 尹偉倫. 胡楊SCL7基因及其啟動子片段的克隆與分析[J]. 北京林業(yè)大學(xué)學(xué)報, 2011, 33(1): 1-10.
張煥欣, 董春娟, 尚慶茂. 辣椒GRAS家族全基因組鑒定與表達(dá)分析[J]. 園藝學(xué)報, 2017, 44(12): 2305-2317.
楊? 輝, 楊曉娜, 王? 翀, 等. 煙草NtGRAS基因的克隆與轉(zhuǎn)錄激活及表達(dá)特性分析[J]. 湖南農(nóng)業(yè)大學(xué)學(xué)報(自然科學(xué)版), 2018, 44(3): 271-277.
殷龍飛, 王朝陽, 吳忠義, 等. 玉米ZmGRAS31基因的克隆及功能研究[J]. 作物學(xué)報, 2019, 45(7): 1029-1037.
韓雯毓, 李國瑞, 風(fēng)? 蘭, 等. 蓖麻GRAS轉(zhuǎn)錄因子家族的全基因組分析及逆境脅迫響應(yīng)[J]. 植物遺傳資源學(xué)報, 2020, 21(1): 252-259.
闕友雄, 許莉萍, 徐景升, 等. 甘蔗基因表達(dá)定量PCR分析中內(nèi)參基因的選擇[J]. 熱帶作物學(xué)報, 2009, 30(3): 274-278.
郭華軍, 焦遠(yuǎn)年, 邸? 超, 等. 擬南芥轉(zhuǎn)錄因子GRAS家族基因群響應(yīng)滲透和干旱脅迫的初步探索[J]. 植物學(xué)報, 2009, 44(3): 290-299.
李亞飛, 陽文龍, 顧晶晶, 等. 小麥GRAS基因家族的全基因組鑒定與分析[J]. 麥類作物學(xué)報, 2019, 39(5): 549-559.
Xu K, Chen S J, Li T F, et al. OsGRAS23, a rice GRAS transcription factor gene, is involved in drought stress response through regulating expression of stress-responsive genes[J]. BMC Plant Biology, 2015(15): 141.
Ma H S, Liang D, Shuai P, et al. The salt- and drought-inducible poplar GRAS protein SCL7 confers salt and drought tolerance in Arabidopsis thaliana[J]. Journal of Experimental Botany, 2010, 61(14): 4011-4019.
Czikkel B E, Maxwell D P. NtGRAS1, a novel stress-induced member of the GRAS family in tobacco, localizes to the nucleus[J]. Journal of Plant Physiology, 2007, 164(9): 1220-1230.
Li P, Zhang B, Su T B, et al. BrLAS, a GRAS transcription factor from Brassica rapa, is involved in drought stress tolerance in transgenic Arabidopsis[J]. Frontiers in Plant Science, 2018(9): 1792.
Liu B L, Sun Y, Xue J N, et al. Genome-wide characterization and expression analysis of GRAS gene family in pepper (Capsicum annuum L.)[J]. Peerj, 2018(6): e4796.
粟莉圓, 李孝哲, 陳淑雯, 等. 核桃JrGRAS2基因響應(yīng)熱脅迫的表達(dá)及功能分析[J]. 植物研究, 2018, 38(1): 125-131.
韓? 菲, 戴洪義, 張玉剛. 柱型蘋果GRAS基因家族MdSCR的克隆與生物信息學(xué)分析[J]. 青島農(nóng)業(yè)大學(xué)學(xué)報(自然科學(xué)版), 2012, 29(3): 196-200.
李阿英, 劉? 洪, 李曉穎, 等. 枳兩個GRAS基因cDNA全長的克隆及其亞細(xì)胞定位分析[J]. 基因組學(xué)與應(yīng)用生物學(xué), 2012, 31(3): 240-248.

