利用范數描述符預測苯衍生物對費氏弧菌(V. fischeri)的毒性效應
史琦玉,賈青竹,王 強
(1. 天津科技大學海洋與環境學院,天津 300457;2. 天津科技大學化工與材料學院,天津 300457)
苯衍生物包括苯胺、苯酚、硫酚、苯甲酸和抗生素(磺胺類與磺胺類增強劑)等,在化工、醫藥和農業等領域廣泛應用,由此導致這些化合物在環境中顯著檢出;同時,此類化合物對水體環境產生生態毒性,對人體健康存在潛在的風險,因此,有必要對此類化合物的生態風險進行評估[1].
有機物對費氏弧菌(V. fischeri)的生態毒性數據是對其進行生態風險評價的主要依據[2].實驗測定生物毒性不僅耗時費力,而且與動物倫理 3R原則(replacement,reduction,refinement)相違背.因此,亟需尋求快速高效獲取生態毒性數據的方法.根據歐洲化學品注冊、評估、授權和限制條例 REACH的規定,為避免和減少化學品對生態環境可能造成的潛在危害,鼓勵使用數學模型預測工具如定量構效關系(QSAR)獲取生態毒性數據.QSAR方法已成為預測化學品生態毒理危害不可或缺的工具[3-4].
許多研究者報道了苯衍生物對V. fischeri的水生毒性評估方法[5-9].Wang等[5]采用逐步回歸分析法建立了 QSAR模型,預測苯酚衍生物和苯胺衍生物(共15種)對V. fischeri的毒性pEC50(EC50取負對數),該模型統計分析結果 R2為 0.89.Melo等[6]運用連續偏最小二乘法(PLS),預測了苯砜基羧酸酯衍生物(41種)對 V. fischeri的毒性,該 QSAR模型的相關系數R2為 0.884.Shi等[7]測定了苯硫酚衍生物(31種)對V. fischeri的毒性,分別使用比較分子場分析(CoMFA)描述符和比較分子相似性指數分析(CoMSIA)描述符,采用逐步多元線性回歸分析法建立模型并成功地預測了苯硫酚衍生物的毒性,R2(0.848~0.928)和 Q2(0.516~0.652)表明這兩個模型均表現出良好的穩定性和預測能力.Liu等[8]基于CoMFA和CoMSIA描述符,采用偏最小二乘法分別建立兩個 QSAR模型,預測了苯砜基羧酸酯衍生物(56種)對 V. fischeri毒性;其中,使用 CoMFA描述符的模型(R2=0.958)優于 CoMSIA 描述符的模型(R2=0.933).綜上,這些模型都取得了令人滿意的預測結果,但是,大部分研究工作是針對單一類型的苯衍生物,同時針對多種類苯衍生物對 V. fischeri的生態毒性的研究工作還很少.
近年來,本課題組提出了范數指數的概念,據此建立了一系列 QSAR模型,并成功應用于離子液體對鼠細胞、細菌的生態毒性[9-10]、有機物的物化性質以及對藻類、魚類的生態毒性[11-16]等領域.
本研究目的是建立一個 QSAR模型預測多種苯衍生物對V. fischeri的生態毒性.
1 方法和理論
1.1 數據集
本工作中,苯衍生物生態毒性實驗值(EC50,單位用 mol/L 表示)收集于 Wang等[17]、Shi等[7]、Aruoja等[18]和 Zhao等[19]的研究工作.參與建模的110種苯衍生物包括苯酚衍生物和苯胺衍生物(共58種)、苯硫酚衍生物(30種)、苯甲酸衍生物(10種)和抗生素(12種).這些化合物對V. fischeri的生態毒性實驗值pEC50等的信息列于表1中.

表1 苯衍生物對V. fischeri的毒性值Tab. 1 Toxicity of benzene derivatives to V. fischeri

續表

續表
1.2 原子分布矩陣
本工作利用 HyperChem7.0軟件(http://www.hyper.com)進行結構優化.優化方法采用量子化學從頭計算方法,在 STO-3G基組下優化計算,得到化合物穩定的結構.根據原子性質和空間分布,建立了原子分布矩陣(M),并對 M 進一步數據挖掘.M 范數計算即為范數指數.范數定義表達為式(1)—式(3).


式中:mij表示矩陣中第 i行 j列的元素;n表示化合物的總數;λi表示矩陣的特征值;MH是矩陣 M 的轉置共軛矩陣.
采用矩陣(D,式(4))和相鄰矩陣(A,式(5))來描述原子的位置分布.原子性質包含范德華半徑(vr)、原子電荷(ac)、電子層數(es)、原子質量(aw)和分子質量(mw),原子性質矩陣(P)表達為式(6)—式(10).本文建立的8個原子分布矩陣列在表2中.

表2 8個原子分布矩陣Tab. 2 8 atomic distribution matrices

式(4)中:dij是原子 i和 j之間的歐氏空間距離.HyperChem7.0軟件中原子的歐氏空間距離以 ?為單位,無量綱.

P1—P5為列向量.原子的范德華半徑以 ? 為單位,無量綱.

1.3 模型驗證
根據 OECD指導原則,良好的 QSAR模型應具有適當的擬合度、穩健性和預測能力.QSAR模型驗證最常用的方法包括內部驗證和外部驗證[20].其中,統計參數為相關性系數的平方(R2)、Fisher值(F)和均方根誤差(RMSE)等.
在內部驗證過程中,留一法交叉驗證(LOO CV)和五折交叉驗證(5-fold CV)的數據擬合質量統計參數分別是和均方根誤差(RMSELOO和RMSE5-fold).為了進行外部驗證,整個數據集以 4∶1的比例分為訓練集(88個有機物)和測試集(22個有機物).對于訓練集(tr)和測試集(te),分別使用外部驗證的統計參數和均方根誤差(RMSEtr,RMSEte)驗證模型的預測能力.另外,本工作通過 Y隨機測試檢查了模型的偶然相關性.
2 結果和討論
2.1 模型建立
本文提出了一個預測多種類苯衍生物對 V.fischeri生態毒性(pEC50)的模型,如式(11)所示,其中模型參數見表3.

此模型:n=110,R2=0.895,F=108,RMSE=0.241,Q2=0.874,RMSELOO=0.264.

表3 模型中8個范數描述符及其對應系數值Tab. 3 8 norm descriptors in the model and their corresponding coefficient values
2.2 模型預測結果
苯衍生物對V. fischeri生態毒性的模型計算值列在表1中,實驗值和模型計算pEC50值的散點圖如圖1所示.由圖 1可知:實驗與計算 pEC50值吻合較好.模型統計結果R2、F、RMSE分別為0.895、108和0.241,說明了該模型能夠準確預測苯衍生物對 V.fischeri的生態毒性.

圖1 實驗值與計算值pEC50散點圖Fig. 1 Scatter diagram of experimental and calculated pEC50 values
2.3 內部驗證
本工作利用留一法和五折交叉驗證法對模型進行內部驗證.圖 2為模型(式(11))與留一法(LOO CV)、五折交叉驗證(5-fold CV)計算結果的誤差分布圖.圖 2結果顯示,本工作模型與留一法、五折交叉驗證的誤差分布具有高度一致性,三者總體誤差分布均在[-0.15~0.15]之間,并符合正態分布.統計結果顯示,都等于 0.874,進一步表明本工作所建立模型具有很好的穩健性.
2.4 外部驗證
訓練集和測試集實驗值與計算值 pEC50的散點圖如3所示.從圖3中可以看出,訓練集和測試集的實驗值和預測值都與對角線相近,外部驗證的分別為 0.873 和 0.938,RMSEtr、RMSEte分別為0.247和 0.242,說明基于范數描述符建立的 QSAR模型穩定性好,具有良好的預測能力.此外,其他驗證參數[20]:CCC為0.965,均說明該模型具有良好的預測能力.

圖2 模型、留一法交叉驗證和五折交叉驗證的誤差分布Fig. 2 Error distribution of the new model,LOO CV and 5-fold CV
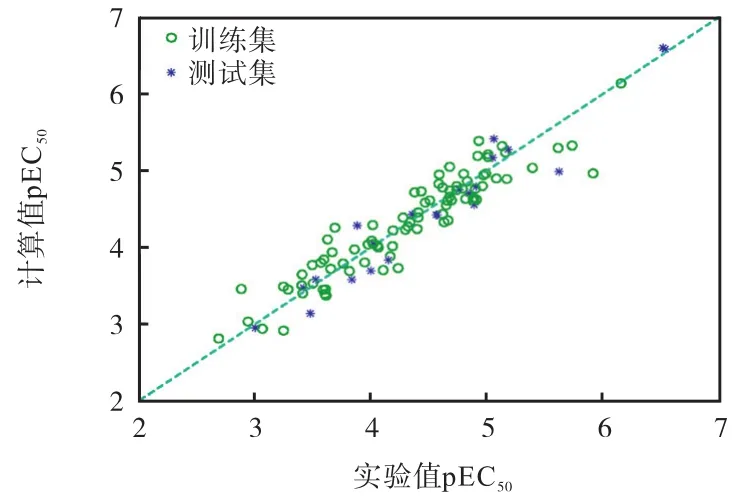
圖3 訓練集和測試集實驗值和計算值pEC50散點圖Fig. 3 Scatter diagram of experimental and calculated pEC50 values of the training set and testing set
2.5 Y隨機驗證
Y隨機化檢驗對 QSAR模型的驗證具有重要意義[21].本工作將有機物的毒性實驗值 pEC50隨機打亂順序 1000次,對模型進行 1000次 Y隨機化檢驗,結果如圖4所示.

圖4 1000次Y隨機檢驗結果Fig. 4 Results of 1000 times of Y-randomization test
2.6 應用域分析
為了獲得可靠的預測效果,有必要驗證 QSAR模型的應用領域.本工作利用三維Williams plot描述pEC50模型的應用域.在該圖中,杠桿(hi)度量化合物離模型適用性范圍的遠近,臨界值(h*)由 3×(p+1)/n計算,其中 p為模型變量數,n為建模過程中訓練集的數量.對于h大于h*的化合物,認為其屬于結構異常的范疇;對于交叉驗證的標準殘差大于3個標準偏差單位的化合物,則認為是響應異常的范疇.
本工作模型應用域的驗證結果如圖5所示,幾乎所有化學物質位于可接受區域內,僅存在一個結構異常和一個響應異常值.由此可見,本工作建立的模型具有廣泛的應用域,可以為苯衍生物對 V. fischeri生態毒性的預測提供可靠數據.
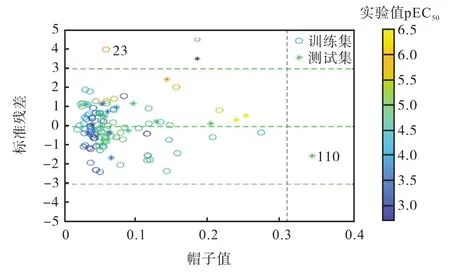
圖5 模型的應用域Fig. 5 Applicability domain of the new model
2.7 與文獻對比
將本工作模型與其他文獻模型進行比較,比較結果見表 4.首先文獻研究基本上都是針對小樣本量的某一類化合物進行 QSAR研究,均取得較好的模型預測精度[6,7,17,22].Guimar?es 等[22]和 Shi等[7]的研究工作得到了較高的 R2(0.848~0.928),但是這些模型交叉驗證的 Q2均較低,由此說明,這些模型的精度較高但是穩定性不高.本工作針對多種類苯衍生物對 V. fischeri的生態毒性進行建模研究,不僅樣本量大,而且模型的穩定性高(Q2=0.874);本工作基于范數描述符建立的 QSAR模型穩定性良好,預測能力較強,表明該模型可以較為成功地預測苯衍生物對V.fischeri的生態毒性.
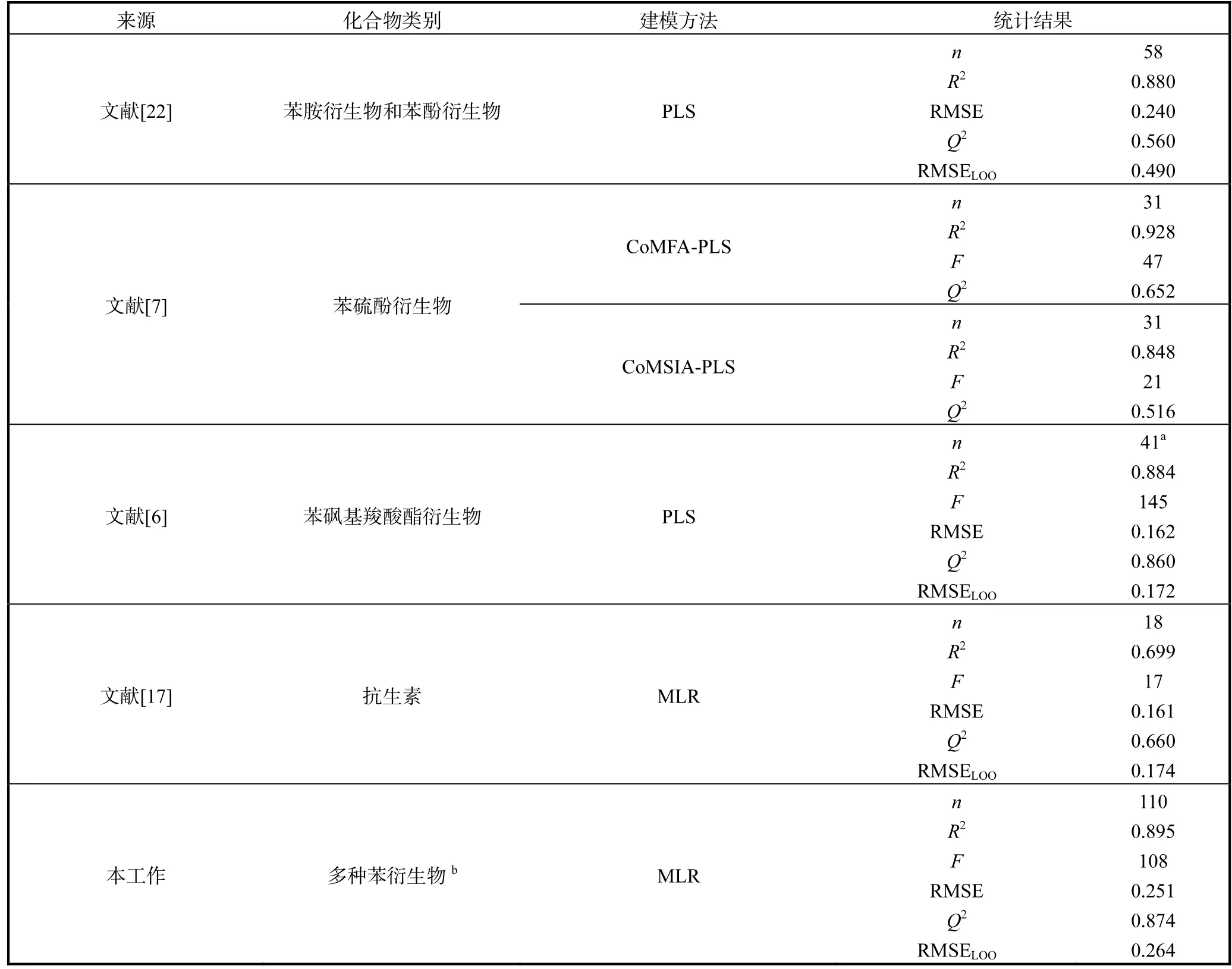
表4 本工作與文獻研究的比較Tab. 4 Comparison of this research with references
3 結 論
本工作提出了范數描述符,據此建立 QSAR模型,對 5類苯衍生物(苯胺、苯酚、硫酚、苯甲酸和抗生素)的費氏弧菌生態毒性進行預測.主要研究結論如下:(1)本工作模型對費氏弧菌生態毒性具有很好的預測精度;(2)統計結果和模型驗證結果表明了本工作模型的穩定性和廣泛適用性;(3)基于原子分布矩陣構建的范數描述符能夠準確描述有機物的分子結構,據此建立的模型對于有機物的生態風險評價具有重要意義.

