基于遷移學習的腫瘤病理學數據分析研究
王晨,王一博,陶子勛,付昂揚,曹玥琦

摘要:為解決癌癥的診斷問題,提高診斷的效率與準確率,增強診斷的可靠性,運用了TensorFlow搭建訓練基于卷積神經網絡的癌癥分類模型,基于VGG16架構,采用深度單類分類算法,使用遷移學習,采集正常細胞病理學數據,來訓練出可以識別癌變數據的模型,從而精準自動地將正常細胞與腫瘤病理學數據分類。結果表明建立的基于遷移學習的分類模型可以很好地幫助病理學家檢測癌癥,縮短診斷時間。
關鍵詞:卷積神經網絡;遷移學習;DOC算法;圖像分類;VGG16模型
中圖分類號:TP391? ? ? 文獻標識碼:A
文章編號:1009-3044(2021)34-0099-03
伴隨著人類社會高速發展的同時,環境的破壞大大提高了惡性腫瘤的發病率,癌癥的防治形勢仍然需要大眾廣泛的努力。癌癥發現得越晚,治愈概率越小,同時,組織病理學圖像在臨床分析中會耗費醫生大量的時間和精力,而且存在判斷錯誤的現象,所以癌癥的及時發現仍是全世界的難題。而利用計算機輔助設計(CAD)[1]自動處理病理學圖像不僅可以提高診斷效率,還可以提供更為準確的診斷結果。深度學習自發展以來,已被運用于多個領域,也使得醫學影像分析有了新的突破。1999年,Golub等人利用基因芯片技術和機器學習將急性白血病進行分類[2],使得機器學習與醫學影像緊密聯系,在機器學習的范疇內,腫瘤數據分類也變為了研究熱門課題之一。應用于病理學圖像分析的深度學習網絡結構有深度置信網絡(DBN)、卷積神經網絡(CNN)、降噪自動編碼器(DAE)等[3]。在現階段,由于資源的限制,腫瘤數據集的樣本量較小,為了解決特征選擇算法只能在各自數據集操作的局限性,Fakoor等人提出了將無監督學習和深度學習結合在一起的方法,利用稀疏自編碼器作為框架,使分類器更有效運用于多個腫瘤數據集。獲得高質量模型保險做法是增加模型深度或寬度,但這樣又會增加模型的復雜度并且有可能造成過擬合。Inception V3模型[4]將卷積核分解降維,節約了大量參數,加速運算并減輕了過擬合,同時增加了非線性擴展模型表達能力。在醫學領域,深度學習已經發揮了重大的作用,但隨著要處理的數據量越來越大,構建更加具有高效率、魯棒性的分類模型依然是需要研究的內容。而且使用大量的標簽數據訓練才會表現良好的監督學習,這在數據采集方面是較為困難的問題。在深度學習的基礎上使用遷移學習的方法,可以直接利用正常細胞圖像數據集建立分類模型,實現兩種圖像的分離[5]。這不僅解決了使用不同數據集存在的數據分布差異、標注數據過期的問題,還使得分類模型更適用于現代醫學環境,保證新的任務上的模型精度。
1相關理論
1.1卷積神經網絡
卷積神經網絡(CNN)不同于一般的神經網絡的是,它將每一個隱藏節點只與圖像的某個特定部分相連接,從而在卷積層用來提取特征時減少參數訓練的數量[6]。并且通過卷積核的權值共享,可以減少參數的數量。在卷積層的線性卷積操作完成后,使用激活函數增加非線性變換,從而使該模型學習到非線性的變換。幾種激活函數如sigmoid函數、ReLU函數、softmax函數、tanh函數等。CNN主要包括數據輸入層,卷積層,激勵層,池化層和全連接層。
由于輸入的數據存在尺寸過大、類型不符合等問題,需要預處理輸入前的數據。常見的數據預處理的方式包括:均值化處理(將輸入數據的每個特征減去其均值)、格式轉換(使原始數據轉為符合模型訓練的格式)、降維(數據集的維度發生改變,將高維數據集投影到達低維坐標軸)等。通過數據預處理可以減少神經網絡訓練中的等待時間,提高模型訓練的準確性和效率。
卷積計算層是CNN的重點,先將整個圖片分割成多個具有重復成分的區域,不斷計算各個部分的值,從而達到增強原始數據的特征值,減少噪聲的目的。在每一層中使用相同的卷積核,保證了一個神經元提取一個特征,多個神經元提取不同的特征。在CNN中,計算是通過對輸入的數據與濾波器(帶固定值的矩陣)做內積來提取數據的特定信息。每計算完一個部分的權重后,數據窗口移動至下個區域,直到計算完全部的圖像信息。設定計算過程中的若干參數能夠獲取不同的卷積神經網絡,參數包括:深度(濾波器個數,決定處理完的數據的厚度),步長(計算過程中窗口一次滑動的長度)與填充值(在原始數據外各個維度填充若干個0以保證總長度能整除步長)。
池化層用來降低數據維度,待處理的圖像過大,對圖像進行池化,即類似于壓縮的過程,通過一個下采樣方式來調整圖像的大小。經過池化操作后,結果相比輸入縮小了,而特征并沒有變形,并且在空間范圍內做了維度約減,從而使模型能夠抽取更加廣范圍的特征。同時減少了下一層的輸入大小,減少計算量和參數個數,在一定程度上防止過擬合。
全連接層在CNN的最后,類似傳統的神經網絡結構,將兩層之間的所有神經元連接。全連接層能夠對特征圖進行分類,將矩陣轉化為列向量,完全連接各個輸入。
1.2 VGG16架構
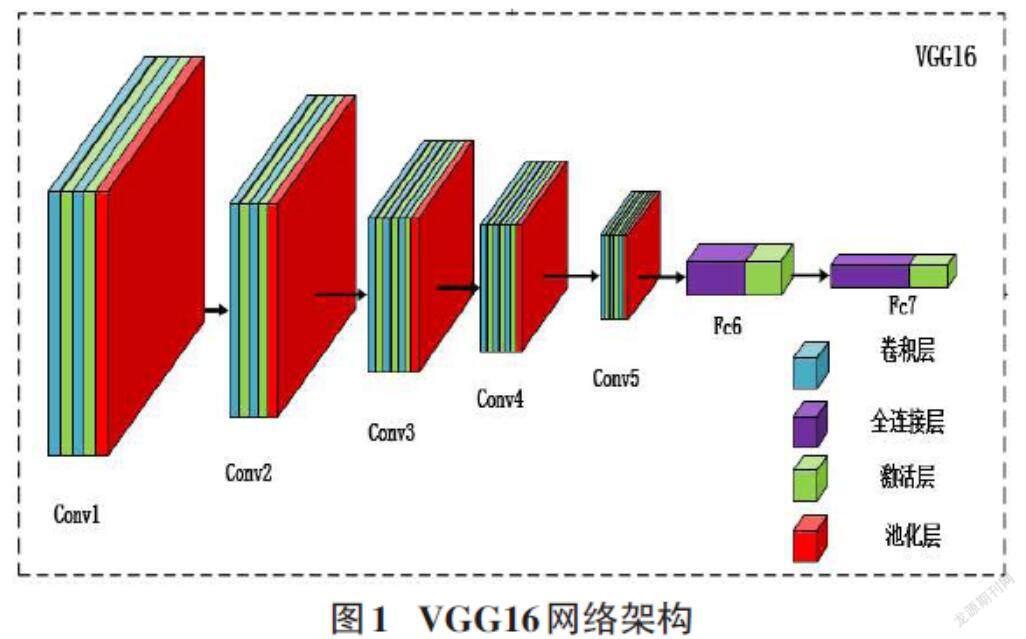
在模型的搭建上適用于VGG16和AlexNet網絡架構,通過截取這兩種主干網絡中的部分卷積層和全連接層來應用。該架構的網絡示意圖如圖1所示。
截取的VGG16模型共有13個卷積層,2個全連接層,5個池化層。所有卷積層的卷積核大小都是3×3,用來檢測某一方面的特征,如形狀,顏色,對比度等。CNN層數的增加使提取的特征更加全面。
VGG16使用最大池化,即選擇特征圖中最大元素進行下采樣,激活層采用修正線性單元(ReLu)激活函數,將卷積層的計算進行非線性映射,由圖1可知,VGG16全連接層主要是指Fc6和Fc7,用于后期檢測。
AlexNet是一種比VGG16更早提出的典型的CNN,它的網絡架構由5個卷積層,2個全連接層和最后一層的標簽層組成。[7]AlexNet和VGG16的主要區別是:1. VGG16的卷積核大小都是統一的3×3,而AlexNet每一層的卷積核大小并不統一。2.VGG16有16層網絡,AlexNet只有8層,但相比之下,VGG16需要的迭代次數更少(迭代器數量多)。所以VGG16是一個具有更深網絡、更多參數、特征提取效果更好的結構。
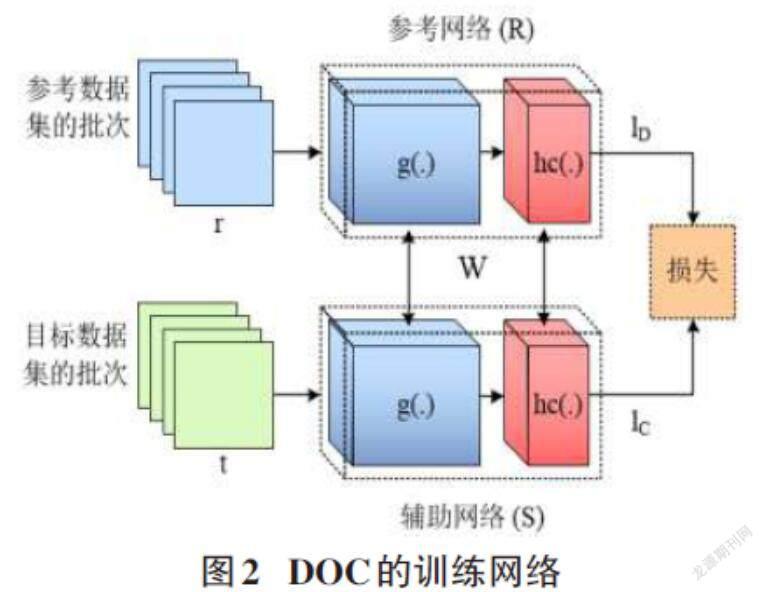
1.3 深度單類分類(DOC)算法[8]
DOC算法的訓練網絡結構如圖2所示,由兩個CNN組成,分為參考網絡R和輔助網絡S,兩類特征提取網絡的權重相互捆綁。DOC的網絡架構主要組件和CNN相同。參考網絡R是預先訓練的網絡,參考網絡則是特征提取網絡的組合。
在經典的多分類問題中,特征學習的目標是最大化與最小化類間的距離。在DOC算法中,用兩個指標來表示單類的特征。緊湊性表示將圖像中提取的一組特征緊湊地放置在特征空間,由類間距離決定。描述性是指給定的特征對不同的圖像有不同的表達,在這種情況下,描述性特征有較大的類間距離。單類分類的目標是找到合適的特征表示,使其最大程度上展現緊湊性與描述性。可以用公式表示為:
[g=maxD(gt+λC(gt)]
t是給定的數據,[λ]為常數,[D(g(t))]表示描述性,[C(g(t))]表示緊湊性。
1.4 遷移學習
在傳統的機器學習中,有許多需要大量數據標注和訓練的情況,為了對已有數據的利用和保證新訓練模型的精度,遷移學習的思想引起了廣泛關注[9-10]。按照遷移的參照分類,可以分為基于實例的遷移,基于特征的遷移與基于參數的遷移,在確定好確定對象后,才能針對具體問題得到解決方法,設計出合適的算法來遷移已有網絡。
為了避免數據過多造成的繁重重復的工作,我們采用遷移學習的方法,其中包括僅訓練最后一個完全連接的層,訓練整個網絡,優化模型從而減少樣本數量。通過遷移學習,我們可以充分利用一切可以應用的現有資源,經過大數據訓練好的模型與樣本結合起來調整模型參數,稍加修改遷移到我們的研究中。
2實驗結果與分析
2.1數據集
實驗對癌癥數據進行分類,數據來自TCGA(The Cancer Genome Atlas),是由美國國家癌癥研究所和國家人類基因組研究所共同提供的大型癌癥基因組數據庫。在TCGA數據庫中下載了1021張肺癌圖像作為數據集。由于直接下載的圖像過大,在訓練前對數據進行預處理,轉換成TensorFlow支持的TFRecord格式,并將處理好的數據按7:3隨機分為訓練和測試集兩類。
2.2實驗設置
實驗采用VGG16模型,輸入預處理完成的數據,大小為224×224×3像素,各個卷積層的卷積核大小均為3×3,各個池化層的池化單元均為2×2[11],Conv1兩層卷積層和64個通道數,輸出為(224,224,64),最大池化后輸出(112,112,64)。Conv2兩層卷積層和128個通道數,經過最大池化后輸出(56,56,128)。Conv3三層卷積層和256個通道數,最大池化后輸出(28,28,256)。Conv4三層卷積層和512個通道數,最大池化后(14,14,512)。Conv5三層卷積層和512個通道數,最大池化后輸出(7,7,512)。全連接層Fc6將神經元全部連接,輸出(1,1,4096),全連接層Fc7完全連接后輸出(1,1,1000)。網絡獲取足夠的特征后將最終矩陣轉化為向量,用于數據分類和預測。
數據的分類結果如表1所示:
共300條數據進入訓練好的模型測試,正常病理學圖像236張,肺癌細胞圖像64張,其中對正常細胞分類正確的有219張,錯誤的有17張,對肺癌細胞分類正確的有58張,錯誤的有6張。結果分析可得,模型的準確率為92.3%,召回率為97.3%。說明訓練的模型在對細胞病理學圖像分類上有較為良好的效果。
3結論
癌癥已經成為當今世界重視的醫學難題,對癌癥圖像快速準確的診斷有利于患者的病情治療。而癌癥圖像相對正常細胞圖像來說更難獲得,在深度學習的基礎上結合遷移學習能夠減少數據的使用,提高數據標簽的利用率。通過數據驗證,本文基于VGG16模型提出的腫瘤分類模型取得了很好的效果,準確率達到92.3%。
參考文獻:
[1] Araújo T,Aresta G,Castro E,et al.Classification of breast cancer histology images using Convolutional Neural Networks[J].PLoS One,2017,12(6):e0177544.
[2] Raderschall E,Golub E I,Haaf T.Nuclear foci of mammalian recombination proteins are located at single-stranded DNA regions formed after DNA damage[J].PNAS,1999,96(5):1921-1926.
[3] 楊鑫,章真.基于深度學習的人工智能在數字病理學中的進展[J].中國癌癥雜志,2021,31(2):151-155.
[4] Dong N,Zhao L,Wu C H,et al.Inception v3 based cervical cell classification combined with artificially extracted features[J].Applied Soft Computing,2020,93:106311.
[5] 莊福振,羅平,何清,等.遷移學習研究進展[J].軟件學報,2015,26(1):26-39.
[6] 周飛燕,金林鵬,董軍.卷積神經網絡研究綜述[J].計算機學報,2017,40(6):1229-1251.
[7] 盧宏濤,張秦川.深度卷積神經網絡在計算機視覺中的應用研究綜述[J].數據采集與處理,2016,31(1):1-17.
[8] 余凱,賈磊,陳雨強,等.深度學習的昨天、今天和明天[J].計算機研究與發展,2013,50(9):1799-1804.
[9] Perera P,Patel V M.Learning deep features for one-class classification[J].IEEE Transactions on Image Processing,2019,28(11):5450-5463.
[10] 石祥濱,房雪鍵,張德園,等.基于深度學習混合模型遷移學習的圖像分類[J].系統仿真學報,2016,28(1):167-173,182.
[11] 馮國徽.基于卷積神經網絡VGG模型的小規模圖像分類[D].蘭州:蘭州大學,2018.
【通聯編輯:唐一東】

