不同培養條件下釀酒酵母菌的轉錄組差異分析
楊新,陳莉,楊雙全,盧紅梅,章之柱
1(貴州大學,貴州省發酵工程與生物制藥重點實驗室,貴州 貴陽, 550025)2(貴州大學 釀酒與食品工程學院,貴州 貴陽, 550025)3(貴州大學 化學與化工學院,貴州 貴陽, 550025)4(開陽縣市場監督管理局,貴州 貴陽, 550300)
硒(selenium,Se)是一種人體生長發育所必需的微量元素,具有“生命火種”、“心臟的守護神”和“抗癌之王”的美譽[1-2]。它是體內某些酶和蛋白的重要組成部分,具有抗衰老、抗氧化、抗腫瘤、影響人和動物的生殖發育、增強機體免疫力、拮抗有害重金屬和預防多種疾病等生物學功能[1,3-5]。硒元素的缺乏會引起心血管疾病、腫瘤、克山病、大骨節病、癌癥、高血壓和免疫系統功能紊亂等疾病[6-9]。
酵母菌對許多微量元素具有較強的富集作用,比如鐵、鋅、硒、鉻及鍺等微量元素,但其富集機制與富集離子的種類有關[10-11]。此外,取決于酵母菌獨特的細胞壁結構,它的主要結構及成分為葡聚糖、蛋白質、甘露聚糖、幾丁質及少量脂類。因酵母菌具有體積小,表面積大,即比表面積大的特點,所以它具有代謝旺盛、繁殖速度快、產率高等優點。此外,還具有培養基原料來源廣泛,易于人工控制培養,不受時間、季節、氣候等條件的影響,且生產成本低、安全無污染、可持續性強等優點。因此,只需要在培養基中加入無機硒,酵母菌在生長繁殖過程中就可以將無機硒轉化為有機硒,主要以蛋白質、氨基酸、多糖等結合而存在。故酵母菌是一種優越的富硒載體,富硒酵母菌作為一種理想的功能性食品添加劑,它不僅能提供硒源,而且還可以提供一定的蛋白質、氨基酸等營養物質。作為一種重要的、食品安全級微生物的釀酒酵母菌(Saccharomycescerevisiae)[12-13],將其作為富硒載體有著巨大的市場前景。
高通量測序技術又稱“下一代”測序技術[14-15],隨著該技術的發展,它在生物體轉錄組基因表達分析中被廣泛應用,而且能夠精確便捷地挖掘出相關功能基因[16-19]。轉錄組測序技術(RNA-sequencing,RNA-Seq)具有通量大、高分辨率、高靈敏度、不需克隆、檢測范圍廣、成本低及操作簡單等優點,因此成為轉錄組研究的主要手段[20-23],RNA-Seq技術已成功應用于多項研究中[24-26]。本實驗采用轉錄組測序技術對釀酒酵母菌在不同硒濃度(0、20 μg/mL)培養條件下進行測序,然后基于轉錄組學采用生物信息學對釀酒酵母菌基因的表達差異進行分析,并對差異基因進行GO功能和KEGG富集分析,為今后對釀酒酵母菌富硒基因的挖掘以及研究提供了一定的理論基礎。
1 材料與方法
1.1 材料與試劑
1.1.1 材料
具有富硒能力的釀酒酵母菌(實驗室保藏)。
1.1.2 藥品試劑
葡萄糖、硫酸鎂,天津市永大化學試劑有限公司;酵母浸出粉、蛋白胨,上海博微生物科技有限公司;瓊脂,北京Solarbio Science & Technology公司;亞硒酸鈉,山東西亞化學股份有限公司;磷酸二氫鉀,成都金山化學試劑有限公司;化學藥品均為分析純。
1.1.3 儀器與設備
SN-CJ-IF潔凈工作臺、YXQ-LS-5DS11立式壓力蒸汽殺菌器,上海博訊實業有限公司醫療設備廠;SPX-250B智能型生化培養箱、DHG—9140B(101-2B)智能型電熱恒溫鼓風干燥箱,上海瑯玕實驗設備有限公司;TG16-WS臺式高速離心機,湖南湘儀實驗室儀器開發有限公司;BCD-290 W冰箱,青島海爾股份有限公司;SZ-96A自動純水蒸餾器,上海嘉措儀器設備有限公司;ZD-2A自動電位滴定儀,上海大普儀器有限公司;ESJ220-4B電子天平,沈陽龍騰電子有限公司。
1.2 實驗方法
1.2.1 培養基配制
YPD培養基(g/L):葡萄糖10.0,蛋白胨20.0,酵母浸出粉10.0,pH自然。
基礎發酵培養基(g/L):葡萄糖20.0、酵母浸出粉10.0、蛋白胨20.0、KH2PO4·3H2O 3.0、MgSO4·7H2O 1.0,121 ℃條件下高壓滅菌20 min,pH 4.5,備用。
1.2.2 菌體收集
將釀酒酵母菌種子液以6%的接種量分別接種于硒質量濃度為0、20 μg/mL(每個硒濃度做3個重復)的富硒培養基中,培養基初始pH值為4.5,加硒時間為培養后6 h,裝液量為100 mL/250 mL錐形瓶,在30 ℃,150 r/min的恒溫搖床上培養72 h。培養結束后,離心收集菌體,分別標記為Kb1、Kb2、Kb3和Se1、Se2、Se3,然后立即置于液氮中保存。
1.2.3 酵母菌總RNA提取與質量檢測
根據制造商的說明,使用TRIzol?試劑盒從釀酒酵母菌細胞中提取總RNA,并使用DNase I去除基因組DNA。對提取得到的RNA樣品,使用1%(質量分數)的瓊脂糖凝膠電泳檢測RNA的純度和完整度,使用NanoDrop2000 RNA檢測RNA的純度及濃度,使用Agilent 2100 Nano檢測RNA的完整值(RNA integrity number,RIN)。僅使用高質量的RNA樣品來構建測序文庫,然后上機測序。
1.2.4 文庫制備和轉錄組測序
RNA純化,反轉錄,文庫構建和測序均根據制造商的指導在上海美吉生物醫藥科技有限公司進行。使用Illumina TruSeqTM RNA Sample Prep Kit試劑盒構建RNA-seq轉錄組文庫。立即根據poly(A)選擇方法,通過oligo-dT磁珠分離mRNA。首先通過片段緩沖液進行片段化,然后使用SuperScript double-stranded cDNA合成試劑盒和隨機六聚體引物合成雙鏈cDNA。再根據文庫構建方案對合成的cDNA進行末端修復,即磷酸化和“A”堿基添加,選擇大小為2%低范圍超瓊脂糖上200~300 bp的cDNA目標片段,然后使用Phusion DNA聚合酶進行15個PCR循環進行PCR擴增。通過TBS380定量后,用Illumina Novaseq 6000(2×150 bp讀長)對雙端RNA-seq測序文庫進行上機測序。
1.3 數據分析
使用軟件SeqPrep(https://github.com/jstjohn/SeqPrep)和Sickle(https://github.com/najosh-i/sickle)以默認參數對原始的末端讀數進行裁剪和質量控制。使用軟件TopHat(http://tophat.cbcb.umd.edu/,版本2.1.1)[27]以定向模式將干凈的讀數分別與參考基因組比對。基于所選參考基因組序列,使用StringTie(http://ccb.jhu.edu/software/stringtie/)軟件對Mapped Reads進行拼接,然后與原有的基因組注釋信息進行比較。使用DESeq2軟件對Raw counts進行統計分析,基于P<0.05 & |log2FC|≥2為篩選條件,篩選獲得實驗組與空白組之間表達差異的基因。
聚類分析是根據不同條件下差異基因TPM值的表達水平,作層次聚類分析,用于判斷DEGs在不同條件下的表達模式。將篩選獲得的差異表達基因在GO數據庫和KEGG數據庫中進行比對,然后根據比對結果進一步分析基因產物的功能及在細胞中的代謝途徑。
2 結果與分析
2.1 RNA樣品質量分析
轉錄組測序的RNA樣品質量要求OD260/OD280值應為1.8~2.2、OD260/OD230值>2時核酸的純度較高,RIN為1~10,數值越接近10完整性越好[28]。由圖1及表1可知,6個樣品RNA的純度、濃度和完整度等質量指標完全符合后續轉錄組測序的要求,因此可以構建cDNA文庫,進行上機測序。
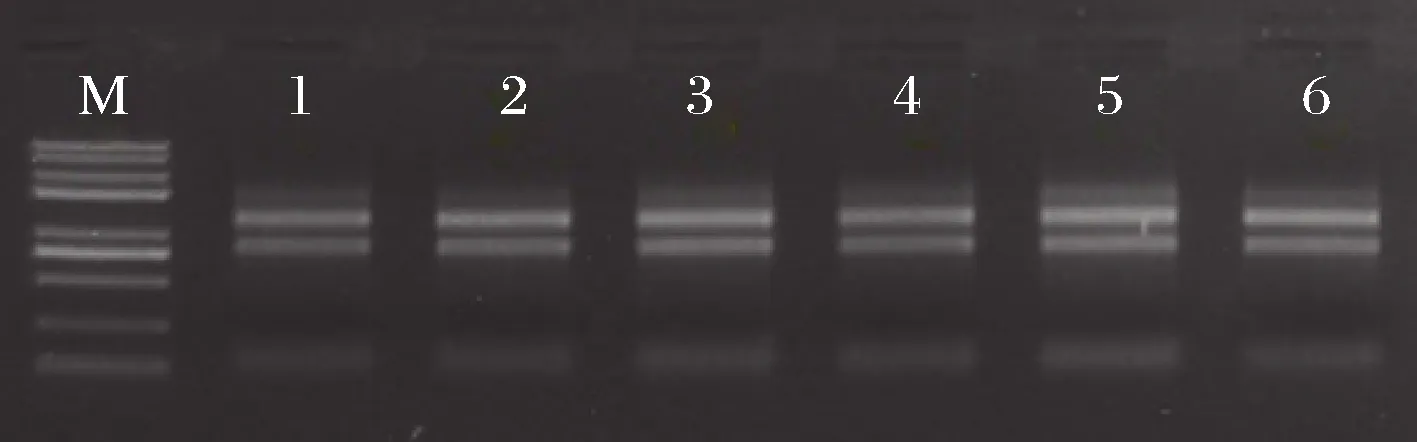
M-marker;1~6-樣品Kb1、Kb2、Kb3、Se1、Se2、Se3圖1 富硒酵母RNA樣品瓊脂糖凝膠電泳圖Fig.1 Agarose gel electrophoretogram of Se-enriched yeast RNA samples
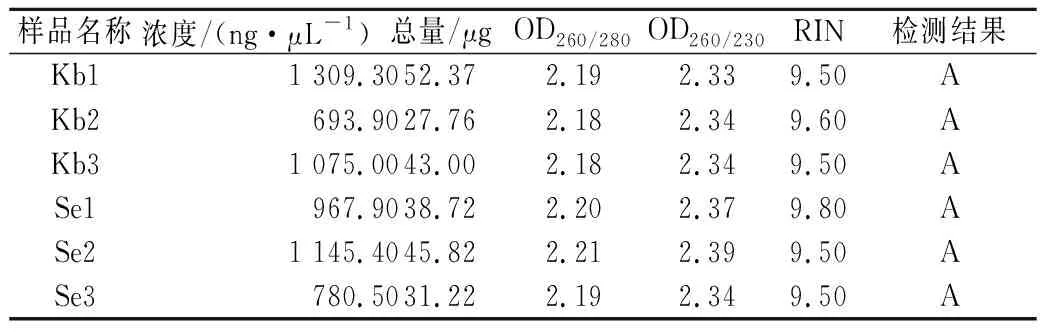
表1 釀酒酵母RNA樣品質量檢測結果Table 1 Quality test results of RNA samples of saccharomyces cerevisiae
2.2 測序數據質量評估與分析
為探究釀酒酵母菌在不同硒濃度培養條件下的變化機制,首先對其進行轉錄組測序,共獲得261 794 672條原始序列,然后對原始序列進行過濾處理,共獲得259 164 884條干凈序列。由表2可知,Kb組和Se組的Q20平均值分別為98.90%和98.89%,Q30平均值分別為96.15%和96.12%,此外GC相對含量分別為42.48%和42.23%。因此可知,轉錄組測序數據質量好,準確性高,可用于后續分析。
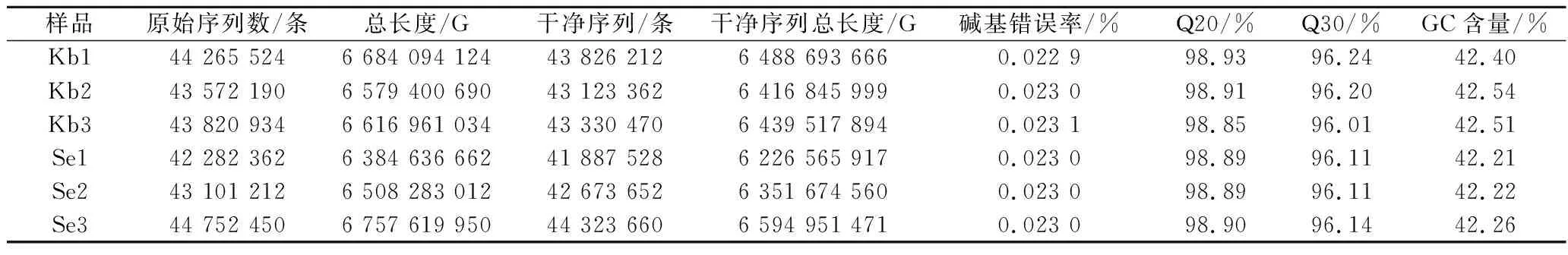
表2 測序數據統計表Table 2 Statistical table of sequencing data
2.3 參考序列比對分析
2.3.1 比對結果統計
本研究選取NCBI中釀酒酵母菌的基因組作為參考基因組。通常情況下,如果參考基因組選擇合適,而且相關實驗不存在污染,實驗所產生的測序干凈序列能定位到基因組上的比率通常會高于65%。本研究用Smalt軟件對高質量數據進行比對,其結果見表3。
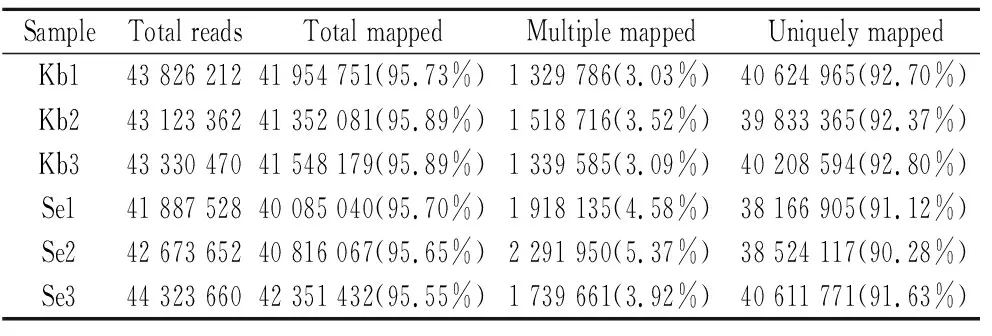
表3 比對結果統計表Table 3 Statistical table of comparison results
由表3可知,Kb組和Se組轉錄組測序獲得的干凈序列中,能定位到基因組上的干凈序列片段平均值分別為41 084 180條和41 618 337條,占比為95.55%~95.89%,在參考序列上有多個比對位置的干凈序列片段平均值分別為1 396 029條和1 983 249條,占比為3.03%~5.37%,在參考序列上有唯一比對位置的干凈序列片段平均值分別為40 222 308條和39 100 931 條,占比為90.28%~92.8%。其中能定位到基因組上的干凈序列片段數均大于95%,而在參考序列上有多個比對位置的干凈序列片段數均小于10%,因此本次研究所測序列沒有受到污染。
2.3.2 測序飽和度分析
轉錄本的表達水平不同,準確定量所需的測序深度也有所區別,在低表達的轉錄本中,為了保證定量的準確性,往往需要更大的測序深度,因而,可以通過飽和度曲線來評估不同測序深度條件下不同表達水平轉錄本是否被準確定量。本研究采用RSeQC-2.3.6軟件對6個樣品的測序飽和度進行分析,其結果如圖2所示。

a-Kb1飽和度;b-Kb2飽和度;c-Kb3飽和度;d-Se1飽和度;e-Se2飽和度;f-Se3飽和度圖2 測序飽和度曲線圖Fig.2 The curve map of sequencing saturation
通常情況下,當TPM值>3.5的基因,在測序序列的40%比對上時接近飽和,即縱軸數值趨近于1,說明飽和度總體質量較高,且轉錄本表達水平越高相對誤差越小。由圖2可知,本研究中所有測序量能夠覆蓋絕大多數的表達基因,因此,該測序深度條件下轉錄本能夠被準確定量。
2.3.3 測序覆蓋度分析
測序覆蓋度是用來評估測序結果的均一性,它是針對樣品中所有基因的5′~3′區域上序列覆蓋情況的綜合分析。本研究采用RSeQC-2.3.6軟件對本研究的6個樣品進行分析,其結果如圖3所示。其中橫坐標為單個基因的堿基長度占總堿基長度的百分比,縱坐標為比對到所有基因的橫軸位置上相應區間內的序列條數的總和。由圖3可知,該測序所得序列在基因上均勻分布,且測序無偏向性。
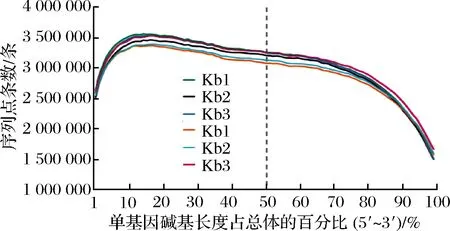
圖3 測序覆蓋度分布圖Fig.3 The distribution map of sequencing coverage
2.3.4 不同區域序列分布
將比對到基因組上的序列在參考基因組不同區域的分布情況進行統計,主要定位區域為5個,即編碼區(coding sequence,CDS)、內含子(intron)、基因間區(intergenic region)和5′和3′非翻譯區(untranslated region,UTR)。如果測序序列被定位到內含子上,通常情況是因為注釋不完全的基因組或是有非成熟的mRNA污染。如果測序序列被定位到基因間隔區域時,通常是因為背景噪音或者注釋不完全的基因組。
圖4分別為每個樣品的序列在參考基因組不同區域的分布情況,可以看出6個樣品的序列定位到編碼區域最多,分別為98.27%、97.82%、98.23%、97.94%、96.93%和98.80%;定位到內含子區域分別為0.13%、0.13%、0.15%、0.15%、0.15%和0.15%;定位到基因間隔區域分別為0.05%、0.05%、0.05%、0.06%、0.06%和0.06%;定位到5′和3′非翻譯區域分別為1.55%、2.01%、1.58%、1.85%、2.87%和0.98%。
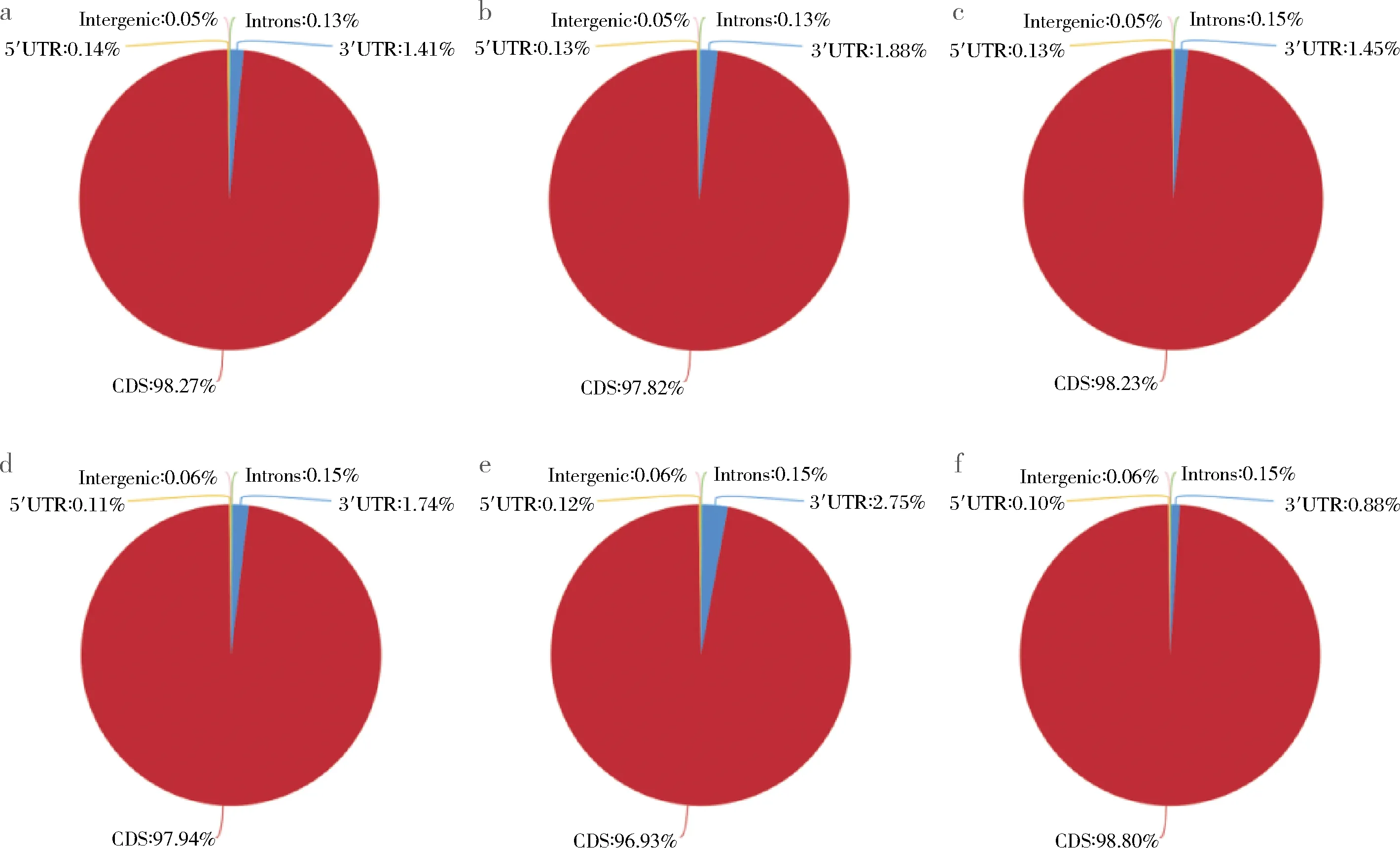
a-Kb1;b-Kb2;c-Kb3;d-Se1;e-Se2;f-Se3圖4 不同區域Reads分布統計餅圖Fig.4 Statistical pie charts of Reads distribution in different regions
2.3.5 不同染色體序列分布
為了從宏觀上了解所測序列在各染色體上的分布情況,因此將比對到基因組上的序列在參考基因組不同染色體上的分布情況進行統計。圖5為6個樣品比對到基因組上的序列在不同染色體上的分布統計柱狀圖,其中橫坐標為不同染色體的名稱,縱坐標為序列在染色體上的數量。由圖5可知,釀酒酵母菌富硒后,NC-001136.10號染色體的序列數變化較大,而其他染色體的變化較小。
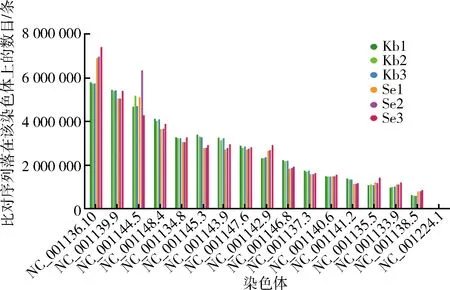
圖5 不同染色體序列分布統計柱狀圖Fig.5 Statistical histogram of reads distribution on different chromosomes
2.4 轉錄組功能注釋
將釀酒酵母菌組裝轉錄本分別在GO、KEGG、COG、NR、Swiss-Prot和Pfam各大數據庫的比對結果進行統計(見表4)。其中有1 401個(21.74%)Unigene被注釋到GO數據庫,有3 665個(56.87%)Unigene被注釋到KEGG數據庫,有5 630個(87.35%)Unigene被注釋到COG數據庫,有6 112個(94.83%)Unigene被注釋到NR數據庫,有6 077個(94.29%)Unigene被注釋到Swiss-Prot數據庫和有5 059個(78.49%)Unigene被注釋到Pfam數據庫。

表4 功能注釋統計表 單位:%Table 4 Functional annotation statistics table
2.5 樣本間相關性分析
在研究過程中,樣品之間基因表達水平相關性通常用皮爾遜相關系數的平方(R2)來表示,相關系數R2的值為0~1,越接近1,表明樣品之間的相關性越大,即相似度越高。由圖6可知,本研究中6個樣品之間的相關系數R2的最小值為0.943,故本研究中結果可靠,同時對樣品的選擇也合理。

圖6 樣品間相關性熱圖Fig.6 Heat map of correlation between samples
2.6 基因表達水平分析
2.6.1 差異表達基因篩選
同一生物體在不同時間和環境條件下,其某些基因的表達會存在顯著性差異。同樣,釀酒酵母菌從無硒環境到有硒環境中,主要通過調節機體內的某些基因的表達,來適應有硒環境。因本研究中有生物學重復的樣品,其基因差異分析應使用Bioconductor軟件包的DESeq2(V1.6.3)進行分析[29-30],設置P≤0.05,上/下調差異倍數為≥2.0倍。共篩選出差異表達的基因994個,其中有498個基因表現為下調,占總差異表達50.10%,有496個基因表現為上調,占總差異表達49.90%,其結果分析見差異基因火山圖及散點圖(圖7)。
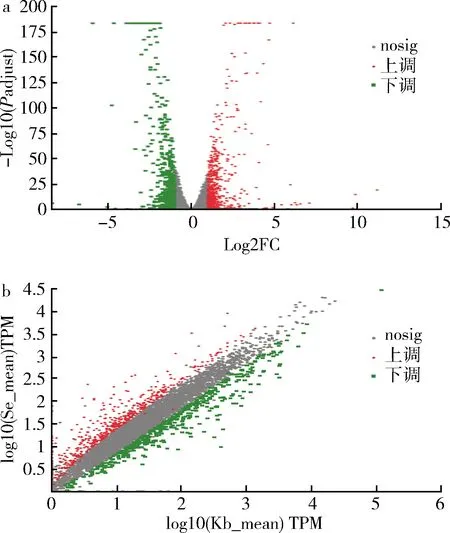
a-火山圖;b-散點圖圖7 表達量差異火山圖及散點圖Fig.7 Expression difference volcanic map and scatter map
通過對基因在各樣品中的表達進行聚類,通常表達模式相似的基因具有功能相關性,因此,可以根據聚類分析來判斷差異基因在不同實驗條件下的表達模式,從而推斷未知基因的功能。本研究采用差異分析軟件DESeq2進行分析,以P≤0.05,上/下調差異倍數為≥10.0倍進行篩選,得到55個差異顯著基因,然后繪制基因熱圖,結果如圖8所示。
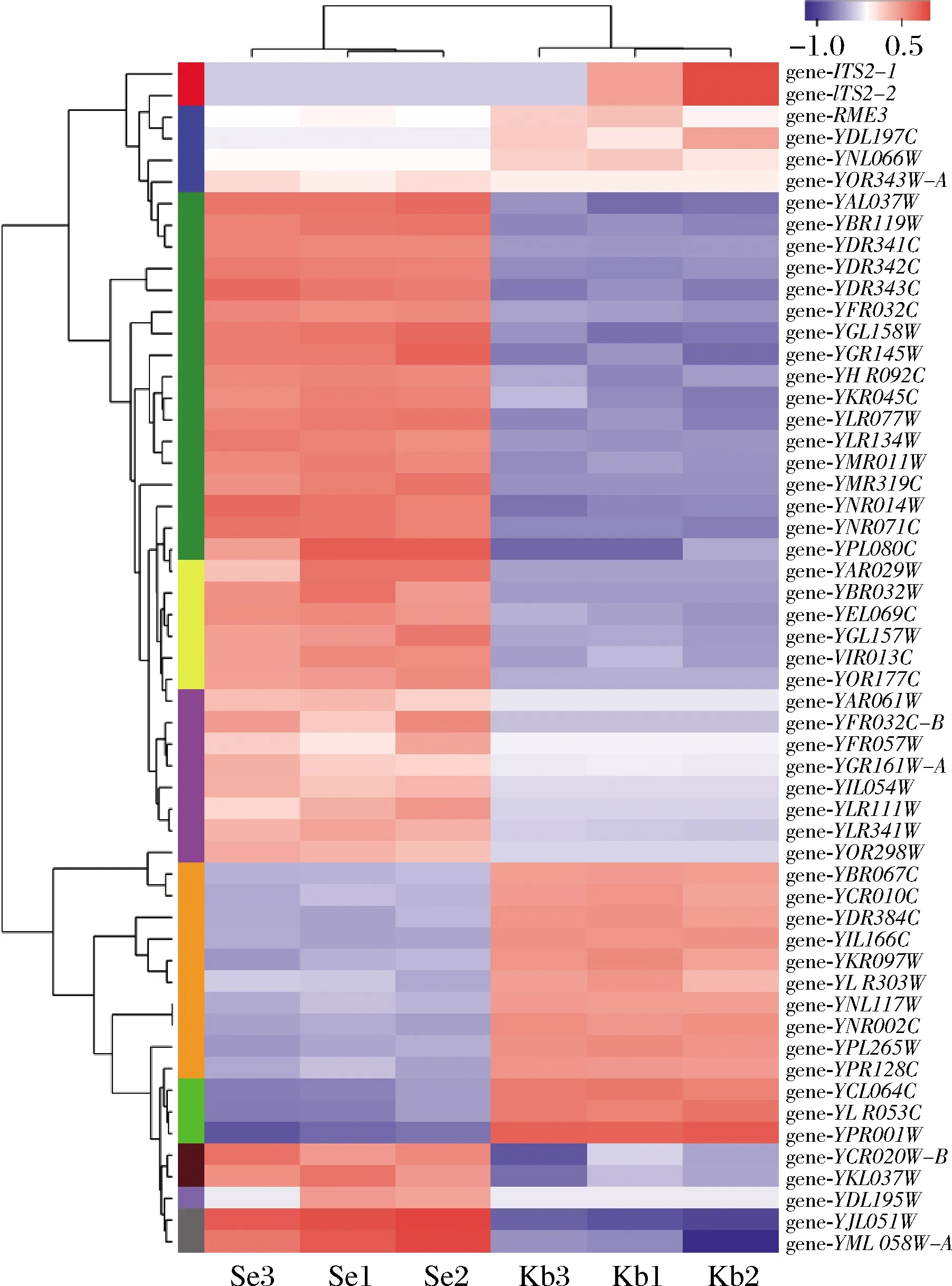
圖8 基因聚類熱圖Fig.8 Heat map of gene cluster
2.6.2 差異基因GO富集分析
對篩選出來的差異表達基因進行GO富集,可以查看其參與的生物學過程、構成細胞的組分及分子功能等信息,因此,對差異表達基因進行GO功能顯著性富集分析,可以得到差異表達基因與哪些生物學功能顯著相關。
釀酒酵母菌在不同硒質量濃度(0、20 μg/mL)培養條件下的差異基因GO富集分類結果如圖9所示,結果顯示共分為41個功能組,其中,生物過程(biological process,BP)有17個功能亞類,主要注釋為細胞過程、代謝過程、單一生物過程、細胞成分的組織或生物發生等亞類;細胞組成有13個功能亞類,主要注釋為細胞、細胞部分、細胞器、細胞器部分、大分子復合物等亞類的組成;分子功能有11個功能亞類,主要注釋為催化活性、捆綁等亞類。

圖9 GO分類統計柱形圖Fig.9 Statistical histogram of GO classification
2.6.3 差異基因KEGG富集分析
生物體內的生物學功能主要通過不同基因的相互協調而發揮作用,通過對差異表達基因進行通路顯著性分析,可以找出差異表達基因相對于所有有注釋的基因顯著富集的通路,同時還可以確定差異表達基因參與的最主要生化代謝途徑和信號轉導途徑。
釀酒酵母菌在不同硒濃度(0、20 μg/mL)培養條件下的差異基因KEGG富集氣泡圖是KEGG富集分析結果的可視化方式。縱坐標表示通路名稱,橫坐標表示比值,其值越大,該代謝通路富集的程度越大,而氣泡的大小表示此通路中基因數量,氣泡的顏色對應于不同的q值范圍。本研究挑選最顯著的20條富集基因進行展示。由圖10可知,富集到“減數分裂-酵母”通路上的基因共有38個,呈顯著富集;其次是“糖酵解/糖異生”、“乙醛酸和二羧酸酯代謝”、“過氧化物酶體”、“淀粉和蔗糖代謝”與“氨基糖和核苷酸糖代謝”通路上的基因分別有24、19、19、17、16個;富集到“半乳糖代謝”與“丙酮酸代謝”通路上的基因均為14個;富集到“甲烷代謝”通路上的基因有12個;富集到“脂肪酸降解”通路上的基因有11個;富集到“果糖和甘露糖代謝”通路上的基因有10個;富集到“谷胱甘肽代謝”與“甘油脂代謝”通路上的基因均有9個;富集到“脂肪酸生物合成”與“丙酸酯代謝”通路上的基因均為7個;富集到“纈氨酸,亮氨酸和異亮氨酸的降解”與“β-丙氨酸代謝”通路上的基因均為6個;富集到“不飽和脂肪酸的生物合成”與“戊糖和葡萄糖醛酸酯的相互轉化”通路上的基因均為5個;富集到“抗壞血酸和藻酸鹽代謝”通路上的基因為3個。

圖10 KEGG富集分析氣泡圖Fig.10 Bubble map of KEGG enrichment analysis
3 結論
本研究采用Illumina HiSeq測序平臺,對釀酒酵母菌在不同硒質量濃度(0、20 μg/mL)培養條件下的轉錄組進行測序分析,測序質量良好。其中對照組是以不富硒的釀酒酵母菌為材料,共獲得131 658 648條原始序列,經過過濾后得到130 280 044條干凈序列;實驗組是以富硒的釀酒酵母菌為材料,共獲得130 136 024條原始序列,經過過濾后得到128 884 840 條干凈序列。基于P<0.05 & |log2FC|≥2為篩選條件,2組樣品之間共篩選獲得994個顯著性差異表達基因,其中有496個基因表達量上調,有498個基因表達量下調。通過差異基因GO富集分析結果可以明確釀酒酵母菌在不同硒濃度培養條件下的細胞組分、生物學過程和分子功能三大分類中的差異表達基因。最后對差異表達基因進行KEGG富集分析,確定了差異表達基因的顯著富集通路,同時找出了差異表達基因參與的最主要生化代謝途徑和信號轉導途徑,最終以氣泡圖的形式對分析結果進行展示。結果表明2組樣品的差異表達基因KEGG代謝通路中顯著富集的通路有:減數分裂-酵母、糖酵解/糖異生、乙醛酸和二羧酸酯代謝、過氧化物酶體、淀粉和蔗糖代謝、氨基糖和核苷酸糖代謝、硫代謝、半胱氨酸和蛋氨酸代謝、谷胱甘肽代謝及硒化合物代謝等。研究結果為進一步分析釀酒酵母菌富硒基因的挖掘和研究提供了一定理論參考及科學依據。

