小麥Glu-3位點基因拷貝數的變異分析
陳璨,韓南南,劉洋,史曉維,司紅起,馬傳喜
小麥位點基因拷貝數的變異分析
陳璨,韓南南,劉洋,史曉維,司紅起,馬傳喜
安徽農業大學農學院/農業農村部黃淮南部小麥生物學與遺傳育種重點實驗室,合肥 230036
【】基因拷貝數變異是一種常見又重要的基因結構變異,往往影響個體表型。低分子量麥谷蛋白(low-molecular-weight glutenin subunit,LMW-GS)是小麥貯藏蛋白的主要組成部分,位于位點。小麥作為異源六倍體,其龐大且復雜的基因組結構導致難以利用傳統方法檢測目的基因的拷貝數,針對小麥基因組,篩選可靠穩定的內參基因和體系,探索適合復雜基因組的拷貝數變異測定技術,測定位點LWM-GS基因拷貝數。以為內參基因,根據基因序列設計內參引物和探針,通過定性和定量PCR測定內參基因在12個普通小麥品種中的拷貝數,分析該基因拷貝數在不同品種間的穩定性;又以小麥品種篙優2018的5個稀釋濃度的基因組DNA為模板,利用qRT-PCR驗證內參系統的重復性和準確性;根據位點LMW-GS基因序列設計特異性引物及探針,利用qRT-PCR和ddPCR 2種方法檢測8個小麥品種位點基因拷貝數,比較后選擇更優的高通量基因拷貝數檢測方法;再根據和位點LMW-GS基因序列設計相應的特異性引物及探針,并利用ddPCR技術檢測和分析了231份小麥品種的、和位點上LMW-GS基因拷貝數。在12個普通小麥品種間、同一品種5個DNA稀釋濃度間的拷貝數測定結果一致,技術重復間的變異系數僅為0.07%—0.77%,所構建的內參系統穩定;比較qRT-PCR和ddPCR 2種拷貝數檢測方法,8個品種所測的位點拷貝數結果一致,分別為3、5、3、4、3、3、3和3;且ddPCR檢測重復間的變異系數為0.30%—1.67%,遠低于qRT-PCR的3.14%—12.72%,更加可靠;利用ddPCR對231份普通小麥品種的、和位點上LMW-GS基因拷貝檢測后分析發現,大多數小麥品種在3個位點上的拷貝數為4,所占頻率分別為51.95%、32.03%和28.57%,位點總拷貝數變異范圍為10—21,變異系數為16.12%。內參系統具有良好的穩定性和重復性,可以用作小麥位點和其他目的基因拷貝數檢測的內參;qRT-PCR和ddPCR均可用于小麥基因拷貝數的檢測,但后者更穩定、可靠,且操作簡單、檢測通量高。
小麥;低分子量麥谷蛋白;微滴式數字PCR;拷貝數變異
0 引言
【研究意義】小麥低分子量麥谷蛋白(low molecular weight glutenin subunit,LMW-GS)約占貯藏蛋白的1/3[1],其組成和結構對小麥面粉品質具有重要影響,尤其是面團面筋強度,并決定面團的粘彈性[2-3]。研究表明,LMW-GS的編碼基因大部分定位于小麥第一同源群染色體1A、1B和1D短臂近端粒處,依次被命名為、和位點,每個位點都包含著多個LMW-GS基因,構成了非常復雜的LMW-GS基因家族[4-6]。LMW-GS基因的組成、拷貝數及其表達情況都影響著小麥品質性狀[7-9]。但由于LMW-GS基因組成復雜,目前缺少有效的分離方法,很大程度上限制了對其拷貝數的研究。因此,探索和構建高通量檢測拷貝數變異的方法尤為重要。【前人研究進展】基因的拷貝數變異(copy number variation,CNV)是一種常見而又重要的基因結構變異,一般認為是由染色體重排等染色體結構變異引起的,會導致基因組遺傳不穩定,對個體性狀會產生一定的影響[10-11]。對于拷貝數的檢測,常用的技術主要包括實時熒光定量PCR(quantitative real-time PCR,qRT-PCR)、熒光原位雜交(fluorescence in situ hybridization,FISH)、Sothern印跡雜交(southern blot)、短片段多重定量PCR(quantitative multiplex PCR of short fragments,QMPSF)以及數字PCR(digital PCR,dPCR)技術。20世紀末,Vogelstein等[12]提出了dPCR的概念,即將有限稀釋法、泊松分布和PCR技術聯合使用的新方法[13-14]。一種新的dPCR分析系統——微滴式數字PCR(droplet digital PCR,ddPCR),用于檢測目的基因的拷貝數,可以使用熒光探針對2個靶基因同時進行檢測,先使用微滴生產儀將一個樣本分成幾萬份(>10 000),分配到不同的微滴單元,每個單元包含一個或多個拷貝數的目標分子(DNA模板),在每個反應單元中分別對目標分子進行PCR擴增,擴增結束后利用微滴分析儀對各個反應單元的熒光信號進行統計學分析,最后根據泊松分布原理及陽性微滴的個數與比例得出靶分子的起始拷貝數或濃度[15-16]。在利用ddPCR進行基因拷貝數檢測時需要一個已知的單拷貝或低拷貝基因作為內參基因,乙酰輔酶A羧化酶(acetyl-CoA carboxylase,ACCase)是植物脂肪酸生物合成中的一種限速酶[17-19]。在禾本科作物基因組中,同時含有編碼質體ACCase()和胞質ACCase()的基因[20-21],其中,被定位于2A、2B和2D染色體靠近端粒的短臂上,且Southern印跡雜交試驗表明普通小麥中在每個染色體組都是單拷貝[22]。因此,可以作為檢測小麥中其他基因拷貝數的內參基因。【本研究切入點】由于六倍體小麥為異源多倍體作物,其基因組龐大且復雜,利用傳統的Sothern印跡雜交等技術檢測小麥基因拷貝數費時費力,并且無法達到高通量測定要求。【擬解決的關鍵問題】本研究擬選擇適當內參基因,通過ddPCR技術構建一種高通量、快速且穩定的小麥基因拷貝數測定方法;并通過構建的內參體系和方法測定普通小麥位點LMW-GS基因拷貝數。
1 材料與方法
1.1 材料
231份不同品種六倍體小麥秋播于合肥高新技術農業園試驗站,每個材料種植2行,行長2 m,行距25 cm,田間管理同大田管理。
1.2 內參的選擇
構建可靠的CNV檢測方法需要選擇特異性的DNA序列作為內參基因,并且需要滿足3個要求:具有物種特異性,具有單個或穩定的低拷貝數,以及不同品種之間拷貝數相同[23-24]。通過查閱文獻,選擇作為內參基因,從NCBI網站上下載已知序列,在保守區域設計引物和探針,構建內參系統。
1.3 定性PCR條件
采用全式金生物公司的TransTaq?Hifi酶在T100TMThermal Cycler (BIO-RAD Laboratories,USA)熱循環儀進行定性PCR分析,反應體系為2.0 μl樣本DNA、0.5 μl上游引物(10 μmol·L-1)、0.5 μl下游引物(10 μmol·L-1)、2.0 μl dNTPs(2.5 mmol·L-1)、2.5 μl 10×Buffer、0.25 μl DNA酶,補加ddH2O至25 μl。PCR擴增程序為95℃ 5 min;95℃ 50 s,50—65℃ 30 s,72℃ 30s,40個循環;72℃ 10min;4℃保存。擴增產物用1.2%瓊脂糖凝膠電泳進行檢測。
1.4 定量PCR條件
qRT-PCR采用愚公生命科技有限公司的Taq-HS Probe qPCR Premix,在CF×96TMReal-Time System(BIO-RAD Laboratories,USA)中進行,反應體系為2.0 μl樣本DNA、1.0 μl上游引物(10 μmol·L-1)、1.0 μl下游引物(10 μmol·L-1)、1.0 μl探針(10 μmol·L-1)和1.25 μl Taq-HS Probe qPCR Premix(10×),補加ddH2O至25 μl。qPCR擴增程序為95℃ 3 min;95℃ 30 s,50—65℃ 30 s,72℃ 30 s,40個循環;在每個循環的延伸階段采集熒光信號。參考Weng等[25]報道的絕對定量法來計算不同品種中目的基因拷貝數。
1.5 基因組DNA的酶切處理
采用NEB公司的HⅠ限制性內切酶對基因組DNA進行酶切處理,50 μl的反應體系包含1 μg基因組DNA、1 μlHⅠ限制性內切酶和5 μl 10×NE Buffer,補加ddH2O至50 μl。在37℃恒溫條件下反應5 h,于4℃保存備用。
1.6 微滴式數字PCR分析基因拷貝數
先配制20×目的基因(或內參基因)引物/探針混合液,包含目的基因(或內參基因)上、下游引物(100 μmol·L-1)各18 μL和目的基因(或內參基因)探針(100 μmol·L-1)5 μL,補加ddH2O至100 μL。再采用BIO-RAD公司的ddPCR Supermix for Probes(No dUTP)制備ddPCR反應液,反應體系為10 μL 2×ddPCR Supermix for Probes(No dUTP)、1 μL 20×目的基因引物/探針混合液、1 μL 20×內參基因引物/探針混合液和30 ng酶切后基因組DNA,補加ddH2O至20 μL。
隨后將反應液通過QX200TM微滴發生器(QX200TMDroplet Generator, BIO-RAD Laboratories,USA)生成納升級別的油包水的液滴(理論上可形成20 000個微滴),緊接著轉移到96孔PCR板(Eppendorf, Germany)中,并利用熱封儀(PX1TMPCR Plate Sealer, BIO-RAD Laboratories,USA)進行封膜。然后轉移到T100TMThermal Cycler(BIO-RAD Laboratories,USA)上進行PCR擴增,擴增程序為95℃ 10 min;94℃ 30 s;50—65℃ 1 min,40個循環;98℃ 10 min;4℃保存待用,升降溫速度為2.0 ℃·s-1。
PCR擴增完成后,將其轉移到QX200TM微滴分析儀(QX200TMDroplet Reader, BIO-RAD Laboratories,USA)對所有樣品孔進行熒光檢測。最后利用QuantaSoft軟件(V1.3.10)對ddPCR數據進行分析,計算每個樣品目的基因拷貝數。
2 結果
2.1 內參基因的選擇及其引物與探針的設計
通過查閱文獻搜索到低拷貝數的候選基因質體乙酰輔酶A羧化酶基因(),在普通小麥中具有3個拷貝。從NCBI網站上下載已公布的序列,運用DNAMAN生物軟件對下載的序列進行序列對比,運用Primer express3.0軟件在的保守區域設計引物(Acc1F和Acc1R)和探針(T-Acc1)(表1)。

表1 特異性引物和探針
2.2 定性和定量PCR檢測Acc1在不同六倍體小麥中的穩定性
合適的內參基因應在同一物種的不同品種之間具有相同的拷貝數,為了檢測不同普通小麥品種之間拷貝數的種內穩定性,選取12個品種的普通小麥提取基因組DNA,每個品種各取30 ng DNA,以Acc1F、Acc1R為引物進行定性PCR擴增,結果表明,不同品種之間獲得相同大小和等效強度的PCR產物,并且沒有顯示出非特異性的條帶(圖1),表明所設計的引物能夠穩定地擴增出序列,特異性較高,并且普通小麥品種間拷貝數沒有明顯差異。
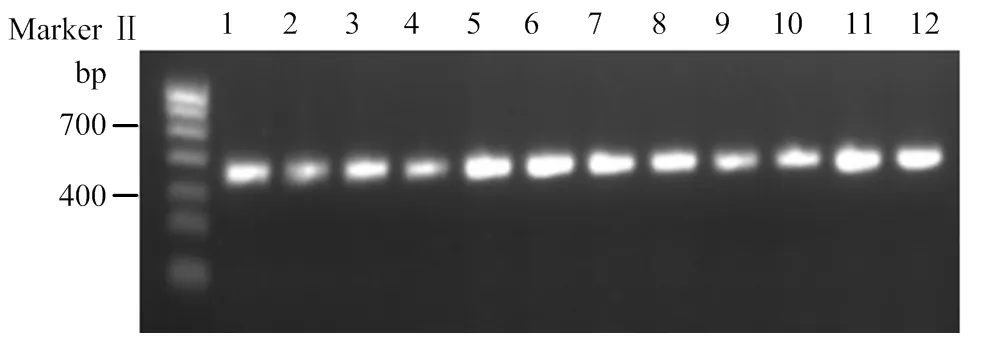
1:黔11240-2;2:鄭麥583;3:中麥14;4:德宏福麥2號;5:瑞泉麥168;6:中合-75;7:益科麥5號;8:內麥836;9:陽光838;10:安農1020;11:新麥31;12:黔090304-4
選取12個不同品種小麥50 ng基因組DNA,以Acc1F、Acc1R為引物,T-Acc1為探針進行實時熒光定量PCR,定量PCR的結果顯示不同品種的小麥擴增曲線極為相似,并且獲得的Ct值只有輕微的變異性(圖2),范圍為29.18—30.00,變異系數為1.03%,拷貝數在供試品種之間被認為是相同的。
2.3 定量PCR檢測Acc1定量系統的重復性
為了進一步驗證定量系統的準確性,選用篙優2018的5個稀釋度的基因組DNA為模板,以Acc1F、Acc1R為引物,T-Acc1為探針進行定量PCR(表2),結果表明,復孔間變異系數為0.07%—0.77%,SD值為0.0208—0.2100,由于這些值相對較小,因此,定量系統被認為是穩定且可靠的,可作為內參基因來測定其他基因的拷貝數。
2.4 Glu-3位點LMW-GS基因的引物與探針設計及其特異性分析
從NCBI網站上下載已知的分別位于、和位點上的LMW-GS基因,通過對LMW-GS基因序列進行比對,并設計相應的特異性引物與探針(表1),其中,Glu-A3T、Glu-B3T和Glu-D3T探針的5′端、3′端所連的熒光集團分別為5′-FAM和3′-BHQ1;T-Acc1探針的5′端、3′端所連的熒光集團分別為5′-HEX和3′-BHQ1。
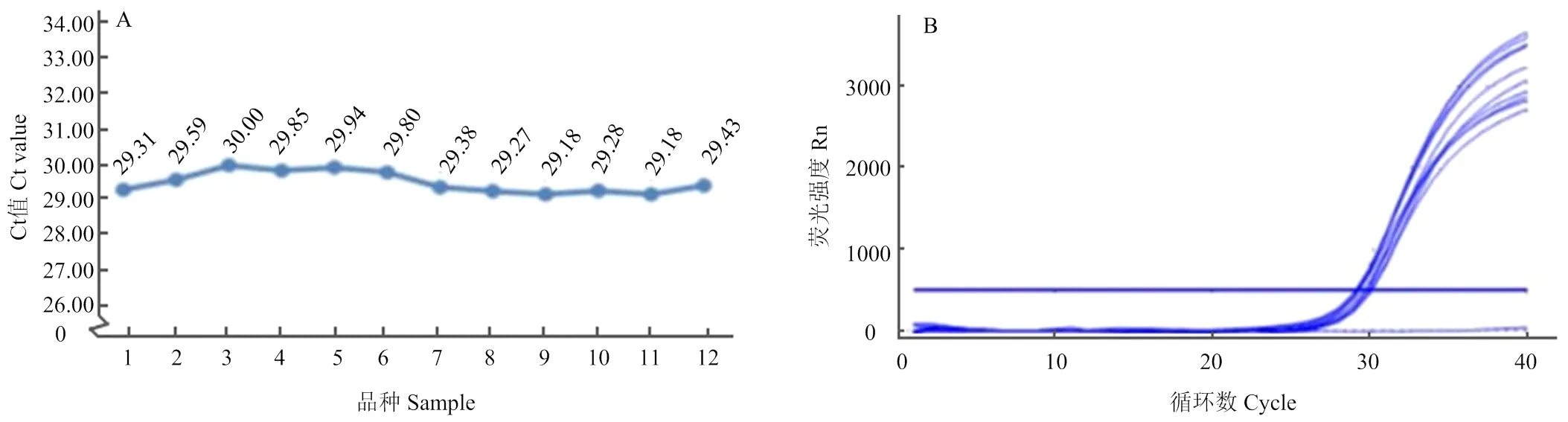
A:12個不同品種的內參基因定量PCR擴增曲線;1:黔11240-2;2:鄭麥583;3:中麥14;4:德宏福麥2號;5:瑞泉麥168;6:中合-75;7:益科麥5號;8:內麥836;9:陽光838;10:安農1020;11:新麥31;12:黔090304-4。B:12個不同普通小麥品種的Ct值

表2 內參基因重復性檢測Ct值變化情況
從普通小麥近緣物種(烏拉爾圖小麥AA、一粒小麥AA、擬斯卑爾脫山羊草BB、節節麥DD和圓錐小麥AABB)及普通小麥AABBDD中國春、安農0711中提取基因組DNA,分別用引物Glu-A3F/R、Glu-B3F/R、Glu-D3F/R進行PCR擴增,結果顯示,Glu-A3F/R只能從含A基因組的材料中擴增出目的基因,Glu-B3F/R只能從含有B基因組的材料中擴增出目的基因,Glu-D3F/R只能從含D基因組的材料中擴增出目的基因(圖3)。
2.5 2種拷貝數變異分析方法的比較
qRT-PCR與ddPCR均能對目的基因拷貝數進行測定,選用8個普通小麥品種,分別采用qRT-PCR和ddPCR 2種方法來測定普通小麥位點上的LMW-GS基因拷貝數。
以煙農19全基因組DNA(1 066.71 ng·μL-1)為模板,稀釋成6個梯度,進行qRT-PCR反應;以水作為空白對照,得出Ct值與DNA初始濃度的對數值之間的標準曲線(圖4),并得到內參基因的標準曲線方程(=-2.780+33.098,2=0.998,E=106.1%)和目的基因的標準曲線方程(=-2.823+33.993,2=0.998,E=108.9%),這兩個基因擴增曲線的相關系數(2)均接近1,且擴增效率(E)也均接近于1,表明所構建的標準曲線可以用于拷貝數的測定。
選取8個小麥品種的基因組DNA(濃度均為200 ng·μL-1),進行qRT-PCR反應,每個樣品3次重復,并且與繪制標準曲線的樣品同時擴增,擴增結束后得到8個樣品的Ct值(表3),結果顯示,8個小麥品種中拷貝數最高為5,最低為3,有6個品種的拷貝數為3,并且利用qRT-PCR測定時重復間的變異系數為3.14%—12.72%。
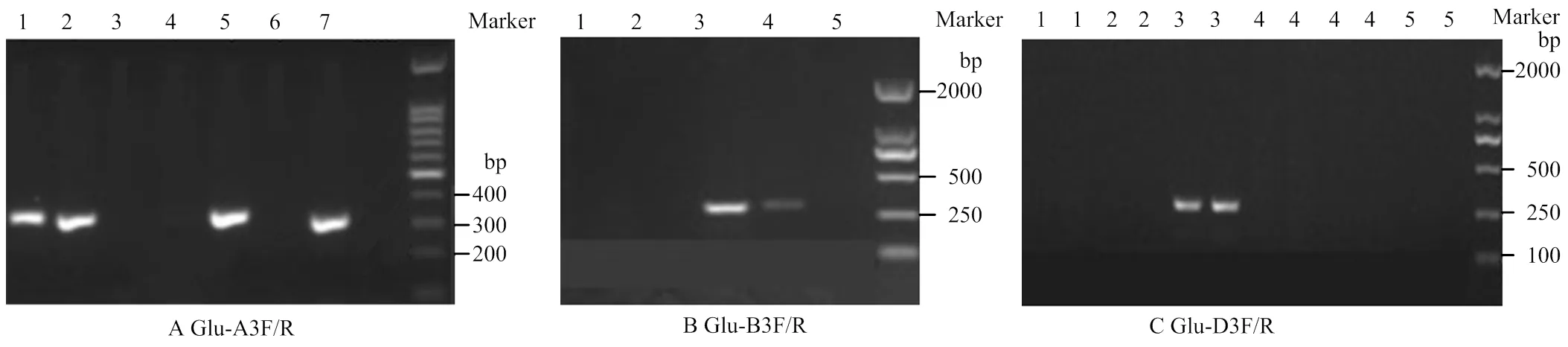
A:Glu-A3F/R引物檢測。1:中國春;2:一粒小麥;3:擬斯卑爾托山羊草;4:節節麥;5:烏拉爾圖小麥;6:水;7:安農0711。B:Glu-B3F/R引物檢測。1:一粒小麥;2:節節麥;3:圓錐小麥;4:中國春;5:水。C:Glu-D3F/R引物檢測。1:烏拉爾圖小麥;2:擬斯卑爾托山羊草;3:節節麥;4:圓錐小麥;5:水

表3 8個不同樣品3次重復的Ct值及目的基因拷貝數變化情況

圖4 目的基因和內參基因標準曲線
采用ddPCR來測定上述8個不同小麥品種的拷貝數。限制性內切酶HⅠ酶切基因組DNA(濃度為200 ng·μL-1)后,再按照ddPCR操作步驟進行試驗,最后得出了8個品種的拷貝數測定結果(圖5),其中,安農92484W拷貝數最高為5,小偃6號拷貝數為4,其余6個品種的拷貝數為3,且重復間的變異系數僅在0.30%—1.67%。
綜上所述,利用qRT-PCR和ddPCR 2種方法對8個樣品目的基因拷貝數的檢測結果是一致的,但是ddPCR重復間的變異系數要遠低于qPCR,表明用ddPCR方法來測定基因拷貝數更穩定、準確。

1:煙農15;2:安農92848W;3:百農64;4:小偃6號;5:周麥16;6:02P67;7:邯鄲6172;8:周麥18
2.6 微滴式數字PCR測定普通小麥Glu-3位點LMW-GS拷貝數
利用微滴式數字PCR對231份不同品種的普通小麥的、和位點LMW-GS基因分別進行拷貝數檢測(電子附表1),對檢測結果進行了統計分析(圖6),結果顯示,在231份材料中,大多數小麥品種的位點LMW-GS基因拷貝數為4,所占頻率51.95%,最高拷貝數為8,最低為3,不同品種間拷貝數變異系數為28.40%;大多數小麥品種的位點LMW-GS基因拷貝數為4,所占頻率為32.03%,拷貝數最高為9個拷貝,最低為3個拷貝,不同品種間拷貝數變異系數為29.30%;大多數小麥品種的位點LMW-GS基因拷貝數為4,所占頻率為28.57%,拷貝數最高為9個拷貝,最低為3個拷貝,不同品種間拷貝數變異系數為30.07%。
綜合、和3個位點的LMW-GS基因拷貝數測定結果,可以計算出小麥位點基因的總拷貝數(圖7)。結果顯示,在231份材料中,大多數小麥品種的位點LMW-GS基因拷貝數為12—18,所占頻率為85.90%,拷貝數最高為21,最低為10,不同品種間拷貝數變異系數為16.10%。
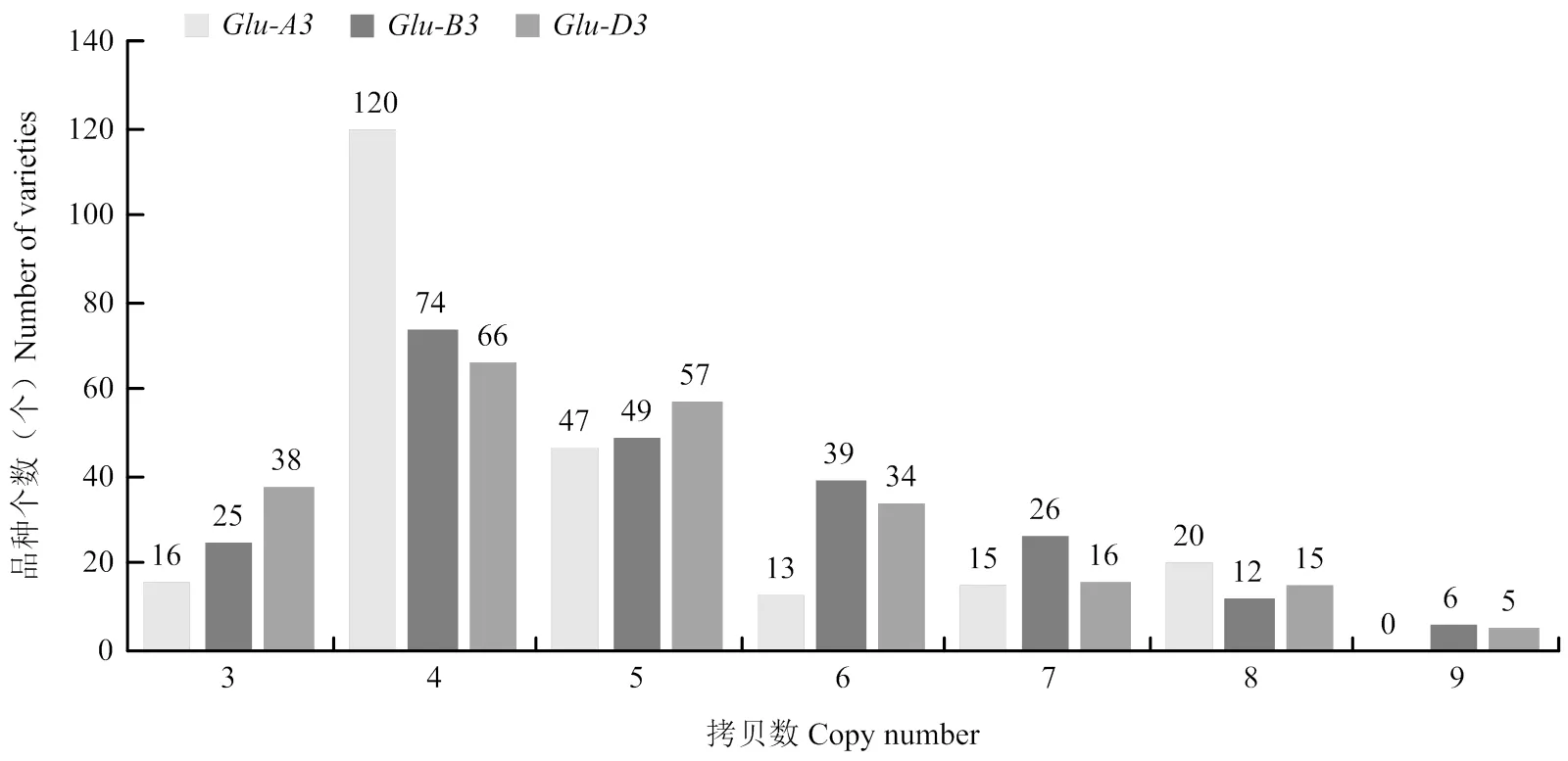
圖6 231份小麥品種Glu-A3、Glu-B3和Glu-D3位點基因拷貝數頻數分布條形圖
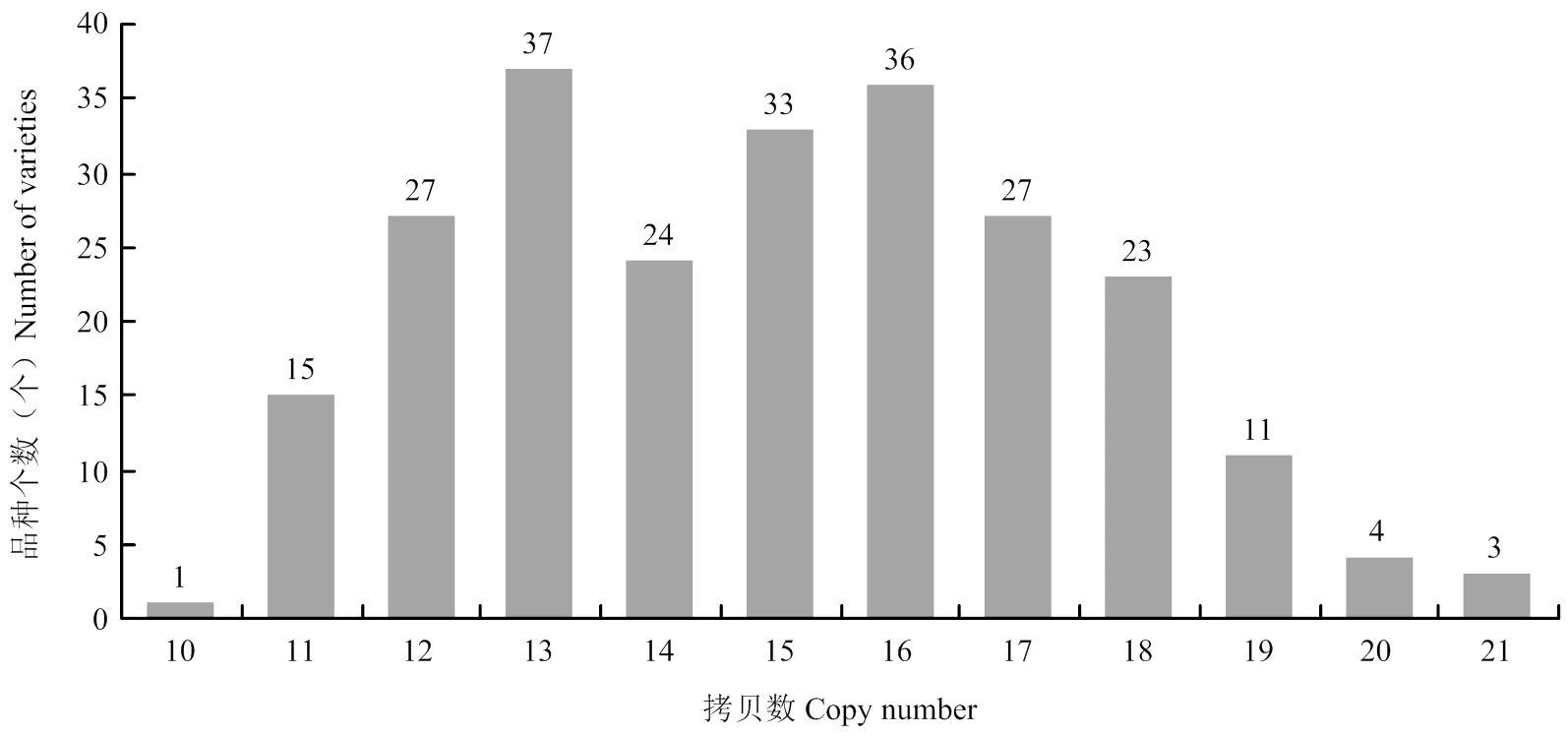
圖7 231份小麥品種Glu-3位點基因拷貝數頻數分布條形圖
3 討論
微滴式數字PCR(ddPCR)技術是近幾年迅速發展起來的第三代PCR技術,已被越來越多地應用于生命科學研究中,比如致病微生物檢測[26]、稀有突變檢測[27]、基因表達分析[28]、基因拷貝數變異分析[29]等。在利用ddPCR來測定目的基因拷貝數時需要一個已知拷貝數的單拷貝或低拷貝基因作為內參基因。一些研究以單拷貝的、和作為內參基因來檢測轉基因小麥外源基因拷貝數[30]。而本研究所選擇的內參基因為質體乙酰輔酶A羧化酶(plastid ACCase)基因()。Gornicki等[31]利用中國春缺四體材料將定位在了第二同源群2A、2B和2D染色體靠近端粒的短臂上,并利用Southen印跡雜交分析得出六倍體小麥中在每個染色體組都為單拷貝,因此,在六倍體小麥中有3個拷貝。作為內參基因,還要求其在不同的品種中拷貝數是穩定的,并且內參基因的擴增還要具有重復性。本研究借鑒Mayu等[32]的研究方法,用不同品種相同濃度的DNA驗證了內參基因拷貝數的穩定性,并用相同品種不同濃度的DNA驗證了內參基因擴增的可重復性。因此,本研究設計的內參系統(Acc1F/R和T-Acc1)可作為檢測六倍體小麥中目的基因拷貝數的內參引物和探針。
目前,研究植物基因拷貝數的方法有很多,但利用ddPCR技術來檢測小麥目的基因拷貝數的報道很少。經典的植物基因拷貝數檢測方法主要有Southern印跡雜交和qRT-PCR 2種[33-34]。其中傳統的Sounthern印跡雜交技術對DNA樣品的質量和純度有一定的要求,其試驗周期長,雜交步驟繁瑣,并且對試驗技術條件要求較高;可能因存在等位基因,而導致檢測結果不能真實反映該基因的拷貝數;另外,試驗中所用的探針標記若采用同位素標記,對人體健康和環境安全存在嚴重威脅[35]。qRT-PCR檢測方法的步驟較為繁瑣,其需要準備標準DNA樣品,再利用標準DNA樣品繪制標準曲線,標準曲線的建立又需對反應條件和反應體系進行不斷摸索與優化,所需試驗周期較長[36];借助標準曲線進行定量本身就是一種相對定量的方法,檢測結果可能不準確[37]。而ddPCR作為一種新興的、準確的絕對定量技術,與qRT-PCR相比,ddPCR的優勢非常明顯,主要體現在以下幾個方面[38-39]:(1)靈敏度可達單個核酸分子,檢測限低至0.001%;(2)無需繪制標準曲線即可對DNA樣品進行絕對定量;(3)終點PCR檢測,不依賴Ct值,不依賴擴增曲線,特別適合基質復雜樣品的檢測;(4)具有更好的準確度、精密度和重復性,可用于精確測定靶基因的拷貝數。本文以為內參基因,通過qRT-PCR和ddPCR 2種技術對8個樣品的位點LMW-GS基因進行拷貝數測定,結果基本一致,但ddPCR重復間的變異系數更小,且不依賴于標準曲線。在本研究中以為內參基因,使用特異性引物和探針系統,采用ddPCR技術分別對231份普通小麥、和位點LMW-GS基因拷貝數進行測定,3個位點的基因拷貝數分別在3—9、4—9、4—9范圍內,位點總拷貝數為10—21,這與前人通過Southen印跡雜交技術研究發現普通小麥的LMW-GS基因在總基因組中的拷貝數可能在10—15個和35—40個范圍內基本相符[40-41]。
當拷貝數可變區域的劑量敏感基因或涉及調控作用的基因發生拷貝數變異時,也會對表型產生顯著的影響[42-43]。關于基因拷貝數變異對表型的影響在一些植物性狀上已有不少報道。小麥矮稈基因()由于拷貝數的增加而導致植株高度降低,其致矮能力是單拷貝的3倍多[44]。在大麥中,硼轉運基因拷貝數的增加會使大麥Sahara具備硼毒性耐受性[45]。(C-重復結合因子)位于小麥和大麥的抗霜性位點2(),其拷貝數變異與低溫耐受性相關,冬小麥的拷貝數要高于春小麥,四倍體硬粒小麥和六倍體面包小麥的位點上大型基因的缺失(包括、和)會導致小麥耐寒性降低[46-49]。在小麥中隨著拷貝數的增加,會延長小麥春化時間導致開花推遲,而大麥中高拷貝數能夠加速開花時間[50-51]。以上研究表明,某些基因的拷貝數變異對相應的表型有著重要的影響。
小麥低分子量麥谷蛋白(LMW-GS)賦予面筋強度和延展性,在小麥的營養品質和面粉加工品質形成中具有重要影響[2-3],已有大量研究表明LMW-GS基因的組成和表達量與小麥加工品質密切相關,不同種質的小麥含有的LMW-GS等位基因不同,其編碼的LMW-GS亞基組合不同,因此對小麥面粉品質產生的影響也不同[7-9]。而關于位點LMW-GS基因拷貝數變異對小麥面粉品質的影響還未有報道。本研究將測定的231份普通小麥的、和位點LMW-GS基因拷貝數與小麥4個品質性狀進行相關性分析,除位點基因拷貝數與2012年小麥面團穩定時間和形成時間達到顯著負相關性外,其余均未達到顯著相關性,說明位點的LMW-GS拷貝數多樣性不是影響上述小麥品質性狀的主要原因。
4 結論
以為內參基因,構建了內參系統(引物和探針),它在品種間和同一品種不同濃度間均具有穩定性和重復性,可用于小麥和其他目的基因拷貝數研究。qRT-PCR和ddPCR均可用于小麥中特定基因或位點的拷貝數檢測,但相較于前者,ddPCR方法變異系數更低,且具有操作簡單、結果更準確、檢測通量高等優點。
[1] 趙獻林, 夏先春, 劉麗, 何中虎, 孫其信. 小麥低分子量麥谷蛋白亞基及其編碼基因研究進展. 中國農業科學, 2007, 40(3): 440-446.
ZHAO X L, XIA X C, LIU L, HE Z H, SUN Q X. Review on low molecular weight glutenin subunits and their coding genes. Scientia Agricultura Sinica, 2007, 40(3): 440-446. (in Chinese)
[2] XIANG L, HUANG L, GONG F Y, LIU J, WANG Y F, JIN Y R, HE Y, HE J S, JIANG Q T, ZHENG Y L, LIU D C, WU B H.Enriching LMW-GS alleles and strengthening gluten properties of common wheat through wide hybridization with wild emmer. 3 Biotech, 2019, 9(10): 355.
[3] HAZARD B, TRAFFORD K, LOVEGROVE A, GRIFFITHS S, UAUY C, SHEWRY P. Strategies to improve wheat for human health. Nature Food, 2020, 1(8): 475-480.
[4] Gupta R B, Shepherd K W. Two-step one-dimensional SDS-PAGE analysis of LMW subunits of glutelin. Theoretical and applied genetics, 1990, 80(1): 65-74.
[5] Singh N K, Shepherd K W. Linkage mapping of genes controlling endosperm storage proteins in wheat. Theoretical and Applied Genetics,1988, 75(4): 628-641.
[6] D'Ovidio R, Masci S. The low-molecular-weight glutenin subunits of wheat gluten. Journal of Cereal Science,2004, 39(3): 321-339.
[7] CHO K, JO Y M, LIM S H, KIM J Y, HAN O, LEE J Y. Overexpressing wheat low-molecular-weight glutenin subunits in rice (L.cv. Koami) seeds.3 Biotech, 2019, 9(2): 1-8.
[8] RAI A, SINGH A M, GANJEWALA D, KUMAR R R, AHLAWAT A K, SINGH S K, SHARMA P, JAIN N. Rheological evaluations and molecular marker analysis of cultivated bread wheat varieties of India. Journal of Food Science and Technology,2019, 56(4): 1696-1707.
[9] BEOM H R, KIM J S, JANG Y R, LIM S H, KIM C K, LEE C K, LEE J Y. Proteomic analysis of low-molecular-weight glutenin subunits and relationship with their genes in a common wheat variety. 3 Biotech, 2018, 8(1): 56.
[10] Shaw-Smith C, Redon R, Rickman L. Microarray based comparative genomic hybridisation (array-CGH) detects submicroscopic chromosomal deletions and duplications in patients with learning disability/mental retardation and dysmorphic features. Journal of Medical Genetics, 2004, 41(4): 241-248.
[11] Maron L G, Guimaraes C T, Kirst M, ALBERT P S, BIRCHLER J A, BRADBYRY P J, BUCKLER E S, COLUCCIO A E, DANILOVA T V, KUDRNA D, MAGALHAES J V, PINEROS M A, SCHATZ M C, WING R A, KOCHIAN L. Aluminum tolerance in maize is associated with highergene copy number. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(13): 5241-5246.
[12] Vogelstein B, Kinzler K W. Digital PCR. Proceedings of the National Academy of sciences of the United States of America, 1999, 96(16): 9236-9241.
[13] Sykes P J, Neoh S H, Brisco M J. Quantitation of targets for PCR by use of limiting dilution.Biotechniques, 1992, 13(3): 444-449.
[14] YANG Q, Xi J, CHEN X X, HU S H, CHEN N, QIAO S L, WAN S G, BAO D K. The development of a sensitive droplet digital PCR for quantitative detection of porcine reproductive and respiratory syndrome virus.International Journal of Biological Macromolecules, 2017, 104(Part A): 1223-1228.
[15] LIN Q, FU X Z, LIU L H, LIANG H R, NIU Y J, WEN Y Y, HUANG Z B, LI N Q. Development and application of a sensitive droplet digital PCR (ddPCR) for the detection of infectious spleen and kidney necrosis virus. Aquaculture, 2020, 529: 735697.
[16] ANTKOWIAK M, NOWACKA-WOSZUK J, SZCZERBAL I, SWITONSKI M, SZYDLOWSKI M. AMY2B gene copy-number variation studied by droplet digital PCR (ddPCR) in three canids: Red fox, arctic fox, and chinese raccoon dog. Folia Biologica, 2020, 68(2): 51-55.
[17] KONISHI T, SHINOHARA K, YAMADA K, SASAKI Y. Acetyl-CoA carboxylase in higher plants: most plants other than gramineae have both the prokaryotic and the eukaryotic forms of this enzyme. Plant and Cell Physiology, 1996, 37(2): 117-122.
[18] CHEN Q, SONG J, DU W P, XU L Y, JIANG Y, ZHANG J, ZHANG M, YU G R. Phylogenetic analyses of four Chinese endemic wheat landraces based on two single copy genes. Cereal Research Communications, 2018, 46(2): 191-200.
[19] 雷映霞. 鵝觀草屬及其近緣屬物種的分子系統與進化研究[D]. 雅安: 四川農業大學, 2018.
LEI Y X. Phylogenetic and evolution analysis of Roegneria and its related genera () [D]. Yaan: Sichuan Agricultural University, 2018. (in Chinese)
[20] GORNICKI P, PODKOWINSKI J, SCAPPINO L A, DIMAIO J, WARD E, HASELKORN R. Wheat acetyl-CoA carboxylase: cDNA and protein structure. Proceedings of the National Academy of Sciences of the United States of America,1994, 91(15): 6860-6864.
[21] Podkowinski J, Sroga G E, Haselkorn R, GORNICKI P. Structure of a gene encoding a cytosolic acetyl-CoA carboxylase of hexaploid wheat.Proceedings of the National Academy of Sciences of the United States of America, 1996, 93(5): 1870-1874.
[22] Gornicki P, Faris J, King I, PODKOWINSKI J, GILL B, HASELKORN R. Plastid-localized acetyl-CoA carboxylase of bread wheat is encoded by a single gene on each of the three ancestral chromosome sets. Proceedings of the National Academy of Sciences of the United States of America, 1997, 94(25): 14179-14184.
[23] 繆青梅, 汪小福, 陳笑蕓, 彭城, 徐曉麗, 魏巍, 徐俊鋒. 基于雙重微滴數字PCR精準定量轉基因水稻G6H1的方法研究. 農業生物技術學報, 2019, 27(1): 159-169.
MIAO Q M, WANG X F, CHEN X Y, PENG C, XU X L, WEI W, XU J F. Studies on accurate quantification of genetically modified rice () G6H1 based on duplex droplet digital PCR. Journal of Agricultural Biotechnology, 2019, 27(1): 159-169. (in Chinese)
[24] 蔡教英, 姚麗鋒, 王小玉, 游淑珠, 丁琦. 基于雙重微滴式數字PCR對轉基因油菜RF1品系的定量方法. 現代食品科技, 2018, 34(6): 282-287.
CAI J Y, YAO L F, WANG X Y, YOU S Z, DING Q.Quantitative analysis of genetically modified rapeseed of RF1 by duplex droplet digital polymerase chain reaction (duplex-ddPCR).Modern Food Science and Technology, 2018, 34(6): 282-287. (in Chinese)
[25] Weng H b, Pan A h, Yang L t, ZHANG C M, LIU Z L, ZHANG D B. Estimating number of transgene copies in transgenic rapeseed by real-time PCR assay with HMG I/Y as an endogenous reference gene.Plant Molecular Biology Reporter, 2004, 22(3): 289-300.
[26] Caviglia G P, Abate M L, Tandoi F, CIANCIO A, AMOROSO A, SALIZZONI M, SARACCO G M, rizzetto m, ROMAGNOLI R, smedile a. Quantitation of HBV cccDNA in anti-HBc-positive liver donors by droplet digital PCR: a new tool to detect occult infection. Journal of Hepatology, 2018, 69(2): 301-307.
[27] Dyavar S R, Ye Z, BYRAREDDY S N, SCARSI K K, WINCHESTER L C, WEINHOLD J A, FLETCHER C V, PODANY A T. Normalization of cell associated antiretroviral drug concentrations with a novel RPP30 droplet digital PCR assay. Scientific Reports, 2018, 8(1): 3626.
[28] Elmahalawy S T, Halvarsson P, Skarin M, H?GLUND J. Genetic variants in dyf-7 validated by droplet digital PCR are not drivers for ivermectin resistance incontortus. International Journal for Parasitology: Drugs and Drug Resistance, 2018, 8(2): 278-286.
[29] YU R L, XUAN W J, ZHOU L, LUO Y, LIU X Y, XIONG P W, REN X Y. Detection of HER2 amplification in formalin-fixed paraffin- embedded breast carcinoma tissue with digital PCR using two TFF3 sequences as internal reference. Experimental and Molecular Pathology, 2018, 104(3): 235-238.
[30] 琚鵬舉, 孫黛珍, 寧蕾, 葛林豪, 許成杰, 史華偉, 梁凱歌, 馬亮, 劉陶然, 陳明. 采用優化的數字PCR方法分析轉基因小麥外源基因拷貝數. 中國農業科學, 2020, 53(10): 1931-1939.
JU P J, SUN D Z, NING L, GE L H, XU C J, SHI H W, LIANG K G, MA L, LIU T R, CHEN M. Analysis of foreign gene copy number in transgenic wheat by optimized digital PCR. Scientia Agricultura Sinica,2020, 53(10): 1931-1939. (in Chinese)
[31] KAUTBALLY S, LEPROPRE S, LERIGOLEUR A, GINION A, BEAULOYE C. Platelet acetyl-coa carboxylase phosphorylation: a risk stratification marker that reveals platelet-lipid interplay in coronary artery disease patients. Archives of Cardiovascular Diseases Supplements, 2019, 11(2): 185-186.
[32] LIda M, Yamashiro S, Yamakawa H, HAYAKAWA K, KURIBARA H, KODAMA T, FURUI S, AKIYAMA H, MAITANI T, HINO A. Development of taxon-specific sequences of common wheat for the detection of genetically modified wheat. Journal of Agricultural and Food Chemistry, 2005, 53(16): 6294-6300.
[33] SINGH R, DUBEY A K, SANYAL I. Optimisation of adventitious shoot regeneration and agrobacterium-mediated transformation in Canna × generalis (). Horticultural Plant Journal, 2019, 5(1): 39-46.
[34] YIN Y C, HOU J M, TIAN S K, YANG L, ZHANG Z X, LI W D, LIU Y. Overexpressing chalcone synthase () gene enhanced flavonoids accumulation inuralensis hairy roots. Botany Letters, 2020, 167(2): 219-231.
[35] Yang L T, Ding J Y, Zhang C M, JIA j W, weng H B, LiN W X, ZHANG D B. Estimating the copy number of transgenes in transformed rice by real-time quantitative PCR.Plant Cell Reports, 2005, 23(10/11): 759-763.
[36] WHALE A S, HUGGETT J F, COWEN S, SPEIRS V, SHAW J, ELLISON S, FOY C A, SCOTT D J.Comparison of microfluidic digital PCR and conventional quantitative PCR for measuring copy number variation. Nucleic Acids Research, 2012, 40(11): e82.
[37] 姜羽, 胡佳瑩, 楊立桃. 利用微滴數字PCR分析轉基因生物外源基因拷貝數. 農業生物技術學報, 2014, 22(10): 1298-1305.
JIANG Y, HU J Y, YANG L T. Estimating the exogenous genes copy number of genetically modified organisms by droplet digital PCR.Journal of Agricultural Biotechnology,2014, 22(10): 1298-1305. (in Chinese)
[38] Gao F g, PFEIFER E, FARAH H, KARAMPINI E, DUA D, KAMAI N, CANE P, ToBAL K, SETHI T, SPICER J, MCCAUGHAN F.Microdroplet digital PCR: detection and quantitation of biomarkers in archived tissue and serial plasma samples in patients with lung cancer. Journal of Thoracic Oncology, 2015, 10(1): 212-217.
[39] Hindson C M, Chevillet J R, Briggs H A, GALLICHOTTE E N, RUF I K, HINDSON B J, VESSELLA R L, TEWARI M. Absolute quantification by droplet digital PCR versus analog real-time PCR. Nature Methods, 2013, 10(10): 1003-1005.
[40] Harberd N P, Bartels D, Thompson R D. Analysis of the gliadin multigene loci in bread wheat using nullisomic-tetrasomic lines. Molecular and General Genetics, 1985, 198(2): 234-242.
[41] Cassidy B G, Dvorak J, Anderson O D. The wheat low molecular weight glutenin genes: characterization of six new genes and progress in understanding gene family structure. Theoretical and Applied Genetics, 1998, 96(6/7): 743-750.
[42] KORBEL J O, KIM P M, CHEN X Y, URBAN A E, WEISSMAN S, SNYDER M, GERSTEIN M B. The current excitement about copy-number variation: how it relates to gene duplications and protein families. Current Opinion in Structural Biology, 2008, 18(3): 366-374.
[43] CHEN C, WANG W, YUAN J X, CHEN J, MOU L M. Analysis of HMW-GS and LMW-GS in spring wheat varieties and key parental materials cultivated in Gansu dryland. Acta Agriculturae Boreali- occidentalis Sinica, 2018, 27(11): 1598-1605.
[44] Li Y Y, Xiao J H, Wu J J, DUAN J L, LIU Y, YE X G, ZHANG X, GUO X P, GU Y Q, ZHANG L C, JIA J Z, KONG X Y. A tandem segmental duplication (TSD) in green revolution generegion underlies plant height variation. The New Phytologist, 2012, 196(1): 282-291.
[45] Sutton T, Baumann U, Hayes J E, COLLINS N C, SHI B J, SCHNURBUSCH T, HAY A, MAYO G M, PALLOTTA M A, TESTER M A. Adelaide research and scholarship: boron toxicity tolerance in barley arising from efflux transporter amplification. American Association for the Advancement of Science, 2007, 318(5855): 1446-1449.
[46] Francia E, Morcia C, Pasquariello M, MAZZAMURRO V, MILC J A, RIZZA F, TERZI V, PECCHIONI N. Copy number variation at thegenomic segment is a major component of frost resistance in barley. Plant Molecular Biology, 2016, 92(1/2): 161-175.
[47] Knox A K, Dhillon T, Cheng H M, TONDELLI A, PECCHIONI N, STOCKINGER E J. CBF gene copy number variation at Frost Resistance-2 is associated with levels of freezing tolerance in temperate-climate cereals. Theoretical and Applied Genetics, 2010, 121(1): 21-35.
[48] Pearce S, Zhu J, Boldizsár á, VáGúJFALVI A, BURKE A, KIMBERLEY G C, GáBOR G, DUBCOVSKY J. Large deletions in the CBF gene cluster at thelocus are associated with reduced frost tolerance in wheat. Theoretical and Applied Genetics, 2013, 126(11): 2683-2697.
[49] AURORA D, MELULEKI Z, ADRIAN S T, PETER L, DAVID A L. Copy number variation affecting theandgenes is associated with altered flowering time in wheat (). PloS ONE,2012, 7(3): e33234.
[50] Armour J A, Sismani C, Patsalis P C, CROSS G. Measurement of locus copy number by hybridisation with amplifiable probes. Nucleic acids research, 2000, 28(2): 605-609.
[51] NITCHER R, DISTELFELD A, Tan C T, YAN L L, DUBCOVSKY J.Increased copy number at thelocus is associated with accelerated flowering time in barley. Molecular Genetics and Genomics, 2013, 288(5): 261-275.
Analysis of copy number variation oflocus in Common wheat
CHEN Can, HAN Nannan, LIU Yang, SHI XiaoWei, SI HongQi, MA ChuanXi
College of Agronomy, Anhui Agriculture University/Key Laboratory of Wheat Biology and Genetic Improvement on Southern Yellow & Huai River Valley, Ministry of Agriculture and Rural Affairs, Hefei 230036
【】The variations in numbers of copies of a gene is a common and important gene structure variation, which often effects individual phenotype. Low-molecular-weight glutenin subunit (LMW-GS) located at theloci, is an essential part of storage protein in wheat. As a hetero-hexaploid, the huge and complex wheat genome makes it difficult to detect numbers of gene copies by classical methods. A limited information is available about CNV analysis of LWM-GS. To screen reliable and stable internal reference genes and systems for complex genome and explore the CNV determination technology suitable in different wheat varieties, the techniques used for detection of gene copy numbers, based on droplet digital PCR (ddPCR) which improve the detection flux of target gene and determine the particular numbers of gene copies ofloci.【】In this paper,was used as the internal reference gene. The internal reference primers and probes were designed by using the corresponding gene sequence. In order to analyze stability of numbers of copies ofqRT-PCR was used to analyze the repeatability and accuracy ofinternal reference system (primers and probes).The corresponding specific primers and probes were designed according to the LMW-GS gene sequence ofloci. Two different methods, qRT-PCR and ddPCR, were used to detect the numbers of copies ofloci in eight wheat varieties, in order to determine which method is more suitable for high-throughput detection ofloci gene. The specific primers and probes were also designed according to the sequences of LMW-GS gene atandloci. The numbers of copies of LMW-GS gene at,andloci of 231 varieties were determined and analyzed by ddPCR.【】The results showed that the gene copy numbers of Acc1 was consistent among varieties and different DNA concentrations of the same variety. The coefficient of variation (CV) between repeats was 0.07%-0.77%. It is also indicated from results that theinternal reference system constructed in this paper has good stability and repeatability. The results of qRT-PCR and ddPCR were consistent in detecting the numbers of copies of LMW-GS gene atloci in 8 wheat varieties, which were 3, 5, 3, 4, 3, 3, 3 and 3, respectively. However, the CV among repeats detected by ddPCR was 0.30%-1.67%, which much lower than that by qRT-PCR. It showed that using ddPCR method to detect the gene copy numbers ofloci gene is more stable and reliable. The numbers of copies of LMW-GS gene were 4 at,andhaving a frequency of 95%, 32.03% 28.57% respectively in 231 wheat varieties. The total variation range in numbers of copies ofwas 10-21, and the CV was 16.12%.【】With good stability and repeatability,could be used as internal reference gene for numbers of gene copies detection in wheat. Both qRT-PCR and ddPCR could be used in gene copy numbers detection with wheat genes, but the ddPCR is more simple, stable, reliable and has high detection flux.
wheat; low-molecular-weight glutenin subunit (LMW-GS); droplet digital PCR (ddPCR); copy number variation (CNV)

10.3864/j.issn.0578-1752.2021.06.002
2020-07-28;
2020-09-27
國家重點研發計劃(2017YFD0100804,2016YFD0101802)、國家小麥產業技術體系專項(CARS-03)、安徽高校協同創新項目(GXXT-2019-033)、江蘇現代作物協同創新中心項目(JCIC-MCP)
陳璨,E-mail:chencan-L@163.com。韓南南,E-mail:1565635050@qq.com。陳璨和韓南南為同等貢獻作者。通信作者司紅起,E-mail:sihq2002@163.com。通信作者馬傳喜,E-mail:machuanxi@ahau.edu.cn
(責任編輯 李莉)

