基于DNA 折紙模板的鐵原子陣列構建及其信息加密應用*
凡洪劍 李江 王麗華 樊春海 柳華杰?
1) (同濟大學化學科學與工程學院, 上海自主智能無人系統科學中心, 先進土木工程材料教育部重點實驗室, 上海 200092)
2) (中國科學院上海高等研究院, 張江實驗室, 上海光源科學中心, 上海 201800)
3) (上海交通大學化學化工學院, 上海 200240)
1 引 言
自20 世紀50 年代以來, 硅基集成電路技術的進步推動了電子計算機的蓬勃發展.然而, 隨著“自上而下”的光刻等手段不斷走向尺寸極限, 電路越來越集成化、電子設備越來越小型化的要求正面臨新的巨大挑戰, 后摩爾時代的來臨已迫在眉睫.突破原有技術極限, 進行原子尺度的精準構筑, 是當前的重大科學問題, 也是解決下一代信息技術發展的關鍵.不同于“自上而下”的光刻、操縱等手段,“自下而上”的自組裝是構成生命體系的基本原理,也充分展示出生命體從原子直到宏觀的跨尺度、跨維度的精準度, 更在此基礎上使生命體展現出了超乎無機材料的智能性.受此啟發, 從原子尺度進行物質的人工自組裝, 通過調控基本構筑單元的物理排布與功能集成, 進而實現器件制造, 是未來的一項前沿發展方向.
DNA 是一類具有原子級精準度的生物大分子.基于精確的A-T, G-C 堿基配對原理, DNA 單鏈之間可以形成具有堿基序列特異性的雙鏈結構—即著名的DNA 雙螺旋結構, 此外DNA 也能夠形成三鏈[1]、四鏈[2]等結構.特定序列的DNA單鏈可通過人工化學合成獲得, 而通過設計并合成DNA 序列, 就能夠實現可編程性自組裝構建人工DNA 納米結構.這些DNA 納米結構可看作由若干DNA 鏈在空間上進行排布, 進而形成的人工框架結構, 由于框架上的每個堿基位置都是可定位的, 因此也為功能基元在框架上的定點修飾提供了可能[3,4].DNA 框架結構中最為著名的是DNA 折紙(DNA origami)結構[5], 組裝原理是由約200 多條20—60 堿基的訂書釘鏈(staple)引導一條7000多堿基的骨架鏈(scaffold)以類似光柵填充的形式折疊而成, 具有良好的結構穩定性和可編程設計性, 能夠設計構建任意二維和三維結構[6].DNA 折紙在結構上可看作一種具有精確尋址功能的模板,經常被用作有機染料分子[7]、核酸[8]、蛋白質[9-11]、無機納米顆粒[12-15]、碳納米管[16,17]等的陣列構建,在光電器件[18]、生物醫藥[19-21]、信息處理[7,22,23]等領域取得了一系列重要應用.
本文首次基于DNA 折紙結構的精準尋址性,進行原子陣列的自組裝構筑.由于原子的化學穩定性問題, 實驗選取二茂鐵分子為研究對象, 通過兩個茂基使鐵原子處于穩定的化學狀態, 再通過化學共價修飾將二茂鐵準確定位在DNA 折紙的指定位置上, 構成受茂基保護的鐵原子陣列圖案.為了展示此材料的應用潛力, 結合前期發展的DNA 折紙加密技術(DNA origami cryptography, DOC)[24],發展了基于原子陣列的加密技術(A-DOC).通過對DNA 序列及自組裝過程的編碼, 將明文加密成密文隱藏于鐵原子陣列中, 并借助單分子成像手段進行密文的讀取, 最后使用正確密鑰進行解密.該策略在原子層面上整合了加密術和隱寫術, 理論上適用于文本、數字、圖片等各類信息的加密, 為信息安全的發展提供了一種具有巨大潛力的生物分子解決方案.
2 “信息預置”思路制備鐵原子陣列圖案
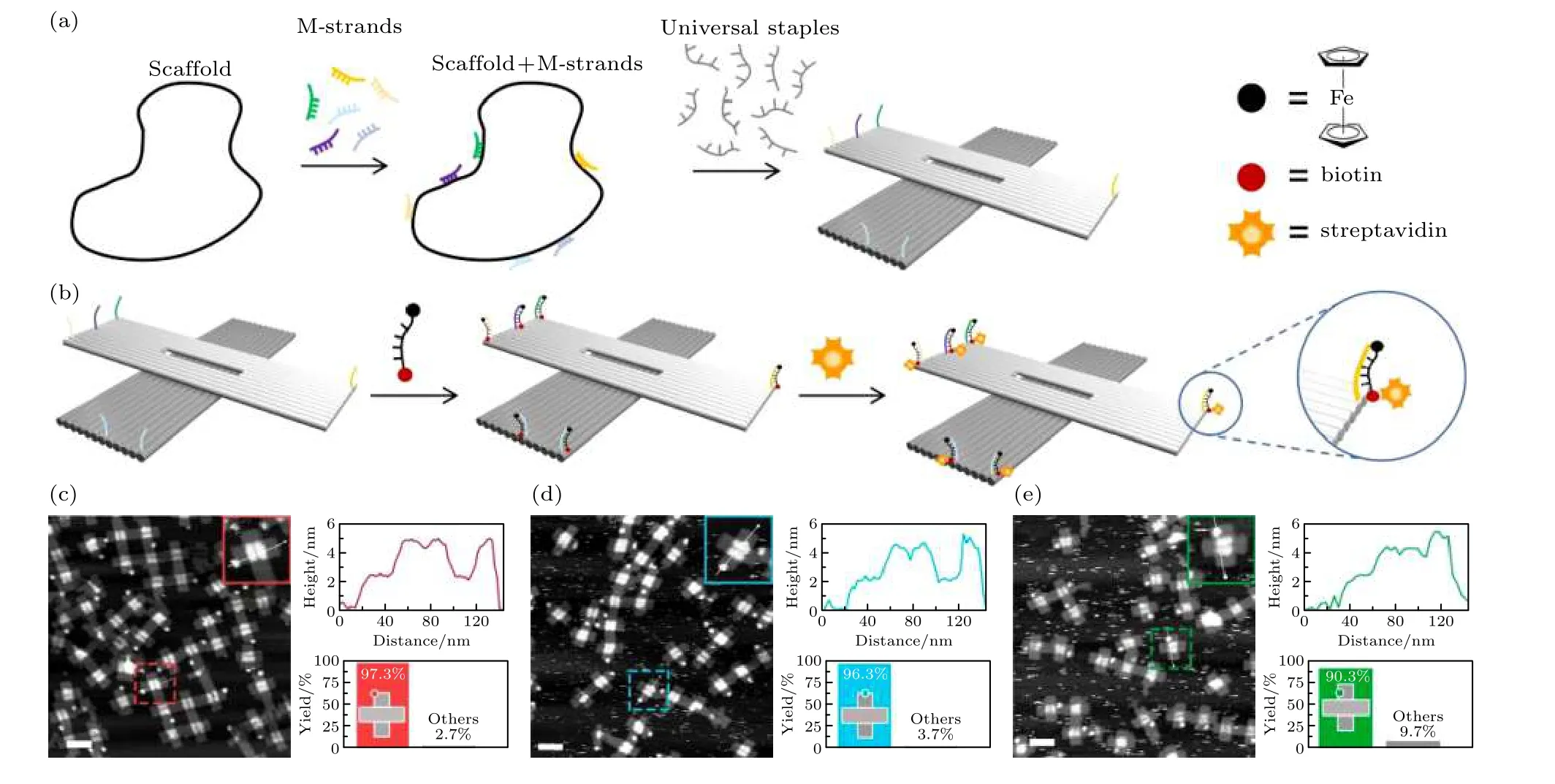
圖1 鐵原子陣列的構建 (a), (b) 信息鏈預置于骨架鏈上的策略形成DNA 折紙并組裝鐵原子陣列, 通過生物素和鏈霉親和素的強結合力將位置顯影; (c)—(e) 3 個位點單個鐵原子圖案組裝原子力表征圖(比例尺: 100 nm)Fig.1.Fabrications of iron atoms arrays.(a), (b) The M-strand strategy forms DNA origami and assembles the iron atoms arrays.The position is visualized by the strong binding force of biotin and streptavidin.(c)—(e) The atomic force characterization diagram of the assembly of a single iron atom at three sites (scale bar: 100 nm).
本文提出的鐵原子陣列圖案構建思路如圖1所示, 與通常的DNA 折紙組裝方法不同, 本文采用了一種具有“信息預置”特點的思路(圖1(a)).首先, 根據最終陣列圖案的要求, 將用于固定二茂鐵基團的訂書釘鏈先與長骨架鏈雜交結合.這個過程等同于將鐵原子陣列信息“預置”于骨架鏈上, 因此, 這些訂書釘鏈被稱為“信息鏈”(M-strand, M 鏈)(補充材料的圖S1 (online)).第二步, 攜帶信息鏈的骨架鏈經過純化后與一套通用訂書釘鏈集合(universal staples)進行退火, 形成折紙結構, 此時的折紙在預定放置鐵原子陣列的位點上具有相同的一段捕獲序列.為了將二茂鐵基團在折紙結構上進行固定, 即形成鐵原子陣列, 實驗中采用了修飾有二茂鐵的DNA 序列, 通過分子識別與折紙上的捕獲序列雜交結合(圖1(b)).在修飾二茂鐵的序列另一端修飾了生物素分子(biotin)使鐵原子陣列能夠以成像的方式被觀測到.這是因為生物素分子能夠特異性地與鏈霉親和素(streptavidin, SA)結合, 而后者能夠被原子力顯微鏡(atomic force microscope, AFM)清晰成像, 從而可通過觀測SA陣列來驗證設計的鐵原子陣列形狀.因此, 修飾有二茂鐵與生物素的序列可被稱為“顯影鏈”(N-strand,N 鏈), 而SA 就是“顯影劑”.M 鏈與骨架鏈結合部分的長度為40 個堿基(補充材料的圖S2 (online)),M 鏈延伸端與N 鏈序列互補.
需要指出的是, 本思路的實現基于對DNA 雜交反應熱力學和動力學過程的嚴格調控.在預置有M 鏈的骨架鏈與通用訂書釘鏈集合共同退火時,M 鏈的長度以及退火溫度決定了能否避免未期望的解離以及設計圖案的生成.一方面, M 鏈的長度可以通過模擬計算確定, 需特別注意的是要避免M 鏈在骨架鏈上形成來回折疊, 以及由于M 鏈的獨特設計對于周圍其他訂書釘鏈的影響.結果顯示, M 鏈與骨架鏈雜交長度設計為40 堿基時, 與普通訂書釘鏈比較, 在能量上具有較大優勢(補充材料的圖S3 (online)).另一方面, 退火溫度的選擇可以進一步避免預置M 鏈的脫落, 根據模擬結果,在起始溫度設置為57 ℃、M 鏈與骨架鏈結合長度為40 個堿基時, 未配對比例僅為3.5%; 而對于普通訂書釘鏈, 與骨架鏈的未配對比例, 相比M 鏈高了至少2 倍以上.
與通常制備DNA 折紙結構的思路不同, 將信息鏈預置于骨架鏈上極大降低了實驗工作量.設單個折紙的訂書釘鏈數量是s, 陣列中的鐵原子數量是m, 按常規做法需要將m 條信息鏈去替代對應的普通訂書釘鏈, 即需要將m 條信息鏈與s-m 條剩余普通訂書釘鏈逐條混合, 然后再和1 條骨架鏈混合, 總計需要s + 1 個混鏈步驟.需要指出的是,如果制備另一個不同的鐵原子陣列, 前述混合的樣品將完全不能使用, 而是再重新經過s + 1 個步驟獲得新的樣品.以圖1 所示的十字形折紙為例, 共有201 條普通訂書釘鏈, 如果用于制備圖1(c)—(e)中的3 個不同圖案, 需要606 個混鏈步驟.但是,使用“信息預置”的方法, 只需混合1 個包含201 條普通訂書釘鏈的通用庫, 然后根據圖案的不同單獨將信息鏈與骨架鏈混合即可, 總計需要207 個混鏈步驟.如果一次制備的通用訂書釘鏈集合的量非常巨大, 不再考慮構建此集合的步驟, 此方法將進一步節省工作量, 僅需6 步就能完成.
為了驗證此方法的有效性, 對鐵原子在十字形折紙的3 個不同位置上的組裝效率進行了測試, 如圖1(c)—(e)所示, 分別是棱角上、棱中點和折紙面上.由于二茂鐵和生物素是同步修飾在N 鏈兩端,AFM 下觀測到SA 的正確組裝即說明生物素和二茂鐵都固定在正確的位置上.結果顯示, 這3 個不同位置的組裝效率均在90%以上, 分別為96.3%,97.3%, 90.3%.最終圖案的高效組裝決定于三方面的高效率: 一是M 鏈在骨架鏈上的高效結合, 二是顯影劑N 鏈高效地與M 鏈延伸段雜交, 三是SA與生物素的結合效率.SA 與生物素的結合被公認為親和能力很強, 以上結果驗證了本方案在M 鏈和N 鏈的結合上也具有高效率, 因此實現折紙上鐵原子陣列構建的有效性.
3 “一鍋法”同時制備不同鐵原子陣列圖案
采用“信息預置”的另一個更為重要的優勢, 是能夠實現以“一鍋法”同時批量制備多種不同的陣列圖案.如圖2(a)所示, 將攜帶不同M 鏈的3 條DNA 骨架鏈進行混合, 然后與通用訂書釘鏈集合進行退火, 可以在一鍋內制備出三種鐵原子點陣圖案.其實現原理是, 信息鏈M 從能量上保護了雜交位置的骨架鏈, 使普通訂書釘鏈難以與骨架鏈雜交, 即替換下M 鏈; 攜帶不同M 鏈的骨架鏈之間同樣也不會發生串擾.從結果看, 該方法成功獲得了所設計的三種點陣圖案, 且觀察到的圖案比例分布與各骨架鏈的比例一致, 這證實了訂書釘鏈對M 鏈與骨架鏈的結合幾乎沒有影響.此外, 也沒有觀察到兩個圖案點出現在同一個折紙上的情況發生, 說明沒有發生不期望的解離并結合到別的骨架鏈的情況(圖2(b)).
這種一鍋法技術, 在實驗工作量上也實現了很大的簡化.仍然設M 鏈數量和普通訂書釘鏈數量分別為m 和s, 需要m 步來構建M 鏈集合, s 步構建通用訂書釘鏈集合.對于常規做法, 由于改變陣列圖案即需要重新混合全部訂書釘鏈, 因此對于每個樣品都需要s + 1 個步驟; 而對于一鍋法, 由于不需要重建預混合的通用訂書釘鏈集合, 對每個樣品僅需m + 1 步即可完成.理論計算可知, 對于最多具有m 個修改位點的圖案, 存在個可能的組合變化, 以及個常規策略的模式變化.由于s 通常比m 大1 個數量級, 因此在快速制作各種圖案方面, M 鏈預置策略比常規策略具有顯著優勢.
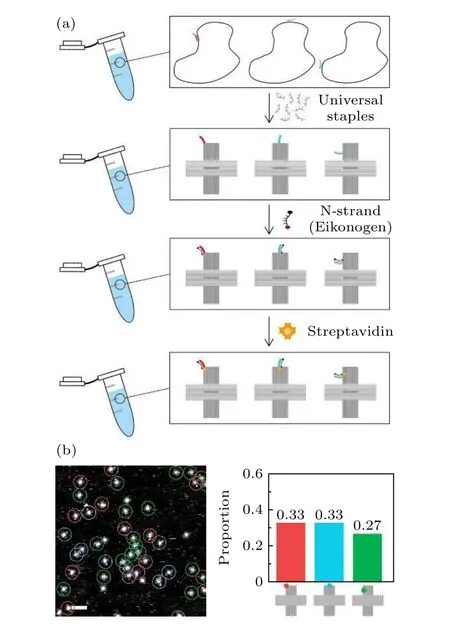
圖2 M 鏈策略“一鍋法”制備多種DNA 折紙納米圖案(a) 多種攜帶不同M 鏈的骨架鏈混合, 一同退火, 在單個離心管中快速制備多種DNA 折紙納米圖案; (b) 原子力表征圖及產率統計圖(比例尺: 200 nm)Fig.2.M-strand strategy to prepare a variety of DNA origami nanopatterns by “one-pot” method: (a) A variety of scaffolds carrying different M-strands are mixed and annealed together to quickly prepare a variety of DNA origami nanopatterns in a single centrifuge tube; (b) AFM diagram and yield statistics (scale bar: 200 nm).
4 基于鐵原子陣列圖案的信息加密
從本文鐵原子陣列的制備和表征過程可以看出, 信息鏈預置于骨架鏈的思路不僅是對骨架鏈的預保護, 實際上也等同于將信息鏈隱藏于骨架鏈上;此外, 信息的寫入(雜交)和讀取(成像)在物理上是分開的, 并且由兩個不同的實體進行.因此, 基于以上技術, 使開發基于鐵原子陣列的信息加密技術成為可能.
在前期開發的DNA 折紙加密(DOC)技術基礎上, 本文利用鐵原子陣列提出了原子陣列DNA折紙加密(A-DOC)技術.如圖3(a)所示, 發送方和接收方(分別命名為Alice 和Bob)利用A-DOC技術進行了文本信息的秘密傳遞, 其原理是對折紙上不同的鐵原子位置編碼, 通過密鑰進行加密, 并結合隱寫術加強保密程度.首先, 使用密鑰對二進制文本進行加密, 轉換成折紙上的原子點陣圖案.在第二步, 將對應該點陣的信息鏈與骨架鏈進行雜交, 得到的樣品即包含了秘密傳遞的信息.Alice 將預置M 鏈的骨架鏈交付給Bob, Bob 再將通用訂書釘鏈庫與骨架鏈進行退火組裝, 獲得折紙結構.值得注意的是, Bob 獲得的折紙雖然已隱藏了秘密信息, 但需要在結合二茂鐵修飾序列, 形成鐵原子陣列后, 才能通過成像手段將隱寫圖案進行讀取.最后, Bob 通過密鑰將讀取的原子點陣圖案進行解密, 獲得明文.
在單個十字折紙上, 13 個可區分的識別位點被選擇作為鐵原子點陣的可選位點, 獨特之處在于, 這13 個位點被分為三種功能: 1)文本信息(紅色位點), 以8 位的二進制ASCLL 碼編碼單個字符; 2)位置信息(黑色位點), 表示此折紙上的字符信息在整個字符串中的位置, 如設置4 位二進制數, 則字符串長度為24= 8; 3)定位標記(綠色位點), 以1 個標記位點打破十字折紙的外在對稱性,使表示文本與位置的位點讀取順序得到明確.在讀取形式上, 通過表征SA 的圖案即可說明鐵原子在對應位置是否存在, 從而讀取位點上的信息為0 或者是1, 有鐵原子的位點為1, 反之為0.
圖3(b)給出了用A-DOC 技術對明文消息“DNA-1954”進行的加密結果.Bob 通過AFM 成像獲得類似盲文的鐵原子點陣圖案后, 查詢密碼表將圖案信息轉換為二進制信息, 再通過分別對位置信息和文本信息進行解碼, 最終可以獲得明文.以位置信息為例, 當讀取到表示“0000”的折紙時, 由于該4 位二進制數表示字符串的第一位字符, 因此再將表示文本信息的8 位二進制數“010000100”解碼, 得知第一位字符為“D”.以此原理, 密鑰大小由(1)式決定:
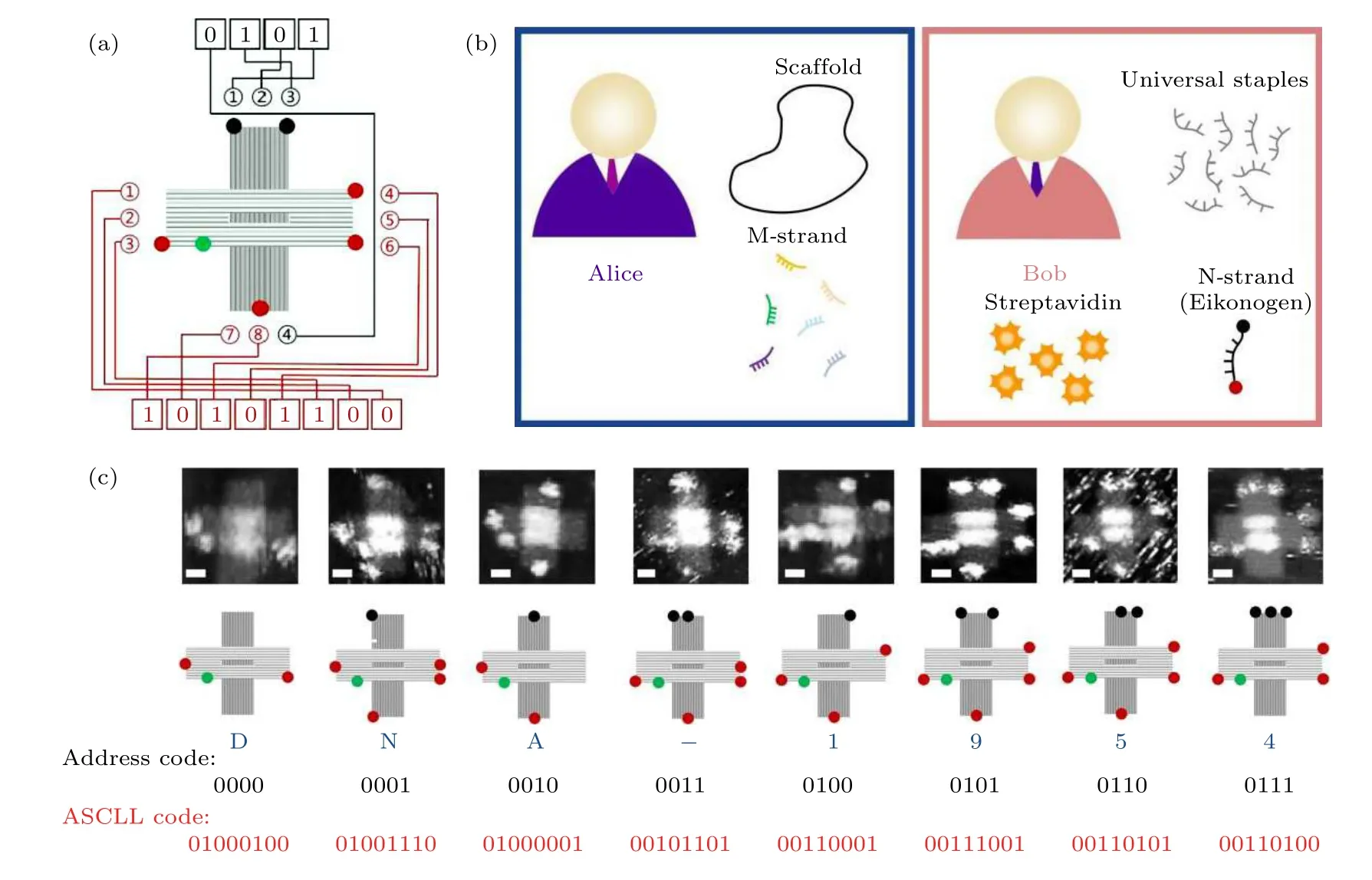
圖3 DNA 折紙加密及編碼原理示意圖 (a) DNA 折紙斑點編碼原理; (b) 發送者(Alice)和接收者(Bob)通信流程; (c) 文本“DNA-1954”的編碼演示(比例尺: 25 nm)Fig.3.Schematic illustration of DNA origami encryption and coding principle: (a) Coding principle of DNA origami spot; (b) the communication procedure between the sender (Alice) and the receiver (Bob); (c) coding demonstration of the text “DNA-1954”(scale bar: 25 nm).
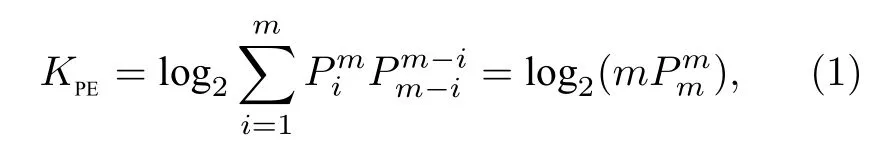
在以上工作基礎上, 進一步提出了多折紙編碼單字符的設計, 對字符串長度和可編碼字符容量進行了提升, 并以加密唐詩《登鸛雀樓》為例進行演示(圖4).根據漢字代碼標準GB2312 的規定, 漢字按94 個“區”和94 個“位”分區索引, 可以看作一個橫豎各有94 個格子的正方形棋盤, 每個格子對應一個漢字或者符號.其中1—9 區為符號區,16—87 區為漢字編碼區, 其他為用戶自定義編碼區.如圖4(a)所示, 本文使用兩個不同標記的十字折紙, 對于第一個折紙, 12 個可識別位點中5 個黑色點位代表在字符串中的位置信息, 容量為25;7 個藍色的位點①—⑦編碼漢字的區, 稱為區碼(section code), 信息容量可達128 位(27), 超過了國標規定94 位的需求.同樣另一個折紙以相同的五個黑色點位代表位置信息, 對應第一個折紙, 紅色的7 個點位①—⑦代表在該區所處的位, 稱為位碼(postion code).通過兩個折紙位置信息匹配讀取信息就可得到漢字信息和這個漢字在文本中所處的位置.在圖4(b)中演示了該編碼規則的實例,對于漢字“流”, 根據區碼表得到它的區碼為33、位碼為87, 代表它在區碼表中處于33 行87 列的位置.然后將區碼和位碼分別轉換為二進制數據, 區碼二進制為“0100001”, 位碼二進制為“1011101”,根據紅色和藍色序號的循序依次寫入.同時根據它在詩中的位置定義了它的位置碼為15, 即“01110”.
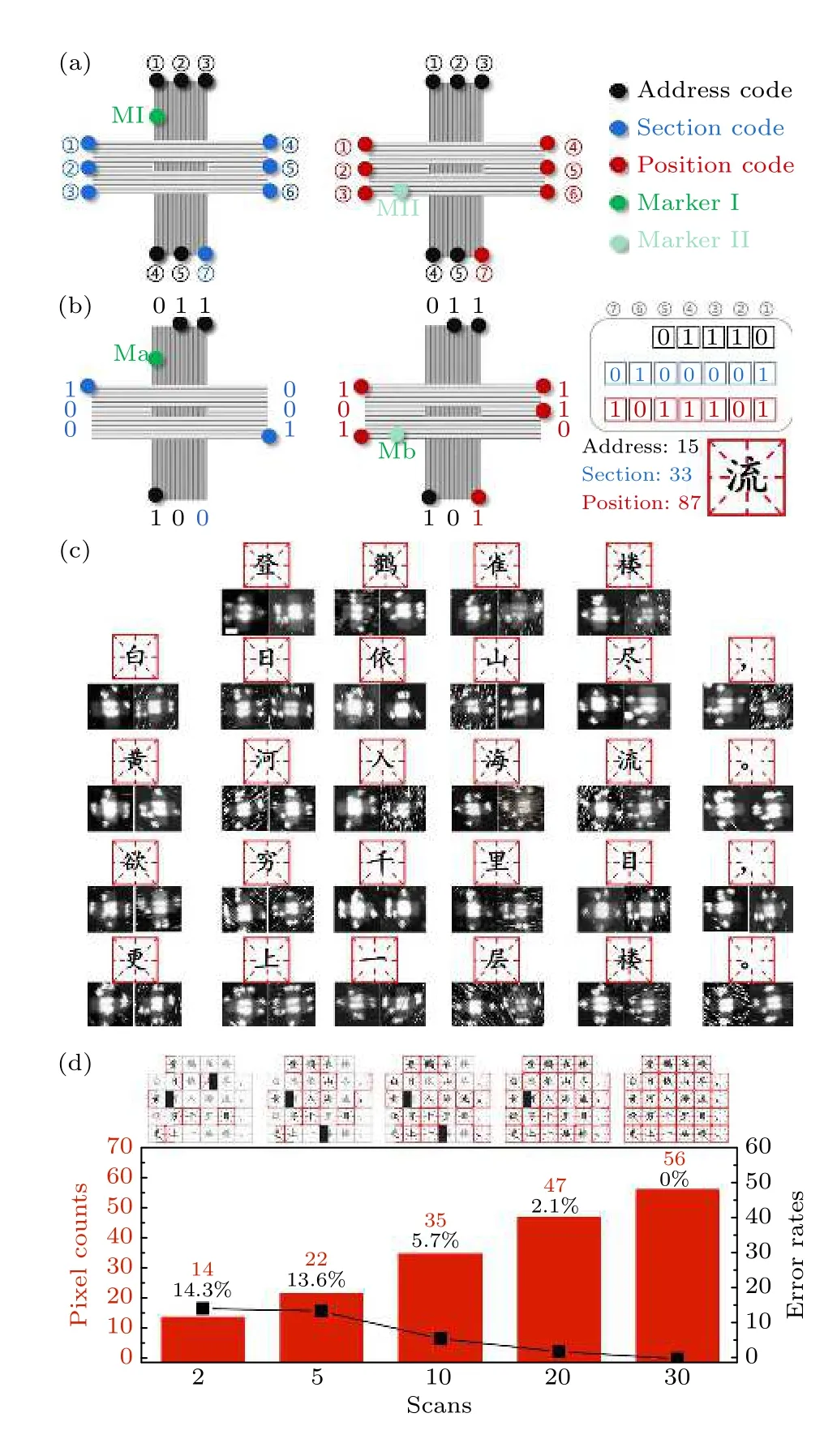
圖4 將漢字在DNA 折紙上的加密方案 (a) 區位碼在折紙上的編碼原理; (b)漢字“流”的編碼演示; (c) 28 個漢字唐詩文本AFM 實驗圖(比例尺: 40 nm); (d)唐詩隨掃描次數收集完成度和錯誤率圖, 正確收集標記為紅色, 單個鏈霉親和素圖案未收集超過20 個的標記為白色Fig.4.Scheme of encoding Chinese characters on DNA origami: (a) Encoding principle of section and position code on origami; (b) demonstration of Chinese encoding Chinese character “流” on DNA origami (scale bar: 100 nm); (c) AFM experimental graph of 28 Chinese characters Tang poetry text (scale bar: 40 nm); (d) collection completion and error rate graphs of Tang poetry with the number of scans completed and error rate graphs.The correct collection is marked as red, and the single streptavidin pattern which is not collected for more than 20 will be marked as white.
如圖4(c)所示, 用該方法對唐詩《登鸛雀樓》進行了加密, 首先將這首唐詩的每一個字符按照區位碼編碼(補充材料的圖S4 (online)), 這首唐詩總共28 個字符, 包含24 個漢字和4 個標點符號.為避免在傳遞信息時, 由于十字折紙中間凹陷不清晰引起的辨識困難, 我們將攜帶區碼和位碼的折紙分別表征(補充材料的圖S5 (online)).此外,為避免由于折紙圖案較大的多樣性帶來的數據可能缺失問題, 可以通過增加讀取次數, 即增加AFM成像數量來消除錯誤, 隨著掃描次數的增多, 同一位置出現圖案最多的信息會被積累并使得初始的錯誤得到校正.如圖4(d)所示, 在獲得30 次掃描結果后, 排除無法識別(聚集或未良好形成)的折紙, 錯誤率由兩次掃描時的14.3%下降到30 次掃描時的0%, 證明了隨著圖案基數的增加, 正確的圖案累積, 在后期更難以出現新的錯誤以替代正確的統計結果.
5 結 論
本文利用DNA 折紙結構的精準定位能力, 通過“信息預置”的思路構建了鐵原子陣列圖案, 并將此應用于對信息的加密.AFM 成像結果顯示, 鐵原子在折紙不同位置的正確定位效率高達90%以上, 驗證了構建原子陣列的可行性.同時, 通過對位置信息的預置, 還極大降低了工作量, 使同時批量制備不同陣列圖案成為可能.在此基礎上, 建立了密鑰長度在700 位以上的原子陣列加密技術, 結合隱寫術將信息隱藏在類似盲文的點陣圖案中, 以唐詩《登鸛雀樓》為例進行了演示.在未來的工作中, 我們將一方面進一步探索對鐵原子陣列的更高分辨率與更直接表征, 另一方面也將在加密原理方面進行優化, 提升可加密字符串長度與字符容量.希望本文的研究能夠推動DNA 自組裝技術在原子制造領域的更多應用, 在利用DNA 原子級精準性方面發揮更多作用, 為發展原子精準性材料與器件提供生物分子解決方案.

