中國美利奴羊羊毛彎曲相關microRNA的篩選
于麗娟, 張艷花,拉扎特·艾尼瓦爾,徐新明,瑪爾孜婭·亞森,狄 江
(新疆畜牧科學院畜牧研究所,烏魯木齊 830011)
0 引 言
【研究意義】彎曲是羊毛最主要的物理性質之一,與毛紡工藝有著密切的關系。研究表明,降低羊毛纖維彎曲度能減小纖維抗彎曲剛性和紗線厚度并進而增加織品柔軟度,其對紡織品柔軟度的影響與纖維直徑有同等重要的作用[1]。了解羊毛彎曲形成的分子機理,將有助于利用分子育種培育符合市場需求羊毛彎曲類型細毛羊新群體。【前人研究進展】羊毛彎曲與毛囊中細胞的不對稱分裂、毛纖維中正副皮質的雙邊排列、不同角蛋白的不對稱組成及角質化過程等相關[2]。對小鼠、人等哺乳動物毛發纖維彎曲的形成機制方面的研究取得了很大進展,Wnt、EDA/EDAR、EGFR、BMP、NF-κB、Shh等經典信號通路與毛發彎曲有著直接或間接的聯系[3]。【本研究切入點】MicroRNA(miRNA)為一類大小在18~24個核苷酸的非編碼單鏈RNA,通過與靶mRNA結合位點的特異性結合,抑制靶mRNA翻譯或降解靶mRNA來發揮其重要的生理調節作用[4]。研究表明,miRNA幾乎參與了機體所有的代謝調節活動,包括肌肉生長發育、細胞周期變化、細胞增殖與凋亡、脂肪代謝、器官形成以及機體的免疫應答等反應[5]。隨著miRNA對皮膚和毛囊發育形成的研究的不斷深入,miRNAs是毛囊發育過程中重要的轉錄后調控因子,參與毛囊的發生和周期性生長[6-9]。目前,miRNA對羊毛彎曲性狀形成的相關調控機制仍不清楚,需要研究miRNAs在羊毛彎曲形成過程中發揮的作用。【擬解決的關鍵問題】通過采用高通量測序技術對羊毛彎曲頻率有極端差異表型值細毛羊個體皮膚的miRNA組進行測序并比較分析,篩選出與細毛羊羊毛彎曲性狀相關的差異表達miRNA,利用miRNAda與TargetScan軟件對差異表達的miRNA進行靶基因預測,并對靶基因進行簡單的Gene Ontology與KEGG Pathway富集分析,分析差異 miRNA的預測靶基因參與的生物學功能及調控途徑,為進一步解析羊毛彎曲形成分子機理、以及借助分子育種手段培育符合市場需求羊毛彎曲類型細毛羊新群體提供理論指導。
1 材料與方法
1.1 材 料
綿羊為新疆拜城種羊場的中國美利奴羊,羊群在基本一致的條件下飼養。采集樣品主要以羊毛彎曲頻率性狀為依據。在羊毛品質鑒定中,羊毛彎曲頻率一般分為大、中、小3個彎曲類型,其中大彎為1 cm 羊毛纖維長度內有小于3 個彎曲、中彎為1 cm 羊毛纖維長度內有3 ~ 5 個彎曲、小彎為1 cm 羊毛纖維長度內大于5 個彎曲。個體鑒定時,選取大彎周歲母羊個體與小彎周歲母羊個體各10只。體側肩胛部皮膚剪毛消毒后,用皮膚采樣器分別采集其肩胛部皮膚組織1 cm2左右,投入裝有RNA保存液(RNAlater)的凍存管中。冷藏條件下帶回實驗室,-80℃冰箱保存備用。對其毛樣進行細度與羊毛彎曲頻率測定,選取羊毛彎曲頻率有極端差異的大彎、小彎個體各4只的皮膚樣品用于后續實驗。
1.2 方 法
1.2.1 RNA提取、小RNA文庫構建及Solexa測序
按照Trizol法提取細毛羊皮膚組織總RNA,采用Qubit 2.0 (美國)試劑盒進行RNA的精確定量;利用NanoPhotometer?分光光度計進行純度檢測,同時利用Bioanalyzer 2100 系統的 RNA Nano 6000 Assay Kit進行質量完整性的檢測。樣品檢測合格后,使用Small RNA Sample Pre Kit構建文庫,利用Small RNA的3’及5’端特殊結構(5’端有完整的磷酸基團,3’端有羥基),以總RNA為起始樣品,直接將Small RNA兩端加上接頭,然后反轉錄合成cDNA。隨后經過PCR擴增,PAGE膠電泳分離目標DNA片段,切膠回收得到cDNA文庫。利用安捷倫2100高靈敏度DNA芯片檢測文庫質量。庫檢合格后,使用 Illumina Hiseq 2500 上機測序。
1.2.2 測序數據
對測序得到的原始數據(raw data)進行質控和過濾,包括去除低質量、有5’接頭污染、沒有3’接頭序列、沒有插入片段及包含poly A/T/G/C的reads。進行質控后得到的數據為clean reads,用于后續分析。采用bowtie軟件與羊的參考基因組(Oar_v4.0)進行比對,分析small RNA在參考序列上的分布情況。且利用miRNA所在的位置對已知(保守)miRNA進行鑒定,而新miRNA則通過miREvo和mirdeep2這2個軟件進行預測。利用DESeq軟件篩選差異表達的miRNA,利用TPM分別計算樣本huACF,huBDE表達量取log2,滿足Pvalue≤0.05篩選2組之間的差異miRNA。利用miRNAda、TargetScan靶基因預測軟件對miRNA進行靶基因預測。根據miRNA與其靶基因的對應關系,對差異miRNA的預測靶基因進行簡單的Gene Ontology與KEGG Pathway富集分析。
1.2.3 實時熒光定量PCR驗證
隨機選擇4個差異表達的miRNA,檢測其在2組樣品的表達量。以U6作為內參基因,分別根據4個miRNAs的成熟序列設計熒光定量PCR的上游引物,下游引物由試劑盒提供,引物由上海生工生物科技有限公司合成。表1

表1 qRT-PCR引物Table 1 The list of primers of qRT-PCR
采用poly(A)加尾法進行cDNA的反轉錄,具體操作步驟參照康為世紀miRNA cDNA Synthesis Kit進行。反應體系為20 L:2×miRcute增強型miRNA定量預混試劑(含SYBR和ROX)10 μL,反向引物(10 μmol/L )0.4 μL,正向引物(10 μL )0.4 μL ,cDNA 2 μL,無核酶ddH2O。補至20 μL。PCR反應條件:95℃ 預變性15 min;94℃ 變性20 s,60℃ 退火延伸34 s,共40個循環。每個樣品每個miRNA均進行3次技術重復,結果采用2-ΔΔCt法計算miRNA的相對表達量,與測序結果對比,檢驗測序結果的可靠性。
2 結果與分析
2.1 總RNA質量檢測
研究表明,提取的總RNA進行質量檢測,其OD260/280值均在1.9~2.0,RIN值均高于8.0,總RNA純度及完整性均達到構建文庫要求。表2
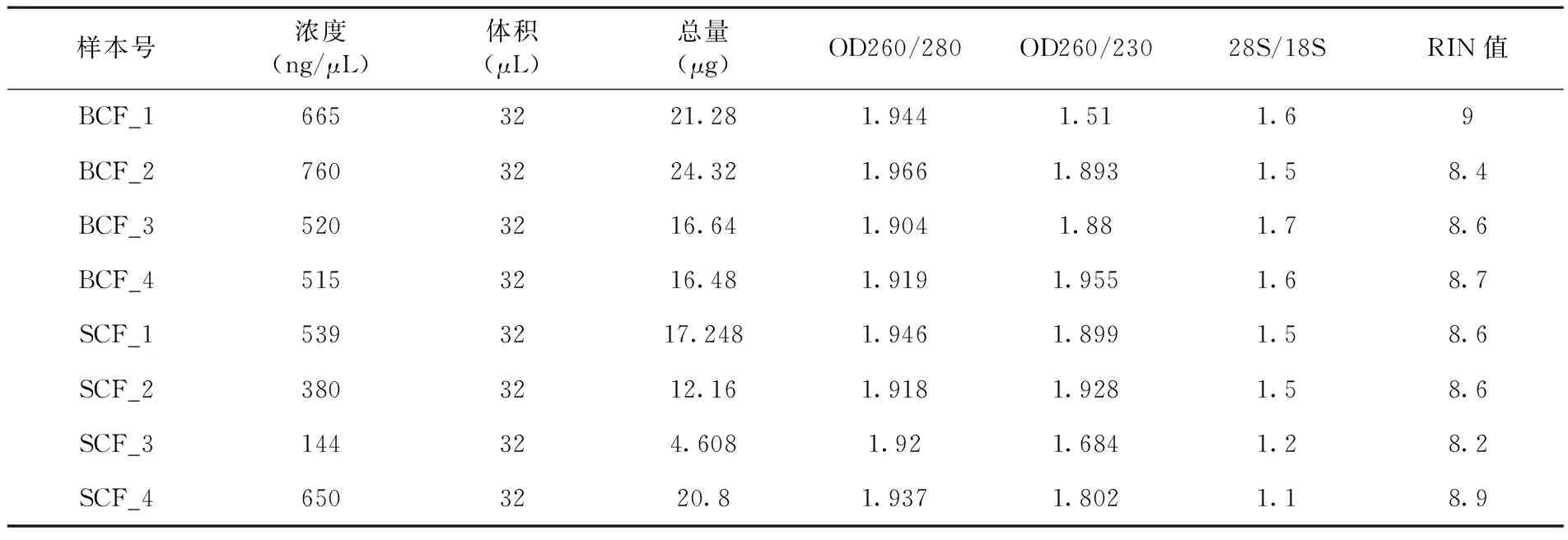
表2 總RNA質量檢測Table 2 The quality test results of total RNA
2.2 中國美利奴羊皮膚組織測序
研究表明,經Solexa測序和生物信息學分析,去除接頭序列以及低質量序列,8個文庫共獲得81 820 492條總序列,經過濾后共獲得79 455 352條純凈序列。細毛羊皮膚組織小RNA序列長度大部分位于21~24 nt,其中長度為22 nt的序列所占比列最大。表3,圖1
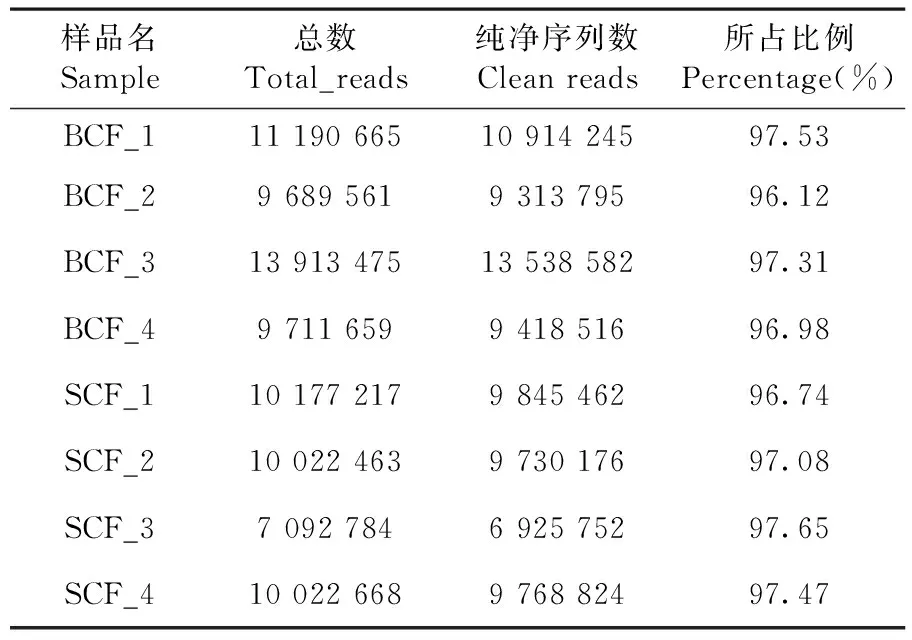
表3 數據過濾信息Table 3 The information of raw data filtering
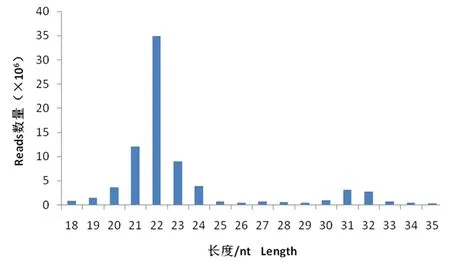
圖1 小RNA的長度分布Fig.1 Length distribution of sRNAs
2.3 小RNA的分類注釋
利用Bowtie 軟件將得到的純凈序列在數據庫中比對后進行分類注釋,得到各種類型sRNA,包含有已知miRNA、新miRNA、rRN A、snRNA、snoRNA、tRNA和其他RNA。其中,各樣品中已知的miRNA所占的比例最大。表4
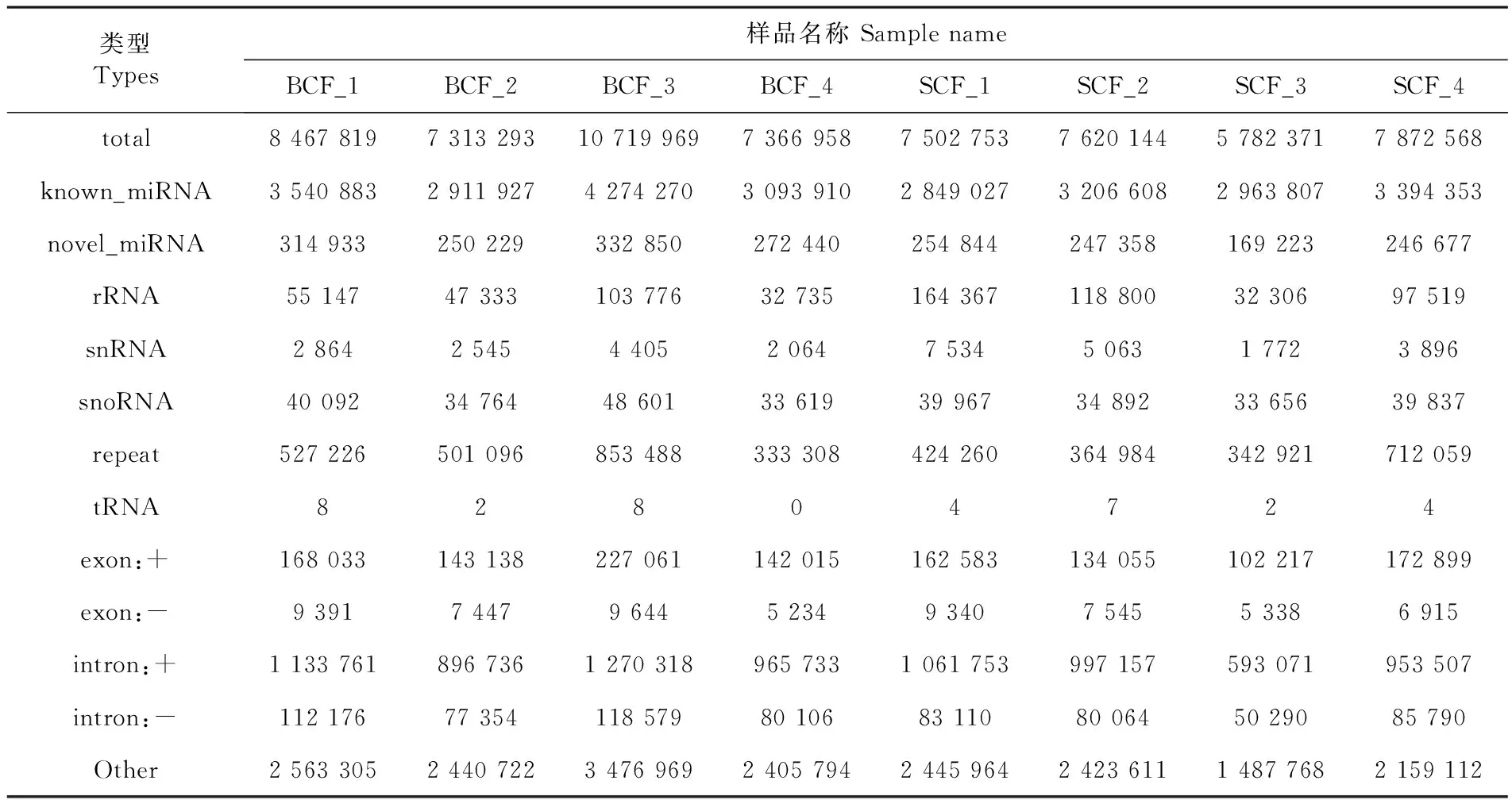
表4 各樣本sRNA分類注釋Table 4 sRNA classification annotation results for each sample
2.4 小RNA序列基因組比對
針對每個樣本預處理后所有序列和去重復后的唯一序列,分別采用Bowtie軟件與羊的參考基因組(Oar_v4.0)進行比對分析。獲取其在參考基因組上的位置信息。比對結果表明81%以上的片段能比對到綿羊基因組上。61%以上的種類比對到綿羊基因組上。各樣品間的比對率存在一定的差異說明樣品間小RNA存在一定差異。表5
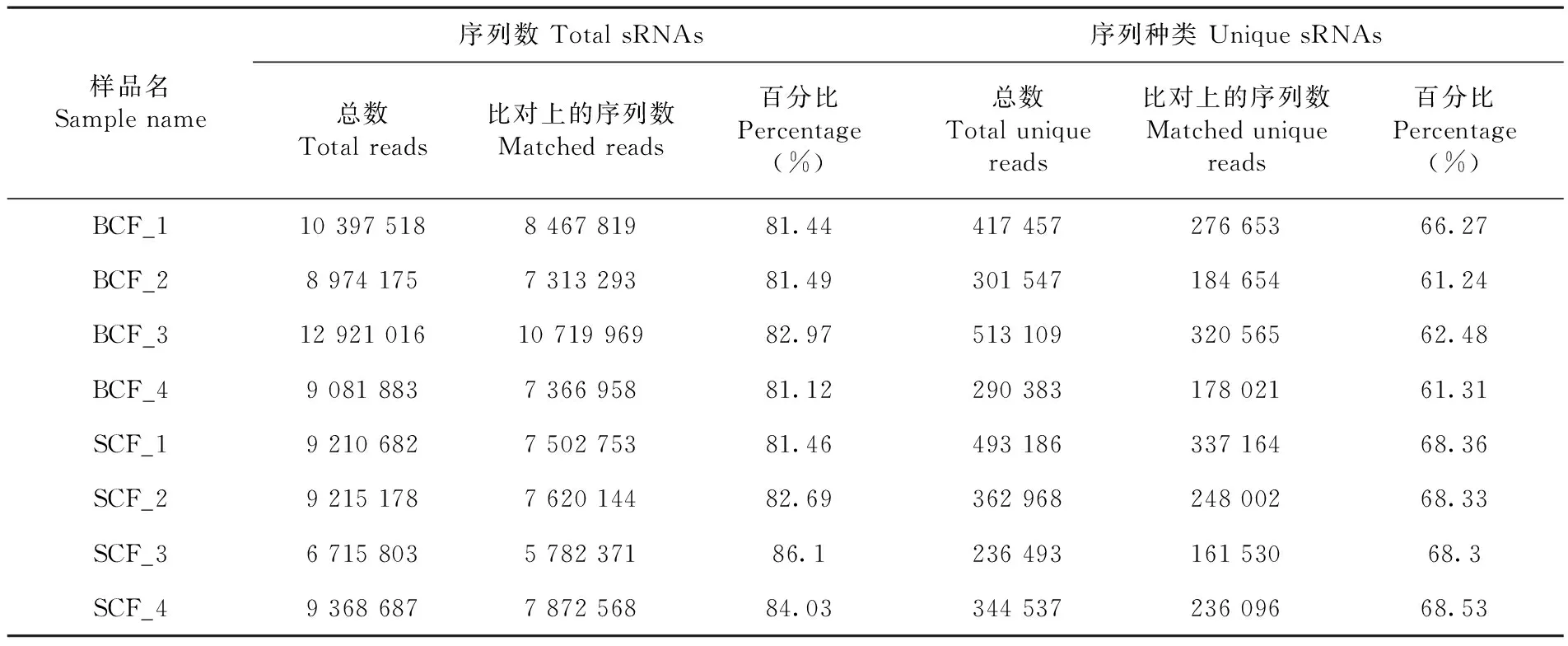
表5 與參考序列比對Table 5 Alignment results of the reference sequence
2.5 已知miRNA分析和新miRNA預測
將mapped到參考序列上的reads,與miRBase中指定范圍序列進行比對,得到各樣品匹配上的sRNA的詳細情況,包括匹配上的已知miRNA的二級結構,各樣本中miRNA的序列、長度、出現的次數等信息。比對上的已知成熟miRNA143條,已知 miRNA前體有106條。
整合miREvo(Wen et al.,2012)和mirdeep2(Friedlander et al.,2011)這些miRNA預測軟件來進行新miRNA的分析。在小RNA文庫中總共預測得到306條miRNA前體,成熟miRNA也有282條。
2.6 miRNA差異表達
利用DESeq軟件篩選差異表達的miRNA,利用TPM分別計算樣本huACF,huBDE表達量取log2,滿足Pvalue≤0.05篩選2組之間的差異miRNA。共鑒定出差異表達miRNA25個,其中上調表達8個,下調表達17個。
2.7 差異miRNA靶基因預測與功能富集
利用miRNAda與TargetScan靶基因預測軟件對差異表達的miRNA進行靶基因預測。預測結果顯示,大彎曲與小彎曲組的25個差異表達的miRNA共預測到了15 761個無重復的靶基因。根據miRNA與其靶基因的對應關系,對差異miRNA的預測靶基因進行簡單的Gene Ontology與KEGG Pathway富集分析。GO分析結果表明,其主要參與跨膜信號受體活動、信號轉導活動、分子轉導活動、膜的組成、細胞交流、生物過程、細胞過程的調節等過程。15 761個靶基因注釋到252個信號通路。其中,107個靶基因參與了Wnt信號通路,93個靶基因參與了AMPK信號通路,34個靶基因參與了Notch信號通路,161個靶基因參與了MAPK信號通路,68個靶基因參與了NF-kappa B信號通路。圖2
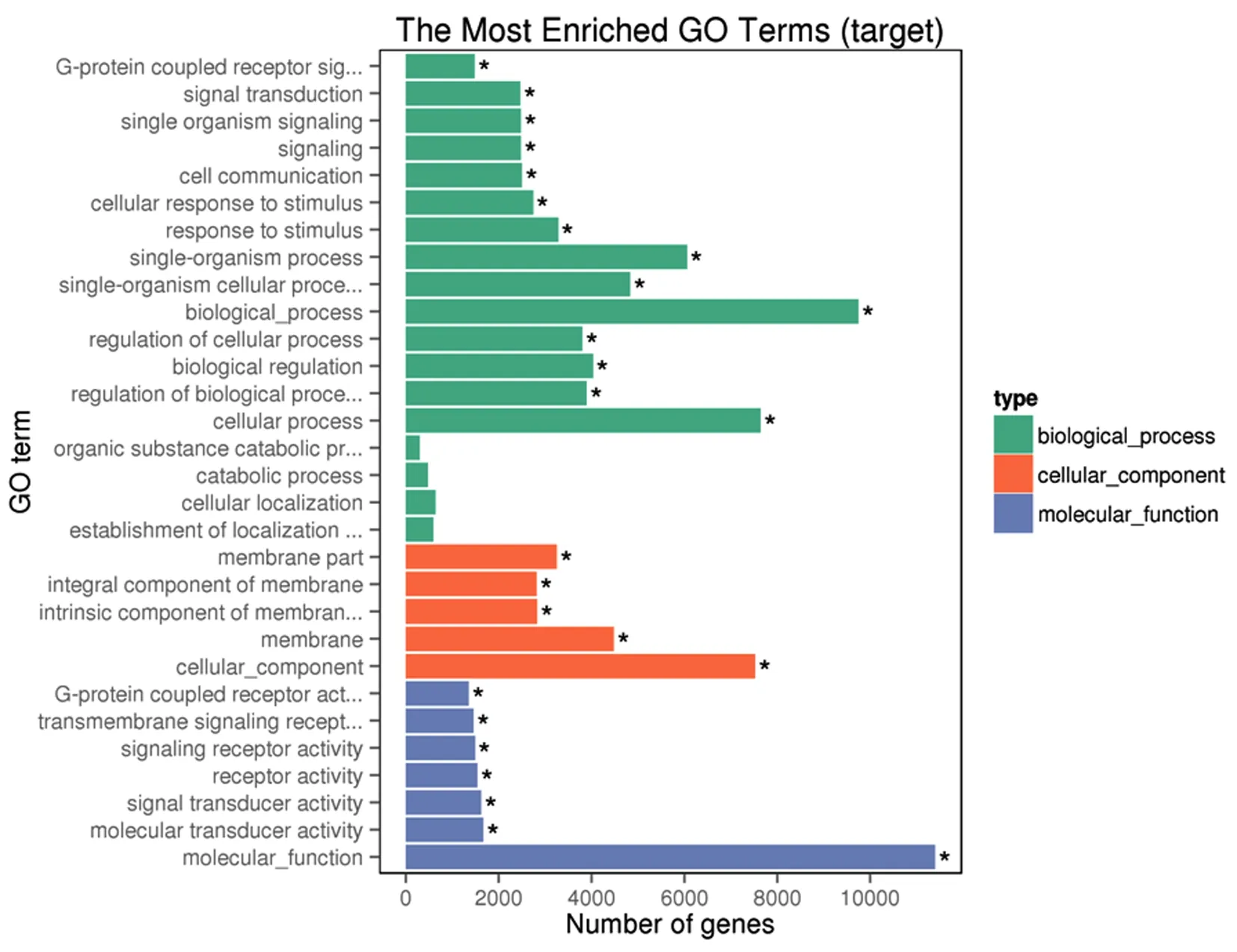
圖2 差異表達miRNA靶基因GO
2.8 miRNA qRT-PCR 驗證
隨機選擇4個差異表達miRNA(novel_503、oar-miR-133、oar-let-7g、novel_9)進行定量驗證,結果表明:novel_503、oar-miR-133、oar-let-7g、novel_9在大彎曲組中上調表達,而在小彎曲組中下調表達,與高通量測序結果一致,證明高通量測序結果的可靠性。圖3,表6
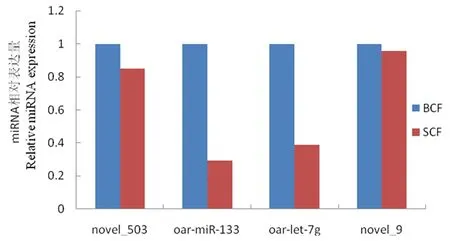
圖3 miRNA 的qRT-PCR 驗證Fig.3 qRT-PCR verification of miRNA
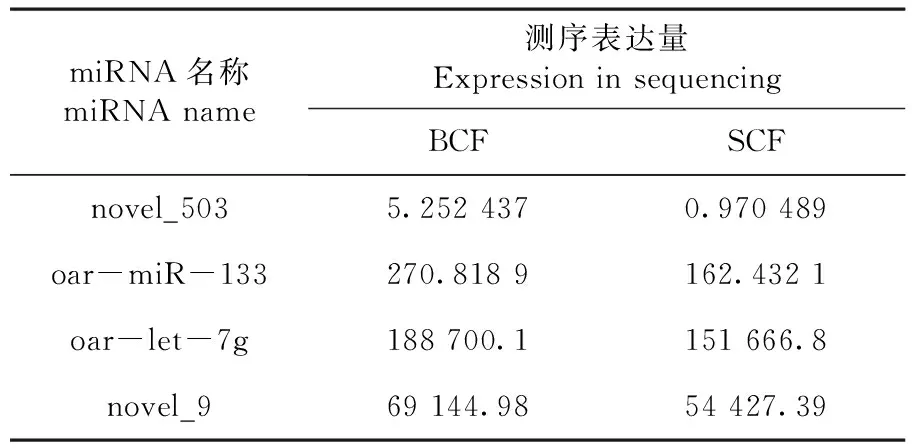
表6 用于qRT-PCR 驗證的miRNA測序表達量數據Table 6 Sequencing data of miRNA verified by qRT-PCR
3 討 論
關于中國美利奴羊羊毛經濟性狀方面的研究主要集中在對羊毛細度、長度等。在中國美利奴羊羊毛彎曲性狀方面的研究僅見狄江等[10]通過全基因組關聯分析檢測與細羊毛彎曲頻率性狀顯著關聯的SNPs 。
高通量測序技術已被廣泛應用于動植物轉錄組測序、小RNA測序等方面,在中國美利奴羊羊毛彎曲性狀形成方面的研究還未見報道。研究運用高通量測序技術對羊毛彎曲頻率有極端差異表型值(大彎與小彎)細毛羊個體皮膚的miRNA組進行測序并比較,共鑒定出425個miRNAs,其中包括143個已知miRNAs和282個新發現的miRNAs。鑒定出的miRNAs進行表達量差異分析發現,在大彎曲組與小彎曲組共篩選到8個上調和17個下調的miRNAs。其中,差異表達miRNA中的oar-miR-133、oar-miR-379-5p的表達量趨勢與Liu的研究結果一致[11]。并且,高水平表達的oar-miR-379-5p在灘羊與藏綿羊皮膚毛囊中也檢測到[12,13]。差異表達miRNA中的oar-miR-21可能通過調控CNKSR2、KLF3和TNPO1的表達來影響毛囊發育[14]。差異表達miRNA中的oar-let-7g屬于let-7家族,該miRNA的靶基因與皮膚組織中毛囊的生長和毛發的質量相關[15],而且研究表明,let-7a、let-7b、let-7c、let-7i等與灘羊被毛卷曲、湖羊羔皮花紋及生長相關[16,17]。推測let-7家族可能參與羊毛彎曲的形成。對于其他差異表達的miRNA,目前還沒有文獻報道這些miRNAs在羊毛彎曲形成中的具體作用,其功能尚需進一步研究。
4 結 論
研究利用高通量測序技術比較了不同羊毛彎曲頻率中國美利奴羊皮膚組織中miRNA差異表達,共鑒定出了25個差異表達的miRNAs,其中,8個上調,17個下調。靶基因預測和功能富集分析表明,差異表達miRNA可能在羊毛毛囊發育相關通路中起作用。

