基于轉錄組的池蝶蚌系統發育及其遺傳分析
周 偉 張萬昌 周 葉 王小敏 胡蓓娟 王軍花 洪一江
(南昌大學生命科學學院, 江西省水產動物資源與利用重點實驗室, 南昌 330031)
池蝶蚌(Hyriopsis schlegelii)和三角帆蚌(Hyriopsis cumingii)是目前在我國養殖的優質淡水珍珠育珠蚌, 它們具有相似的外形和生理功能, 但人們在分類學上對它們有不同的認識。有的認為它們是同屬不同種[1], 有的認為是同種不同亞種[2]。隨著分子生物學和生物化學的發展, 有研究者對池蝶蚌和三角帆蚌的同工酶和ITS1基因等進行了分析, 這些研究表明池蝶蚌和三角帆蚌的親緣關系很近, 達到了種內分化的水平[2,3]。此外, 已有的研究表明: 相比于三角帆蚌, 池蝶蚌在生長速度、育珠性能和抗病性等方面更具優勢[4,5]。然而, 鮮有基于基因組或轉錄組對二者進行比較分析的報道。
雖然同工酶和ITS1基因等分析表明池蝶蚌和三角帆蚌具有更近的親緣關系, 但是它們的種間關系還存在爭議, 因為這些傳統的分子進化研究方法幾乎都是選取一個或多個大分子的序列為標準以研究其在各物種同源序列之間的差異, 如16S rRNA和同工酶等。然而, 一個物種的基因編碼了數以萬計的序列, 以其中一個序列的差異代表整個生物體的差異是不全面的。因此, 有必要從生物體基因組或轉錄組水平來探討池蝶蚌和三角帆蚌的親緣關系。此外, 池蝶蚌生長、育珠和抗病等優勢性能相關的分子機制也尚不清楚。然而, 池蝶蚌和三角帆蚌的基因組和轉錄組信息是未知的。
比較轉錄組學是對不同物種或近緣物種的轉錄組進行比較分析。在缺乏基因組信息時, 比較轉錄組學是分析近緣物種系統發育和同源基因家族的有力手段, 基于比較轉錄組學的分析主要應用于近緣物種的進化關系和一些動植物適應性進化相關的研究[6,7]。在本研究中, 我們對包括池蝶蚌和三角帆蚌在內的4種淡水珠蚌轉錄組進行了二代測序(Next Generation Sequencing, NGS)和de novo組裝。在此基礎上, 通過比較轉錄組學的方法對池蝶蚌及其近緣物種進行了系統發育分析, 這旨在更深入地理解池蝶蚌和三角帆蚌的親緣關系。隨后, 我們對池蝶蚌中的特有基因進行了富集分析, 這有助于我們進一步研究池蝶蚌在生長發育、育珠和抗病性等方面的分子機制。
1 材料與方法
1.1 樣品收集、總RNA提取和轉錄組測序
池蝶蚌取自江西省撫州市池蝶蚌良種場, 三角帆蚌取自浙江省金華三角帆蚌良種場, 褶紋冠蚌(Cristaria plicata)和背角無齒蚌(Anodonta woodiana)分別采集于江西鄱陽湖和山東省微山湖。對這4種蚌每種隨機選取健康的三齡蚌10只, 置于充分曝氣的水族箱內暫養一周, 水溫控制在18—25℃。
從水族箱中分別取出4種淡水珠蚌, 每種蚌為一組, 每組各3只。分別取每組每只蚌的多種組織(包括閉殼肌、心臟、腎臟、鰓、斧足、性腺、肝臟、腸胃和外套膜)各100 mg, 并分別將取出的三份相同組織混合。隨后采用Trizol試劑法提取4種淡水珠蚌的總RNA。以每種蚌各組織提取到的總RNA濃度為依據,取等量的各組織總RNA進行混合, 最后分別得到4種淡水珠蚌混合組織的總RNA樣品。本研究采用的建庫類型為真核鏈特異性文庫(250—300 bp), 之后在Illumina HiseqXten平臺上進行雙端測序。
1.2 De novo組裝和功能注釋
用Trimmomatic[8]移除reads中的Illumina測序接頭和引物序列, 去除質量過低的序列(Phred數值小于20), 過濾過短(序列長度小于50 bp)和不合格的序列。采用Trinity軟件[9]分別對4種淡水珠蚌轉錄組測序數據進行de novo組裝, 所有參數設置為默認參數。首先用CD-HIT軟件去除冗余轉錄本序列[10],再用Corset軟件進行分層聚類[11], 將分層聚類后得到的轉錄本序列進行后續的分析。對轉錄本序列進行統計并對其拼接質量進行評估。
首先用TransDecoder軟件(Find Coding Region Within Transcripts, https://transdecoder.github.io/)對轉錄本序列進行ORF框預測并翻譯成蛋白序列, 然后用EggNOG-mapper將4種淡水珠蚌的蛋白序列分別比對到EggNOG數據庫, 依據同源序列的相似性進行基因功能注釋[12—14]。
1.3 直系同源基因鑒定和基因家族聚類
用Orthofinder對4種淡水育珠蚌的蛋白序列進行同源基因家族分析[15], 參數設置使用Diamond[14]以默認的Evalue值1e-3進行all-vs-all搜索, 尋找潛在的同源基因。使用馬爾可夫聚類算法(Markov Cluster Algorithm, MCL)對基因進行聚類, 劃分直系同源簇。提取4種淡水珠蚌中各自特有的基因家族, 結合之前得到的注釋信息獲取4種淡水珠蚌特有基因對應的功能。基因家族聚類的結果可以直接從orthofinder分析結果中獲取。
1.4 系統發育分析
對4種蚌的單拷貝同源基因進行多序列聯配(Multiple sequence alignment, MSA), 在MEGA軟件中采用pairwise uncorrected p-distance模型對多序列聯配得到的長片段序列的遺傳距離進行計算[16]。從Uniprot數據庫(https://www.uniprot.org/proteomes/)下載光滑雙臍螺(Biomphalaria glabrata)、太平洋牡蠣(Crassostrea gigas)、霸王蓮花青螺(Lottia gigantea)、蝦夷扇貝(Mizuhopecten yessoensis)和加利福尼亞雙斑章魚(Octopus bimaculoides)這5個物種的蛋白質組數據, 結合本研究中的4種淡水珠蚌蛋白序列數據共9個貝類物種以供進一步的分析。隨后利用Orthofinder對這9個物種蛋白質組序列篩選出的1317個共有單拷貝同源基因重建了貝類的系統發育。默認使用mafft[17,18]進行多序列聯配, 用fasttree推斷系統發生樹[19]。
通過生命進化時間的公共信息庫TimeTree網站(http://www.timetree.org/)查找到霸王蓮花青螺與光滑雙臍螺分歧時間大約在471(423—519)百萬年前(Millions of years ago, Mya), 太平洋牡蠣與蝦夷扇貝分歧時間大約在455(421—497)百萬年前。以上述2個已知的分歧時間作為固定值和校正值, 根據上一步得到的系統發生樹用最大似然法(r8s軟件)預測它們的分歧時間[20]。
1.5 池蝶蚌特有基因分析
首先用AnnotationForge[21]依據池蝶蚌功能注釋信息構建OrgDb數據包, OrgDb包用于存儲不同數據庫基因ID之間的對應關系及基因與GO、KEGG等注釋的相對關系。根據同源基因家族的分析結果提取池蝶蚌中特有基因, 隨后基于本研究構建的池蝶蚌OrgDb數據包用clusterprofiler[22]對池蝶蚌中特有基因進行KEGG和GO富集分析。
2 結果
2.1 轉錄組組裝和注釋
通過Illumina測序獲得了池蝶蚌、三角帆蚌、背角無齒蚌和褶紋冠蚌的轉錄組數據, 質控后分別得到12.97、13.58、13.41和11.36 Gb轉錄組測序數據, 各含有clean reads約4323萬、4525萬、4471萬和3785萬條。對這4種蚌的轉錄組數據進行了de novo組裝, 其中池蝶蚌轉錄組組裝后得到138948個轉錄本和104803個Unigenes, reads回貼率(reads回貼到轉錄本的比率)達85.62%, Unigenes的平均長度為1373.20 bp, N50長度為1928 bp(表 1)。以后生動物單拷貝同源基因集作為比對數據庫, 使用Busco軟件對轉錄組完整性進行分析, 結果表明: 在4種淡水珠蚌的轉錄本中, 超過98%都是完整的, 只有不到2%的轉錄本是部分比對或比對不上, 這說明我們組裝結果完整度較好, 轉錄本序列可用于后續分析(表 2)。
將得到的池蝶蚌、三角帆蚌、背角無齒蚌和褶紋冠蚌基因翻譯成蛋白序列后比對到EggNOG數據庫, 基于相似性(E-value<10–3), 這4種蚌分別有34143、28180、28609和24778個基因得到了注釋,在蛋白質直系同源簇(Clusters of orthologous groups of proteins, COG)分類統計表明, 這4種育珠蚌的COG類別分布大致相同, 其中, 富集基因最多的2個分類是“功能未知(S. Function unknown)”和“信號轉導機制(T. Signal transduction mechanisms)”(圖 1)。
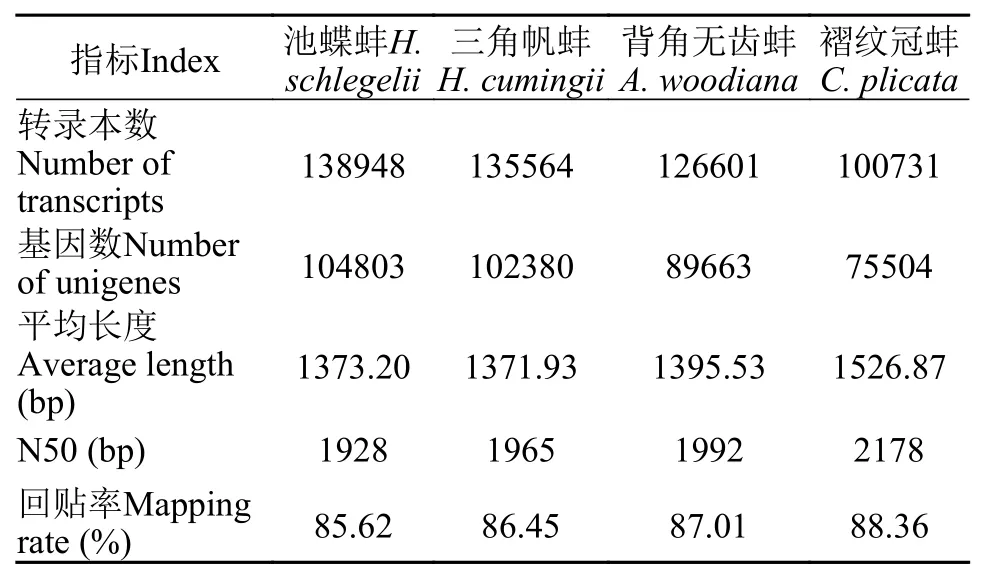
表 1 轉錄組組裝數據統計Tab. 1 The statistic of transcriptome assembled data

表 2 組裝轉錄組的BUSCO分析結果Tab. 2 Results of BUSCO analysis on the assembled transcriptomes

圖 1 池蝶蚌(a)、三角帆蚌(b)、背角無齒蚌(c)和褶紋冠蚌(d)的蛋白質直系同源簇(COG)分類Fig. 1 Clusters of Orthologous Group of proteins (COG) classifications for H. schlegelii (a), H. cumingii (b), A. woodiana (c) and C.plicata (d)
2.2 直系同源基因的鑒定及基因家族聚類
通過Orthofinder在4種淡水珠蚌中共鑒定到29468個潛在的同源基因家族, 其中4個物種共享的16001個同源基因家族構成了淡水珠蚌核心基因集,這些核心基因參與廣泛的基礎活動, 幾乎在每一個GO功能中都有分布。只存在于池蝶蚌和三角帆蚌中的同源基因家族有5175個(圖 2)。另外, 池蝶蚌、三角帆蚌、褶紋冠蚌和背角無齒蚌4種育珠蚌間各自特有的基因家族數量分別為12、11、9和15個。注釋結果表明, 池蝶蚌特有基因主要與生長發育(TRIM45、TRIM33和JAG1等)和免疫(ABCB9)等過程相關, 三角帆蚌的特有基因與抗腫瘤(CUZD1)和甾醇代謝(HDLBP)等功能有關, 褶紋冠蚌特有基因主要與離子平衡(SLC12A2)和免疫(CEACAM1)功能相關, 背角無齒蚌特有基因主要與疾病(TMPRSS12和CLCA2)、細胞內通訊和信號傳遞(FLNB)等功能相關(表 3)。

圖 2 池蝶蚌、三角帆蚌、褶紋冠蚌和背角無齒蚌中的基因家族聚類韋恩圖Fig. 2 Gene families clustering venn diagram in H. schlegelii, H.cumingii, C. plicata and A. woodiana

表 3 四種淡水珠蚌中物種特有基因列表Tab. 3 The list of species-specific genes in four freshwater mussels
2.3 系統發育分析
我們對池蝶蚌、三角帆蚌、背角無齒蚌和褶紋冠蚌的單拷貝同源基因進行了多序列聯配, 采用pairwise uncorrected p-distance模型對多序列聯配得到的長片段序列計算遺傳距離(E-value<1e–3), 結果表明池蝶蚌和三角帆蚌的遺傳距離小于1%, 而池蝶蚌與其他物種的遺傳距離幾乎是它與三角帆蚌的10倍(表 4)。結合Uniprot數據庫中已發布的光滑雙臍螺、太平洋牡蠣和蝦夷扇貝等5個物種的蛋白質組數據和本研究中獲得的4種淡水珠蚌的Unigenes對應的蛋白質組數據, 我們篩選了包含1317個共有單拷貝直系同源基因家族的蛋白集, 隨后利用這些單拷貝同源蛋白集重建了貝類的系統發育。結果表明, 池蝶蚌與三角帆蚌的親緣關系最近, 2個物種聚為一支(帆蚌屬)后再與冠蚌屬、無齒蚌屬聚在一起, 瓣鰓綱的真瓣鰓目與異柱目聚為一支(瓣鰓綱), 然后它們再與腹足綱、頭足綱聚在一起(圖 3)。隨后, 我們對這9個物種進行了分歧時間評估, 結果表明, 池蝶蚌與三角帆蚌大約在424萬年前發生分歧, 帆蚌屬與冠蚌屬、無齒蚌屬發生分歧的時間大約在5144萬年前, 真瓣鰓目與異柱目大約在5.72億年前發生分歧, 瓣鰓綱、腹足綱和頭足綱大約在6.18億年前發生分歧(圖 4)。此外, 我們還對它們的同源基因家族擴張和收縮情況進行了分析, 發現池蝶蚌在淡水珠蚌中發生了最多的同源基因家族擴張。
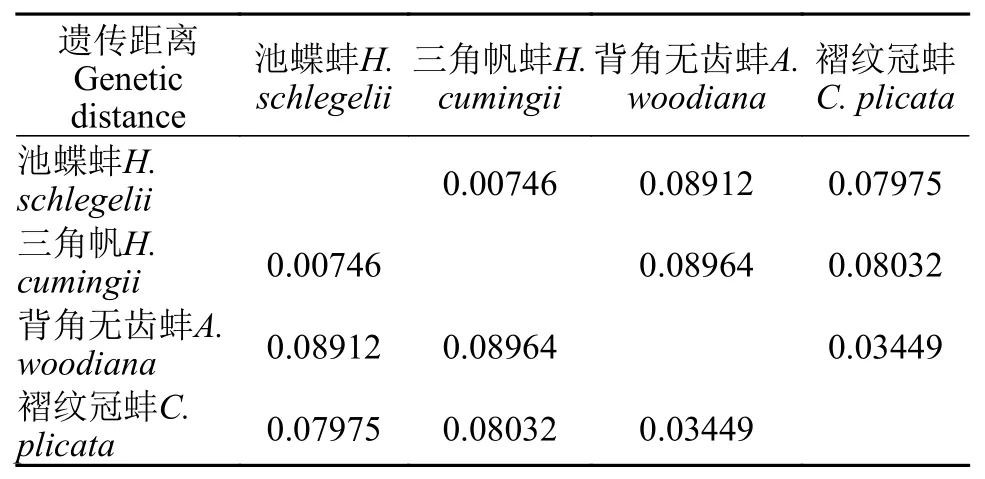
表 4 基于4種淡水珠蚌單拷貝同源基因序列的遺傳分化距離Tab. 4 Genetic differentiation distance based on single copy ortholog sequences of four freshwater mussel
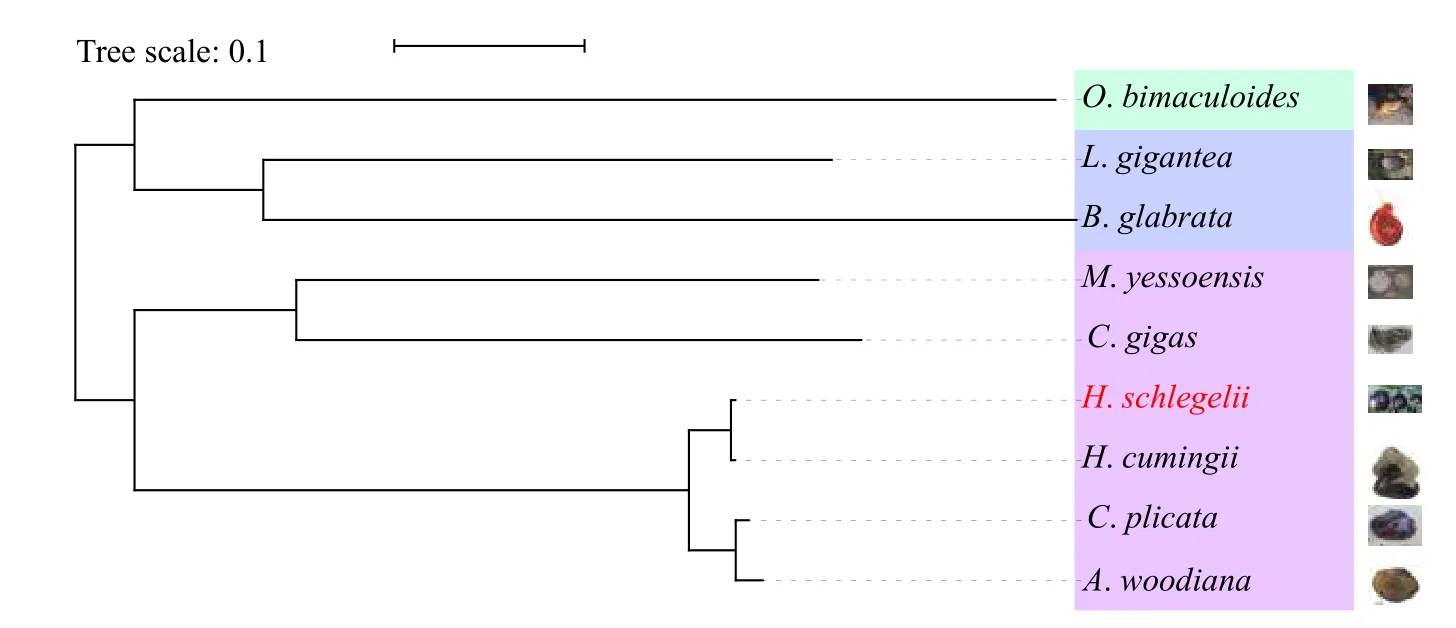
圖 3 九種貝類動物的系統發育樹Fig. 3 Phylogenetic tree of nine species in mollusca

圖 4 九種貝類動物的分歧時間及其基因家族擴張/收縮情況Fig. 4 Time of divergence of nine species in mollusca and their expansion/contraction of genefamilies
2.4 池蝶蚌相對于三角帆蚌的特有基因
通過比較池蝶蚌和三角帆蚌的轉錄組, 得到了池蝶蚌轉錄組中相對于三角帆蚌特異性表達的基因3721條, 其中345個特有基因得到了注釋。這些特有基因可能與池蝶蚌的生長發育、育珠及抗病能力相關。我們對這些特有基因進行了KEGG和GO富集分析。KEGG 通路富集分析結果表明, 池蝶蚌特有基因主要富集在免疫細胞膜分子(CD molecules)和蛋白消化吸收(Protein digestion and absorption)等通路上(圖 5)。此外, 池蝶蚌特有基因GO注釋結果表明: 有很多特有基因與池蝶蚌的優良性狀相關, GO類別包括生長發育過程(91個), 系統發育(69個), 動物器官發育(51個), 免疫系統過程(25個), 抗原的處理和呈遞(4個, 圖 6)。
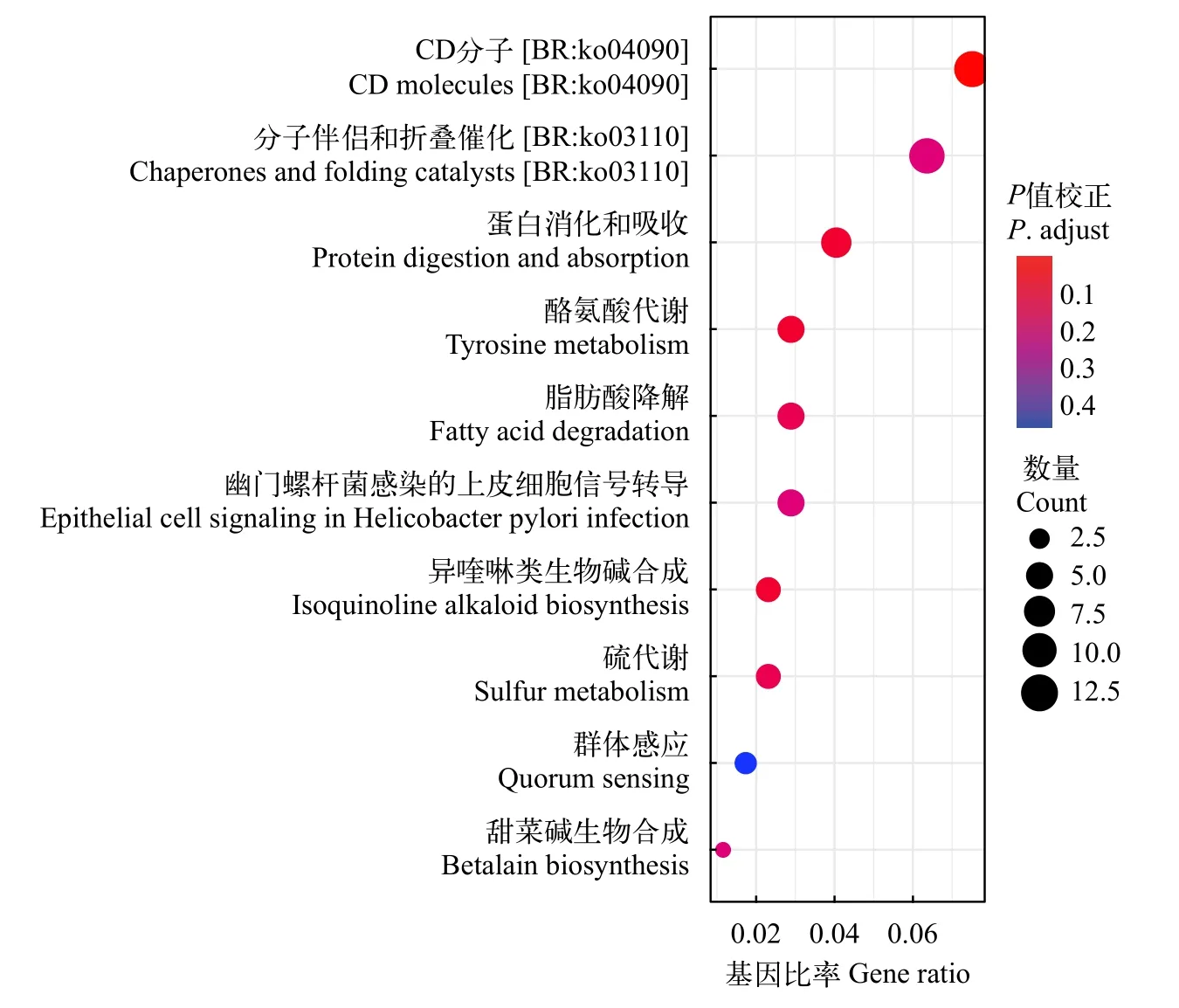
圖 5 池蝶蚌特有基因的KEGG富集分析Fig. 5 KEGG enrichment analysis of species-specific genes in H. schlegelii
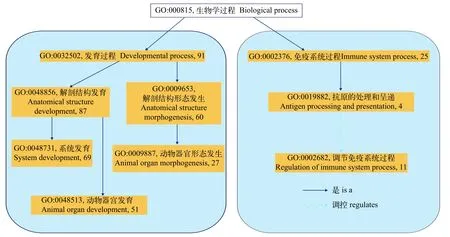
圖 6 池蝶蚌優勢性能相關的特有基因GO富集Fig. 6 GO enrichment of specific genes related to ecological adaptation in H. schlegelii
3 討論
3.1 池蝶蚌與三角帆蚌的親緣關系
池蝶蚌和三角帆蚌的親緣關系一直存在爭議,這是由于我國的淡水雙殼類蚌科分類系統主要依據傳統的形態學分類, 而蚌科動物存在趨同現象且貝殼具有高度可塑性, 因此傳統的形態學分類并不能完全反映各類群之間的親緣關系。近年來, 分子生物學技術常用于解決一些分類學上的疑難問題。在淡水珠蚌中的分子進化研究方法, 大多采用某個大分子的序列為標準, 通過比較相應物種的同源序列分析物種的進化關系。以往池蝶蚌和三角帆蚌的分子進化研究主要是基于同工酶[2]、ITS1[3]和線粒體DNA[23]的比較分析, 但這樣單個基因或少數基因的比較不能代表整體差異, 而且可能由于遺傳變異存在一定的偶然性, 不足以使人信服。比較基因組或比較轉錄組可以最大限度地上對不同物種的遺傳差異進行分析, 在缺乏基因組信息的非模式生物中, 往往可以基于轉錄組數據解決物種的進化關系[24,25]。我們基于轉錄組水平的系統發育分析表明池蝶蚌與三角帆蚌的遺傳距離不到1%, 大大低于其他淡水珠蚌間的遺傳距離, 這與傳統的形態學分類有所不同, 但與Sakai等[2]的研究結果一致,很大程度上支持了池蝶蚌與三角帆蚌可能是不同亞種或不同種群的假設, 二者的形態學差異可能是由于生存環境差異造成的。進一步的分析表明, 小方蚌亞科與無齒蚌亞科大約在5144萬年前發生分歧, 而池蝶蚌和三角帆蚌發生分化的時間大約在424萬年前, 這表明可能約在424萬年前由于某些環境變化使池蝶蚌和三角帆蚌發生地理隔離導致它們產生一些遺傳上的差異, 但這些差異還處于種內分化的水平。
3.2 池蝶蚌中的特有基因
特有基因的分析能幫助我們理解表型性狀產生的分子機制, 同時可以揭示基因表達模式與表型形成之間的關系。有研究表明, 池蝶蚌生長速度快,其殼寬、外套膜的厚度、晶桿的長度和直徑都大于三角帆蚌[4]。個體的生長及其形態與生長發育過程息息相關, 如外套膜的生長與發育。本研究對池蝶蚌和三角帆蚌轉錄組中的基因進行比較分析, 結果表明, 根據GO分類, 多達51個池蝶蚌特有基因被標注為動物器官發育。其中, 基因α-Ⅰ型膠原質(COL1A1)主要負責編碼Ⅰ型膠原的主要成分, 即大部分結締組織中的纖維膠原, 包括軟骨[26]。另有基因JAG1和Notch信號的調控有關, 該基因在胚胎發育中起著重要作用, 可能涉及造血過程中的細胞命運決定和抑制成肌細胞的分化[27]。結締組織中的纖維膠原可能與池蝶蚌外套膜的厚度相關, 而胚胎發育對有機體的生長發育至關重要。這表明池蝶蚌中可能存在一些特有基因或者調控這些特有基因表達的相關基因, 使得池蝶蚌在生長發育方面優于三角帆蚌。
此外, 有研究表明池蝶蚌對蚌瘟病和嗜水氣單胞菌的抵抗力明顯強于三角帆蚌[5,28]。有機體的抗病能力主要依賴免疫系統發揮防御作用, 這個過程包括抗原的識別和免疫系統的調控。GO富集結果表明有25個池蝶蚌特有基因涉及免疫系統過程, 這個結果與富集在免疫細胞膜分子通路中的13個基因相一致。作為免疫系統過程的重要環節, 抗原的處理和呈遞發揮著重要的作用。例如, 基因ABCB9是ATP依賴的低親和力肽轉運體, 它將廣譜的多肽從胞漿轉移到溶酶體腔[29]。類似地, 基因SEC24A編碼衣殼蛋白復合物Ⅱ的組成成分, 促進內質網轉運囊泡的形成。衣殼主要有兩種功能, 一是內質網膜在囊泡中的物理變形, 二是選擇轉運到高爾基體的貨物分子, 這與異質蛋白在內質網-高爾基體界面被識別、分揀和捕獲有關。抗原的有效識別和處理決定了免疫系統能否有效地發揮作用[30]。基于以上結果, 我們推測池蝶蚌優良的抗病能力可能與池蝶蚌中特有的免疫基因有關。
4 結論
本研究通過轉錄組學分析對池蝶蚌和三角帆蚌進行了比較, 為厘清池蝶蚌和三角帆蚌的親緣關系提供了重要的證據和幫助, 并為研究池蝶蚌優良性狀相關的分子機制提供了新的視角和有力的支持。在本研究中, 我們的計算表明池蝶蚌和三角帆蚌的遺傳距離不到1%, 大約只有其他蚌種間遺傳距離的十分之一, 這與種內分化水平相當。另外,本研究對包括池蝶蚌、三角帆蚌、背角無齒蚌和褶紋冠蚌在內的9種貝類重建了貝類系統發育分析,結果表明, 池蝶蚌和三角帆蚌發生分歧的時間大約在424萬年前。池蝶蚌產自日本, 而三角帆蚌是中國本土物種, 可以推斷, 它們可能在那個時間點由于某些事件發生了地理上的隔離。池蝶蚌在一些性能上優于三角帆蚌, 這很可能是遺傳差異導致的。池蝶蚌轉錄組中特有基因富集分析結果說明池蝶蚌具有潛在的優勢性能, 如生長發育和免疫。這很可能是池蝶蚌在生長速度、外套膜厚度和抗病能力等方面具有優勢的關鍵所在。由于轉錄組學分析難以獲得全部和完整的遺傳信息, 因此需要進一步的研究。盡管如此, 本研究依然為池蝶蚌和三角帆蚌的親緣關系提供了重要參考價值, 為進一步研究池蝶蚌生長發育和免疫等相關的分子機制提供了有益線索。

