氨氮脅迫下菲律賓蛤仔肝胰腺內參基因的篩選
徐宏超,邢榮蓮,李源美,叢明
煙臺大學,煙臺 264005
過去30年,我國海水養殖規模迅猛發展,產量得到快速增長。其中,海洋軟體動物的產量最高,達到海水養殖總產量的72.6%~78.6%,因此成為我國海水養殖產量的主要貢獻者[1]。海岸帶區域是海洋生物特別是海洋軟體動物的重要養殖區域[2],海岸帶環境的健康狀況與軟體動物尤其貝類養殖的健康發展密切相關。隨著現代工農業的快速發展和人民生活水平的大幅提高,大量污染物未經過嚴格的凈化處理就排放到環境中,并隨著徑流進入海洋環境中。由于海岸帶位于河流和海洋的交匯處,因此陸源污染導致海岸帶的污染狀況尤其嚴重。國家海洋水質質量標準規定,養殖海域非離子氨的限定濃度為0.02 mg·L-1(GB11607—89)。然而,2019年中國生態環境報告顯示,入海河流的氨氮濃度范圍為0.02~26.4 mg·L-1,平均濃度為0.64 mg·L-1,遠遠超過養殖區域內海水氨氮的安全濃度[3]。已有研究表明,海洋環境中的氨氮對軟體動物的生長發育產生嚴重影響,但是對其致毒機制還不是很清楚,因此有必要從基因水平對其毒性進行分析研究[4-6]。
實時熒光定量PCR是檢測基因表達的重要方法之一,可以很好地反映受試生物體中相關基因表達的變化情況。通常可以選擇一個內參基因作為其他基因表達的對照,如果內參基因的表達不穩定或者是不同的實驗條件下發生改變,則無法檢測其他基因表達的微小變化,甚至可能導致錯誤的實驗結果。因此,為特定的實驗目的篩選合適的內參基因非常重要。理論上,內參基因的表達對維持組織細胞的生命活動是必不可少的,在細胞中的表達比較恒定。但是,研究發現在生物體不同的發育階段、不同的組織以及不同的實驗條件下基因表達水平存在很大差異[7-8]。李迪等[9]以鱖魚6個不同組織和5個不同發育階段為研究對象,從GAPDH、β-Actin和18SrRNA這3個備選內參基因中篩選最適內參基因。結果表明,在胚胎發育階段β-Actin表達最穩定;不同組織樣品中,GAPDH表達最穩定。鮑相渤等[10]研究Actin、GAPDH、Tubu、Cytb、Tbp和EF-1α等6個備選內參基因,在饑餓脅迫下各組織、致病菌感染前后和水環境升溫前后不同時段血樣中mRNA的表達情況,結果表明,饑餓脅迫條件下,在鰓、腎和血淋巴中Actin的表達穩定,但是在外套膜、肝胰臟和閉殼肌中的表達不穩定;致病菌感染條件下,Actin的表達最穩定;而在水溫升高的條件下,Actin的穩定性降低;GAPDH在饑餓脅迫中的表達穩定性較差,但是在致病菌感染和溫度改變的條件下相對穩定。
海洋軟體動物的鰓和肝胰腺組織在免疫應答和解毒過程中都發揮重要作用,前期我們已經對無機氮脅迫條件下菲律賓蛤仔鰓組織的內參基因做了深入研究[11-12],但是對肝胰腺中內參基因的表達情況不甚了解。因此,本研究以菲律賓蛤仔為研究對象,采用環境相關濃度(0.1 mg·L-1和0.5 mg·L-1)的氨氮暴露菲律賓蛤仔,在暴露3 h、1 d、3 d、7 d和14 d后分別取肝胰腺組織作為實驗材料,篩選不同濃度氨氮暴露后,菲律賓蛤仔肝胰腺組織穩定表達的內參基因。本實驗選擇了肌動蛋白基因(β-Actin、Actin)、轉錄延伸因子基因(elongation factor 1-alpha,EF-1α)、微管蛋白基因(beta-tubulin,Tubu)、核糖體(18S RNA, 18S)、泛素蛋白基因(ubiquitin,Ubi)和親環素基因(cyclophilin A,CyPA)作為備選內參基因,以肝胰腺cDNA為模板,利用qRT-PCR檢測備選基因的表達情況,并結合geNorm、NormFinder和BestKeeper程序評估其穩定性,最后通過RefFinder軟件綜合分析不同氨氮濃度暴露后菲律賓蛤仔肝胰腺組織的最適內參基因,為后續研究不同氨氮濃度條件下菲律賓蛤仔肝胰腺組織的基因表達提供定量分析基礎。
1 材料與方法(Materials and methods)
1.1 材料
本實驗所用的菲律賓蛤仔于2019年10月購自煙臺萊山區佳世客超市,選取健康、生命力強、體態均勻(長3.77 cm±0.12 cm)的個體。氨氮暴露實驗開始前,將菲律賓蛤仔放入室內裝有40 L過濾海水的養殖池(60 cm×60 cm×30 cm)里暫養7 d,水溫(18±1) ℃,pH 8.0,鹽度30‰,24 h曝氣供氧;按照100只菲律賓蛤仔稱取1.0 g螺旋球藻喂食,每天喂食1次,喂食前和喂食后2 h各換水一次。從第8天開始,根據海水的體積、溫度和pH值,向養殖池中加入對應體積的1 mg·L-1分析純氯化銨配制的母液(國藥集團,上海),使其氨氮濃度分別達到0.1 mg·L-1和0.5 mg·L-1。
我們的前期研究表明,0.1 mg·L-1和0.5 mg·L-1氨氮濃度脅迫菲律賓蛤仔21 d,能夠導致菲律賓蛤仔鰓組織出現膜系統損傷、攝食功能下降、鰓組織結構損傷甚至死亡等嚴重毒性效應[13-14]。因此,我們同樣采用相同的氨氮濃度梯度進行14 d暴露實驗,即設置0.1 mg·L-1暴露組、0.5 mg·L-1暴露組和空白對照,每個實驗組設置3個重復,每個重復組30只菲律賓蛤仔。當蛤仔暴露3 h、1 d、3 d、7 d和14 d后,從每個實驗重復組中隨機取2個蛤仔,摘取肝胰腺組織,濾紙吸干水分后迅速放入Trizol試劑中,-80 ℃保存。
1.2 總RNA提取及可用性檢測
采用Trizol試劑盒(TaKaRa,大連),提取肝胰腺組織的總RNA。勻漿管中加入0.8 mL預冷的Trizol;取菲律賓蛤仔的肝胰腺組織放入勻漿管中,置于勻漿機(Bertin Precellys 24,法國)勻漿20 s;轉移0.8 mL的勻漿液于1.5 mL EP管中,冰上放置5 min;加入200 μL的氯仿并用力震蕩30 s,冰上放置5 min后,4 ℃ 12 000g離心10 min;取300 μL的上清液,加入等體積的異丙醇并-20 ℃放置15 min后,4 ℃ 12 000g離心5 min;倒掉上清液,RNA沉淀用1.0 mL 75%的乙醇洗滌2次,4 ℃ 7 500g離心5 min;倒掉乙醇放置超凈臺干燥,待乳白色沉淀變為無色膠透明狀,加入30 μL RNase free水并放置于冰盒中溶解RNA沉淀;使用1%瓊脂糖凝膠電泳檢測RNA的完整性,利用Nanodrop 2000(Thermo Scientific,美國)檢測其純度和濃度,當OD260/OD280在1.8~2.2范圍內時,認為RNA的純度滿足qRT-PCR的實驗條件。
1.3 cDNA合成及擴增濃度確定
普通PCR反轉錄RNA合成cDNA,按照TransScript?One-Step gDNA Removal and cDNA Synthesis SuperMix (Transgen Biotech)提供的試劑和操作方案反轉錄合成cDNA。反應體系為:總體積20 μL,包括2 μL RNA、1 μL Anchored Oligo(dT)18Primer(0.5 μg·μL-1)、10 μL 2×TS Reaction Mix、1 μL TransScript?RT/RI Enzyme Mix、1 μL gDNA Remover、5 μL RNase-free water;反應條件為:42 ℃孵育15 min;85 ℃加熱5 s失活TransScript?RT/RI與gDNA Remover。
1.4 實時熒光定量PCR
根據前期研究基礎[11-12],本次實驗選取Actin、EF-1α、Tubu、18S、Ubi和CyPA作為備選內參基因,其定量引物如表1所示,以菲律賓蛤仔肝胰腺組織的cDNA作為模板進行實時熒光定量PCR擴增。

表1 內參基因的引物序列Table 1 Primer sequences for housekeeping genes
實時熒光定量PCR反應在ABI 7500 Real-Time Detection System (Applied Biosystems,美國)中進行,根據TransStart Tip Green qPCR SuperMix(Transgen Biotech)提供的試劑和操作方法擴增目的基因。其擴增的體系為:總體積20 μL,包括6 μL Template cDNA、0.4 μL Forward Primer(10 μmol·L-1)、0.4 μL Reverse Primer(10 μmol·L-1)、10 μL 2×TransStart tip green qPCR superMix、0.4 μL Passive Reference Dye Ⅰ、2.8 μL Nuclease-free water;反應條件為94 ℃ 30 s;(94 ℃ 5 s、58 ℃ 15 s、72 ℃ 10 s)×40 cycles,擴增結束后進行熔解曲線的數據分析。
1.5 內參基因穩定性分析
對備選內參基因進行qRT-PCR檢測獲得CT值,用-2-ΔΔCT法[15](ΔCT=CTsample-CTmin)計算每個基因的相對表達量。利用geNorm[16]、NormFinder[17]和BestKeeper[18]軟件3種方法對6個內參基因的表達穩定性值(expression stability value, M)進行評價,并且最終根據RefFinder軟件[19]確定合適的內參基因。根據數值越小穩定性越好的原則,篩選出表達最穩定的內參基因。
geNorm軟件是一款用于微軟Excel平臺的VBA宏程序,分析Actin、18S、Ubi、CyPA、EF-1α和Tubu的-2-ΔΔCT,輸出平均穩定性值(M),M<1.5的基因被認為是可行性的基因。根據M值越小基因表達穩定性越好的原則,確定備選內參基因表達穩定性大小。另外據M值還可以得出配對差異值(Vn/Vn+1),系統默認配對差異值為0.15,若Vn/Vn+1<0.15,則最適內參基因適配數為n,若Vn/Vn+1>0.15,則最適內參基因適配數為n+1。
NormFinder是根據差異性來評估內參基因穩定性的軟件,它的算法和geNorm相似,也是分析內參基因數據得出M值,根據M值越小其穩定性越好的原理篩選最佳內參基因,但是它無法確定最適內參基因的數目。
BestKeeper軟件輸入每個備選內參基因的CT值,并且基于標準差(SD)和變異系數(CV)值進行計算,確定內參基因的穩定性,基因的穩定性與SD和CV值成反比。SD值<1的備選內參基因被認為是可接受作為內參基因的;而SD值>1被認為是不可接受的。
RefFinder是一款基于Web的內參基因綜合評估工具,根據大量的CT值評估和選擇內參基因。這個軟件集合geNorm、NormFinder、BestKeeper軟件和ΔCT4種內參基因的評估方法,比較和排列備選內參基因。每個內參基因分配適當的權重,計算它們權重的幾何平均值,最終根據賦值越小穩定性越好的原則,確定最穩定的內參基因。
2 結果(Results)
2.1 RNA、cDNA和引物的質量檢測
提取的總RNA進行1%的瓊脂糖凝膠電泳,有28S、18S和5S條帶(圖1),并且用Nanodrop 2000檢測純度,OD260/OD280在1.8~2.2范圍內。結果表明,提取樣品RNA的完整性和純度良好,符合qRT-PCR的質量要求。備選內參基因的qRT-PCR結果顯示,熔解曲線呈單一的峰并且重復性好(圖2)。因此,所設計的引物特異性好,適合作為qRT-PCR擴增引物。

圖1 對照組、氨氮脅迫組菲律賓蛤仔的肝胰腺組織總RNA電泳圖譜注:C1~C6,Control組;M,Marker;A1~A6,0.1 mg·L-1氨氮脅迫組;B1~B6,0.5 mg·L-1氨氮脅迫組。Fig. 1 Gel electrophoresis of total RNA samples from the hepatopancreas tissues of R. philippinarum in the control and ammonia nitrogen exposed groupsNote: C1~C6 denote control groups; M denotes Marker; A1~A6 denote 0.1 mg·L-1 of ammonia nitrogen-exposed groups; B1~B6 denote 0.5 mg·L-1 of ammonia nitrogen-exposed groups.

圖2 6個備選內參基因擴增產物的熔解曲線Fig. 2 Melting curves of PCR products from 6 reference genes
2.2 cDNA的濃度確定
對樣品cDNA模板進行梯度稀釋,稀釋倍數為10倍、20倍、100倍、200倍和1 000倍。qRT-PCR結果表明,10倍和20倍稀釋濃度下CT值在20.0~25.0之間,其他的稀釋濃度的CT值>25.0。因此,10倍和20倍稀釋濃度的cDNA模板適合作為檢測模板,本次實驗采用稀釋20倍的cDNA為定量模板。
2.3 內參基因穩定性分析
2.3.1 geNorm軟件分析
通過geNorm軟件分析(表2),得到Actin、18S、Ubi、CyPA、EF-1α和Tubu的平均表達穩定性值M值分別為1.029、1.207、1.029、1.085、1.861和1.403。根據M值越小基因的穩定性越好的原則,6個備選內參基因的穩定性大小為Actin=Ubi>CyPA>18S>Tubu>EF-1α。因此,geNorm分析表明Actin和Ubi是表達相對最穩定的內參基因。
內參基因的數目通過geNorm軟件分析得到的配對差異值Vn/Vn+1來確定,一般默認值為0.15,當Vn/Vn+1<0.15時,選擇n個基因作為內參基因,不必添加基因來提高基因表達分析的準確性;當Vn/Vn+1>0.15時選擇n+1個基因作為內參基因。在本研究中Vn/Vn+1均>0.15(圖3),無法通過geNorm確定內參基因的數目。

圖3 geNorm分析氨氮暴露后內參基因表達的穩定性Fig. 3 Stability value of reference gene expression after ammonia nitrogen exposure by geNorm
2.3.2 NormFinder軟件分析
NormFinder分析結果顯示(表2),6個備選內參基因的表達穩定值分別為Actin(0.075)、18S(0.416)、Ubi(0.363)、CyPA(0.284)、EF-1α(0.932)和Tubu(0.475),即NormFinder分析表明表達最穩定的基因是Actin。
2.3.3 BestKeeper軟件分析
BestKeeper軟件分析結果顯示(表2),Actin、18S、CyPA和Ubi的SD值分別為0.99、0.67、0.81和0.47,均<1;Tubu和EF-1α的SD值分別為1.11和2.19,均>1。根據BestKeeper軟件的規則,SD值<1的基因可接受作為內參基因,否則不可接受為內參基因。因此,BestKeeper軟件分析可知Actin、18S、CyPA和Ubi可接受為內參基因,其中Ubi是表達最穩定的內參基因。
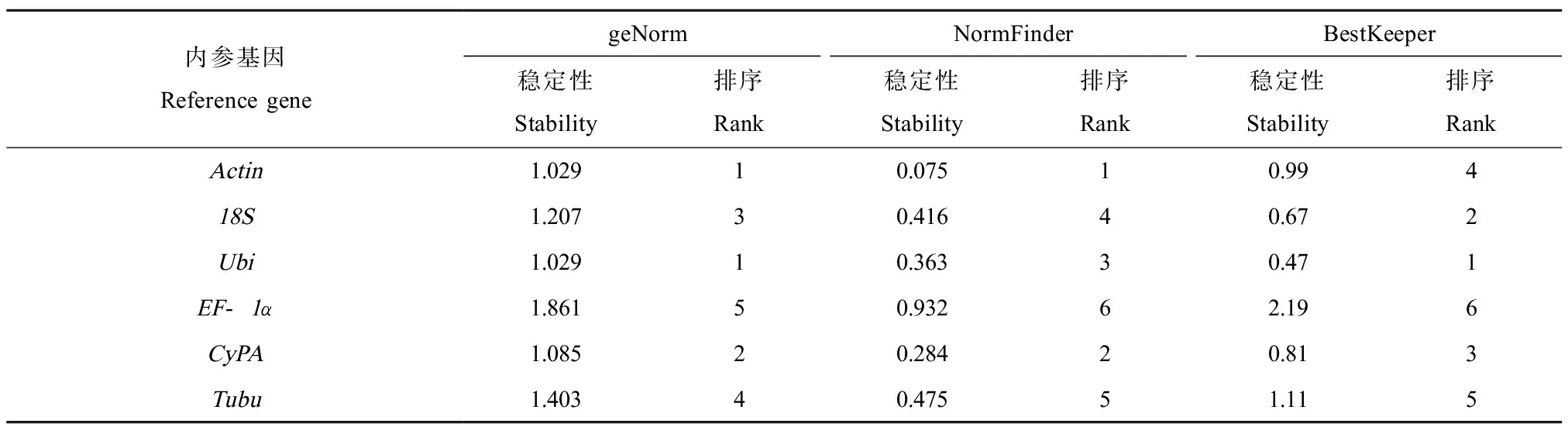
表2 geNorm、NormFinder和BestKeeper分析穩定值排名Table 2 Stability ranking by geNorm、NormFinder and BestKeeper analysis
2.3.4 綜合排名分析
通過在線軟件RefFinder集合geNorm、NormFinder、BestKeeper軟件和ΔCT的內參基因評估方法,將各基因的CT值輸入程序中,程序為每個基因賦值排序,賦值越小則表達越穩定。RefFinder綜合分析6個備選基因表達穩定性大小依次為(表3):Actin(1.32)、18S(1.83)、Ubi(2.00)、CyPA(2.71)、Tubu(5.00)和EF-1α(6.00)。因此,不同濃度氨氮暴露菲律賓蛤仔的肝胰腺組織中,表達相對最穩定的內參基因是Actin。
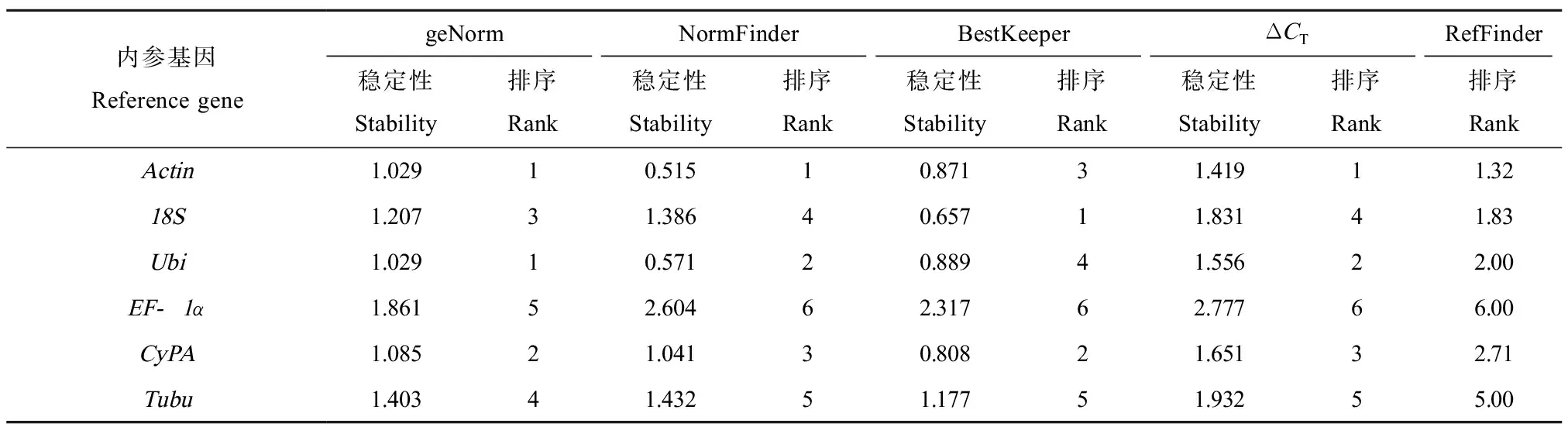
表3 RefFinder分析穩定值排名Table 3 Stability ranking by RefFinder analysis
3 討論(Discussion)
目前,熒光定量PCR是快速、可靠定量目標基因表達水平的常用方法。為了準確比較不同實驗條件下mRNA的轉錄水平,需要選擇一個表達穩定的內參基因作為對照[20]。閆含笑等[21]在研究池蝶蚌β-連環蛋白基因cDNA的克隆及表達特征分析時,采用Actin作為內參基因,對不同組織中的β-連環蛋白基因進行表達定量分析;程雪艷等[22]利用qRT-PCR技術,分析谷胱甘肽過氧化物酶(glutathione peroxidase, GPx)基因在泥蚶的不同組織以及重金屬刺激后的表達量時,選取18S作為內參基因。關于菲律賓蛤仔內參基因的篩選,牟政強等[23]以菲律賓蛤仔的不同發育階段和不同組織為研究材料,運用qRT-PCR技術結合3個內參基因篩選方法geNorm、NormFinder及△CT對12個備選內參基因進行穩定性分析,發現菲律賓蛤仔在不同發育時期表達較穩定的內參基因是CYTC、CYTB5和RPS23,菲律賓蛤仔成體不同組織中表達穩定的內參基因是CYTB5和GFRP2,Actin在菲律賓蛤仔的不同組織和不同發育階段表達皆最不穩定。曹滕飛等[11]以經0.1 mg·L-1氨氮暴露后的菲律賓蛤仔鰓組織為研究材料,篩選穩定表達的內參基因,發現EF-1α是表達最穩定的內參基因。王愛云等[12]以經亞硝態氮暴露的菲律賓蛤仔為研究對象,發現Actin可作為研究亞硝態氮對菲律賓蛤仔鰓組織基因功能的內參基因。綜上可見,即使是同一種生物,受到不同的脅迫處理、同一暴露物的不同暴露濃度、不同發育階段和不同組織等條件下,其內參基因表達的穩定性存在極大差異。因此,為了確保檢測目的基因表達水平的準確性,當實驗條件發生改變時,相應的內參基因篩選工作需要重新進行。
本研究中,使用geNorm、NormFinder和BestKeeper程序分別評估不同氨氮濃度(0.1 mg·L-1和0.5 mg·L-1)暴露下,菲律賓蛤仔肝胰腺組織內參基因的表達穩定性,最后通過RefFinder程序綜合評估內參基因的穩定性。根據geNorm軟件對6個備選內參基因表達的分析結果,備選內參基因穩定性大小為,Actin=Ubi>CyPA>18S>Tubu>EF-1α;NormFinder分析結果表明,菲律賓蛤仔肝胰腺組織中備選內參基因穩定性大小為Actin>CyPA>Ubi>18S>Tubu>EF-1α;BestKeeper軟件分析SD值<1的18S(0.67)、CyPA(0.81)、Actin(0.99)和Ubi(0.47)可作為內參基因,其穩定性大小為Ubi>18S>CyPA>Actin。
由于geNorm、NormFinder和BestKeeper軟件評估的側重點不同,其分析結果存在差異。因此,我們利用包含上述3種方法以及ΔCT的RefFinder程序綜合評估內參基因的穩定性,為每個內參基因分配適當的權重,并且計算它們權重的幾何平均值,最終確定內參基因穩定性的大小。RefFinder分析結果表明(表3),6個備選內參基因穩定性通過geNorm和NormFinder軟件單獨分析的結果與RefFinder的基本一致,BestKeeper軟件單獨分析的結果與RefFinder稍有差異。但是通過RefFinder綜合分析6個備選內參基因的穩定性,其結果為Actin>18S>Ubi>CyPA>Tubu>EF-1α。
另外,單獨通過geNorm軟件計算內參基因的配對差異(Vn/Vn+1)值可確定最適內參基因的數目。不同氨氮濃度暴露菲律賓蛤仔中配對變異值均>0.15(圖3),無法通過配對變異值確定內參基因的數目。但是,根據geNorm提供的手冊,系統默認的配對變異值0.15并非嚴格的標準。大量的研究表明,實驗過程中存在配對變異值>0.15的情況。Zhang等[24]利用qRT-PCR篩選百合中用于基因表達研究的內參基因時,所有的實驗條件下Vn/Vn+1>0.15,無法確定內參基因的數目,但是作者通過geNorm、NormFinder和BestKeeper的分析結果確定適合的內參基因;Li等[25]通過qRT-PCR鑒定合適的內參基因用于人類卵巢腫瘤的基因表達研究,備選內參基因的配對變異值都>0.15,但是根據配對變異值的趨勢,作者推薦GUSB、PPIA和TBP為人類卵巢腫瘤的基因表達研究的內參基因。Penning等[26]研究內參基因在貓的牙(牙根、牙冠)、心(左心室)、腎、肝、肺和乳腺組織里表達的穩定性時,在所有組織中備選的10個內參基因的配對差異分析值均>0.15,需要同時選用6個才能對檢測的所有組織基因表達進行最佳標準化。潘暢等[27]確定RPS23可以作為孟氏隱唇蟲不同發育階段和不同組織的基因表達分析的內參基因,而該研究的Vn/Vn+1>0.15。鮑相渤等[10]用定量PCR研究內參基因在蝦夷扇貝中表達穩定性時,發現饑餓的條件下,血淋巴、腎和閉殼肌中出現了Vn/Vn+1>0.15的情況,這可能時由于饑餓對內參基因的表達產生了影響;此外,該研究在預試驗中還發現,正常喂食的條件下候選內參基因在各種組織間的Vn/Vn+1值也>0.15,作者指出這可能是由于組織的細胞組成類型和比例不同及試驗中的候選基因有限所致。國外的研究者也指出,在某些情況下默認值0.15可能過于嚴格,它受實驗條件、個體間的差異以及基因數量等因素的影響[28-29],因此不必拘泥于Vn/Vn+1值是否<0.15。綜合3個評估方法的分析結果和RefFinder的綜合分析結果,我們認為在當前氨氮濃度(0.1 mg·L-1和0.5 mg·L-1)暴露條件下,Actin是研究菲律賓蛤仔肝胰腺組織內基因表達的最適內參基因。

