抗乳腺癌基質金屬蛋白酶9類抑制劑的分子設計研究*
張茹玉,葉 潘,李月潞,周 迎,陳秀奎,劉 樺
1.成都醫學院 臨床醫學院(成都 610050);2.成都醫學院 生物醫學實驗教學中心(成都 610050)
乳腺癌是女性常見的惡性腫瘤,它起源于乳腺導管內上皮或乳腺腺泡上皮細胞惡變,遠處轉移是其致死的主要原因之一[1-2]。上皮間質轉化(epithelial-mesenchymaltransition,EMT)是乳腺癌細胞獲得轉移能力的關鍵起始步驟[3]。EMT途徑可幫助乳腺癌細胞獲得間充質表型[4],使癌細胞遠離原發病灶并轉移到遠處淋巴結或其他器官[5]。該途徑是乳腺癌細胞獲得侵襲能力及發生遠處轉移的重要生物學過程[6],是乳腺癌細胞啟動轉移最重要的機制[7]。在乳腺癌EMT過程中,基質金屬蛋白酶(matrixmetallo-proteinases,MMPs)發揮了重要作用[8]。MMPs是一類鋅離子依賴性的蛋白水解酶,可降解細胞外基質(extracellularma-matrix,ECM)[9],誘發EMT,為乳腺癌的侵襲轉移創造條件[10]。MMP-9蛋白又稱明膠酶,是MMPs家族的重要成員之一,可降解ECM中的明膠、Ⅳ型和Ⅴ型膠原等主要成分[11],破壞ECM和基底膜結構,促進癌細胞向周圍組織浸潤,幫助腫瘤細胞的侵襲和轉移[12],協同EMT途徑[13]。因此,MMP-9抑制劑為乳腺癌的治療開辟了一條新途徑。傳統抗癌藥物開發周期長,投入資金量大[14],若能利用現有小分子藥物配體進行藥物設計,可提高新藥物開發效率[15]。三維定量構效關系(three-dimensional quantitative structure-relationship,3D-QSAR)研究可利用藥物分子結構和生物學活性[16],通過計算機實現模擬運算和預測,指導新型藥物的設計、改造和優化,相比傳統藥物的研制更有針對性、高效性[17],可為MMP-9抑制劑的研發及治療乳腺癌提供思路。
目前,關于氧肟酸類MMP-9抑制劑3D-QSAR與分子設計相關研究還鮮見報道。本研究將經典的3D-QSAR技術即比較分子力場分析(comparative molecular field analysis,CoMFA)和第二代3D-QSAR技術即Topomer-CoMFA兩種方法聯合運用構建3D-QSAR模型,確保新建模型的穩定性與可靠性。依托Topomer search技術的側鏈相似性搜索[18],參考3D-QSAR模型提供的模型預測和三維等勢圖信息,構建新的氧肟酸類MMP-9抑制劑分子,以證明此分子設計的方法,可為新型高效抑制劑的研發、抑制劑結構的優化與設計研究提供參考。
1 材料與方法
1.1 數據來源與分子結構的優化
本文用于分子設計研究的氧肟酸類MMP-9抑制劑均參考文獻[19],各化合物結構與活性如下所示。使用SYBYLX 1.1軟件中的Sketch模塊和Compute模塊對小分子抑制劑進行結構與數據表單構建,選擇相應的電荷與能量進行優化,將表示分子活性值的半抑制濃度(IC50)轉化成pIC50(pIC50=-lgIC50)(圖1、表1)[20]。

圖1 氧肟酸類MMP-9抑制劑的骨架結構

表1 氧肟酸類MMP-9抑制劑的結構與活性

(續表)
1.2 CoMFA和Topomer-CoMFA的建模
分別采用CoMFA和Topomer-CoMFA技術對氧肟酸類MMP-9抑制劑進行定量構效研究,從構建的分子表單中隨機選取24個抑制劑作為訓練集,8個抑制劑作為測試集。CoMFA模型選取特征性的公共骨架進行疊合,Topomer-CoMFA模型進行R基團片段切割。
1.3 3D-QSAR模型的驗證
用新建CoMFA模型和Topomer-CoMFA模型分別對訓練集化合物和測試集化合物活性進行內部測試和外部測試,計算實驗值與預測值之間的活性差值。根據兩個模型訓練集的實驗活性值、預測活性值和測試集的預測活性值構建活性擬合圖,以驗證新建3D-QSAR模型的穩定性、內部和外部預測能力[21]。
1.4 虛擬篩選
基于Topomer-CoMFA的split模塊進行分子切割,獲得分子側鏈基團(R1基團)和分子骨架(R2基團)。以R1基團作為分子活性特征性基團,結合Topomer search技術對ZINC數據庫(2012)中的Fragment-like類(34 292個)分子進行分子結構相似性搜索。根據Topomer distance值(默認設為185)來判斷并篩選出有特定貢獻的R1基團[22],以活性最高的21號分子為模版進行過濾,挑選貢獻值明顯高于R1基團的側鏈基團。將挑選出的側鏈基團交替取代模版分子對應的R1基團,以構建新的抑制劑分子。
1.5 統計學方法
采用偏最小二乘法進行交叉驗證系數計算q2值,留一法進行擬合驗證系數計算r2值,得到最佳組成成分(n)、標準偏差(SEE)和Fisher檢驗值(F)[23]。
2 結果
2.1 3D-QSAR模型結果
分子疊合和分子切割是建立3D-QSAR模型的關鍵步驟,CoMFA模型的分子疊合圖和Topomer-CoMFA模型的分子切割圖(圖2~3)。兩種技術所建立3D-QSAR模型的各統計學參數匯總分析結果顯示,兩種模型都具良好的穩定性和較高的可信度(q2>0.5,r2>0.9,F>100,SEE<0.1)(表2)。

圖2 CoMFA模型的分子疊合圖

圖3 Topomer-CoMFA模型的切割方式

表2 CoMFA和Topomer-CoMFA建模結果


圖4 模型的線性回歸圖
2.2 三維等勢圖結果
選取CoMFA模型中21號分子作為模版進行立體場與靜電場的三維等勢圖分析。在立體場三維等勢圖中,分子側鏈旁的綠色模塊表明在此處引入大空間占位基團更有利于增加抑制劑化合物活性;黃色模塊表明在此處引入大空間占位基團不利于增加抑制劑化合物活性。在苯基3號位旁有較大的綠色模塊,提示此處若有較大空間占位的基團有利于增加化合物活性。
靜電場三維等勢圖中,紅色模塊表明在此處引入負性基團有利于抑制劑化合物活性的增加;藍色模塊表明在此處引入正性基團有利于抑制劑化合物活性的增加。在苯基4號位旁邊有較大的紅色模塊,提示此處有負性作用強的基團有利于化合物活性的增加(圖5)。

圖5 三維等勢圖
2.3 分子設計結果
依托Topomer search技術的虛擬篩選,并結合新建模型的三維等勢圖和活性預測能力,以活性最高21號分子結構為基礎,進行梯度分子設計。根據分子結構的Topomer distance值和貢獻度值(>1)來評價化合物與訓練集化合物結構的相似度,以篩選出的基團替換模板分子原有的R1基團,初步重構6個新抑制劑化合物。再依據分子結構的三維等勢圖,分析新建分子R1基團的立體場和靜電場對分子活性的影響。用新建CoMFA模型和Topomer-CoMFA模型對新抑制劑進行分子活性預測,保留分子活性>21號分子的化合物結構,最終使用梯度分子設計的方法構建出2個高活性的氧肟酸類MMP-9化合物。新抑制劑的分子結構與預測活性值如下(表3)。
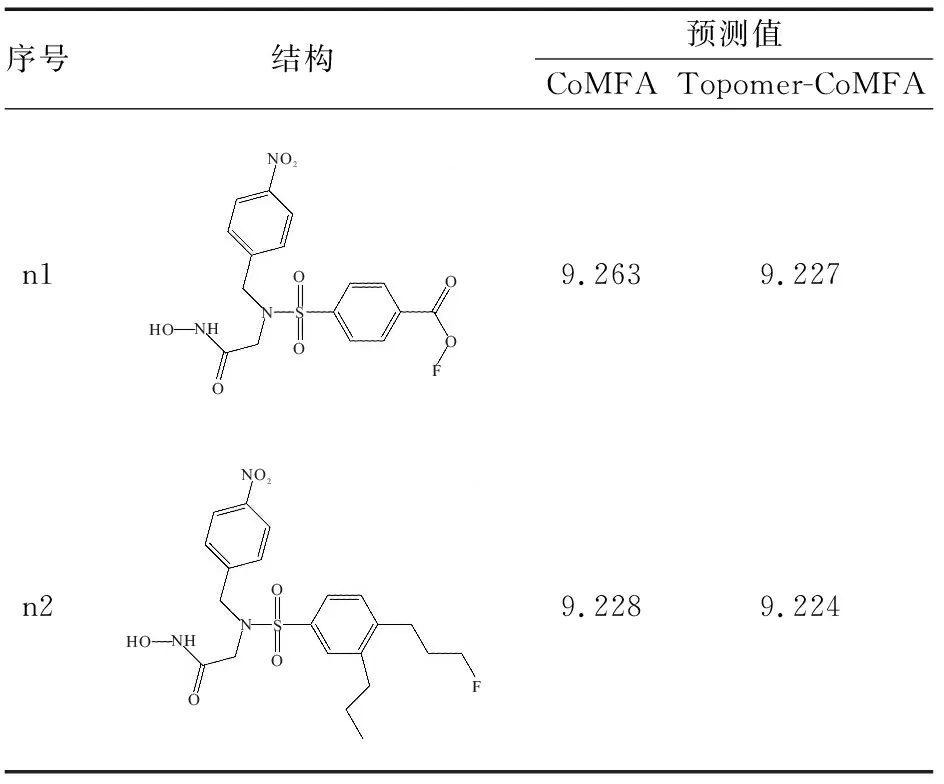
表3 新設計分子的結構及活性預測值
3 討論
乳腺癌發病率逐年增長,其發病率已占女性惡性腫瘤發病率的第一位[24]。乳腺癌不僅對女性造成身心痛苦,而且給患者家庭、社會帶來經濟負擔。乳腺癌遠處轉移是乳腺癌惡化的致命因素,而EMT途徑與MMP-9的協同作用可加速乳腺癌惡化[25]。因此,尋求安全、經濟、有效的抗乳腺癌藥物(如MMP-9類抑制劑)治療方法,已成為醫學界以及乳腺癌患者非常關心的問題。
為獲得高效、新穎的分子設計方法,本研究運用第一代CoMFA技術和第二代Topomer-CoMFA技術對氧肟酸類MMP-9抑制劑進行3D-QSAR研究[26]。結果顯示,新建3D-QSAR模型具有良好的穩定性和可信度。將CoMFA和Topomer-CoMFA模型所測的內部預測活性值、外部預測活性值和化合物實驗活性值進行線性回歸,實驗值與預測值有良好的擬合度,模型外部預測能力比較理想,可用于后續MMP-9抑制劑的分子設計與活性預測。
模型的三位等勢圖可從分子立體場、靜電場等方面對分子設計提供幫助。結合獲得的立體場三位等勢圖分析,15、17、27號分子(pIC50=8.1~8.6)有較高的活性,得益于苯基3號位引入了立體場作用更大的硝基、羧基等基團,在增加立體場同時,也有利于抑制劑化合物活性的增加。從靜電場三位等勢圖研究發現,21號分子有5個氟基團,4號、5號分子也有多個氟基團,這些氟基團提供了強大的負性作用,同時也提高了分子活性。17、26、28、29號分子(pIC50=8.16~8.6)有較高的活性,得益于苯基3號位引入了負性作用強的硝基、甲氧基等基團,增加了負性靜電場及抑制劑化合物的活性。
本研究進一步發現,分子結構多樣性、活性上的差異主要來自于R1側鏈基團的變化。采用Topomer search技術,從小分子數據庫進行基于側鏈基團的虛擬篩選,參考3D-QSAR模型給出的靜電場與立體場三維等勢圖信息,結合CoMFA模型、Topomer-CoMFA模型的外部預測能力,設計了2個高活性的氧肟酸類MMP-9抑制劑分子。該結果發現,新抑制劑分子n1保留了模板21號分子R1基團的苯環結構,在苯環4號位的氟酸基團不僅增加了n1分子的負性作用,也增加了R1基團的立體場作用,這使得抑制劑活性增加。n2分子的苯環3號、4號位均加入正丙基基團,增加了分子的整體空間結構,提高了分子活性,但是活性稍低于n1分子。三維等勢圖信息顯示,靜電場對分子活性影響大于立體場,這與3D-QSAR模型給出的提示一致。結合CoMFA和Topomer-CoMFA模型建模結果可知,靜電場的貢獻度分別為63.6%、67.3%,立體場貢獻度分別為36.4%、32.7%,靜電場對活性的貢獻度占主導地位。對小分子進行分子設計和結構修飾改造時,研究者應充分考慮靜電場與立體場的相互影響,分析基團電荷性、側鏈空間結構與能量匹配等因素。
綜上所述,本研究基于氧肟酸類MMP-9抑制劑進行的CoMFA和Topomer-CoMFA定量構效關系分析和Topomer search虛擬篩選的方法可用于MMP-9抑制劑的分子設計研究,為小分子藥物活性的提高、優質化合物結構的改造與設計提供新的思路,為新型抗乳腺癌藥物篩選提供參考。

