基于生物信息基因表達譜的乳腺癌轉移基因標志物的分析研究
吳奇橋 張樹民 鄭婷婷 劉 娟 胡 永 林登強 戴夢婷 孫 菁▲
1.復旦大學附屬中山醫院廈門醫院放療科,福建廈門 361006;2.復旦大學附屬中山醫院放療科,上海 200030;3.復旦大學附屬中山醫院廈門醫院放射科,福建廈門 361006;4.復旦大學附屬中山醫院廈門醫院泌尿外科,福建廈門 361006
乳腺癌是威脅女性健康的最常見惡性腫瘤,是美國第二大最常見的癌癥相關死亡[1]。乳腺原位癌通常具有良好的預后,然而,乳腺癌若出現遠處轉移往往會導致危及生命的結果[2]。總體而言,乳腺癌的5年平均生存率為90%,但如果存在遠處轉移,則降至26%[3]。
乳腺癌進展的分子機制尚未完全了解。鑒于其高死亡率,迫切需要弄清乳腺癌轉移的潛在分子機制。既往的研究已經調查乳腺癌轉移的相關基因,如Chen 等[4]揭示了ECM-受體相互作用可能有助于乳癌骨轉移;Cai 等[5]表明CDCA8、CCNA2 與乳癌遠處轉移有關;Zheng 等[6]鑒定了幾種與乳腺癌轉移相關的基因。然而上述研究僅對單個數據集進行分析,目前仍然沒有研究結合相似數據集進行基因分析。本研究分析了三個數據集中與乳腺癌轉移相關的差異表達基因(differentially expressed genes,DEGs),目的是更好地了解潛在乳腺癌轉移的機制,并找到潛在生物標志物和治療靶標。
1 資料與方法
1.1 一般資料
GSE32489、GSE14776 和GSE103357[7]使 用 基 因表達綜合數據庫(Gene Expression Omnibus,GEO,http://www.ncbi.nlm.nih.gov/geo)得到的三個基因數據集,均使用Illumina HumanRef 平臺芯片,根據平臺中的注釋信息將探針轉換為相應的基因符號。GSE32489包含非轉移尸檢組織19 個,淋巴結轉移組織90 個。GSE14776 包含8 個非轉移細胞樣本和6 個骨轉移樣本。GSE103357 包含2 個非轉移細胞樣本和3 個骨轉移樣本。
1.2 DEGs 的鑒定
Network Analyst(版本號:10.0)[8-9](http://www.networkanalyst.ca)用于提取數據集乳腺腫瘤樣本和轉移樣本之間的DEGs。具有調整P 值<0.05 和|log2FC|>1.0的基因被認為是DEGs。通過維恩(Venn)工具(http://bioinformatics.psb.ugent.be/webtools/Venn/)檢測重疊以得到共差異表達的基因(co different-expressed genes,Co-DEGs)。調整P 值、Benjamini 和Hochberg 假陽性發現率,用于在統計顯著基因的發現和假陽性之間提供平衡。沒有相應基因符號的探針組或具有多于一個探針組的基因被去除。識錯率(false discovery rate,FDR)的計算公式如下:q-value(i)=p(i)length(p)/rank(p),表明當樣本量增大時,檢出假陽性率的概率增高,FDR 值越高,表明大樣本數據富集后出現的假陽性概率越高。
1.3 基因本體和京都基因與基因組百科全書途徑富集通路分析
基因本體(gene ontology,GO)分析用于對基因集進行功能研究[10]。京都基因與基因組百科全書途徑富集(Kyoto encyclopedia of genes and genomes,KEGG)[11]是處理基因組和生物通路的數據庫集合。注釋可視化和 集 成 發 現 數 據 庫(DAVID,http://david.ncifcrf.gov)(6.8 版)[12]用于解開已識別共基因的GO 和KEGG 途徑。
1.4 DEGs 和模塊選擇的蛋白質-蛋白質交互網絡分析
STRING(版本號:11.5)用于闡明蛋白質-蛋白質交互(protein-protein interaction,PPI)相互作用[13]。使用Cytoscape(版本號:3.8.2)[14]進行可視化PPI 網絡。選擇>0.4 的組合分數作為閾值。PPI 網絡可以協助從蛋白層面識別參與乳腺癌轉移的重要基因模塊。此外,應用分子模塊檢測(molecular complex detection,MCODE)[15]插件對重要模塊進行驗證。
1.5 核心基因的熱圖繪制、GO 和臨床分析
使用cytoHubba(版本號:3.8.2)插件和最大集團中心性(maximum group centrality,MCC)方法來識別前20 個中樞基因。核心基因的熱圖是通過使用R 中的熱圖包繪制的,使用的聚類方法為離差平和法(Ward),標準化方法為正態標準化(Z-score scaling)。使用Kaplan Meier-plotter(KM plotter,http://kmplot.com/analysis/)實現生存分析,Kaplan-Meier plotter 是一種生存分析軟件[16]。通過Cytoscape 的BiNGO 插件(版本號:3.8.2)評估核心基因的GO 功能。
1.6 DEG 的生存分析
乳腺癌患者根據特定基因的表達分為兩組(高表達與低表達)。通過Kaplan-Meier 生存圖比較兩個組患者的隊列,并計算具有95%置信區間和對數等級P 值的風險比(HR),其中,HR=1 意味著基因低表達與高表達的等效性,若低表達處理優于高表達,則HR<1;若低表達處理劣于高表達,則HR>1。GEPIA[17]是基于TCGA 數據庫的在線基因表達譜分析工具,用于驗證樞紐基因與臨床分期之間的相關性,其中,F value 代表單因素分析的F 值,這個值越大,表示組間差異越大,且當P<0.05 時,表示該基因在腫瘤不同分期中差異是有統計學意義的。
2 結果
2.1 三個基因數據集中DEG 和co-DEG 的鑒定
三個基因數據集中,韋恩圖見圖1A 及圖1B,基因表達熱圖見圖1C。結果顯示,295 個基因被鑒定為co-DEGs。其中上調151 個,下調144 個。調整P<0.05和|log2FC|>1 被設置為截止標準。
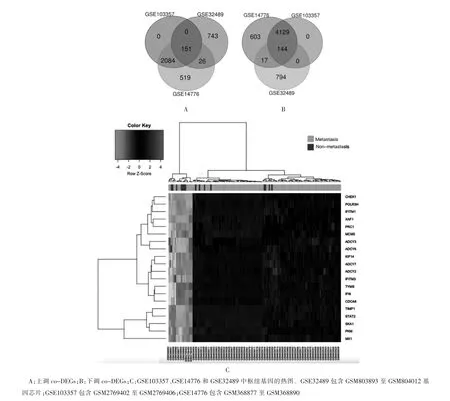
圖1 維恩圖及基因熱圖
2.2 DEGs 的GO 富集分析及KEGG 通路分析
DAVID 在線工具用于闡明富集的co-DEGs 中的GO 和KEGG 通路。結果表明,對于BP,上調的基因主要富集在Ⅰ型干擾素信號通路、凋亡過程中,而下調的基因主要集中在DNA 修復、先天免疫反應等方面。KEGG 通路結果顯示,DEGs 顯著富集在與癌癥發展的信號通路,包括絲裂原活化蛋白激酶(mitogen-activated protein kinase,MAPK) 信號通路和Rap1 信號通路(表1~2)。

表1 Co-DEGs 中上調基因的GO 分析
2.3 DEGs 和模塊選擇的PPI 網絡分析
基于STRING 數據庫得到PPI 網絡分析結果,見圖2A,結果顯示,DEG 的PPI 網絡由280 個節點和357 條邊構成。使用MCC 方法,總共選擇了20 個基因作為樞紐基因,分別為:CHEK1、POLR3H、IFITM1、XAF1、MCM5、ADCY3、ADCY6、KIF14、ADCY7、ADCY2、IFITM3、TYMS、IFI6、CDCA8、TIMP1、STAT2、SKA1、PKM、MX1、PRC1(ASE1)。
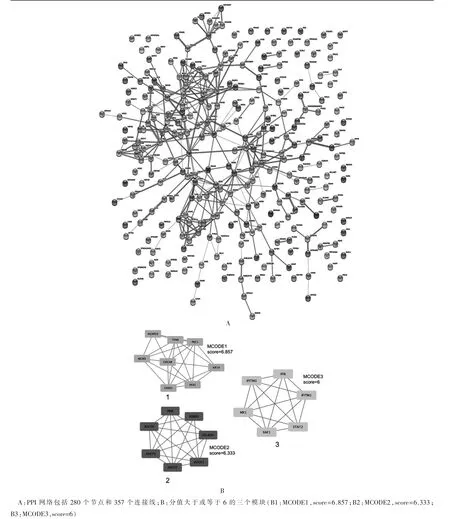
圖2 PPI 網絡分析結果和樞紐基因的鑒定結果
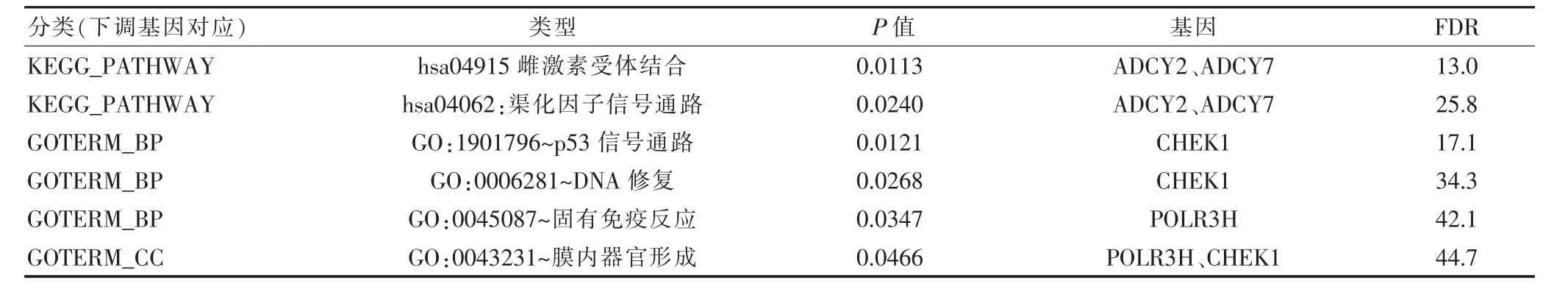
表2 Co-DEGs 中下調基因的GO 分析
樞紐基因的鑒定結果見圖2B,結果顯示,使用MCODE 從DEG 的PPI 網絡中獲得了3 個分值大于或等于6 的重要模塊。
2.4 中樞基因的生存分析
根據每個基因的表達,繪制乳癌患者的無遠處轉移生存(distance metastasis free survival,DMFS)和總生存(overall survival,OS)曲線,結果分別見圖3A 和圖3B。結果顯示,TYMS 的低表達[HR=0.52(0.29~0.91),P=0.021]、SKA1 的低表達[HR=0.57(0.32~1.01),P=0.049]、ADCY7 的低表達[HR=0.45(0.30~0.68),P<0.001]、MX1 的高表達[HR=2.07(1.17~3.66),P=0.011]與較差的OS 相關(圖3A)。POLR3H 的低表達[HR=0.71(0.51~0.98),P=0.039]、CDCA8 的高表達[HR=1.67(1.37~2.04),P<0.001]、ASE1 的高表達[HR=2.1(1.72~2.57),P<0.001]、KIF14 的高表達[HR=1.86(1.33~2.59),P<0.001]、MX1 的高表達[HR=1.31(1.08~1.6),P=0.006]與較差的DMFS 相關(圖3B)。
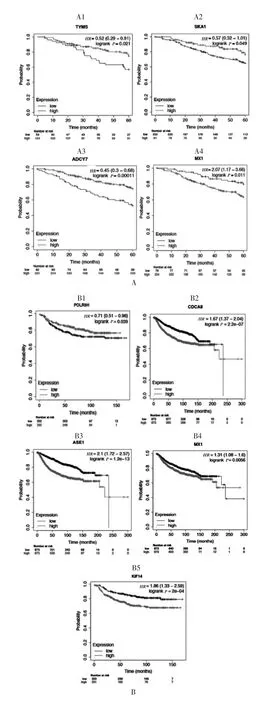
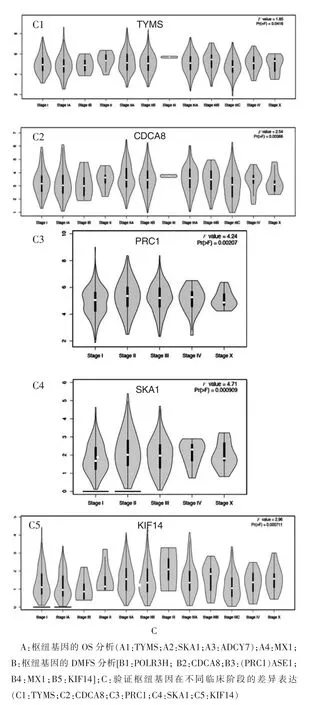
圖3 樞紐基因的臨床分析
選擇數據集TCGA-BRCA 來驗證20 個核心基因與乳腺癌臨床分期之間的相關性。筆者比較了不同臨床分期乳腺癌樣本中核心基因的表達,結果見圖3C,結果顯示,在乳腺癌較晚分期中,TYMS 呈現低表達(P=0.0416),CDCA8 呈現高表達(P=0.003 66),PRC1(ASE1)呈現高表達(P=0.002 07),SKA1(P=0.000 909)呈現低表達,KIF14(P=0.000 711)呈現高表達。
3 討論
本研究中分析了三個包含乳腺癌轉移患者的基因數據集,進行了功能富集分析,表明了樞紐基因通過某些途徑在轉移的進展中發揮了作用。
結果表明,上調基因主要參與Ⅰ型干擾素信號通路、凋亡過程、粘著斑、蛋白質同二聚化活性Rap1 信號通路[18]和MAPK 信號通路[19-21],抗原加工和呈遞,細胞粘附分子,而下調基因主要富集在p53 類介質、雌激素信號通路和趨化因子信號通路。這些發現與已報道的研究[19-29]高度一致,表明細胞凋亡、細胞遷移和粘附在乳腺癌進展過程中的重要作用。
先前的研究[19-29]已經為本研究所篩選的一些樞紐基因在乳腺癌癥進展中的功能提供了大量證據。例如,TYMS 因其作為胸苷酸合酶的功能而被認為是5-氟尿嘧啶的靶標[23-24]。它與晚期乳腺癌[25]患者對化療的耐藥性和敏感性有關。CDCA8 是是有絲分裂的調節因子,SKA1 與有絲分裂有關,均被鑒定為乳癌遠處轉移的樞紐基因[5,26]。KIF14 通過負調節Rap1a-Radil 信號通路促進乳腺癌進展。參與細胞抗病毒的MX1與乳腺癌對淋巴結的侵襲有關[28-29]。盡管有報道稱,PRC1(ASE1)促進了肺腺癌的發生和肝癌的早期復發[30-31],也和鼻咽癌的轉移[32]和乳癌患者較差的無遠處轉移生存期相關[33],POLR3H 與促腫瘤作用相關[34-35]。它們在乳腺癌腫瘤轉移中的確切功能仍然知之甚少,值得進一步深入研究。
綜上所述,本研究確定了乳腺癌轉移過程中的優勢基因及其PPI 網絡。其中一些基因從未被報道過影響乳腺癌的進展,因此可能作為潛在的藥物靶點或生物標志物。然而,本研究仍具有局限性,即需要進一步的實驗驗證。需要檢測更多基因來豐富網絡,以便更全面地了解乳腺癌的轉移通路。

