基于 COI 基因的入侵物種舊金山鹵蟲遺傳多樣性與遺傳分化研究
付春正 王暉 Alireza Asem 郭慧芳 錢彬彬 劉文茹 沈春陽
摘要:伴隨著中國水產養殖業的快速發展,原產美國的舊金山鹵蟲(Artemiafranciscana)被引入至我國渤海灣地區。為研究舊金山鹵蟲被引入中國后其基因組遺傳多樣性改變情況,本研究利用COI線粒體分子標記,評估舊金山鹵蟲在非原生棲息地,即唐山曹妃甸南堡鎮(渤海灣,河北省)定殖的舊金山鹵蟲的遺傳多樣性現狀。研究分析發現:相比于美國原產地區的舊金山鹵蟲種群(大鹽湖種群和舊金山灣種群),舊金山鹵蟲南堡種群表現出較為豐富的遺傳多樣性。而且,舊金山鹵蟲南堡種群與美國原產地區種群(大鹽湖種群和舊金山灣種群)存在顯著的遺傳分化,表明舊金山鹵蟲南堡種群并非是從美國大鹽湖蟲種群和舊金山灣種群直接引入。
關鍵詞:舊金山鹵蟲;COI基因;遺傳多樣性;群體遺傳結構
外來物種入侵是指生物物種由原產地通過人為或非人為的方式遷移到新的生態環境的過程[1]。外來物種的入侵不僅會影響當地自然環境的生物多樣性,還會影響當地的生物群落結構[2]。雖然只有約1%的外來物種會在新的生態環境中成功定殖,但這些成功定殖的外來物種將會給當地的生態環境帶來深遠的影響和巨大的經濟損失[3]。外來物種適應新環境的能力主要取決于該物種的遺傳多樣性,在入侵新的生態環境過程中,外來物種很可能由于入侵種群個體數量較少引發遺傳漂變,進而造成外來物種種群遺傳多樣性降低,甚至可能會造成外來物種的入侵失敗[4-5]。因此,外來物種的遺傳多樣性是其能否在新的環境中成功入侵和擴張的一個重要因素[4]。
鹵蟲(BrineShrimp)又名豐年蟲,是一種小型甲殼動物,隸屬于節肢動物門(Arthropoda),甲殼綱(Crustacea),鰓足亞綱(Branchiopoda),無甲目(Anostraca),鹵蟲科(Artemiidae),鹵蟲屬(Artemia)[6]。鹵蟲具有獨特的滲透調節機制,可以在高鹽(7.0~340g/L)和復雜離子成分等極端環境條件下生存,因此主要棲息于高鹽生境中,如內陸鹽湖、沿海鹽場、鹽池和瀉湖等,在世界各大陸的鹽湖、鹽田、海洋等高鹽水域均具有廣泛的分布[7]。鹵蟲廣泛應用于水產養殖業,是幼魚、幼蝦的理想開口飼料[6,8]。二十一世紀初,伴隨著水產養殖業的迅速發展,我國年需鹵蟲卵超過2000t(粗品),而當時我國年產鹵蟲卵僅為1200~1500t(粗品)[9]。自1950年起,美國主要鹵蟲產區(大鹽湖和舊金山灣)的鹵蟲,開始被出口到世界各地的漁業孵化場用于幼魚、幼蝦的養殖。舊金山鹵蟲(Artemiafranciscana)具有較高的繁殖率和較強的適應性,被許多國家及地區引入以滿足水產養殖業的需求,除美洲大陸外,亞歐大陸的諸多區域也有舊金山鹵蟲的分布[10-12]。
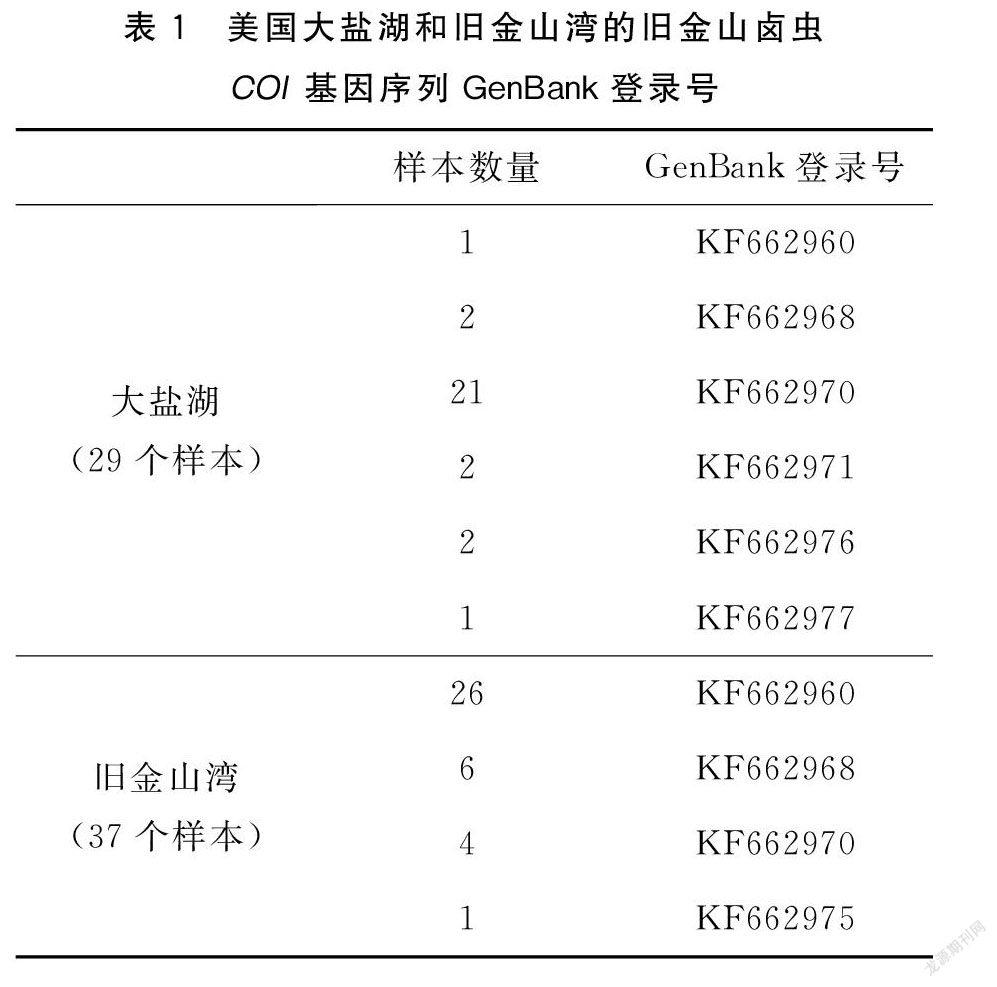
為滿足我國水產養殖業的發展需求,舊金山鹵蟲于1991年首次被引種至我國渤海灣地區,包括:輕工業部制鹽研究所實驗場、塘沽鹽場、漢沽鹽場和南堡鹽場,使當地鹵蟲產量提高了30%~100%[13]。歷時20余年,舊金山鹵蟲已遍及我國東部多個沿海地區,其不僅存在于渤海灣地區,在黃海地區及東海地區也有發現舊金山鹵蟲分布[14]。前人的研究多只利用形態學特征、同工酶遺傳標記、微衛星分子標記等研究不同鹵蟲品種、品系的群體多樣性及遺傳分化,目前尚未有研究報道利用線粒體細胞色素C氧化酶Ⅰ(COI:CytochromeCoxidasesubunitⅠ)基因研究入侵至中國渤海灣地區的舊金山鹵蟲種群的遺傳多樣性及遺傳分化。本研究通過對河北省唐山市曹妃甸區南堡鎮銀海食鹽有限公司鹽田的舊金山鹵蟲種群的線粒體COI基因序列進行測序,分析該種群的遺傳多樣性及遺傳結構,并與美國的舊金山鹵蟲大鹽湖種群(TheGreatSaltLake)和舊金山灣種群(SanFranciscoBay)的COI基因序列數據進行比較分析,從而了解入侵至中國渤海灣地區的舊金山鹵蟲在新環境下的種群遺傳分化及適應性變化,為進一步將舊金山鹵蟲引入至我國其它地區提供理論依據。
1材料與方法
1.1樣本來源與基因組DNA提取
本研究使用的舊金山鹵蟲樣本,計20個,采集于河北省唐山市曹妃甸區南堡鎮銀海食鹽有限公司(39°06′N,118°36′E)(以下簡稱舊金山鹵蟲南堡種群)(圖1)。按照Chelex-100(Bio-Rad,USA)基因組DNA提取方法提取鹵蟲基因組DNA[15-16]。
1.2COI基因序列擴增及測序
擴增線粒體COI基因引物為1/2COI_Fol、F:5-ATTCTACGAATCACAAGGATATTGG–3、R:5-TACACTTCAGGATGGCCAAAAAATCA–3,引物由北京擎科生物科技有限公司合成[17]。PCR的反應體系為20μL,包括2×EasyTaqPCRSuperMix10μL,ddH2O8μL,正反向引物各0.5μL,DNA模板1μL。PCR反應程序為:95℃預變性3min;94℃變性45s,55℃退火60s,72℃延伸60s,35個循環;72℃終延伸5min,4℃保存。PCR產物經1%的瓊脂糖凝膠電泳檢測后,送至北京擎科生物科技有限公司進行測序。
1.3數據分析
測序獲得的COI基因序列利用MEGAv7026采用默認參數進行序列比對,計算序列堿基組成、變異位點、簡約信息位點、自裔位點等參數[18]。利用核苷酸BLAST(https://blast.ncbi.nlm.nih.gov/Blast.cgi)驗證測序樣本為舊金山鹵蟲。利用Networkprogramv5.0.0.3中的median-joining算法進行單倍型網絡分析,檢測樣本的遺傳分化,從而確定舊金山鹵蟲南堡種群與美國大鹽湖種群及舊金山灣種群的遺傳分化關系[19]。舊金山鹵蟲大鹽湖種群及舊金山灣種群的線粒體COI基因序列下載自NCBI核苷酸數據庫(https://www.ncbi.nlm.nih.gov/nuccore,表1)[20]。利用軟件Arlequinv3.5計算每個群體的多態位點數(S)、單倍型數(h)、單倍型多樣性(Hd)、單倍型比(Hr)、核苷酸(基因)多樣性(Pi)、平均核苷酸差異數(k)、期望雜合度、群體遺傳分化指數(FST)和分子方差分析AMOVA[21]。
2結果與分析
2.1COI基因序列特征
本研究共測序獲得20條COI基因序列,利用核苷酸BLAST比對,確定本研究采集的20個鹵蟲樣本均為舊金山鹵蟲。舊金山鹵蟲南堡種群樣本的COI基因序列具有9個變異位點,包括簡約信息位點7個、自裔位點2個。而舊金山鹵蟲大鹽湖種群樣本的COI基因序列具有11個變異位點,包括簡約信息位點7個、自裔位點4個;舊金山鹵蟲舊金山灣種群樣本的COI基因序列含有7個變異位點,包括簡約信息位點6個、自裔位點1個。
2.2種群遺傳多樣性
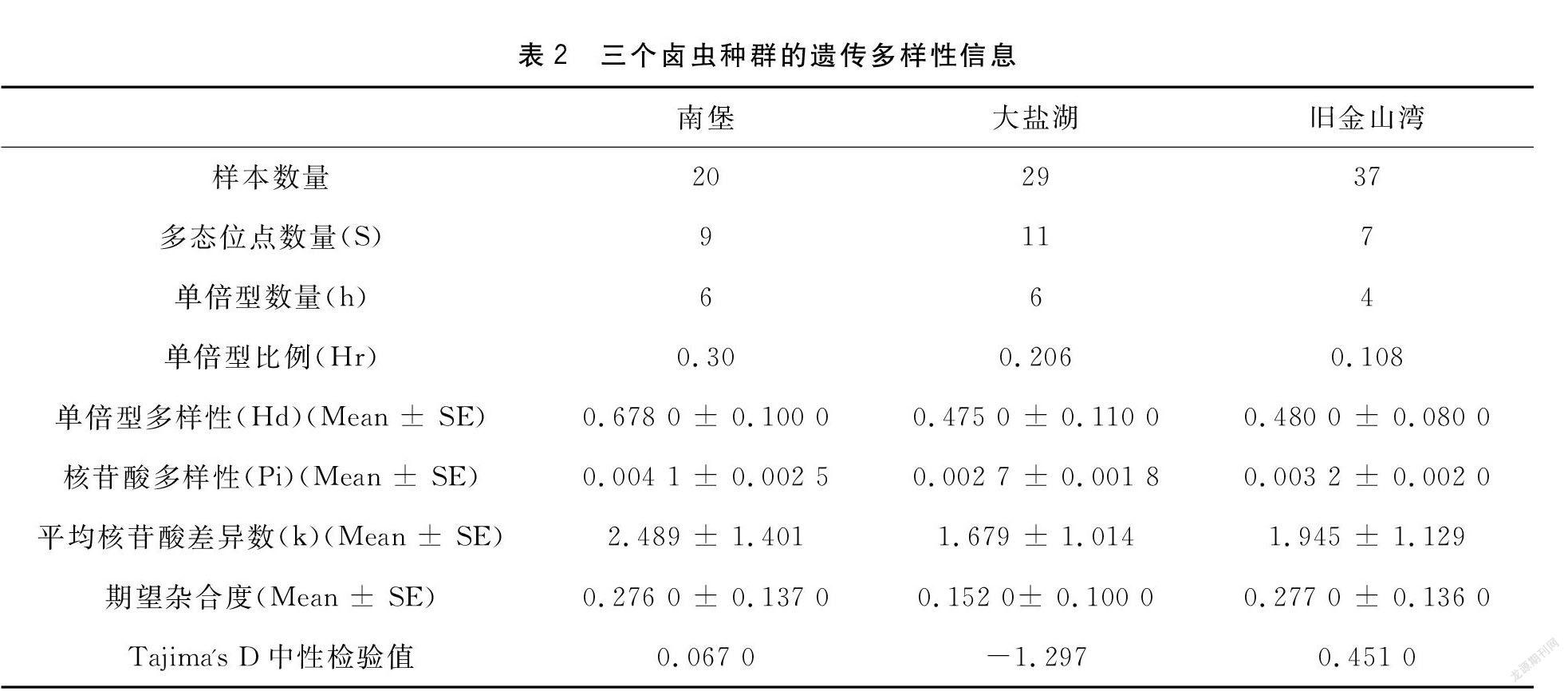
舊金山鹵蟲南堡種群、大鹽湖種群及舊金山灣種群的遺傳多樣性信息見表2。相比于舊金山鹵蟲大鹽湖種群和舊金山灣種群,舊金山鹵蟲南堡種群具有較高的單倍型多樣性和核苷酸多樣性,這表明,相比于美國本土的舊金山鹵蟲種群,入侵并定殖于中國唐山市南堡地區的舊金山鹵蟲種群具有更豐富的遺傳多樣性。
2.3種群遺傳結構
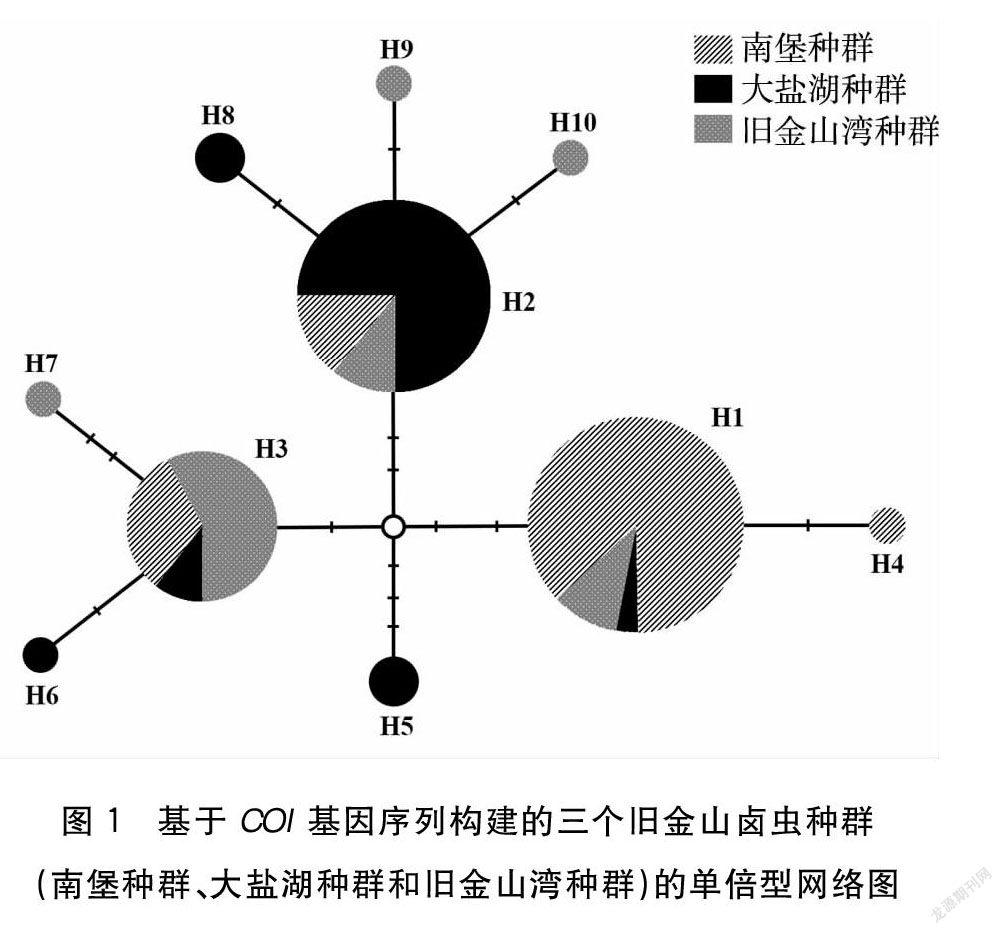
基于舊金山鹵蟲南堡種群、大鹽湖種群及舊金山灣種群共計86個樣本的COI基因序列共檢測到10種單倍型,其中H1、H2和H3為主要單倍型,分別占總數的34.88%(30個樣本)、3256%(28個樣本)和22.10%(19個樣本),見圖1、表3。舊金山鹵蟲南堡種群樣本分布在6個單倍型中,主要單倍型為H3型(55.00%),見圖1、表4。H3型分別由57.89%的南堡種群樣本、10.52%的大鹽湖種群樣本和31.59%的舊金山灣種群樣本組成(見圖1、表5)。舊金山鹵蟲大鹽湖種群樣本分布在6個單倍型中,主要單倍型為H2型(72.40%)。H2型分別由75.00%的大鹽湖種群樣本、10.72%的南堡種群樣本和1428%的舊金山灣種群樣本組成(見圖1、表4、表5)。舊金山灣種群分布在4個單倍型中,主要單倍型為H1型(70.27%)。H1型分別由8666%的舊金山灣種群樣本、10.00%南堡種群樣本和3.34%大鹽湖種群樣本組成(見圖1、表4、表5)。
舊金山鹵蟲南堡種群、大鹽湖種群及舊金山灣種群三個種群兩兩之間均存在顯著的遺傳分化(P=0.0001),南堡種群和大鹽湖種群的遺傳分化指數值(FST)最低,為0.29;大鹽湖種群和舊金山灣種群的遺傳分化指數值(FST)最高,為0.56(見表6)。三個舊金山鹵蟲種群的分子方差分析(AMOVA)表明,舊金山鹵蟲的遺傳變異主要來源于種群內部個體之間,占總變異的55.01%,種群之間的遺傳變異占44.99%(見表7)。
3討論與結論
3.1舊金山鹵蟲南堡種群的遺傳多樣性
外來物種在被引入至新環境后,通常由于奠基者效應或者種群定殖過程中的群體遺傳瓶頸效應等因素,會導致引入種群的遺傳多樣性較低[15,22]。如越南永州地區引入的舊金山鹵蟲種群,相比于種源地舊金山灣的舊金山鹵蟲種群,其表現出更低的種內遺傳變異和更少的單倍型多樣性[23]。然而相比于美國大鹽湖地區的舊金山鹵蟲種群,亞洲地區引入的舊金山鹵蟲種群表現出相對較高的遺傳多樣性[24]。地中海鹵蟲的引入種群中也發現了相似的結果[25-26]。本研究也發現,相比舊金山鹵蟲種源地種群(大鹽湖種群和舊金山灣種群),引入并定殖于中國渤海灣地區的南堡種群表現出較為豐富的遺傳多樣性。在新環境中引入并定殖的種群可能是由于自身具有較強的適應能力和生理可塑性,在不同選擇壓力的作用下,引入種群的遺傳分化模式受到一定影響,進而表現出了相對較高的遺傳多樣性[3,4,24,26-27]。
3.2舊金山鹵蟲南堡種群的引種來源
線粒體基因組DNA具有進化速率快、母系遺傳和缺乏重組的特征,因此線粒體基因組分子標記對于分析在新環境中定殖的外來物種的來源和系統分化具有重要的作用[28-29]。利用線粒體COI基因作為分子標記進行的系統地理分析表明:地中海區域的入侵舊金山鹵蟲種群來源于美國的大鹽湖種群和舊金山灣種群,而亞洲引入的舊金山鹵蟲種群的定殖過程經歷了多次種源引入,即首先由美國的種源地群體引入,而后又經歷了歐洲群體的二次引入[24,26,28]。本研究中,舊金山鹵蟲南堡種群、大鹽湖種群及舊金山灣種群共檢測到10種單倍型,其中H1、H2和H3為主要單倍型,分別以舊金山灣種群(86.66%)、大鹽湖種群(75%)和南堡種群(57.89%)為主。這一結果表明舊金山鹵蟲南堡種群并非來源于大鹽湖種群和舊金山灣種群。舊金山鹵蟲南堡種群與大鹽湖種群及舊金山灣種群具有顯著的群體遺傳分化指數FST值也表明:舊金山鹵蟲南堡種群并非來源于大鹽湖種群和舊金山灣種群。舊金山鹵蟲南堡種群可能從其他舊金山鹵蟲入侵地區間接引入的,如東亞的湄公河三角洲(越南)和渤海灣(中國)地區,這些地區舊金山鹵蟲已經完全商品化并可在水產養殖市場上購買獲得[15,26,30]。
本研究通過利用河北省唐山市曹妃甸區南堡鎮銀海食鹽有限公司鹽田的舊金山鹵蟲種群的線粒體COI基因進行測序,分析該種群的遺傳多樣性及遺傳結構,并與美國的大鹽湖種群和舊金山灣種群的線粒體COI基因測序數據進行比較分析,發現相比于美國本土的舊金山鹵蟲種群,入侵并定殖于中國唐山市南堡地區的舊金山鹵蟲種群具有更豐富的遺傳多樣性。而且舊金山鹵蟲南堡種群與美國大鹽湖種群和舊金山灣種群存在顯著的遺傳分化,表明舊金山鹵蟲南堡種群并非是從美國大鹽湖種群和舊金山灣種群直接引入的。與陸地、海洋及淡水環境相比,高鹽生境系統的物種種類和數量通常有限,這使得高鹽生境內的生物多樣性更加脆弱,而外來物種的引入將對高鹽生境內的生物多樣性帶來更大的威脅。舊金山鹵蟲作為渤海灣區域高鹽生境的外來物種,對于渤海灣的高鹽環境有較好的適應,表現出較為豐富的遺傳多樣性,因此后續研究可將舊金山鹵蟲作為高鹽生境中入侵生物的模式生物,研究外來物種對高鹽生境的生物效應,尤其是研究舊金山鹵蟲的引入對于高鹽生境內其它生物遺傳變異模式的影響。
參考文獻:
[1]付保榮,崔曉男,褚闊,等.遼寧省外來物種入侵防控對策與建議[J].環境保護與循環經濟,2021,41(1):44-49.
[2]OLDENJD,POFFNLR,DOUGLASMR,etal.Ecologicalandevolutionaryconsequencesofbiotichomogenization[J].TrendsinEcology&Evolution,2004,19(1):18-24.
[3]PIMENTELD,ZUNIGAR,MORRISOND.Updateontheenvironmentalandeconomiccostsassociatedwithalien-invasivespeciesintheUnitedStates[J].EcologicalEconomics,2005,52(3):273-288.
[4]DLUGOSCHKM,PARKERIM.Foundingeventsinspeciesinvasions:geneticvariation,adaptiveevolution,andtheroleofmultipleintroductions[J].MolecularEcology,2008,17(1):431-449.
[5]LAVERGNES,MOLOFSKYJ.Increasedgeneticvariationandevolutionarypotentialdrivethesuccessofaninvasivegrass[J].ProceedingsoftheNationalAcademyofSciences,2007,104(10):3883-3888.
[6]畢久勤.開發鹽田新資源—鹽田鹵蟲[J].海湖鹽與化工,1992,21(2):22-24.
[7]VANSTAPPENG.Zoogeography[M].Dordrecht:Springer,2002:171-224.
[8]SORGELOOSP,DHERTP,CANDREVAP.Useofthebrineshrimp,Artemiaspp.,inmarinefishlarviculture[J].Aquaculture,2001,200(1-2):147-159.
[9]沈海燕.鹵蟲資源的開發利用[J].特種經濟動植物,2002,5(8):14.
[10]AMATF,HONTORIAF,NAVARROJC,etal.BiodiversitylossinthegenusArtemiainthewesternMediterraneanregion[J].Limnetica,2007,26(2):387-404.
[11]AMATF,HONTORIAF,RUIZO,etal.TheAmericanbrineshrimpasanexoticinvasivespeciesinthewesternMediterranean[J].BiologicalInvasions,2005,7(1):37-47.
[12]HORVATHZ,LEJEUSNEC,AMATF,etal.EasternspreadoftheinvasiveArtemiafranciscanaintheMediterraneanBasin,withthefirstrecordfromtheBalkanPeninsula[J].Hydrobiologia,2018,822(1):229-235.
[13]張波,郭金昌,商占恒,等.舊金山灣鹵蟲(Artemiafranciscana)在中國渤海灣地區的引種[J].海鹽湖與化工,1993,22(3):7-9.
[14]ZHENGB,SUNS-C.ReviewofthebiogeographyofArtemiaLeach,1819(Crustacea:Anostraca)inChina[J].InternationalJournalofArtemiaBiology,2013,3(1):20-50.
[15]ASEMA,EIMANIFARA,LIWD,etal.Phylogeographyandpopulationgeneticstructureofanexoticinvasivebrineshrimp,ArtemiaLeach,1819(Crustacea:Anostraca),inAustralia[J].AustralianJournalofZoology,2018,66(5-6):307-316.
[16]ASEMA,EIMANIFARA,SUNSC.GeneticvariationandevolutionaryoriginsofparthenogeneticArtemia(Crustacea:Anostraca)withdifferentploidies[J].ZoologicaScripta,2016,45(4):421-436.
[17]MUOZJ,G MEZA,GREENAJ,etal.PhylogeographyandlocalendemismofthenativeMediterraneanbrineshrimpArtemiasalina(Branchiopoda:Anostraca)[J].MolecularEcology,2008,17(13):3160-3177.
[18]KUMARS,STECHERG,TAMURAK.MEGA7:Molecularevolutionarygeneticsanalysisversion7.0forbiggerdatasets[J].MolecularBiologyandEvolution,2016,33(7):1870-1874.
[19]BANDELTHJ,FORSTERP,ROHLA.Median-joiningnetworksforinferringintraspecificphylogenies[J].MolecularBiologyandEvolution,1999,16(1):37-48.
[20]MUOZJ,AMATF,GREENAJ,etal.BirdmigratoryflywaysinfluencethephylogeographyoftheinvasivebrineshrimpArtemiafranciscanainitsnativeAmericanrange[J].PeerJ,2013,1(2):e200.
[21]EXCOFFIERL,LISCHERHEL.Arlequinsuitever3.5:anewseriesofprogramstoperformpopulationgeneticsanalysesunderLinuxandWindows[J].MolecularEcologyResources,2010,10(3):564-567.
[22]GOLANID,AZZURROE,CORSINI-FOKAM,etal.Geneticbottlenecksandsuccessfulbiologicalinvasions:thecaseofarecentLessepsianmigrant[J].BiologyLetters,2007,3(5):541-545.
[23]KAPPASI,ABATZOPOULOSTJ,VANHOAN,etal.GeneticandreproductivedifferentiationofArtemiafranciscanainanewenvironment[J].MarineBiology,2004,146(1):103-117.
[24]EIMANIFARA,VANSTAPPENG,MARDENB,etal.ArtemiabiodiversityinAsiawiththefocusonthephylogeographyoftheintroducedAmericanspeciesArtemiafranciscanaKellogg,1906[J].MolecularPhylogeneticsandEvolution,2014,79(1):392-403.
[25]HONTORIAF,RED NS,MACCARIM,etal.ArevisionofArtemiabiodiversityinMacaronesia[J].AquaticBiosystems,2012,8(1):25.
[26]MUOZJ,G MEZA,FIGUEROLAJ,etal.ColonizationanddispersalpatternsoftheinvasiveAmericanbrineshrimpArtemiafranciscana(Branchiopoda:Anostraca)intheMediterraneanregion[J].Hydrobiologia,2014,726(1):25-41.
[27]VIKASPA,SAJESHKUMARNK,THOMASPC,etal.AquaculturerelatedinvasionoftheexoticArtemiafranciscanaanddisplacementoftheautochthonousArtemiapopulationsfromthehypersalinehabitatsofIndia[J].Hydrobiologia,2012,684(1):129-142.
[28] ASHTONGV,STEVENSM,HARTMC,etal.MitochondrialDNArevealsmultipleNorthernHemisphereintroductionsofCaprellamutica(Crustacea,Amphipoda)[J].MolecularEcology,2008,17(5):1293-1303.
[29]BOOREJL.Animalmitochondrialgenomes[J].NucleicAcidsRes,1999,27(8):1767-1780.
[30]VANSTAPPENG,YUHY,WANGXM,etal.OccurrenceofallochthonousArtemiaspeciesintheBohaiBayarea,PRChina,asconfirmedbyRFLPanalysisandlaboratoryculturetests[J].FundamentalandAppliedLimnology,2007,170(1):21-28.
StudyongeneticDiversityandgeneticdifferentiationofinvasiveArtemiafranciscanabasedonCOIgene
FUChunZheng1,WANGHui1,AlirezaASEM2,GUOHuiFang3,QIANBinBin4,LIUWenRu4,SHENChunyang3
(1.InstituteofSericulture,ChengdeMedicalUniversity,Chengde067000,China;2.CollegeofFisheriesandLifesciences,HainanTropicalOceanUniversity,Sanya572000,China;3.SchoolofBasicMedicalSciences,ChengdeMedicalUniversity,Chengde067000,China;4.JinmingmiddleschoolofTangshan,Tangshan063000,China)
Abstract:WiththerapiddevelopmentofaquacultureinChina,Artemiafranciscana,whichoriginatedintheUnitedStates,hasbeenintroducedtotheBohaiBayareaofChina.InordertostudythechangesofgenomicgeneticdiversityofArtemiafranciscanasinceitwasintroducedintoChina,COImitochondrialmolecularmarkerwasusedtoevaluatethegeneticdiversityofArtemiafranciscanacolonizedinNanbaoTown,Caofeidian,Tangshan(BohaiBay,HebeiProvince).ItwasfoundthatcomparedwithArtemiafranciscanapopulationinGreatSaltLakeandSanFranciscoBay,ArtemiafranciscanainNanpushowedrichgeneticdiversity.Moreover,therewassignificantgeneticdifferentiationbetweenArtemiafranciscanainNanpuandAmericannativepopulation(GreatSaltLakeandSanFranciscoBaypopulation),indicatingthatArtemiafranciscanainNanpuwasindirectlyintroducedfromAmericanGreatSaltLakeSanFranciscoBay.
Keywords:Artemiafranciscana;COIgene;Geneticdiversity;Populationgeneticstructure
(收稿日期:2021-11-27)

