環境DNA宏條形碼在生物多樣性研究與監測中的應用
康子清 張銀龍,2 吳永波,2 謝冬 薛建輝 華建峰
(1. 南京林業大學生物與環境學院,南京 210037;2. 南京林業大學江蘇省南方現代林業協同創新中心,南京 2100037;3. 中國科學院江蘇省植物研究所(南京中山植物園),南京 210014)
生物多樣性是地球上的生物與其生存環境形成的生態復合體以及與此相關的各種生態過程的總和,維持著全人類所依賴的所有尺度上的生態系統,其變化會對生態系統功能產生影響,并通過生態服務系統進而影響人類獲取福利的大小[1]。由于人類活動加劇,對自然環境干擾加強,全球生物多樣性持續下降成為了21世紀人類面臨的挑戰之一[2]。保護生物多樣性,做到防患于未然的第一步就是要做好生物多樣性監測。目前幾乎所有防止外來物種入侵、保護生物多樣性的措施都依賴于物種監測,物種監測是監測生物多樣性與生態環境變化中應用最為廣泛的方法[3-4]。然而物種監測傳統上依賴于物理識別,即通過物種其獨特的形態特征來進行目視調查計數。這種傳統的物理識別有兩個弊端,其一是需要非常專業的分類學知識[5];其二是部分傳統的調查方法會對當地的自然環境有所破壞,尤其是海洋調查[5-6]。可見傳統的物種監測方法已然限制了全球生物多樣性的研究與環境保護工作,因此急需新技術來進行大規模的生物多樣性監測與研究。
隨著高通量測序技術不斷的發展,誕生了可以快速進行生物多樣性監測的環境DNA宏條形碼技術,對傳統監測技術過程中存在的難題提供了一種方便快捷的解決方法,在大規模的生物多樣性監測與研究中展現出了很好的應用前景。本文概述了環境DNA宏條形碼技術操作流程,并從水環境、土壤環境與空氣環境的角度對國內外環境DNA宏條形碼技術研究進行了總結,以期能為后續生物多樣性研究與監測研究提供新的思路和啟發。
1 環境DNA與環境DNA宏條形碼技術
環境DNA是指生物體內活細胞和胞外DNA衰落死亡或細胞結構破壞,最終排放到環境中且不對任何目標生物進行分離的DNA[7-8]。最早于19世紀80年代由美國科學家Ogram等[9]提出,他在沉積物實驗中首次提取出微生物DNA,但直到20世紀后環境DNA的概念才得到學者的廣泛認可。
基于DNA的物種鑒定方法的出現也為后續環境DNA宏條形碼技術的發明與發展打下了堅實的基礎。Hebert等[10]于2003年首次提出了DNA條形碼技術,認為可以通過一種基因序列來鑒別不同物種,他利用動物線粒體中的細胞色素c氧化酶第一亞基(COI)基因對13 320個動物物種進行分析研究,發現其基因序列可以區分所研究物種。隨后DNA條形碼的概念被引入植物學、微生物學中。DNA條形碼技術是利用不同物種在特定基因上存在的差別來進行物種鑒別的技術[10-11]。區別于傳統的物種監測技術,DNA條形碼技術更加快捷、準確以及對操作者要求更低,因此該技術在物種鑒別方面得到了廣泛的應用。
雖然DNA條形碼技術提供了一種新的物種鑒定技術手段,但也存在著比較大的局限性:首先DNA條形碼技術是利用完整的DNA來進行單個物種鑒定,這是耗時且在某些情況下困難的;其次DNA條形碼在操作過程中也會出現DNA降解等問題,導致測試結果準確度降低[12-13]。隨著高通量測序技術不斷發展,誕生出了可以快速進行生物多樣性監測的環境DNA宏條形碼技術,在降低監測工作量的基礎上,提升了監測效率。例如,在湖泊中要檢測到相同數量的魚類,使用88個刺網需要3 d,而使用環境DNA宏條形碼只需要4 h;對于地中海池塘中兩棲動物的檢測,為獲得與單一環境DNA宏條形碼相似的檢測結果需要進行4次檢測[14]。環境DNA宏條形碼技術通過提取水體、土壤、空氣中的環境DNA,使用引物進行PCR擴增并高通量測序得到上千至上百萬的讀數數據,并利用這些數據確定物種存在,進行生物多樣性評估[15]。
2 環境DNA宏條形碼技術的研究方法
環境DNA宏條形碼技術操作主要包括:樣品采集、DNA提取、PCR擴增、高通量測序、序列對比與物種注釋等環節。
2.1 樣品采集
環境DNA樣本主要來源于水體、土壤以及空氣等。樣品采集的核心問題是要盡可能的獲取并保存目標物的環境DNA。但由于環境DNA的分布會受季節[16]、周圍自然環境[17]壓力等因素的影響,不同的采樣方案與采樣方式對不同來源的環境DNA有著不同的收集能力[18]。因此不同特征的環境類型需要設計針對性的采樣方案。對于流動水體,例如溪流,由于其具有混合良好的湍流,因此表層水即可用于環境DNA檢測[19];對于類似湖泊這樣的靜止系統,沿河岸線進行采樣可以捕獲大多數的環境DNA[18]。目前各環境中樣品采集還沒有統一采樣標準,例如空氣環境微生物采集可以采取慣性撞擊類、過濾阻留類、靜電沉著類和溫差迫降類等多種方法[20]。這些方法各有利弊,需根據研究目的選擇合適的采樣方法。
采樣之后要對環境DNA進行保存,有研究證明環境DNA降解速度很快,一般在48 h內超出檢測范圍,同時在野外條件下環境DNA的降解速度將更快[21]。但在低溫[22]、低 UV-B[23]、堿性[24]條件下環境DNA分解速度較慢。因此在采樣結束后通常加入乙醇或Longmire緩沖液,前者通過使細菌細胞脫水和蛋白質變性來保存環境DNA;后者利用EDTA與SDS抑制酶活性、疊氮化鈉抑制細菌生長[25]。近年也有學者提出用丙二醇防凍劑代替乙醇,可以繞過漫長的蒸發步驟,離心后的樣品可以直接、迅速地提取出DNA樣品,以避免在處理樣本過程中抑制DNA提取或PCR擴增[26]。最后將樣品保存于-20℃的冰箱待測。
2.2 DNA提取
有研究對水樣中環境DNA的提取進行了統計分析,除少部分實驗使用溴代十六烷基三甲胺(CTAB)、酚-氯仿-異戊醇(PCI)或高鹽法提取環境DNA,大多數研究采用DNA提取試劑盒進行環境DNA提取[27]。DNA提取試劑盒是依照氯化芐法所構造的,讓核酸溶于特定溶液中,使其在固相介質內部相互吸附并洗滌去除雜質,通過變更溶液環境使DNA能夠在TE緩沖液或純水中溶解[28]。
土壤DNA提取的主要方法有SDS高鹽提取法、裂解酶法、TENS法與試劑盒法等。但與其他環境中的DNA提取不同,土壤中含有大量的腐殖酸類物質,這些物質在提取過程中無法被去除,基因組中殘留的腐殖酸類物質會抑制Tap聚合酶活性,進而降低PCR擴增的準確性,需要進行DNA純化[29]。由于試劑盒法兼顧DNA的提取與純化,因此在土壤環境DNA宏條形碼中使用最為廣泛。
空氣環境DNA提取同水體、土壤類似,主要使用DNA提取盒。研究證明不同的試劑盒會影響細菌[30]、花粉[31]模擬群落的組成和數量,而對真菌[32]沒有影響。溶解細胞提取DNA時,由于花粉外壁層具有高度結構完整性,而真菌對細胞壁有抗性,無效的細胞破壞只能增加脆弱單核細胞的DNA提取量,而不是增加抗性細胞的DNA提取量,所以需根據實際設計一種有效方法最大限度的提高DNA產量[33]。
2.3 PCR擴增
聚合酶鏈式反應(PCR)是一種體外核酸擴增技術,用于放大與擴增特定的DNA片段,因此PCR擴增的第一步就是確定要擴增的DNA片段。在理想情況下,DNA片段應該滿足如下的標準:(1)特異性:同一物種中DNA片段需幾乎相同,不同物種之間不同;(2)一致性:該DNA片段需標準化,同一片段可以應用于不同的分類群中;(3)穩定性:標記需具有穩定的引物結合位點,允許其在大量的群體中進行擴增和測序[34]。盡管科學家多年來一直在各分類單元中尋找共同的DNA條形碼,但為所有物種找到共同的條形碼是不可能的,動物、植物、微生物在PCR擴增過程中有著相應的DNA條形碼。
Hebert等[10]于2003年首先將線粒體基因細胞色素C氧化酶第一亞基(COI)基因片段用作動物的DNA條形碼擴增區域。從此擴增線粒體COI基因作為研究動物的DNA條形碼得到了廣泛的應用。但隨著研究的深入,單一基因序列導致物種分辨率不足的問題逐漸引起了人們的重視,COI基因在兩棲爬行類種內歧化程度高,PCR擴增過程中會使種間與種內遺傳距離發生重疊現象[35]。且相比于單個DNA條形碼,多種DNA條形碼聯用可以提高物種檢出率[36]。目前動物DNA條形碼的研究多采用COI基因,Cyt b 基因,18S rDNA 和 12S rRNA 等[37-39]。
由于植物的線粒體基因遺傳變異較慢、遺傳分化較小,導致COI種間差異較小,不適用于植物的DNA條形碼擴增,而4個編碼基因區(matK、rbcL、rpoB和rpoC1)與3個非編碼間隔基因(atpF-atpH、trnH-psbA和psbK-psbI)都是可以用于植物DNA條形碼擴增的區域[40]。2009年國際生命條形碼植物工作組從普遍性、序列質量和物種鑒別水平評價的基礎上將植物葉綠體中的核酮糖-1,5-二磷酸羧化酶/加氧酶大亞基(rubisco large subunit,rbcL)和trnK基因中的成熟酶編碼(maturase K,matK)作為植物的標準DNA條形碼片段[40]。此外,在實際運用中核糖體基因內轉錄間隔區(internal transcribed spacer,ITS)也得到了廣泛的使用[41]。
微生物DNA條形碼研究主要集中于真菌,經過各國真菌學家對不同真菌類群的不同基因序列研究篩選過后,于第四屆國際生命條形碼大會確定了ITS作為真菌的首選DNA條形碼,這是由于其是當前真菌物種鑒別能力最高的單一DNA片段,分辨率可達72%,并且大小合適,擴增成功率高[42]。
2.4 高通量測序
傳統的測序方法是由Sanger等于1977年發明的雙脫氧核苷酸末端終止法,但其只能對樣本進行單獨測序,不宜處理復雜的環境樣本[43]。直到2005年,出現了可以在大量環境樣本中恢復DNA序列,同時從多個模板讀取DNA的第二代高通量測序技術,相比于第一代測序技術速度更快,成本更低[44]。
2.5 序列對比與物種注釋
DNA宏條形碼技術的生物信息學分析包含5個核心步驟,即樣品分解、合并雙端測序讀數、質量過濾、OTU管理和分配物種注釋。宏條形碼技術為了最有效的利用測序平臺的高容量流通池,通常將許多樣品復用合并到單個測序文庫中,首先使用測序適配器中的獨特寡核苷酸索引將序列分配回原始樣本[45]。之后需要將測序適配器與PCR引物等非生物信息移除,串聯重復序列,但在數十億堿基的情況下難免會產生錯誤[46]。質量過濾就是降低噪音,即去除低質量、嵌合體以及短于150 bp等的噪音序列。質量過濾時的參數非常重要,序列質量過濾嚴格可消除錯誤讀數,維持正確的生物多樣性與物種豐度估計,但過于嚴格的參數會刪除過多的讀數,導致低豐度的物種難以監測,不利于入侵物種的早期檢測,產生假陰性的錯誤,通常最佳參數一般取決于研究目標[47]。之后這些序列將會集中于一個指定的相似性閾值范圍內(通常為97%),形成一個操作分類單元(OTU),利用BLAST等工具查詢序列與參考數據庫中相似性最高的序列分類進行對比分析,分配物種注釋。
3 環境DNA宏條形碼技術的應用
環境DNA宏條形碼其快捷、準確以及對自然環境破壞小的特點,擴大了生物多樣性的研究范圍,讓更多的科研人員進行生物多樣性研究與監測,在水體[38,48-50]、陸地[51-53]、空氣[54-56]環境中得到了廣泛的應用(圖1)。目前該技術已用于新西蘭生物安全和農業的政府監測項目[6],歐洲標準委員會也將DNAqua-Net作為其一個常設工作組[57]。
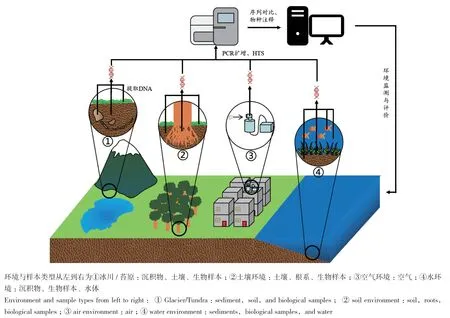
圖1 環境DNA宏條形碼技術在水體、土壤、空氣中的應用流程Fig.1 Application process of environmental DNA macrobarcoding technology in water,soil and air
3.1 水環境
水環境是環境DNA宏條形碼技術運用最廣泛、最成熟的領域。早期研究集中于環境DNA宏條形碼技術與傳統調查方法的比較,發現無論是淡水環境還是海洋,環境DNA宏條形碼技術監測結果更加準確,操作相對便捷,對物種與棲息地的干擾也更小[37,39,58-59]。Ortega 等[60]在研究大型水生植物對海岸沉積物碳匯的貢獻率時,對比了環境DNA宏條形碼技術與傳統的穩定同位素方法,調查表明環境DNA宏條形碼技術與穩定同位素在大型植物碳匯貢獻方面結果相似,但在區分海洋大型植物類群方面具有明顯優勢。Rivera等[61]使用海龜殼上的附生硅藻生物膜來推斷海龜的行為,發現相對于傳統監測方法,環境DNA宏條形碼技術不僅可以應用于生態系統監測,也可應用于監測大型脊椎動物的活動行為。
水環境中的環境DNA宏條形碼研究主要是通過水樣、生物樣本、沉積物這3種方式。水樣采集是水環境中環境DNA宏條形碼測試最常見的方法。正是水體中環境DNA的快速降解的特性,使其在瀕危物種保護與監測入侵物種方面非常有用,因為陽性檢測與當前存在的物種與種群有關,而過去存在物種的環境DNA將不會被發現。目前被用于監測瀕危的巴西青蛙(Hylodes)[62]、鰻鱺[63],與入侵物種亞洲鯉魚[64]、克氏原螯蝦[65]等。利用環境DNA宏條形碼技術,部分地區建立了快速有效和環境友好的監測方案,為生態保護政策的制定提供了科學依據。
除了采集水樣,還可通過直接采集生物樣本與生物膜,并將其分類混合,從混合物中提取環境DNA進行研究。Chain等[49]用大量浮游動物樣本鑒定了加拿大海岸線和五大湖沿岸的海洋和淡水港口約400個后生動物科的生物,其中30種之前從未被報道過。Sohlberg等[50]對地下296 m-798 m的地下水中的真菌群落進行了探索,發現所有深度下均有真菌存在,且多樣性高于預期。環境DNA宏條形碼技術提高了分類學分辨率,產生了更多對已鑒定物種的檢測結果。該技術通過生物樣本可以表征任何規模生物體的復雜群落,并且可使用多個引物實現最佳監測覆蓋范圍。
相較水樣和生物樣本而言,沉積物多被作為古代DNA的主要來源,研究過去的生態環境與生物多樣性[66-67],且可追溯至 12.6 萬年以前[68]。相比于通過花粉與化石研究古代生態系統的傳統方法更加省時省力、準確。這些對過去生態系統的研究在制定未來保護規劃、建立氣候變化模型,以及評估生物多樣性的人為影響等方面具有重要作用[15]。同時環境DNA宏條形碼技術的運用,使得之前傳統方法調查非常困難的海洋沉積物變得簡單了起來[69],且在生物多樣性評價中有著巨大的潛力[70-71]。
如今該技術被用來研究環境中的物種和群落結構,進而評價環境質量與健康狀況。例如通過研究真核生物和細菌指示類群的變化研究河口與沿海環境的營養負荷[72];通過分析細菌、原生生物和后生動物的群落演替,研究河北石油公司溢油事故對沿海生態系統造成的長期的生態影響[73];通過監測浮游植物的生物多樣性與完整性指數,綜合評價太湖水生生態系統的環境質量與健康[74]等。這些研究工作提供了一個全面的、標準化的方法,能夠以更快的時間和更低的成本進行生態系統健康評估。
3.2 土壤環境
土壤環境DNA宏條形碼起始于2009年,Buée等[75]利用土壤樣本評估了6種不同森林土壤中真菌的豐度與多樣性,結果均高于預設,展現出了環境DNA宏條形碼技術在土壤環境中良好的研究前景。
陸地的環境DNA宏條形碼技術研究可以通過土壤、根系、生物樣本等方式,但主要都集中在生物頻繁接觸的土壤樣本上[15]。Bienert等[51]通過環境DNA宏條形碼技術設計2個特異性引物(約30 bp和70 bp),對法國阿爾卑斯山發現的14種蚯蚓建立了線粒體DNA(mtDNA)16S基因的參考數據庫,可以更容易地從土壤樣本中識別出蚯蚓的種類。Andersen等[76]利用丹麥野生動物園、動物園和農場的全pH范圍[(6.2-8.3)±0.2]的土壤樣本監測脊椎動物生物多樣性,為傳統的生物多樣性調查提供了一種快速的替代方法。
植物根系研究中最困難的環節是對不同植物根系的精準識別[77]。隨著高通量技術發展,環境DNA宏條形碼技術被用于植物根系研究。Lamb等[78]對trnL引物進行了修改,成功對溫帶草原與北極苔原生態的根系樣本進行了識別。根系也在構建微生物群落中起著重要作用,Khaliq等[79]分別通過傳統方法與環境DNA宏條形碼技術監測疫霉菌,結果在檢測到的30個疫霉菌系統類型中只有7個是通過傳統方法檢測到的,研究表明根系是檢測疫霉菌群落的最佳樣本。這些研究均體現了環境DNA宏條形碼技術巨大的應用潛力,不僅為土壤根系研究提供了新方法,也為進一步研究植物根系的生態功能開啟了新視角。
土壤環境中,環境DNA宏條形碼技術也可利用生物樣本進行研究。Horton等[52]結合COI基因與核糖體18S rRNA基因設計引物分析無脊椎動物樣本,成功鑒定了馬尼斯蒂國家森林無脊椎動物的生物多樣性。Singer等[80]利用18S rRNA的V9區域,通過凋落物或苔蘚樣本調查高山環境中頂復動物亞門的生物多樣性,進而推導生態系統中后生動物尤其是無脊椎動物的生物多樣性。該研究也表明基于環境DNA宏條形碼技術推斷寄主-寄生物關系,是一種探索未知類群多樣性、推斷潛在寄主-寄生物或其他生物相互作用的有效方法。推導寄主-寄生物和群落的評估,正是一個對生物多樣性監測至關重要,且很難用傳統方法辨別的研究領域[15]。
在土壤環境的生態監測中特別值得關注的是環境DNA宏條形碼技術的一種獨特的監測方法,利用該方法可以通過監測某種生物間接對其他生物進行監測,可監測一些隱蔽與稀有的物種,結果可靠的同時成本也十分低廉。例如Schnell等[81]通過水蛭所吸食的血液在越南熱帶雨林環境中探索了哺乳動物的生物多樣性,并發現了5種新的哺乳動物。Calvignac等[53]將腐肉蠅的源DNA作為一種工具,對哺乳動物生物多樣性進行全面、經濟且有效的評估。Thomsen等[82]通過對野生花卉進行監測來調查陸生節肢動物,發現這些節肢動物來自67個科和14個目。如果沒有環境DNA宏條形碼技術,這樣的監測是不可實現的。
土壤環境中環境DNA宏條形碼技術也可通過監測環境中的物種與群落結構,綜合評價環境質量與健康狀況。Yan等[83]利用環境DNA宏條形碼技術對土壤里的真菌進行定量監測分析,研究真菌對生態系統恢復的反應,監測土壤恢復效果,強調環境DNA宏條形碼技術是一種非常有用的恢復監測工具。Wangensteen等[84]利用多基因(18S和COI)分析3種入侵海藻增殖對西班牙國家公園沿海巖石群落的影響,并制定保護區的管理計劃用來防止或抵消它們的影響。眾多研究表明,相對于傳統技術,環境DNA宏條形碼技術在污染反應研究中顯得更加敏感[85-87]。
3.3 空氣環境
傳統空氣生物學主要依靠培養基和顯微鏡評估生物多樣性與物種豐度。而環境DNA宏條形碼是一種無需培養的方法,可以檢測出不能在體外培養以及死亡或休眠的微生物[88]。因此提供了對空氣中微生物進一步監測、評估、比較和分類的可能性。
空氣中環境DNA宏條形碼主要研究通過空氣傳播導致人與動物過敏甚至感染的物質,例如花粉和病原體等,近年多用于監測城市屋頂和田間中捕獲的孢子樣本的組成[54-55,89-90]。Yamamoto 等[54]以植物病原微生物為研究對象,在美國康涅狄格州紐黑文一座5層建筑的屋頂上進行了4個不同季節的空氣采樣,首次對空氣中真菌的組成進行了研究,發現大氣中真菌多樣性比以往報道的要高,可與土壤環境的真菌多樣性相媲美,同時證明了環境DNA宏條形碼技術在大氣真菌生態學中的巨大潛力。Kraaijeveld等[56]研究了空氣中花粉粒的種類組成,他們在荷蘭萊頓大學醫學中心主樓屋頂通過Burkard空氣顆粒采樣器收集空氣中的花粉,分別使用顯微鏡與環境DNA宏條形碼技術對花粉進行鑒定與定量分析,結果表明環境DNA宏條形碼技術比顯微鏡更易識別出致人過敏的植物花粉的來源,并且隨著該技術的進一步發展,會允許更長的trnL片段被測序,將進一步提高分辨率與降低成本。
雖然大部分空氣環境的環境DNA宏條形碼研究發生在自然環境中,但該方法也可以用于室內環境,特別是類似于垃圾處理廠[91]、農舍[92-93]這些工人每天接觸生物氣溶膠的場所,以及醫院[94]這種人口密集、空氣中存在大量致病微生物的地方。Tong等[94]就利用傳統培養基法和環境DNA宏條形碼方法對醫院空氣中的微生物多樣性進行了研究,測定了真菌、古菌、細菌和病毒的相對豐度,發現與傳統培養基法一次只能鑒定少量病原體不同,環境DNA宏條形碼技術可以提供醫院環境中空氣傳播的微生物的完整分布情況。公共衛生機構也常通過空氣中微生物群落結構對室內的空氣質量進行監測和管理,尤其是大學教學樓與兒童福利機構[33]。Korpelainen等[95]對室內導致人與動物過敏的花粉多樣性與季節關系進行了研究,在冬季與夏季都發現了其具有較高的多樣性。Coombs等[96]檢測了美國綠色環保房屋與非綠色環保房屋中空氣真菌的多樣性,結果顯示綠色環保并不改變室內真菌群落。相信隨著高通量技術的發展,環境DNA宏條形碼技術與其他互補方法的整合,必將提高空氣生物學研究的質量和水平。
4 問題與展望
環境DNA宏條形碼技術作為一種快捷、準確且對環境干擾小的物種監測方法已經受到了生態學家的廣泛應用,雖然仍然存在著一些技術與應用上的挑戰,但隨著環境DNA宏條形碼技術進一步發展,該技術有望成為一種標準的工具[97]。
4.1 采樣方案
樣品采集是進行環境DNA宏條形碼研究的第一步,也是最易受影響的一步。環境DNA的有效性會隨季節變化[16],分布會受到周圍自然環境的限制[17]。不同的采樣方案也對不同來源的eDNA有著不同的收集能力,空間采樣設計深刻影響eDNA檢測,無論是定性還是定量[18]。因此我們要在采用中加入試點研究和適應性抽樣,進一步探究環境條件如何影響環境DNA對物種的檢測,并根據當地環境因地制宜的設計采樣方案。
4.2 偏向性
在進行PCR擴增過程中會產生偏向性[98],出現未能監測到當前存在的生物類群(假陰性),以及對缺失生物類群的錯誤觀察(假陽性)等問題[99],使物種檢測結果準確度降低,對生物多樣性與物種豐富度產生錯誤的評估。建議對eDNA樣本進行優化分析,以嚴格監測污染,提高檢測概率;增加PCR復制次數并將其合并,降低出現假陰性的風險[63]。使用過濾筒和注射器的現場過濾方法也可減少產生假陰性錯誤,因為這種方法將獲得更完整的eDNA[100]。此外,牛津納米孔技術公司(Oxford Nanopore Technologies)于2014年推出的納米孔測序,被廣泛認為是“真正的”第三代測序技術(thirdgeneration sequencing,TGS)[101]。相比于第二代測序技術,第三代測序技術無需進行PCR擴增,可解決PCR擴增偏向性等問題。所以今后要大力發展第三代測序技術,并將環境DNA宏條形碼技術與之結合。
4.3 DNA條形碼數據庫
在目標序列對比分析時,環境DNA宏條形碼技術需要一個高質量、高準確度的參考數據庫。許多研究證明數據庫的錯誤或缺失,都會導致調查結果準確度的下降[102-105]。而構建這樣一個高質量、高準確度的數據庫是一項非常艱難的任務。此外,在全球范圍內對一個大規模的參考數據庫進行對比分析可能需要很長的時間[106]。因此今后要不斷的去豐富并完善DNA條形碼數據庫,提高其準確度與可信度。
4.4 從定性到定量
對于目標生物定量研究一直是環境DNA宏條形碼技術所面臨的主要挑戰[5],自然環境中的生物與非生物因素都會影響環境DNA的來源與降解。盡管有研究在小規模上對目標生物進行了定量與半定量研究[107-109],在大尺度上仍不適用[110]。但這些半定量研究也為我們提供了一種思路,若生物周圍環境DNA的分布規模隨降解力的變化而變化,那么參數化影響過程將是環境DNA宏條形碼技術定量研究的關鍵[111]。也有研究建議使用存在-缺失作為物種相對豐度的度量標準[112]。目前利用環境DNA宏條形碼技術進行定量研究仍有著巨大的挑戰性。
4.5 從研究到應用
要將環境DNA宏條形碼技術長期用于生態環境監測,其成本是一個重要的考慮因素。盡管某些情況下多物種環境DNA宏條形碼技術可以通過評估目標物種數量來彌補高成本這一缺點,但對于單個樣本的測序研究,其成本將大大高于傳統的定量PCR方法(qPCR、ddPCR)[113]。此外,若讓公眾參與到環境DNA宏條形碼技術的調查中,需要更多“用戶友好”型的樣本收集與數據探索方法[97]。隨著大數據、云計算和人工智能等領域的快速發展,可將環境DNA宏條形碼技術與機器學習、衛星遙感等跨學科相結合,形成智能化的生態監測與全球化的監測網絡[114],使環境DNA宏條形碼技術向下兼容,得到更為廣泛的應用。

