利用MiSeq技術研究調味面制食品中優勢菌群及細菌多樣性分析
譚艷妮,施法,李欣蔚,李勇,張健,孫佳星,韓世寧,劉宏生
(1.遼寧大學 生命科學院,沈陽 110036;2.沈陽市市場監管事務服務與行政執法中心,沈陽食品藥品檢驗所,沈陽 110136;3.遼寧省糧食科學研究所,沈陽 110032;4.遼寧省生物大分子模擬計算與信息處理工程研究中心,沈陽 110036)
調味面制食品,俗稱“辣條”、“面筋”、“素牛筋”,主要消費群體是青少年兒童,特別是中小學生[1],年銷售額可高達500億元。它的主要原料是小麥粉,經配料食鹽、食糖、麻辣香辛料以及食品添加劑,通過食品加工機械擠壓、膨脹、蒸煮、成型、調味、包裝等工藝后制成的一種常見的即食方便食品[2-3]。據悉,2015年5月國家食藥監總局發布了關于嚴格加強調味面制品等休閑食品監管工作的通知。總局通過組織專項監測發現,調味面制品普遍存在食品添加劑超范圍、超限量使用,微生物指示菌菌落總數也極易超高超標[4-5]。
長期以來,傳統的微生物菌群多樣性研究主要面臨耗時長、分離鑒定工作量較大等問題[6-8],還有某些特定種群的微生物無法通過傳統的手段分離純化并培養出來[9],令研究成果產生較大的偏差。
高通量宏基因組測序技術,是研究微生物菌群結構多樣性的革命性技術,通過直接對樣品中微生物總的DNA進行提取測序,客觀地反映樣品中低豐度和不可培養的微生物。檢測樣品類別包括食品[10]、醫藥[11]、環境[12]、土壤樣品[13]、含菌空氣、植物果實、動物糞便[14-18]等,檢測結果準確快速。其中二代測序以Illumina MiSeq平臺應用最為廣泛[19],微生物菌群結構多樣性分析主要選擇合適的測序平臺和測序區域。16S rDNA被認為是細菌分類研究和鑒定的指標,通常作為“分子鐘”,16S rDNA 擴增子測序是對高變區進行測序分析和菌種鑒定,其序列包含9個可變區(variable region)和10個保守區(constant region)。可變區因微生物而異,不同的可變區域,分析微生物的能力也不同。目前已涉及一個可變區(V3、V4、V6),兩個可變區(V1-V2、V3-V4、V4-V5),以及3個可變區(V1-V3、V3-V5、V4-V6)。因此,可根據研究選擇特定的可變區域進行PCR擴增,通過檢測目的區域的序列變異和豐度,對特定樣本中微生物種群結構多樣性等方面進行分析比較和研究。
本文采用目前國標方法中的均質拍擊取樣法,選擇Illumina MiSeq測序平臺以及可變區域V3-V4進行測序,分析調味面制品中細菌多樣性,以及對未來改進加工工藝和控制微生物污染以及調味面制食品新國標的擬定提供基礎數據信息。
1 材料與方法
1.1 試驗材料
采用隨機抽樣方式,于沈陽市學校周邊小食店選購調味面制食品1(樣品規格28 g,產地沈陽市)、2(樣品規格26 g,產地重慶市)、4(樣品規格30 g,產地廣州市)。均為預包裝食品,來自不同產地相同的流通環節,并且存放條件相同。由于調味面制品國標缺失,樣品均執行企業標準,購買后保證樣品存儲條件符合要求,并在保質期內對樣品進行檢驗。
1.2 儀器與設備
拍擊式均質機;恒溫水浴鍋;渦旋振蕩器;Eppendorf 5417R冷凍臺式高速離心機、BioPhotometer Plus核酸蛋白測定儀 德國艾本德公司;Model 200/2.0 Power Supply水平電泳儀、Bio-Rad S1000聚合酶鏈式反應儀、Versa Doc凝膠成像儀 美國Bio-Rad公司。
1.3 試驗方法
1.3.1 樣品預處理
稱取25 g樣品置于盛有225 mL生理鹽水的無菌均質袋內,使用拍擊式均質拍打1 min,制成1∶10的樣品勻液。靜置后,吸取上清液40 mL于離心管內,2000 r/min離心10 min,去樣品雜質沉淀;再吸取上清液20 mL,12000 r/min離心5 min,緩慢棄去上清,獲得菌體沉淀。每個樣品均設3個平行,樣品編號分別為1號、2號和4號。
1.3.2 提取樣品基因組DNA
使用細菌基因組DNA快速提取試劑盒(Bacterial DNA Kit,Omega Bio-Tek公司),根據說明書進行細菌總基因組DNA的提取。提取后,檢測DNA提取的濃度,并利用1%瓊脂糖凝膠電泳檢測菌體DNA的完整性。
1.3.3 MiSeq高通量測序
9. D詞義辨析。A.副詞,太,也。B. 副詞,如此的;連詞,因此。C. 副詞,非常,很;形容詞,恰好是,十足的。D. 形容詞,如此的。結合語境可知此處指的是,你是如此了不起的一位父親,故選D。
在大量數據分析中16S rDNA包含高度保守、中度保守和高度變化的序列區域,大小適中,與全長序列測序相比較足以體現不同菌屬之間的差異,同時可降低成本,提高效率。故采用細菌16S rDNA可變區域V3-V4通用引物:
正向引物為341F(5′-CCTACGGGNGGCWGCAG-3′),
反向引物為805R(5′-GACTACHVGGGTATCTAATCC-3′)。
PCR反應體系:15 μL 2×Taq Master Mix,1 μL Primer F,1 μL Primer R,20 ng DNA模版,加入ddH2O至30 μL。PCR反應條件:94 ℃預變性3 min,94 ℃變性20 s,55 ℃延伸20 s,72 ℃退火30 s,25個循環,72 ℃延伸5 min。
1%瓊脂糖凝膠電泳檢測PCR產物,等濃度混樣,使用膠回收試劑盒回收純化產物,用于Illumina MiSeq PE250上機測序。
1.3.4 數據分析
為了更加準確地分析測序得到的數據,去除干擾數據,首先根據Barcode將下機數據(raw data)拆分為不同樣品數據,并截去Barcode序列和PCR擴增引物序列,利用Flash軟件進行過濾拼接,得到高質量的有效數據[20-23]。并對上述數據也就是Effective Tags進行操作分類單元(operational taxonomic units,OTUs)聚類分析(相似性97%)[24]。
本次研究中主要針對的是細菌分析,將OTUs代表序列用RDP Classifier(Version 2.2,http://sourceforge.net/projects/rdp-classifier/)[25]與Greengene 數據庫 (http://greengenes.lbl.gov/)[26]進行物種注釋分析,得到相應的物種及物種豐度信息。
同時計算Chao1指數、ACE指數、Shannon指數、Simpson指數以評估α-多樣性(Alpha diversity),用豐度分布曲線(OTU rank curve)評價豐度及均度,用Venn圖展示不同樣品間特有以及共有OTUs,繪制微生物菌群分布圖和樣品聚類圖[27]。
2 結果與分析
2.1 測序結果及多樣性分析
本試驗將3個產地的調味面制品進行了16S rDNA測序,結果見表1。
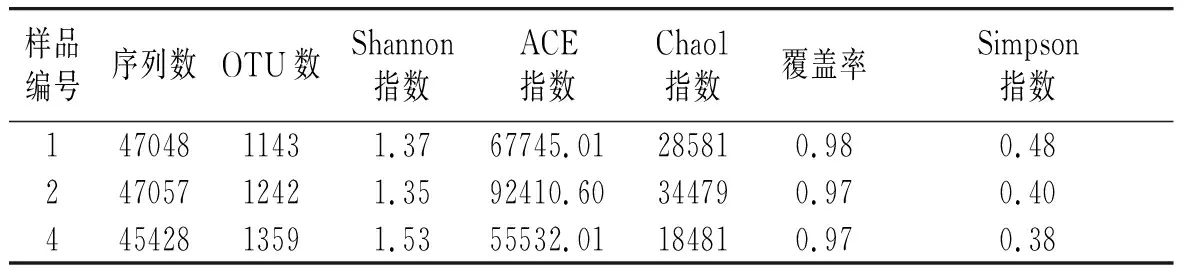
表1 調味面制品樣品16S rDNA測序情況Table 1 16S rDNA sequencing situation of flavored flour foods
經拼接、過濾[28]等處理,本次試驗采集的3個調味面制品樣品總共獲得139533條細菌序列,在97%相似度條件下進行劃分后,共得到3744個OTUs,每個樣品平均為1248個。由表1可知,2號樣品的Chao1指數為34479,即含有最多細菌物種種類,物種豐度最高。而1號樣品的Simpson指數為0.48,即物種均勻度最好。本次試驗覆蓋率(coverage)均在0.97~0.98之間,基本涵蓋樣品中絕大多數菌群,測序數據量滿足分析的需要。
將數據做成指數箱型圖(見圖1)更可直觀看出,這3個樣品的Chao1指數和ACE指數均較高,而Shannon指數和Simpson指數則較低。因此,這3個樣品中的細菌豐度高,而多樣性低。
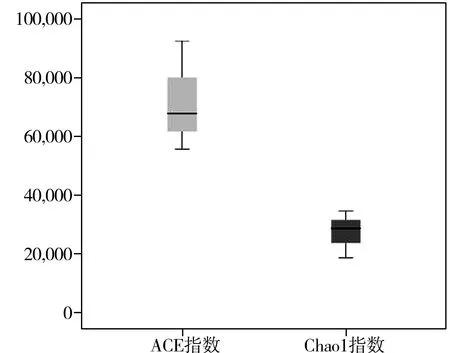

圖1 多樣性指數箱型圖Fig.1 The boxplots of diversity indexes
由圖2可知,來自廣州市的4號樣品物種豐度較高,并且它的物種組成也較為均勻。

圖2 豐度分布曲線Fig.2 OTU rank curve
2.2 樣品中核心細菌菌群及相對含量分析
本試驗測序經過優化平均序列讀長為429 bp,將所得到的全部OTU比對數據庫,共得到10門、14綱、23目、29科、56屬。本試驗中平均相對含量大于1%的細菌屬為芽孢桿菌屬(Bacillus)、明串珠菌屬(Leuconostoc)、銅綠假單胞菌屬(Pseudomonas)、泛菌屬(Pantoea)、布特菌屬(Buttiauxella)、勒克菌屬(Leclercia)。3個樣品中,1號樣品與2號樣品中芽孢桿菌屬(Bacillus)的相對含量均較高,分別為88.08%和50.64%。由圖3可知,調味面制品樣品初始菌群結構中的優勢細菌為芽孢桿菌屬和銅綠假單胞菌屬。

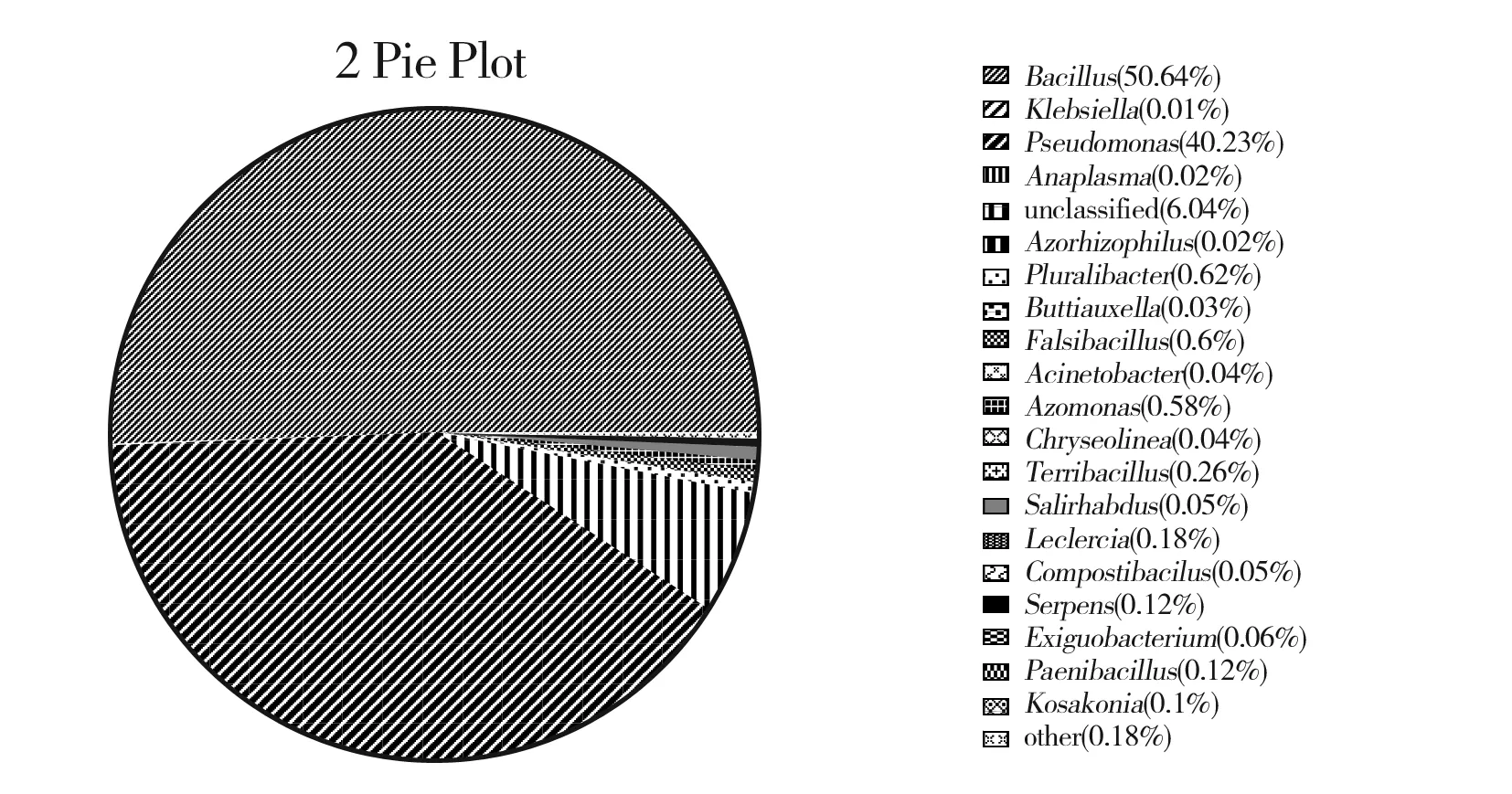

圖3 屬水平所有樣品菌群結構分布圖Fig.3 The distribution of microflora structure of all samples at genus level
2.3 Venn圖分析
在相似度為97%的條件下,進一步統計了3個樣品中OTU出現的次數,利用Venn圖直觀展示了樣品間OTU的重疊情況。結合它所代表的物種,就可以發現不同產地樣品中的核心微生物。
由圖4可知,出現1次和2次的OTU數為3621個和54個,分別占OTU總數的96.71%和1.44%,3組樣品的核心OTU數為15個,占OTU總數的0.40%。1號樣品與2號樣品共有26個OTU數;2號樣品與4號樣品共有34個OTU數;1號樣品與4號樣品共有24個OTU數,由此可見,不同產地樣品之間細菌的多樣性具有很大差異,但是又存在共同的OTU數,并且核心微生物大致相同,可見在加工工藝以及加工過程中存在相似的微生物污染風險。

圖4 OTU樣本分布韋恩圖Fig.4 OTU sample distribution Venn diagram
2.4 樣品中菌群在屬水平上的分類組成
本試驗自定義相對豐度≥10%的菌群為優勢菌群。從屬的水平描述調味面制食品中微生物菌群結構變化,包括59個菌屬:芽孢桿菌屬(Bacillus)、銅綠假單胞菌屬(Pseudomonas)、明串珠菌屬(Leuconostoc)、泛菌屬(Pantoea)、布特菌屬(Buttiauxella)、勒克菌屬(Leclercia)、假芽孢桿菌屬(Falsibacillus)、氮單胞菌屬(Azomonas)、Pluralibacter、微小桿菌屬(Exiguobacterium)、特布爾布爾西菌屬(Trabulsiella)、不動桿菌屬(Acinetobacter)、拮抗菌(Terribacillus)、冷桿菌屬(Psychrobacter)、克呂沃爾菌屬(Kluyvera)、類芽孢桿菌屬(Paenibacillus)、Serpens、沙門氏菌(Salmonella)、陰溝腸桿菌(Enterobacter)、Kosakonia、Salirhabdus、Allobacillus、嗜氮根瘤菌屬(Azorhizophilus)、堆肥芽孢桿菌屬(Compostibacillus)、短小芽孢桿菌(Lysinibacillus)、腸球菌屬(Enterococcus)、克雷伯氏桿菌(Klebsiella)、Chryseolinea、少鹽芽孢桿菌屬(Paucisalibacillus)、無形體屬(Anaplasma)、Ornithinibacillus、約克氏菌(Yokenella)、索氏菌屬(Thauera)、支原菌屬(Mycoplasma)、中華芽孢桿菌屬(Sinibacillus)、Lelliottia、Siccibacter、枝芽孢桿菌(Viridibacillus)、康氏鹽漬芽孢桿菌(Salinibacillus)、太平洋海洋桿菌(Oceanobacillus)、Phenylobacterium、糖芽孢桿菌屬(Saccharibacillus)、弗氏檸檬酸桿菌屬(Citrobacter)、Samsonia、新衣原體屬(Neochlamydia)、海洋紅樹林菌屬(Mangrovibacterium)、弓形桿菌(Arcobacter)、鞘氨醇單胞菌屬(Sphingomonas)、乳球菌屬(Lactococcus)、Thiopseudomonas、Halalkalibacillus、馬賽菌屬(Massilia)、雙歧桿菌屬(Bifidobacterium)、Fontibacillus、奇異單胞菌(Allomonas)、生絲微菌屬(Hyphomicrobium)、根瘤菌屬(Mesorhizobium)、嗜麥芽窄食單胞菌屬(Stenotrophomonas)、Endozoicomonas。進化關系見圖5。

圖5 屬水平所有樣品群落結構分布圖Fig.5 The distribution of microflora structure of all samples at genus level
而利用傳統的微生物培養法檢驗調味面制品,僅發現假單胞菌屬1個優勢菌屬,包括糞嗜冷桿菌(Psychrobacterfaecalis)以及黃褐假單胞菌(Pseudomonasfulva)2株菌,雖然調味面制品產地不同,但是它們的優勢菌中均包括假單胞菌屬,本次試驗共檢出50余種菌,由此可見,傳統的微生物培養法無法反映出樣本中低豐度物種,可能存在對樣本微生物的菌群及優勢菌片面的分析和判斷[29]。
3 討論
本次試驗樣品均購于同一超市,流通環節以及存放方式相同,但產地不同。由圖5可知,各樣品之間的主要菌群結構和比例不盡相同。很可能由于不同樣品的加工工藝以及產地環境各不相同。從門的水平上,3種調味面制食品中的微生物菌群主要為厚壁菌門(Firmicutes)與變形菌門(Proteobacteria),占總菌數的50%~90%,此外還存在少量的擬桿菌門(Bacteroidetes)和螺旋體門(Spirochaetes)。3種調味面制食品在屬的水平上表現出一定的差異,圖3展示了樣品中物種豐度較高的前50個屬水平序列信息。在這些物種豐度較高的50個序列中,選取的3種調味面制食品樣品在菌群結構上存在一定的相似性,3種均包含芽孢桿菌屬(Bacillus)、銅綠假單胞菌屬(Pseudomonas)、布特菌屬(Buttiauxella)、微小桿菌屬(Exiguobacterium)、不動桿菌屬(Acinetobacter)、類芽孢桿菌屬(Paenibacillus)。芽孢桿菌屬、泛菌屬、不動桿菌屬廣泛分布于自然界,而乳球菌屬和雙歧桿菌屬為益生菌,常用于乳制品發酵。值得注意的是,其中銅綠假單胞菌屬、沙門氏菌屬為致病菌,在調味面制食品中發現礦泉水中常見的致病菌——銅綠假單胞菌屬,極有可能是生產加工環節產生了交叉污染、用水不衛生等。可見,為保證產品的衛生質量,加強生產規范化,管理標準化,有效監控微生物風險尤為重要。還有某些菌屬可能處于“活的不可培養狀態(viable but nonculturable state, VBNC)” ,即喪失細菌可培養的特征,但是細胞膜完整,有一定的代謝活力,遇到適宜條件,可恢復到可培養的狀態,例如生長溫度在37 ℃的泛菌屬、明串珠菌屬和不動桿菌屬。
高通量測序技術可以跳過傳統微生物耗時較長的培養步驟,直接提取多個樣品中微生物基因組信息,更全面客觀地反映樣品中微生物菌群結構和多樣性,具有高效、準確、低成本的優勢,但它同時具有相對的局限性。首先,高通量測序過程中產生海量的數據信息,要將這些數據加以總結歸類,建立微生物多樣性的模型,需要生物信息學的專業背景;其次,高通量測序技術雖然可以檢測出樣品中不可培養的微生物,但是并不能確定此微生物是否存活,需要結合傳統培養方法進行比較。
本文采用均質拍擊法提取食品樣品的微生物基因組,利用高通量測序技術(MiSeq)對可變區域V3-V4區進行測序,為后續食品中微生物研究提供了新思路和方法。
研究表明,調味面制食品中微生物多樣性較低,3種調味面制食品中微生物菌群結構具有一定相似性,其中值得引起重視的是致病菌銅綠假單胞菌屬的發現,由于此次未對真菌類進行測序,但仍有部分未確定的菌株,需要進一步研究,在食品安全問題可能發生之前,采取有效的預防措施,將危害系數降到最低。同時加強食品生產工藝流程衛生標準化,保證食品質量安全。

