果蔗Ty3-gypsy類反轉錄轉座子RT基因的多樣性分析
劉俊仙 陽太億 劉菁 張榮華 盧曼曼 雷敬超 高軼靜 丘立杭 李松 熊發前


劉俊仙(1982-),副研究員,廣西知識產權中青年專家和廣西標準化專家,廣西大學校外碩士生導師,主要從事甘蔗生物技術與分子生物學研究工作,近期專注甘蔗全基因組轉座子挖掘、開發應用及其功能研究。主持國家自然科學基金青年科學基金項目1項、地區科學基金項目1項,主持廣西自然科學基金重點項目1項、面上項目2項、青年科學基金項目1項,獲廣西科技進步三等獎1項(排名第四),連續兩次被評為廣西農業科學院有功人員,以第一發明人獲授權國家專利17項,以第一著作權人登記計算機軟件著作權15項,主要參與育成果蔗新品種桂果蔗1號,主要參與制定廣西地方標準5項,以第一作者、共同第一作者或通訊作者發表學術文章近20篇,其中SCI文章4篇。
摘要:【目的】分析Ty3-gypsy類反轉錄轉座子反轉錄酶基因(RT)序列特征、多樣性、系統進化關系及轉錄活性,為深入研究果蔗Ty3-gypsy類反轉錄轉座子全長序列、轉錄轉座活性及生物學功能提供理論參考。【方法】以果蔗品種拔地拉為試材,根據Ty3-gypsy類反轉錄轉座子RT基因保守區設計簡并引物對,利用其進行PCR擴增,將目的條帶回收純化后連接至pMD18-T載體,并轉化大腸桿菌DH5α感受態細胞,挑取陽性克隆進行測序。依據測序結果對RT基因序列進行生物信息學分析。【結果】從果蔗拔地拉中擴增獲得51條序列(SoRT4-1~SoRT4-51),去除重復序列和非RT基因序列后共獲得44條RT基因序列,長度為423~433 bp,AT含量為56.84%~64.97%,AT∶GC比值為1.32~1.85,核苷酸序列間相似性為44.4%~99.3%,其編碼的氨基酸序列間相似性為10.8%~100.0%,說明氨基酸序列比核苷酸序列表現出更高異質性。44條RT基因序列被劃分為4個家族,其中Ⅰ和Ⅲ為主要家族。44條RT基因編碼的氨基酸序列中有20條發生了無義突變,說明其突變率較高。44條RT基因編碼蛋白序列存在5種保守基序,其中,有29條序列同時包含Motif 1~Motif 4,其余15條序列的保守基序變異較大,說明保守基序存在一定異質性。4個家族代表性序列編碼蛋白的三級結構在氫鍵和轉角的數量方面差異較大;系統發育進化樹分析結果顯示,44條RT基因序列被分為七大類,Ⅰ類和Ⅱ類中的果蔗RT基因序列分別占序列總數的40.91%和27.27%,Ⅱ類和Ⅵ類中的部分果蔗RT基因序列與擬南芥的BAB40828.1、粳稻的BAB40824.1、菠菜的BAB40833.1和大豆的BAB40834.1親緣關系較近,推測這些物種在進化過程中發生了Ty3-gypsy類反轉錄轉座子的橫向傳遞。初步發現1條Ty3-gypsy類反轉錄轉座子(SoRT4-40)具有轉錄活性。【結論】獲得44條果蔗Ty3-gypsy類反轉錄轉座子RT基因序列,其中僅有1條序列具有轉錄活性,這些RT基因序列可用于開發基于Ty3-gypsy類反轉錄轉座子的甘蔗分子標記。
關鍵詞: 果蔗;Ty3-gypsy類反轉錄轉座子;反轉錄酶;拔地拉;多樣性
中圖分類號: S566.1? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ?文獻標志碼: A 文章編號:2095-1191(2022)02-0287-12
Diversity analysis of Ty3-gypsy-like retrotransposonsRT gene sequences in fruit sugarcane
LIU Jun-xian YANG Tai-yi LIU Jing ZHANG Rong-hua LU Man-man LEI Jing-chao GAO Yi-jing QIU Li-hang LI Song XIONG Fa-qian
(1Sugarcane Research Institute, Guangxi Academy of Agricultural Sciences/Key Laboratory of Sugarcane Biotechnology and Genetic Improvement (Guangxi), Ministry of Agriculture and Rural Affairs/Guangxi Key Laboratory of Sugarcane Genetic Improvement, Nanning? 530007, China; 2Cash Crops Research Institute, Guangxi Academy of
Agricultural Sciences, Nanning? 530007, China)
Abstract:【Objective】To analyze the characteristics, diversity, phylogenetic evolution and transcriptional activity of Ty3-gypsy-like retrotransposons in fruit sugarcane so as to provide a theoretical reference for the in-depth study of the full-length sequence, transcriptional transposition activity and biological function of Ty3-gypsy-like retrotransposons (RTs) in sugarcane. 【Method】Using the fruit sugarcane variety Badila as the test material, degenerate primers were designed corresponding to the conserved region of Ty3-gypsy-like RT for PCR amplification. The amplified targets were recovered, purified and ligated into pMD18-T vectors. After transformation into Escherichia coli competent cells DH5α, positive clones were picked for sequencing. Ty3-gypsy-like retrotransposon sequences were analyzed by bioinformatics. 【Result】Fifty-one sequences (SoRT4-1-SoRT4-51) were amplified from fruit sugarcane Badila. After removing repetitive sequences and non-RT sequences, a total of forty-four RT sequences were obtained, with lengths of 423-433 bp,an AT content of 56.84%-64.97%,and an AT∶GC ratio of 1.32-1.85. The similarity between the nucleotide sequences was 44.4%-99.3%,whereas the similarity between the encoded amino acid sequences was 10.8%-100.0%, indicating that the amino acid sequences showed higher heterogeneity than nucleotide sequences. The forty-four RT gene sequences were divided into four families,of which I and III were the main families. Nonsense mutations occurred in twenty of the forty-four RT genes, indicating a high mutation rate. There were five conserved motifs in the forty-four RT protein sequences, of which twenty-nine sequences contained motifs 1-4 while the conserved motifs of the remaining fifteen sequences varied greatly. The protein tertiary structures of representative RT gene sequences in four families differ in the number of hydrogen bonds and corner. A phylogenetic tree analysis showed that the forty-four RT gene sequences were divided into seven classes. The sugarcane RT gene sequences in class I and II accounted for 40.91% and 27.27% of the total sequences,respectively. Some su-garcane RT gene sequences in class II and VI were closely related to those of BAB40828.1 of Arabidopsis thaliana,BAB40824.1 of Japonica rice,BAB40833.1 of spinach and BAB40834.1 of soybean. It is speculated that the lateral transmission of Ty3-gypsy-like retrotransposons occurred in the evolution of these species. One Ty3-gypsy-like retrotransposon (SoRT4-40) was initially found to have transcriptional activity. 【Conclusion】Forty-four Ty3-gypsy-like retrotransposons RT gene sequences in fruit sugarcane were obtained, of which only one sequence had transcriptional activity. Molecular markers based on these Ty3-gypsy-like retrotransposons can be used in future analyses of sugarcane genetics.
Key words: fruit sugarcane; Ty3-gypsy-like retrotransposons; reverse transcriptase; badila; diversity
Foundation items:National Natural Science Foundation of China(31960416); Guangxi Natural Science Foundation Program(2018GXNSFDA294004,2020GXNSFAA297081);Guangxi Academy of Agricultural Sciences Fund Project (Guinongke 31960416)
0 引言
【研究意義】甘蔗是我國重要的糖料作物、能源作物和經濟作物。2000年以來,甘蔗種植面積和產糖量均占我國糖料作物總種植面積和食糖總量的90%以上,甘蔗產業對于保障國家蔗糖供給安全發揮舉足輕重的作用(劉俊仙等,2019)。但我國甘蔗生產品種存在單一化且長期連作,導致種性退化嚴重等問題,種性恢復最經濟有效的辦法之一就是培育甘蔗優良新品種,但傳統雜交育種手段選擇效率低下、選擇時間長。隨著生物技術的發展,分子標記輔助育種技術被廣泛應用于作物的新品種選育,該技術可實現定向選擇,從而提高育種效率,但限制甘蔗分子標記輔助育種進展的主要因素之一是缺乏簡單、實用、高效的DNA分子標記。根據長末端重復序列(LTR)反轉錄轉座子的高拷貝數和豐富DNA插入多態性,可開發簡單實用高效的分子標記,對甘蔗分子標記輔助育種具有重要的意義。【前人研究進展】目前,ISSR(余愛麗等,2002)、RAPD(王英等,2009)、SRAP(廖詩童等,2012)、SCoT(Que et al.,2014)和AFLP(昝逢剛等,2015)分子標記技術已在甘蔗上應用,且均可檢測出DNA多態性。但這些技術均存在一些弊端,如RAPD分子標記技術的重復性差;AFLP技術操作流程較繁瑣,且對科研人員及實驗儀器設備上要求較高;ISSR、SRAP和SCoT技術雖然操作不復雜,但結果易受擴增條件和環境的影響,導致穩定性和重復性不高。由于SSR分子標記能揭示豐富的DNA多態性,現已被廣泛應用于甘蔗QTLs定位(Aljanabi et al.,2007)、遺傳圖譜構建(劉新龍等,2010;Andru et al.,2011)和遺傳多樣性分析(劉新龍等,2015)。雖然已開發出一定數量的甘蔗SSR分子標記,但數量遠遠不夠,且擴增效率較低。近年來主要通過高通量轉錄組測序或簡化基因組測序對甘蔗SSR標記進行規模化開發(Huang et al.,2016),但對于基因組龐大(約10 Gb)且復雜的甘蔗而言,設計出總SSR及多態性SSR標記引物還遠遠不夠。另外,SSR分子標記在甘蔗上擴增特異性不強,擴增條帶數較多,擴增條帶模式復雜。LTR反轉錄轉座子具有豐富的插入多態性、高拷貝等特性,根據這些特性已開發建立了4種主要分子標記技術,包括S-SAP、IRAP、REMAP和RBIP(Waugh et al.,1997;Kalendar et al.,1999;Flavell et al.,1998)。在其他物種上,LTR反轉錄轉座子已被廣泛研究,但在甘蔗上,鮮見相關研究報道。Rossi等(2001)以轉座元件、轉座酶、DNA轉座子和反轉錄轉座子為搜索關鍵詞,搜索參數設置為期望值低于e-50,在甘蔗EST庫中進行廣泛搜索,結果顯示,總共獲得了276個轉座元件,DNA轉座子和反轉錄轉座子分別有148和128個,分別占總數的54%和46%,Ty3-gypsy類反轉錄轉座子明顯少于Ty1-copia類反轉錄轉座子,既無LINE類和SINE類反轉錄轉座子,也無MITE轉座子。Raza等(2011)以栽培種甘蔗BL4為材料,一方面使用已知兼并引物對其進行PCR擴增,另一方面根據與Activator(Ac)和Mutator(Mu)DNA轉座子同源的甘蔗表達序列標簽(EST)序列設計引物對其進行PCR擴增,最終從甘蔗BL4中分離出甘蔗Ty1-copia類反轉錄轉座子及Ac和Mu類DNA轉座子;Zhang等(2016)利用多種手段從甘蔗屬96個細菌人工染色體(BAC)克隆鑒定出了各類轉座子。吳子鶯等(2020)通過簡并PCR技術從甘蔗屬大莖野生種中擴增分離出60條Ty1-copia類反轉錄轉座子反轉錄酶基因(RT)序列。劉俊仙等(2021)首次從甘蔗品種新臺糖22中分離出36條Ty3-gypsy類逆轉座子RT基因序列。【本研究切入點】目前未見對果蔗Ty3-gypsy類反轉錄轉座子RT基因序列多樣性分析的研究報道。【擬解決的關鍵問題】克隆果蔗品種拔地拉的Ty3-gypsy類反轉錄轉座子RT基因序列,并分析其序列特征、多樣性、系統進化關系及轉錄活性,不僅為研究RT基因的轉錄活性、轉座活性及調控功能提供原始序列,還為開發果蔗基于Ty3-gypsy類反轉錄轉座子的分子標記打下基礎。
1 材料與方法
1. 1 試驗材料
以目前我國種植面積最大的果蔗品種拔地拉為供試材料。主要試劑:rTaq酶和載體pMD18-T購自寶生物工程(大連)有限公司;大腸桿菌DH5α感受態細胞購自北京全式金生物技術有限公司;其他生化試劑購自生工生物工程(上海)股份有限公司。主要儀器設備:Biometra TOne PCR擴增儀(Analytik Jena AG,德國)和核酸電泳系統(Bio-Rad,美國)。
1. 2 試驗方法
1. 2. 1 DNA提取 參考劉俊仙等(2019)的方法提取果蔗品種拔地拉的總DNA。
1. 2. 2 基因克隆 利用Kumekawa等(1999)設計的簡并引物對Gyrt1和Gyrt2進行RT基因擴增。PCR的反應體系和擴增程序參考劉俊仙等(2019)進行。PCR反應體系:10×PCR Buffer 2.0 μL,50 ng/μL DNA模板1.0 μL,2.5 mmol/L dNTPs 0.5 μL,10 pmol/μL上、下游引物各1.0 μL,5 U/μL rTaq酶0.2 μL,ddH2O補充至20.0 μL。擴增程序:94 ℃,4 min;94 ℃ 30 s,45 ℃ 30 s,72 ℃ 30 s,進行35個循環,72 ℃延伸10 min。取6 μLPCR產物加入4 μL上樣緩沖液并充分混合,利用1.2%瓊脂糖凝膠電泳(電泳緩沖液為1×TAE)進行檢測,凝膠用溴化乙錠(EB)染色后在凝膠成像系統下觀察。用無菌刀片迅速切下目的條帶并回收純化,將其連接至pMD18-T載體上,轉化大腸桿菌DH5α感受態細胞,聯合使用氨芐青霉素、IPTG和X-gal進行篩選,挑取在37 ℃下培養12~16 h后的單菌落接種至LB培養基(含50 mg/L 氨芐青霉素)中培養4~6 h后進行菌液PCR鑒定,將陽性菌液送到生工生物工程(上海)股份有限公司進行測序(熊發前等,2019)。
1. 2. 3 生物信息學分析 利用BLAST對擴增序列進行同源性比對,利用BioEdit對擴增序列進行統計分析,綜合使用DNAMAN、Jalview和Weblogo進行序列多重比對及其Logo生成,利用Phyre2對蛋白的二、三級結構進行預測分析,綜合使用RasMol和MEME計算蛋白的三級結構的轉角、氫鍵并預測其保守基序(陽太億等,2019)。利用MEGA 6.0的鄰接法和No. of differences模型構建系統發育進化樹;通過與甘蔗EST庫比對鑒定Ty3-gypsy類反轉錄轉座子的轉錄活性。
2 結果與分析
2. 1 果蔗RT基因克隆結果
簡并PCR擴增結果(圖1)顯示,從果蔗拔地拉中擴增出一條約430 bp的目的條帶,經過克隆、測序及序列分析,共獲得51條序列(SoRT4-1~SoRT4-51)。去除重復序列和非RT基因序列后,共獲得44條RT基因序列,長度為423~433 bp。對44條RT基因序列采用多重比對分析并生成Logo,如圖2和圖3所示。
2. 2 果蔗RT基因序列的相似性分析結果
由表1可知,44條RT基因序列的長度為423~433 bp,其中,SoRT4-1為423 bp,相較于其他序列,該序列表現為在第285~293 bp處缺失了9 bp,其次是SoRT4-20,該序列表現為在第238~242 bp處缺失了6 bp,但多數序列長度為432 bp。A和T的數量分別為109~147個和110~156個,G和C的數量分別為80~110個和55~92個,AT含量為56.84%~64.97%,AT∶GC比值為1.32~1.85(表1)。44條RT基因的核苷酸序列相似性為44.4%~99.3%,其中,SoRT4-33與SoRT4-38間的相似性最低(44.4%),SoRT4-1與SoRT4-41、SoRT4-8與SoRT4-10間的相似性均最高(99.3%)。44條RT基因編碼的氨基酸序列間相似性為10.8%~100.0%。
2. 3 RT基因序列的聚類分析結果
44條RT基因序列被劃分為4個家族,家族Ⅰ包含14條序列,約占序列總數的31.82%,家族Ⅱ包含9條序列,約占序列總數的20.45%,家族Ⅲ包含14條序列,約占序列總數的31.82%,家族Ⅳ包含7條序列,約占序列總數的15.91%(圖4)。結合圖2推測不同程度的點突變和堿基替換引起家族間及家族內的差異,最終導致果蔗品種Ty3-gypsy類反轉錄轉座子具有較高異質性及多拷貝性。
2. 4 RT基因編碼的氨基酸序列分析結果
對44條RT基因編碼的氨基酸序列進行多重比對分析,結果(圖5)發現有20條序列存在1~14個無義突變。發生1個無義突變的分別是SoRT4-21(第137個aa)、SoRT4-23(第30個aa)和SoRT4-30(第116個aa)、SoRT4-47(第43個aa);發生2個無義突變的分別是SoRT4-8(第65、129個aa)和SoRT4-10(第65、129個aa);發生4個無義突變的是SoRT4-4(第23、56、131、133個aa):發生5個無義突變的分別是SoRT4-33(第30、52、108、121、125個aa)和SoRT4-48(第70、87、95、99、107個aa);發生6個無義突變的分別是SoRT4-26(第23、68、93、97、105、125個aa)、SoRT4-42(第71、88、96、100、108、125個aa)、SoRT4-44(第23、68、93、97、105、125個aa)和SoRT4-50(第23、92、93、97、105、125個aa);發生7個無義突變的是SoRT4-29(第107、108、109、117、121、128、135個aa);發生8個無義突變的分別是SoRT4-4(第77、87、88、101、108、121、123、128個aa)、SoRT4-34(第23、54、92、93、97、105、125、140個aa)和SoRT4-38(第23、44、68、93、97、105、125、140個aa);發生10個無義突變的是SoRT4-20(第88、95、96、103、104、117、127、130、134、137個aa);發生無義突變最多的是SoRT4-5和SoRT4-28,均達到了14個,SoRT4-5突變位點分別是在第23、25、26、29、41、69、90、97、105、112、114、122、129和141個aa,SoRT4-28突變位點分別是在第11、29、33、35、41、54、58、105、106、114、119、122、130和136個aa;SoRT4-29發生了3個連續無義突變。推測無義突變引起反轉錄轉座子失去轉錄活性,導致果蔗品種拔地拉Ty3-gypsy類反轉錄轉座子表現出較高異質性和多拷貝性。
根據44條RT基因核苷酸序列的聚類分析結果,選擇各家族中的代表序列,預測其編碼蛋白的二、三級結構,結果(表2)顯示,這些蛋白的二級結構含有5個α-螺旋、8或9個β-折疊;三級結構含有轉角16~19個及氫鍵68~94個,還含有明顯的螺旋結構5個和折疊結構5個(紅色為N端,藍色為C端)。圖6為家族Ⅰ中代表序列SoRT4-11編碼蛋白的二、三級結構。
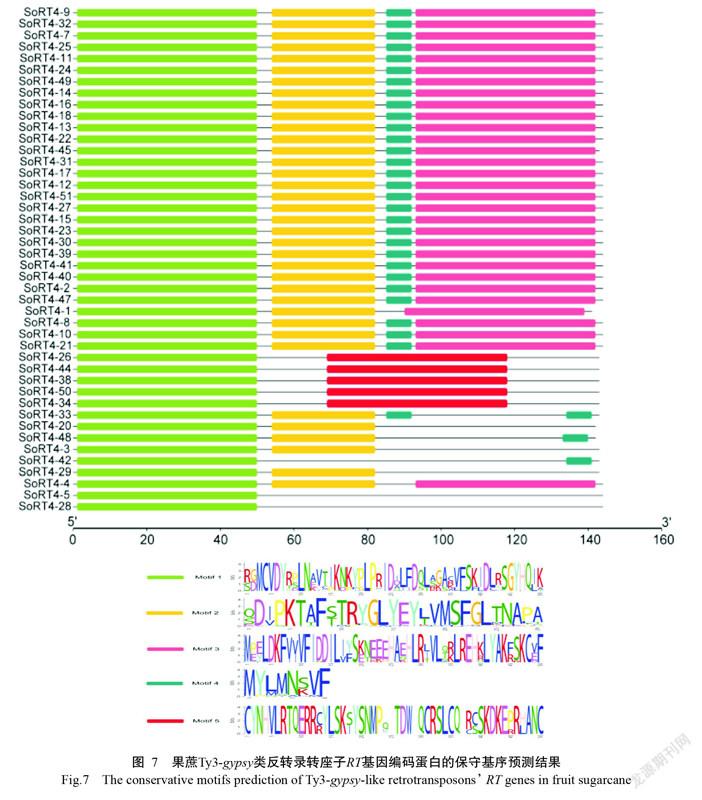
2. 5 RT基因編碼蛋白的保守基序預測結果
由圖7可知,44條RT基因編碼的蛋白序列存在5種保守基序(Motif 1~Motif 5),其中,有29條序列同時包含Motif 1、Motif 2、Motif 3和Motif 4,約占序列總數的65.91%,尤其是Motif 1在44條序列中均存在,高度保守,該保守基序決定了44條序列的編碼基因均為RT基因;其余15條序列中,SoRT4-26、SoRT4-44、SoRT4-38、SoRT4-50和SoRT4-34這5條序列出現了不同的保守基序即Motif 5、SoRT4-33、SoRT4-48和SoRT4-42這3條序列的Motif 4均出現在序列下游。
2. 6 系統發育進化分析結果
將44條果蔗RT基因序列和GenBank中已發表的18條來源于單子葉、雙子葉和裸子植物RT基因序列(表3)翻譯成氨基酸序列,用于構建系統發育進化樹(圖8)。根據果蔗和其他植物RT基因編碼的氨基酸序列,RT基因可劃分為七類(Ⅰ~Ⅶ),Ⅰ類包含18條果蔗RT基因序列,Ⅱ類包含12條果蔗RT基因序列和擬南芥的BAB40828.1,Ⅰ類和Ⅱ類中的果蔗RT基因序列分別占到序列總數的40.91%和27.27%,表明果蔗RT基因序列的保守性較高;Ⅲ類中的14條RT基因序列均來自其他物種植物,但與Ⅰ類和Ⅱ類中的序列親緣關系相對較近;Ⅳ類只包含SoRT4-20,Ⅴ類和Ⅶ類僅包含2條果蔗RT基因序列;Ⅵ類包含果蔗的7條RT基因序列及禾本科粳稻(Oryza sativa japo-nica,BAB40824.1)、藜科菠菜(Spinacia oleracea,BAB 40833.1)和豆科大豆(Glycine max,BAB40834.1)的RT基因序列,表明這些物種親緣關系較近。
2. 7 RT基因序列的轉錄活性初步分析
將RT基因序列與甘蔗EST庫進行比對,結果(表4)顯示,當查詢覆蓋度均為87%時,SoRT4-40與GE325024.1和GE325034.1的一致性均為91.78%,暗示這2條EST序列為果蔗品種拔地拉Ty3-gypsy類反轉錄轉座子的部分轉錄序列,推測SoRT4-40具有轉錄活性。
3 討論
本研究發現,利用簡并引物對PCR擴增同一種質,擴增出的目的條帶是由很多條序列組成的混合體,長度約430 bp,與在其他作物中的擴增結果(侯小改等,2013;張文波等,2016;彭磊等,2017;白楊等,2018;王慶竹等,2018)一致,且這些Ty3-gypsy類反轉錄轉座子RT基因序列在長度、堿基含量和核苷酸相似性上存在較大的差異,表現出較高異質性。另外,在核苷酸序列間相似性和無義突變發生率上,44條果蔗RT基因異質性較高,而在氨基酸序列相似性上,44條果蔗RT基因異質性更高。本研究44條果蔗RT基因編碼氨基酸序列的主要保守基序為Motif 1~Motif 4,表明44條果蔗RT基因保守性較高,但有15條果蔗RT基因序列的保守基序變異較大,表明44條果蔗RT基因序列也存在一定程度的異質性。反轉錄轉座子的轉座高頻突變、自然突變、同源重組及橫縱向傳遞均會導致序列異質性的產生(陽太億等,2019)。
本研究基于RT基因的核苷酸序列進行聚類分析,結果顯示44條果蔗RT基因序列可劃分為四大家族,主要家族是Ⅰ和Ⅲ,表明這些果蔗RT基因呈現一定程度的保守性,初步推測具有轉錄活性的家族是Ⅰ和Ⅲ,它們存在的歷史愈久遠(Tang et al.,2005)。另外,4個家族代表性RT基因編碼的蛋白在氫鍵數量和轉角數量等三級結構上存在差異,家族Ⅳ中的7條RT基因序列均存在多個無義突變,其中SoRT4-26的氫鍵數量明顯少于其他3個家族中的代表性序列,而蛋白質結構的穩定性依靠氫鍵維持,推測該家族RT基因編碼的蛋白已失去活性。
本研究系統發育進化樹分析結果顯示,Ⅱ類中的12條果蔗RT基因序列與擬南芥的BAB40828.1親緣關系較近;Ⅵ類中的7條果蔗RT基因序列與粳稻的BAB40824.1、菠菜的BAB40833.1和大豆的BAB40834.1親緣關系最近,推測橫向傳遞曾發生在果蔗Ty3-gypsy類反轉錄轉座子與這些物種植物之間。Ⅳ類只包含SoRT4-20,Ⅴ類和Ⅶ類中的RT基因序列均來自果蔗,但所包含的RT基因序列較少,且這3類與其他家族的親緣關系較遠,推測這3類RT基因在起源和進化上較古老,為果蔗所特有,特異性較強。
4 結論
從果蔗品種拔地拉克隆獲得44條Ty3-gypsy類反轉錄轉座子RT基因序列,這些RT基因在序列長度、AT含量、核苷酸及氨基酸序列間相似性、無義突變發生率、保守基序異質性及蛋白結構上呈現出多樣性,44條中的1條RT基因序列SoRT4-40具有轉錄活性。
參考文獻:
白楊,林曉飛,張文波. 2018. 雜交構樹Ty3-gypsy類逆轉座子逆轉錄酶序列的多樣性分析[J]. 分子植物育種,16(22):7429-7437. [Bai Y,Lin X F,Zhang W B. 2018. Diversity analysis of Ty3-gypsy-like retrotransposon reverse transposasesin Broussonetia papyrifera L. Vent[J]. Molecular Plant Breeding,16(22):7429-7437.] doi:10. 13271/j.mpb.016.007429.
侯小改,郭大龍,黃燕梅,張曦. 2013. 牡丹Ty3-gypsy類反轉錄轉座子反轉錄酶序列的克隆及分析[J]. 園藝學報,40(1):98-106. [Hou X G,Guo D L,Huang Y M,Zhang X. 2013. Cloning and analysis of reverse trancriptase of Ty3-gypsy-like retrotransposonsin Tree Peony(Paeonia)[J]. Acta Horticulturae Sinica,40(1):98-106.]doi:10. 16420/j.issn.0513-353x.2013.01.013.
廖詩童,賢武,周會,梁強,桂意云,楊榮仲. 2012. 不同耐寒甘蔗品種的SRAP標記分析[J]. 西南農業學報,25(4):1171-1176.[Liao S T,Xian W,Zhou H,Liang Q,Gui Y Y,Yang R Z. 2012. Assessment of cold tolerance in different sugarcane varieties using SRAP markers[J]. Southwest China Journal of Agricultural Sciences,25(4):1171-1176.]doi:10.16213/j.cnki.scjas.2012.04.034.
劉俊仙,劉菁,陽太億,高軼靜,段維興,雷敬超,劉麗敏,劉紅堅,張榮華,何為中,李松,熊發前. 2021. 甘蔗Ty3-gypsy類逆轉座子RT基因的克隆及分析[J]. 福建農業學報,36(9):989-998.[Liu J X,Liu J,Yang T Y,Gao Y J,Duan W X,Lei J C,Liu L M,Liu H J,Zhang R H,He W Z,Li S,Xiong F Q. 2021. Cloning and analysis of reverse transcriptase gene of Ty3-gypsy-like retrotransposons in sugar-cane[J]. Fujian Journal of Agricultural Sciences,36(9):989-998.]doi:10.19303/j.issn.1008-0384.2021.09.001.
劉俊仙,熊發前,劉菁,羅麗,丘立杭,劉麗敏,吳建明,劉紅堅,劉欣,盧曼曼,何毅波,李松. 2019. 用于克隆及分子標記分析的甘蔗高質量基因組DNA提取方法[J]. 分子植物育種,17(2):545-552. [Liu J X,Xiong F Q,Liu J,Luo L,Qiu L H,Liu L M,Wu J M,Liu H J,Liu X,Lu M M,He Y B,Li S. 2019. High quality sugarcane DNA extraction methods for cloning and molecular marker analysis[J]. Molecular Plant Breeding,17(2):545-552.]doi:10.13271/j.mpb.017.000545.
劉新龍,李旭娟,劉洪博,馬麗,徐超華,范源洪. 2015. 云南甘蔗常用親本資源遺傳多樣性的SSR分析[J]. 植物遺傳資源學報,16(6):1214-1222. [Liu X L,Li X J,Liu H B,Ma L,Xu C H,Fan Y H. 2015. Genetic diversity analysis of Yunnan commonly-used parents by using SSR marker[J]. Journal of plant genetic resources,16(6):1214-1222.] doi:10.13430/j.cnki.jpgr.2015.06.011.
劉新龍,毛鈞,陸鑫,馬麗,Aitken K S,Jackson P A,蔡青,范源洪. 2010. 甘蔗SSR和AFLP分子遺傳連鎖圖譜構建[J]. 作物學報,36(1):177-183.[Liu X L,Mao J,Lu X,Ma L,Aitken K S,Jackson P A,Cai Q,Fan Y H. 2010. Construction of molecular genetic linkage map of sugarcane based on SSR and AFLP markers[J]. Acta Agronomica Sinica,36(1):177-183.]doi:10.3724/SP.J.1006. 2010.00177.
彭磊,吳艷,劉小翠,楊鵾,范付華,文曉鵬. 2017. 火龍果Ty3-gypsy類反轉錄轉座子反轉錄酶序列的克隆及分析[J]. 果樹學報,34(2):186-195. [Peng L,Wu Y,Liu X C,Yang K,Fan F H,Wen X P. 2017. Cloning and analysis of reverse transcriptase of Ty3-gypsy retrotransposon in Hylocereus undatus[J]. Journal of Fruit Science,34(2):186-195.]doi:10.13925/j.cnki.gsxb.20160286.
王慶竹,李慧平,文曉鵬,范付華. 2018. 桂花LTR類反轉錄轉座子RT序列的克隆及分析[J]. 園藝學報,45(2):309-320. [Wang Q Z,Li H P,Wen X P,Fan F H. 2018. Clo-ning and analysis of reverse transcriptase of LTR retrotransposons in Osmanthusfragrans[J].Acta Horticulturae Sinica,45(2):309-320.]doi:10.16420/j.issn.0513-353x.2017-0310.
王英,莊南生,黃東益,高和瓊,吳文嬙. 2009. 甘蔗祖親種RAPD標記的序列特征性片段擴增區域(SCAR)轉化分析[J]. 農業生物技術學報,17(4):713-721.[Wang Y,Zhuang N S,Huang D Y,Gao H Q,Wu W Q. 2009. Sequence characterized amplified region(SCAR) translation analysis of the RAPD markers in sugarcane parental germplasm[J]. Journal of Agricultural Biotechnology,17(4):713-721.]doi:1006-1304(2009)04-0713-09.
吳子鶯,楊善,錢旺,吳嘉云,陳柯,張珂,李佩婷,鄧祖湖. 2020. 甘蔗屬大莖野生種Ty1-copia反轉錄轉座子逆轉錄酶序列克隆與特點分析[J]. 植物遺傳資源學報,21(2):466-476. [Wu Z Y,Yang S,Qian W,Wu J Y,Chen K,Zhang K,Li P T,Deng Z H. 2020. Cloning and characterisation of reverse transcriptase sequences of Ty1-copia retrotransposon in Saccharum robustum[J]. Journal of Plant Genetic Resources,21(2):466-476.]doi:10.13430/j.cnki.jpgr.20190506001.
熊發前,劉俊仙,劉菁,賀梁瓊,蔣菁,唐秀梅,黃志鵬,吳海寧,鐘瑞春,韓柱強,唐榮華. 2019. 花生DNA的五種改良CTAB提取方法的比較分析及其應用[J]. 分子植物育種,17(7):2207-2216. [Xiong F Q,Liu J X,Liu J,He L Q,Jiang J,Tang X M,Huang Z P,Wu H N,Zhong R C,Han Z Q,Tang R H. 2019. Comparative analysis and application of five improved CTAB extraction methods for peanut DNA[J]. Molecular Plant Breeding,17(7):2207-2216.]doi:10.13271/j.mpb.017.002207.
陽太億,劉俊仙,劉菁,蔣菁,韓柱強,賀梁瓊,唐秀梅,鐘瑞春,黃志鵬,吳海寧,唐榮華,熊發前. 2019. 四倍體野生種花生Ty1-copia類逆轉座子逆轉錄酶基因的克隆與分析[J].山東農業科學,51(9):9-20. [Yang T Y,Liu J X,Liu J,Jiang J,Han Z Q,He L Q,Tang X M,Zhong R C,Huang Z P,Wu H N,Tang R H,Xiong F Q. 2019. Clo-ning and analysis of reverse transcriptase of Ty1-copia-like retrotransposons in Arachis monticola[J].Shandong Agricultural Sciences,51(9):9-20.]doi:10.14083/j.issn. 1001-4942.2019.09.002.
余愛麗,張木清,陳如凱. 2002. ISSR分子標記在甘蔗及其近緣屬分類上的應用[J]. 福建農林大學學報(自然科學版),31(4):484-489.[Yu A L,Zhang M Q,Chen R K. 2002. Applicability of inter-simple sequence repeat polymorphisms in sugarcane and its related genera as DNA markers[J]. Journal of Fujian Agriculture and Forestry University(Natural Science Edition),31(4):484-489.]doi:1006-7817(2002)04-0484-06.
昝逢剛,應雄美,吳才文,趙培方,陳學寬,馬麗,蘇火生,劉家勇. 2015. 98份甘蔗種質資源遺傳多樣性的AFLP分析[J]. 中國農業科學,48(5):1002-1010. [Zan F G,Ying X M,Wu C W,Zhao P F,Chen X K,Ma L,Su H S,Liu J Y. 2015. Genetic diversity analysis of 98 collections of sugarcane germplasm with AFLP markers[J]. Scientia Agricultura Sinica,48(5):1002-1010.]doi:10.3864/j.issn. 0578-1752.2015.05.18.
張文波,陳凌,李雪輝,白玉娥,林曉飛. 2016. 興安落葉松Ty3-gypsy類逆轉座子逆轉錄酶的多樣性分析[J]. 分子植物育種,14(5):1098-1106. [Zhang W B,Chen L,Li X H,Bai Y E,Lin X F. 2016. Sequence diversity analysis of reverse transcriptases of Ty3-gypsy-like retrotransposons in Larix gmelinii[J]. Molecular Plant Breeding,14(5):1098-1106.]doi:10.13271/j.mpb.014.001098.
Aljanabi S M,Parmessur Y,Kross H,Dhayan S,Saumtally S,Ramdoyal K,Autrey L J C,Dookun-Saumtally A. 2007. Identification of a major quantitative trait locus(QTL) for yellow spot(Mycovellosiella koepkei) disease resistance in sugarcane[J]. Molecular Breeding,19(1):1-14.doi:10.1007/s11032-006-9008-3.
Andru S,Pan Y B,Thongthawee S,Burner D M,Kimbeng C A. 2011. Genetic analysis of the sugarcane(Saccharum spp.) cultivar ‘LCP 85-384. I. Linkage mapping using AFLP,SSR,and TRAP markers[J]. Theoretical and Applied Genetics,123(1):77-93.doi:10.1007/s00122-011-1568-x.
Flavell A J,Knox M R,Pearce S R,Ellis T H N. 1998. Retrotransposon-based insertion polymorphisms(RBIP) for high throughput marker analysis[J]. The Plant Journal,16(5):643-650. doi:10.1046/j.1365-313x.1998.00334.x.
Huang D L,Gao Y J,Gui Y Y,Chen Z L,Qin C X,Wang M,Liao Q,Yang L T,Li Y R. 2016. Transcriptome of high-sucrose sugarcane variety GT35[J]. Sugar Tech,18(5):520-528.doi:10.1007/s12355-015-0420-z.
Kalendar R,Grob T,Regina M,Suoniemi A,Schulman A. 1999. IRAP and REMAP:two new retrotransposon-based DNA fingerprinting techniques[J]. Theoretical and Applied Genetics,98(5):704-711. doi:10.1007/s00122005 1124.
Kumekawa N,Ohtsubo E,Ohtsubo H. 1999. Identification and phylogenetic analysis of gypsy-type retrotransposons in the plant kingdom[J]. Genes & Genetic Systems,74(6):299-307. doi:10.1266/ggs.74.299.
Que Y X,Pan Y B,Lu Y H,Yang C,Yang Y T,Huang N,Xu L P. 2014. Genetic analysis of diversity within a chinese local sugarcane germplasm based on start codon targeted polymorphism[J]. BioMed Research International,11:468375.doi:10.1155/2014/468375.
Raza S,Anjum S,Qamarunisa S,Jamil I,Azhar A,Qureshi J A. 2011. Genome analysis of sugarcane(cultivar BL4) to investigate transposable elements[J]. Pakistan Journal of Biochemistry and Molecular Biology,44(2):68-72.
Rossi M,Araujo P G,Van Sluys M A. 2001. Survey of transposable elements in sugarcane expressed sequence tags(ESTs)[J]. Genetics and Molecular Biology,24(1-4):147-154.doi:10.1590/S1415-47572001000100020.
Tang Y M,Ma Y Z,Li L C,Ye X G. 2005. Identification and characterization of reverse transcriptase domain of transcriptionally active retrotransposons in wheat genomes[J]. Journal of Integrative Plant Biology,47(5):604-612. doi:10.1111/j.1744-7909.2005.00055.x.
Waugh R,McLean K,Flavell A J,Pearce S R,Kumar A,Thomas B B T,Powell W. 1997. Genetic distribution of Bare-1-likeretrotransposable elements in the barley genomerevealed by sequence-specific amplification polymorphisms (S-SAP)[J]. Molecular and General Genetics,253(6):687-694. doi:10.1007/s004380050372.
Zhang J S,SharmaA,Yu Q Y,Wang J P,Li L T,Zhu L,Zhang X T,Chen Y Q,Ming R. 2016. Comparative structural analysis of Bru1 region homeologs in Saccharum spontaneum and S. officinarum[J]. BMC Genomics,17:446. doi:10.1186/s12864-016-2817-9.
(責任編輯 陳 燕)

