2種不同生長規格墨瑞鱈肌肉組織轉錄組測序分析
尤宏爭 肖蕊 劉肖蓮 郝爽 肖俊 郭忠寶
摘要:【目的】掌握不同生長規格墨瑞鱈個體間差異表達基因的表達特點,為其功能相關基因深度挖掘及分子遺傳育種提供科學依據。【方法】挑選同一養殖條件下極大個體和極小個體的墨瑞鱈,構建肌肉組織cDNA文庫后,采用Illumina HiSeqTM 4000測序平臺對存在生長差異的墨瑞鱈肌肉組織進行轉錄組測序分析,獲得的Unigenes在Nr、Nt、Pfam、KOG/COG、Swiss-Prot、KEGG和GO等數據庫中進行比對;通過FPKM及DEGseq篩選出差異表達基因,以GOseq和KOBAS對差異表達基因分別進行GO功能注釋及KEGG信號通路富集分析,并采用MISA進行SSR鑒定分析。【結果】從墨瑞鱈肌肉組織中共測序獲得39749條Unigenes,其長度范圍在301~55230 bp,平均長度為1705 bp。注釋到Nt、Nr、Swiss-Prot、Pfam數據庫的Unigenes分別有27046、20824、18268和17772條,在7個數據庫中均得到注釋的Unigenes共計6742條,占Unigenes總數的16.96%。根據差異表達基因篩選條件P<0.05且|log2Fold Change|>1,共篩選出722個差異表達基因,其中上調基因308個、下調基因414個。差異表達基因GO功能注釋分析結果表明,注釋基因數目較多的GO功能條目包括細胞過程、代謝過程、膜、細胞器及結合等;KEGG信號通路富集分析發現,差異表達基因被成功富集到234條信號通路上,主要涉及磷脂酰肌醇3激酶/蛋白激酶信號通路、MAPK信號通路、胰島素信號通路及FoxO信號通路等。在39749條Unigenes中鑒定篩選出22120個SSRs,占Unigene總數的55.65%,SSR的平均間距為3063 bp。【結論】基于轉錄組測序分析獲得的墨瑞鱈肌肉組織差異表達基因以發揮結合、細胞過程及代謝過程等功能為主,且主要富集在PI3K-Akt信號通路、核糖體信號通路、FoxO信號通路及細胞凋亡等能量代謝相關通路上,通過共同協調而對墨瑞鱈的生長發育起調控作用。
關鍵詞: 墨瑞鱈;肌肉;生長分化;差異表達基因;微衛星(SSR);轉錄組測序
中圖分類號: S917;S965.321? ? ? ? ? ? ? ? ? ? ? ? ? ?文獻標志碼: A 文章編號:2095-1191(2022)03-0768-08
Analysis of muscle transcriptome of two different growth specifications of Maccullochella peelii
YOU Hong-zheng XIAO Rui LIU Xiao-lian HAO Shuang XIAO Jun GUO Zhong-bao
(1Tianjin Fisheries Research Institute, Tianjin? 300221, China; 2Tianjin Counterpart Support Gansu Front Headquarters, Lanzhou, Gansu? 730030,China; 3Guangxi Academy of Fishery Science, Nanning, Guangxi? 530021, China)
Abstract:【Objective】To identify differentially expressed genes(DEGs) between small and large individuals of Maccullochella peelii, to provide a scientific basis for the deep study of genes related to growth and development, molecular genetics and breeding. 【Method】Significantly large and small M. peelii individuals obtained under the same breeding conditions were selected and their muscle tissue sampled to construct cDNA libraries. Transcriptome sequencing of M. peelii muscle tissues was conducted using the Illumina HiSeqTM 4000 sequencing platform. The obtained unigenes were compared in Nr, Nt, Pfam, KOG/COG, Swiss-Prot, KEGG and GO databases. The differentially expressed genes were screened using FPKM and DEGseq. GOseq and KOBAS were used to perform GO function annotation and KEGG signaling pathway enrichment, then identified by SSR by MISA. 【Results】A total of 39749 unigenes were generated from muscle tissue of M. peelii, with lengths ranging from 301 to 55230 bp and an average length of 1705 bp. There were 27046, 20824, 18268 and 17772 unigenes annotated to Nt, Nr, Swiss-Prot and Pfam databases, respectively. In total, 6742 unigenes were annotated by seven databases, accounted for 16.96% of the total unigenes. Differentially expressed genes were defined as those displaying an adjusted P<0.05 and |log2Fold Change|>1. A total of 722 DEGs were selected, consisting of 308 up-regulated genes and 414 down-regulated genes. The results of GO function annotation showed that the GO function items with a large number of annotation genes including cellular process, metabolic process, membrane, organelle and binding. KEGG signaling pathway enrichment revealed that DEGs were enriched in 234 signal pathways, mainly involving phosphatidylinositol 3 kinase / protein kinase signal pathway, MAPK signal pathway, insulin signal pathway and the FoxO signal pathway. A total of 22120 SSRs were identified from 39749 unigenes, accounted for 55.65% of the total number of unigenes. The average spacing of SSRs was 3063 bp. 【Conclusion】The obtained DEGs in M. peelii muscle tissue based on transcriptome sequencing mainly play the functions of binding, cellular process and metabolic process, and mainly enriched in PI3K Akt signal pathway, ribosomal signal pathway, FoxO signal pathway, apoptosis and other energy metabolism related pathways, which together regulate the growth and development of M. peelii.014C7B63-A0F3-407B-A447-0D6F456619EF
Key words: Maccullochella peelii; muscle; growth and differentiation; differentially expressed genes; microsatellite (SSR); transcriptome sequencing
Foundation items: National Modern Agriculture Industry Technology System Construction Project(CARS-46); Tianjin Science and Technology Planning Project (21YDTPJC00070); Guangxi Characteristic Freshwater Fish Industry Innovation Team Project of National Modern Agriculture Industry Technology System Construction Project (nycytxgxcxtd-2021-08-03)
0 引言
【研究意義】墨瑞鱈(Maccullochella peelii)又名蟲紋鱈鱸、澳洲龍紋斑、河鱈、東洋鱈和澳洲淡水鱈鱸等,隸屬于鱸形目(Perciforme)鮨鱸科(Percichthyidae)鱈鱸屬(Maccullochella)。墨瑞鱈原產于澳大利亞東南部墨累—達令盆地,因其肉質細嫩、味道鮮美,且富含EPA(二十碳五烯酸)和DHA(二十二碳六烯酸),在澳大利亞素有“國寶魚”之稱(羅欽等,2020)。目前,在我國天津、江浙及山東等地已引進養殖,是一個具有良好發展前景的淡水養殖優質品種(饒秋華等,2020);但在養殖過程中,常出現相同養殖環境中規格不一的情況,嚴重制約著養殖效益的進一步提高。關于墨瑞鱈生長緩慢、規格不一的原因至今尚未明確,因此,亟待對存在生長差異的墨瑞鱈肌肉組織進行轉錄組測序分析,為解決其生長緩慢的問題提供思路,進而保障墨瑞鱈養殖業持續高質量發展。【前人研究進展】轉錄組測序是通過高通量測序技術對特定條件下機體某組織的所有轉錄本數據進行比對分析及功能注釋,旨在挖掘與性狀相關的功能基因和信號通路(白獻曉等,2017;崔凱等,2019)。隨著高通量測序技術的快速發展及測序成本的不斷降低,轉錄組測序技術在水產研究領域中已得到廣泛應用(張毓霞等,2018;趙彥花等,2019;黃新芯等,2021)。嚴璐琪等(2016)采用Illumina高通量測序對黑殼與金殼葡萄牙牡蠣進行轉錄組分析,通過功能注釋與比對分析鑒定出14個與殼色表達有關的貝殼基質蛋白基因,其中5個下調基因可能與金殼基因表達相關。劉思嘉等(2018)采用轉錄組測序技術檢測低溫耐受與低溫敏感鯉品種在低溫脅迫下的差異表達基因,發現編碼糖酵解/糖質新生途徑中的多個關鍵酶基因可能與其低溫適應過程有關。孟瑋等(2021)基于高通量測序技術對不同鹽度條件下的日本黃姑魚幼魚肌肉組織進行轉錄組測序,結果表明日本黃姑魚對急性鹽度脅迫的適應可能是一個涉及多組織和多基因的復雜過程,鹽度變化會影響肌肉組織的離子通道、離子轉運體及蛋白降解和免疫系統功能。關于魚類生長優勢分子機制的研究成果也取得長足進展,林明德(2019)基于高通量測序獲得的雜交石斑魚和褐點石斑魚腦組織、肝臟和肌肉轉錄組數據,篩選出一批生長相關的差異表達基因;羅志嘉等(2019)以生長存在顯著差異的快長組和慢長組角鱉肝臟為材料,通過RNA-Seq測序分析探索了角鱉生長發育的分子機制;王登東等(2019)通過轉錄組測序在云龍石斑魚腦組織、垂體、肝臟和肌肉樣本中發現成纖維生長因子、表皮生長因子、血管生成因子等相關基因表達差異顯著,并預測了云龍石斑魚生長優勢的重要貢獻因素;馮培哲(2020)通過對金錢魚垂體和肝臟組織的轉錄組分析,為進一步研究金錢魚的生長調控機制和分子特征提供了理論依據;李雅慧等(2021)通過轉錄組測序對比2種不同規格羅氏沼蝦基因表達量的差異,探究其生長差異的分子機理;王紅明等(2021)通過轉錄組數據分析了飼料添加劑對龍膽石斑魚的生長性能等影響。可見,在水產動物中,高通量測序已廣泛應用于生長、發育等相關生命活動的分子機制研究。【本研究切入點】目前,關于墨瑞鱈的研究主要集中在遺傳多樣性檢測(Loughnan et al.,2010)、資源恢復(Forbes et al.,2016)及飼料營養(Francis et al.,2019)等方面。此外,Downing和Litvak(2001)研究表明光照強度能增加黑線鱈幼魚的攝食量,進而影響其生長;狄正凱(2020)通過探究光照因子對墨瑞鱈生長性能、肌肉營養成分及應激反應影響的變化規律,證實合理選擇光照條件對促進墨瑞鱈生具有重要意義;但尚無針對墨瑞鱈生長差異產生機理闡釋及轉錄組學分析的研究報道。【擬解決的關鍵問題】挑選同一養殖條件下極大個體和極小個體的墨瑞鱈,通過高通量測序對其肌肉組織進行轉錄組分析,篩選出差異表達基因,并利用GO功能注釋及KEGG信號通路富集進行差異性分析,旨在掌握不同規格個體間差異表達基因的表達特點,為墨瑞鱈功能相關基因深度挖掘及分子遺傳育種提供科學依據。
1 材料與方法
1. 1 試驗材料
供試墨瑞鱈為天津盛億養殖有限公司飼養的5月齡苗種,在同一養殖池中隨機選取極大個體和極小個體各3尾,對應的體長分別為13.37±0.29和7.75±0.06 cm,體質量分別為41.23±3.10和9.57±0.21 g。采集墨瑞鱈肌肉組織,經液氮速凍后-80 ℃保存備用。
1. 2 cDNA文庫構建及高通量測序
使用TRIzol Reagent試劑盒(Invitrogen)對提取肌肉組織總RNA,分別采用瓊脂糖凝膠電泳檢測RNA是否降解,NanoDrop檢測OD260/280比值,Qubit檢測RNA濃度,Agilent 2100 Bioanalyzer檢測RIN值(RNA integrity number,分子完整數)。根據TruSeq DNA Library Prep Kit (Illumina)說明,進行cDNA合成、末端修復、連接接頭及擴增純化,最終獲得墨瑞鱈肌肉組織cDNA文庫。構建好的cDNA文庫委托天津諾禾致源生物信息科技有限公司使用Illumina HiSeqTM 4000測序平臺完成測序。014C7B63-A0F3-407B-A447-0D6F456619EF
1. 3 序列拼接及功能注釋
將測序所得原始序列(Raw reads)中帶接頭且低質量的序列刪除,即得到有效序列(Clean reads)。應用Trinity對Cleans reads進行拼接,選擇最長的轉錄本作為Unigenes。將所得的Unigenes在Nr、Nt、Pfam、KOG/COG、Swiss-Prot、KEGG和GO等數據庫中進行比對,以獲得Unigenes的功能注釋信息。
1. 4 差異基因表達分析
利用Bowtie將每個樣品的Clean reads比對到組裝出來的轉錄本上,使用RSEM統計每個樣品比對到每個基因上的Read count數目,根據FPKM估算基因的表達水平。采用DESeq篩選差異表達基因,差異表達基因篩選條件:P<0.05且|log2 Fold Change|>1。采用GOseq和KOBAS進行差異表達基因的GO功能注釋分析及KEGG信號通路富集分析。
1. 5 SSR鑒定分析
采用MISA對獲得的Unigenes進行微衛星位點(SSR)查找,其篩選參數設為:單核苷酸重復、二核苷酸重復、三核苷酸重復、四核苷酸重復、五核苷酸重復、六核苷酸重復的最少重復次數分別為10、6、5、5、5和5。
2 結果與分析
2. 1 轉錄組測序與組裝結果
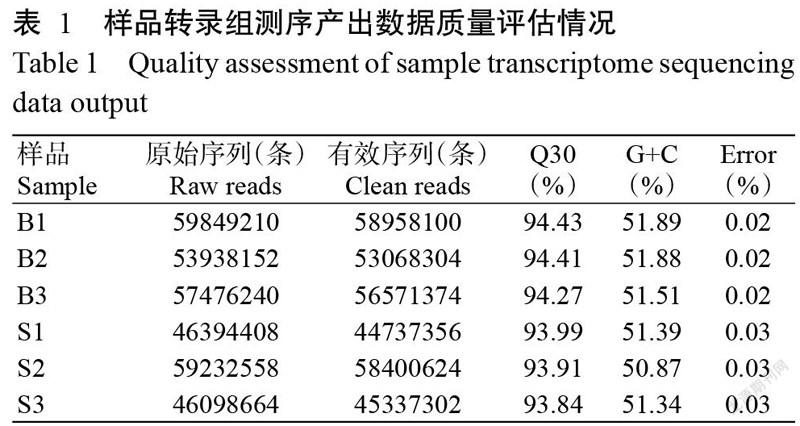
采用Illumina HiSeqTM 4000測序平臺對極大個體和極小個體(各3尾)進行轉錄組測序,測序產出數據的質量評估情況詳見表1。6個樣品共測序獲得322989232條Raw reads,去除低質量序列后獲得317073060條Clean reads。各樣品的Q30均在93.00%以上,GC含量在50.00%以上,表明轉錄組測序產出量豐富且質量良好。通過數據組裝共得到39749條Unigenes,其長度范圍在301~55230 bp,平均長度為1705 bp。
2. 2 Unigenes注釋結果
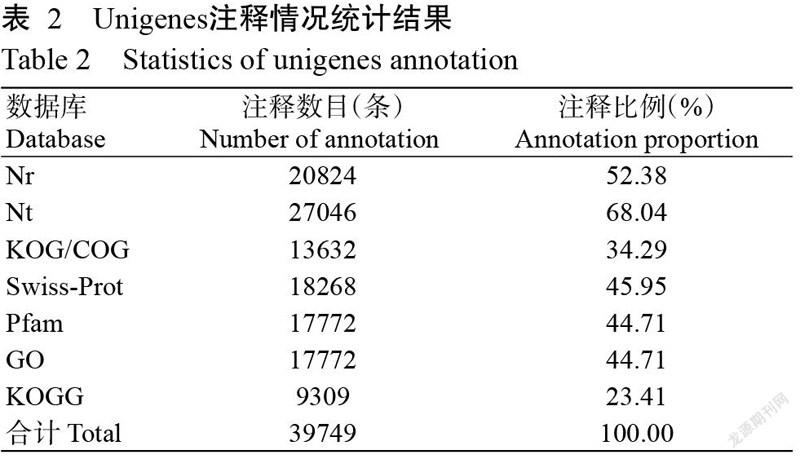
拼接獲得的39749條Unigenes在Nr、Nt、Pfam、KOG/COG、Swiss-Prot、KEGG及GO等數據庫中進行比對,結果如表2所示,注釋到Nt、Nr、Swiss-Prot和Pfam數據庫的Unigenes分別有27046、20824、18268和17772條,其中注釋到Nr數據庫的Unigenes占Unigenes總數的52.38%(表2)。在7個數據庫中均得到注釋的Unigenes共計6742條,占Unigenes總數的16.96%;至少在1個數據庫中得到注釋的Unigenes共計29069條,占Unigenes總數的73.13%,剩下未注釋的Unigenes可能是未知基因。
2. 3 差異表達基因篩選結果
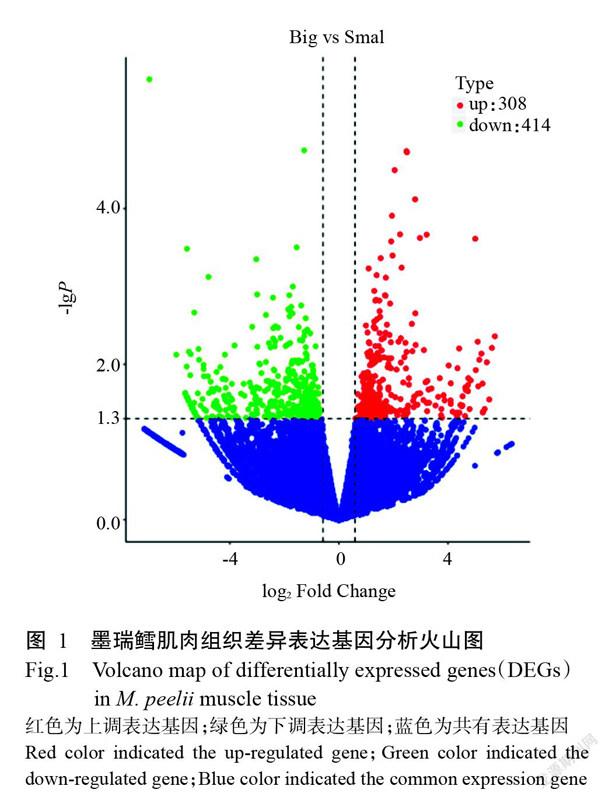
根據差異表達基因篩選條件P<0.05且|log2 Fold Change|>1,采用DESeq進行篩選差異表達基因,結果篩選得到722個差異表達基因,其中上調基因308個、下調基因414個(圖1)。
2. 4 差異表達基因GO功能注釋分析結果
對722個差異表達基因進行GO功能注釋分析,結果表明差異表達基因分別注釋到生物學過程(Biological process)、細胞組分(Cellular component)及分子功能(Molecular function)三大功能分類(圖2)。在生物學過程中,差異表達基因主要注釋到細胞過程(Cellular process)、代謝過程(Metabolic process)、單有機體過程(Single organism process)、有機物代謝過程(Metabolic process of organic matter)及初級代謝過程(Primary metabolic process)等方面;在細胞組成中,差異表達基因主要注釋到膜(Membrane)、細胞器(Organelle)及高分子復合物(Polymer complex)等方面;在分子功能中,差異表達基因主要注釋到結合(Combination)、催化活性(Catalytic activity)、水解酶活性(Hydrolase activity)及GTPase活性(GTPase activity)等方面。
2. 5 差異表達基因KEGG信號通路富集分析結果
差異表達基因KEGG信號通路富集分析結果(圖3)顯示,722個差異表達基因被成功富集到234條KEGG信號通路上,且主要富集在磷脂酰肌醇3激酶/蛋白激酶信號通路(PI3K-Akt signaling pathway)、MAPK信號通路(MAPK signaling pathway)、半胱氨酸和蛋氨酸代謝(Cysteine and methionine metabolism)、PPAR信號通路(PPAR signaling pathway)、B細胞受體信號通路(B cell receptor signaling pathway)等信號通路上。
2. 6 SSR鑒定分析結果
在39749條Unigenes中鑒定篩選出22120個SSRs,占Unigenes總數的55.65%,SSR的平均間距為3063 bp。其中,復合型SSR有2163個,單核苷酸重復SSR有9483個,二核苷酸重復SSR有6742個,三核苷酸重復SSR有3326個,四核苷酸重復SSR有356個,五核苷酸重復SSR有36個,六核苷酸重復SSR有14個。在單核苷酸重復類型中,數量最多的重復基元為A/T(8535個),二核苷酸重復中出現次數最多的為AC/GT(2569個)。
3 討論
生長分化是魚類生長過程中的普遍現象,同一群體中的個體由于受到遺傳、環境及營養等因素的協同影響,通常會出現生長分化現象(石軍等,2013)。為揭示墨瑞鱈的生長分化機理,本研究針對同一群體中不同生長規格的墨瑞鱈肌肉組織進行轉錄組測序,結果共獲得39749條Unigenes,其長度為301~55230 bp,平均長度為1705 bp,數據產出與質量均滿足轉錄組分析及差異表達基因挖掘的基本需求。通過與Nr、Nt、Pfam、KOG/COG、Swiss-Prot、KEGG和GO等數據庫中的序列進行比對及功能注釋,結果表明,有29069條Unigenes與其他物種的已知基因具有不同程度的同源性,占Unigenes總數的73.13%;其中在GO和KEGG數據庫中注釋富集到722條編碼氨基酸次生代謝途徑關鍵酶基因。014C7B63-A0F3-407B-A447-0D6F456619EF
高通量轉錄組測序技術在豐富物種基因資源的同時,也為分子標記的開發提供了有利條件(賈昌路等,2015;宋尚橋等,2020;彭金英和伊成器,2021)。與傳統的磁珠富集法相比,利用轉錄組測序進行分子標記開發具有更周期短、費用低廉、標記量大、標記與功能基因連鎖等優勢(黃思婕,2020),目前已在水產研究領域得到廣泛應用(龔詩琦等,2016;趙彥花等,2019;Francis et al.,2019;方軍等,2020;李喜蓮等,2020)。本研究在39749條Unigenes中篩選出22120個SSRs,其發生率為55.65%,遠高于龔詩琦等(2016)在黃姑魚(39.30%)、方軍等(2020)在青蛤(10.76%)、李喜蓮等(2020)在紅螯螯蝦(33.74%)中的篩選效果,說明本研究轉錄組數據中墨瑞鱈的SSR分子標記含量更豐富,可為下一步開展群體遺傳結構分析、親緣關系鑒定、種質資源保護及良種選育提供技術支撐。
本研究的差異表達基因GO功能注釋分析結果表明,注釋基因數目較多的GO功能條目包括細胞過程、代謝過程、膜、細胞器及結合等;同時部分差異表達基因富集在肌鈣蛋白復合物(Troponin complex)、橫紋肌細絲(Striated muscle filament)、肌原纖維(Myofibril)、肌節(Sarcomere)、肌絲(Myofilament)、收縮纖維(Shrinkage fiber)、收縮纖維部分(Shrin-kage fiber part)、蛋白質分解代謝過程調節(Regulation of protein catabolism)、組織再生(Tissue regene-ration)、發育生長(Development and growth)、肌動蛋白結合(Actin binding)、生長因子活性(Growth factor activity)、胰島素樣生長因子結合(Insulin like growth factor binding)、生長因子結合(Growth factor binding)、細胞生長調節(Cell growth regulation)及細胞生長(Cell growth)等GO功能條目,這些基因可能參與墨瑞鱈的生長調控過程,可進一步進行基因功能與表達模式研究。KEGG信號通路富集分析結果顯示,差異表達基因被成功富集到234條信號通路上,富集度最高的是磷脂酰肌醇3激酶/蛋白激酶信號通路。此外,本研究篩選出部分與生長過程密切相關的KEGG信號通路,包括胰島素信號通路(Insulin signaling pathway)、MAPK信號通路、細胞凋亡(Apoptosis)、FoxO信號通路(FoxO signaling pathway)、TGF-β信號通路(TGF-beta signaling pathway)等,與Zhang等(2020)的研究結果一致。磷脂酰肌醇3激酶/蛋白激酶信號通路可被多種生長因子和信號傳導復合物,包括成纖維細胞生長因子(FGF)、血管內皮生長因子(VEGF)、人生長因子(HGF)、血管位蛋白 I(Ang1)和胰島素等激活,活化的Akt通過磷酸化作用調控一系列下游因子以實現對細胞增殖、分化、凋亡和葡萄糖轉運等功能的調控(狄正凱,2020)。該結論為進一步闡釋墨瑞鱈的生長發育機理和調控網絡提供了基礎數據支持。
4 討論
基于轉錄組測序分析獲得的墨瑞鱈肌肉組織差異表達基因以發揮結合、細胞過程及代謝過程等功能為主,且主要富集在PI3K-Akt信號通路、核糖體信號通路、FoxO信號通路及細胞凋亡等能量代謝相關通路上,通過共同協調而對墨瑞鱈的生長發育起調控作用。
參考文獻:
白獻曉,張子敬,王璟,徐照學. 2017. 轉錄組測序技術在家畜遺傳育種中的應用研究進展[J]. 河南農業科學,46(4):6-9. [Bai X X,Zhang Z J,Wang J,Xu Z X. 2017. Application of transcriptome sequencing technology in genetic breeding of livestock[J]. Journal of Henan Agricultural Sciences,46(4):6-9.] doi:10.15933/j.cnki.1004-3268. 2017.04.002.
崔凱,吳偉偉,刁其玉. 2019. 轉錄組測序技術的研究和應用進展[J]. 生物技術通報,35(7):1-9. [Cui K,Wu W W,Diao Q Y. 2019. Application and research progress on transcriptomics[J]. Biotechnology Bulletin,35(7):1-9.] doi:10.13560/j.cnki.biotech.bull.1985.2019-0374.
狄正凱. 2020. 光照對循環水系統中墨瑞鱈生長、肌肉營養成分及應激反應的影響[D]. 上海:上海海洋大學. [Di Z K. 2020. Effects of light factor on the growth,muscle nutrition and stress response of Murray cod in recirculating aquaculture[D]. Shanghai:Shanghai Ocean University.] doi:10.27314/d.cnki.gsscu.2020.000469.
方軍,沈彥鵬,張雷雷,李騰騰,邵艷卿. 2020. 基于轉錄組數據的青蛤微衛星標記開發與驗證[J]. 應用海洋學學報,39(2):214-220. [Fang J,Shen Y P,Zhang L L,Li T T,Shao Y Q. 2020. Development and validation of SSR mar-kers in Cyclina sinensis[J]. Journal of Applied Oceanography,39(2):214-220.] doi:10.3969/J.ISSN.2095-4972. 2020.02.008.014C7B63-A0F3-407B-A447-0D6F456619EF
馮培哲. 2020. 金錢魚生長抑素(Somatostatin)在生長調控中的功能研究[D]. 湛江:廣東海洋大學. [Feng P Z. 2020. Functions of somatostatin(SST) in growth regulation of Scatophagus argus[D]. Zhanjiang:Guangdong Ocean University.] doi:10.27788/d.cnki.ggdhy.2020.000145.
龔詩琦,王志勇,肖世俊,林愛強,謝仰杰. 2016. 黃姑魚轉錄組SSR的開發與驗證[J]. 集美大學學報(自然科學版),21(4):241-246. [Gong S Q,Wang Z Y,Xiao S J,Lin A Q,Xie Y J. 2016. Development and validation of SSR based on transcriptome of Yellow Drum,Nibea albiflora[J]. Journal of Jimei University(Natural Science),21(4):241-246.] doi:10.3969/j.issn.1007-7405.2016.04.001.
黃思婕. 2020. 持續性高溫對雌雄羅非魚肌肉生長的影響機制研究及可變剪接分析[D]. 上海:上海海洋大學. [Huang S J. 2020. Study on the growth mechanism of muscle after persistent high temperature to male and female Nile tilapia and alternative splicing analysis[D]. Shanghai:Shanghai Ocean University.] doi:10.27314/d.cnki.gsscu.2020. 000392.
黃新芯,蔣艷琳,蔣小姿,楊天燕. 2021. 基于高通量轉錄組測序技術的龍頭魚微衛星信息分析[J]. 浙江海洋大學學報(自然科學版),40(3):189-197. [Huang X X,Jiang Y L,Jiang X Z,Yang T Y. 2021. Analysis of microsatellite markers in harpadon nehereus based on transcriptome sequencing Illumina HiseqTM 2500[J]. Journal of Zhejiang Ocean University(Natural Science),40(3):189-197.] doi:10.3969/j.issn.1008-830X.2021.03.001.
賈昌路,張瑤,朱玲,張銳. 2015. 轉錄組測序技術在生物測序中的應用研究進展[J]. 分子植物育種,13(10):2388-2394. [Jia C L,Zhang Y,Zhu L,Zhang R. 2015. Application progress of transcriptome sequencing technology in biological sequencing[J]. Molecular plant Breeding,13(10):2388-2394.] doi:10.13271/j.mpb.013.002388.
李喜蓮,郭建林,李倩,施偉達,黃振遠,顧志敏. 2020. 紅螯螯蝦轉錄組中的SSR位點信息分析[J]. 湖北農業科學,59(7):207-211. [Li X L,Guo J L,Li Q,Shi W D,Huang Z Y,Gu Z M. 2020. The information analysis of SSR loci in the red claw crayfish(Cherax quadricarinatus) transcriptome[J]. Hubei Agricultural Sciences,59(7):207-211.] doi:10.14088/j.cnki.issn0439-8114.2020.07.043.
李雅慧,劉志偉,戴習林. 2021. 生長滯緩與正常羅氏沼蝦轉錄組差異分析[J]. 基因組學與應用生物學,40(1):89-100. [Li Y H,Liu Z W,Dai X L. 2021. Transcriptome analysis of growth retardation and normal Macrobrachium rosenbergii[J]. Genomics and Applied Biology,40(1):89-100.] doi:10.13417/j.gab.040.000089.
林明德. 2019. 基于轉錄組測序對雜交石斑魚及其母本褐點石斑魚的比較分析[D]. 湛江:廣東海洋大學. [Lin M D. 2019. Comparative analysis of hybrid grouper (Epinephelus fuscoguttatus ♀×E. Polyphekadion ♂) and its female parent tiger grouper(E. Fuscoguttatus) based on transcriptome sequencing[D]. Zhanjiang: Guangdong Ocean University.] doi:10.27788/d.cnki.ggdhy.2019.000082.
劉思嘉,田菲,張存芳,喬志剛,趙凱. 2018. 鯉在低溫脅迫下肝胰腺轉錄組測序分析[J]. 生物技術通報,34(11):168-178. [Liu S J,Tian F,Zhang C F,Qiao Z G,Zhao K. 2018. Transcriptome sequencing and analysis of hepatopancreas from carps under cold stress[J]. Biotechnology Bulletin,34(11):168-178.] doi:10.13560/j.cnki.biotech.bull.1985.2018-0503.014C7B63-A0F3-407B-A447-0D6F456619EF
羅欽,李冬梅,黃敏敏,饒秋華,劉洋,翁伯琦,潘葳,羅土炎. 2020. 不同生長階段墨瑞鱈脂肪酸組成及主成分分析[J]. 核農學報,34(4):788-795. [Luo Q,Li D M,Huang M M,Rao Q H,Liu Y,Weng B Q,Pan W,Luo T Y. 2020. Fatty acid compositions and principal component analysis in muscle of Murray cod at different growth stages[J]. Journal of Nuclear Agricultural Sciences,34(4):788-795.] doi:10.11869/j.issn.100-8551.2020.04.0788.
羅志嘉,李瀟,曾丹,王佩,彭娜,王曉清. 2019. 角鱉RNA-seq轉錄組分析及生長相關基因篩選[J]. 基因組學與應用生物學,38(4):1480-1487. [Luo Z J,Li X,Zeng D,Wang P,Peng N,Wang X Q. 2019. RNA-seq transcriptome analysis and growth related gene screening of Apalone spinifera[J]. Genomics and Applied Biology,38(4):1480-1487.] doi:10.13417/j.gab.038.001480.
孟瑋,徐開達,李振華,史會來,周永東. 2021. 急性鹽度脅迫對日本黃姑魚肌肉組織轉錄組的影響[J]. 水產學報,45(5):649-660. [Meng W,Xu K D,Li Z H,Shi H L,Zhou Y D. 2021. Transcriptome analysis of Nibea japonica under acute salinity stress[J]. Journal of Fisheries of China,45(5):649-660.] doi:10.11964/jfc.20200612317.
彭金英,伊成器. 2021. 蓬勃發展的表觀轉錄組學[J]. 中國生物化學與分子生物學報,37(4):407-418. [Peng J Y,Yi C Q. 2021. Flourishing epitranscriptomics research[J]. Chinese Journal of Biochemistry and Molecular Biology,37(4):407-418.] doi:10.13865/j.cnki.cjbmb.2021.03.1076.
饒秋華,劉洋,張志燈,羅欽,羅土炎. 2020. 加快墨瑞鱈新興水產養殖業發展的對策[J]. 科技導報,38(15):52-58. [Rao Q H,Liu Y,Zhang Z D,Luo Q,Luo T Y. 2020. Countermeasures for speeding up the emerging Murray cod (Maccullochella peelii) industry development[J]. Science & Technology Review,38(15):52-58.] doi:10.3981/j.issn.1000-7857.2020.15.006.
石軍,褚武英,張建社. 2013. 魚類肌肉生長分化與基因表達調控[J]. 水生生物學報,37(6):1145-1152. [Shi J,Chu W Y,Zhang J S. 2013. Muscle growth,differentiation and gene expression regulation in fish[J]. Acta Hydrobiologica Sinica,37(6):1145-1152.] doi:10.7541/2013.155.
宋尚橋,馬圍圍,張超龍,曾素先,孫翠翠,李鑫,嚴瑾,黎宗強. 2020. 基于轉錄組測序生物信息學分析的研究進展[J]. 中國畜牧獸醫,47(2):392-398. [Song S Q,Ma W W,Zhang C L,Zeng S X,Sun C C,Li X,Yan J,Li Z Q. 2020. Advances in bioinformatics analysis based on transcriptome sequencing[J]. China Animal Husbandry & Veterinary Medicine,47(2):392-398.] doi:10.16431/j.cnki. 1671-7236.2020.02.009.
王登東,楊玉鵬,鄭樂云,孫愷輝,何家瑞,王崇偉,鄧賢銘,李水生,張勇,林浩然. 2019. 云龍石斑魚生長優勢的轉錄組研究[J]. 海南熱帶海洋學院學報,26(2):1-8. [Wang D D,Yang Y P,Zheng L Y,Sun K H,He J R,Wang C W,Deng X M,Li S S,Zhang Y,Lin H R. 2019. Transcriptomic studies of a novel hybrid“Yunlong grouper”on the growth superiorities[J]. Journal of Qiongzhou University,26(2):1-8.] doi:10.13307/j.issn.2096-3122.2019.02.01.
王紅明,丁雪婧,陳儉,宋守鋼,譚北平,章雙. 2021. 飼料中添加甘露寡糖對珍珠龍膽石斑魚生長性能、血清免疫指標、轉錄組及腸道菌群的影響[J]. 動物營養學報,33(12):6982-6998. [Wang H M,Ding X J,Chen J,Song S G,Tan B P,Zhang S. 2021. Effects of dietary mannan-oligosaccharides on growth performance,serum immune indices,transcriptome and intestinal microflora of Epinephelus fuscoguttatus ♀×Epinephelus lanceolatus ♂[J]. Chinese Journal of Animal Nutrition,33(12):6982-6998.] doi:10.3969/j.issn.1006-267x.2021.12.039.014C7B63-A0F3-407B-A447-0D6F456619EF
嚴璐琪,郭香,巫旗生,祁劍飛,寧岳,王曉清,曾志南. 2016. 葡萄牙牡蠣外套膜轉錄組測序及殼色基因挖掘[J]. 湖南農業大學學報(自然科學版),42(4):409-414. [Yan L Q,Guo X,Wu Q S,Qi J F,Ning Y,Wang X Q,Zeng Z N. 2016. Transcriptome analysis and shell gene mining on Crassostrea angulata[J]. Journal of Hunan Agricultural University(Natural Sciences),42(4):409-414.] doi:10. 13331/j.cnki.jhau.2016.04.012.
張毓霞,石戈,王日昕,宮延斌,范美華,廖智. 2018. 大彈涂魚皮膚轉錄組測序及抗菌肽基因分析[J]. 生命科學研究,22(1):26-35. [Zhang Y X,Shi G,Wang R X,Gong Y B,Fan M H,Liao Z. 2018. Transcriptome of Boleophthalmus pectinirostris skin and analysis of antimicrobial peptide related unigenes[J]. Life Science Research,22(1):26-35.] doi:10.16605/j.cnki.1007-7847.2018.01.005.
趙彥花,區又君,溫久福,李加兒,周慧. 2019. 基于轉錄組測序技術的黃唇魚SSR分子標記篩選[J]. 南方農業學報,50(9):2078-2087. [Zhao Y H,Ou Y J,Wen J F,Li J E,Zhou H. 2019. Development of SSR markers in Bahaba flavolabiata by transcriptome sequencing[J]. Journal of Southern Agriculture,50(9):2078-2087.] doi:10.3969/j.issn.2095-1191.2019.09.26.
Dowming G,Litvak M. 2001. The effect of light intensity and spectrum on the incidence of first feeding by larval haddock[J]. Journal of Fish Biology,59(6):1566-78. doi:10.1111/j.1095-8649.2001.tb00221.x.
Forbes J,Watts R J,Robinson W A,Baumgartner L J,McGuffie P,Cameron L M,Crook D A. 2016. Assessment of stocking effectiveness for Murray cod (Maccullochella peelii) and golden perch (Macquaria ambigua) in ri-vers and impoundments of south-eastern Australia[J]. Marine and Freshwater Research,67(10):1410-1419. doi:10.1071/MF15230.
Francis D S,Cleveland B J,Jones P L,Turchini G M,Conlan J A. 2019. Effects of PUFA-enriched Artemia on the early growth and fatty acid composition of Murray cod larvae[J]. Aquaculture,513:734362. doi:10.1016/j.aquaculture.2019.734362.
Loughnan S R,Baranski M D,Robinson N A,Jones P L P,Burridge C P. 2010. Microsatellite loci for studies of wild and hatchery Australian Murray cod Maccullochella peelii peelii(Percichthyidae)[J]. Molecular Ecology Notes,4(3):382-384. doi:10.1111/j.1471-8286.2004.00660.x.
Zhang J H,Shen Y B,Xu X Y,Dai Y F,Li J L. 2020. Transcriptome analysis of the liver and muscle tissues of black carp (Mylopharyngodon piceus) of different growth rates[J]. Marine Biotechnology,22:706-716. doi:10.1007/ s10126-020-09994-z.
(責任編輯 蘭宗寶)014C7B63-A0F3-407B-A447-0D6F456619EF

