普通煙草熱激轉錄因子家族的全基因組鑒定與分析
郭 存,王 奇,李曉旭,張震彪,文利超,鄧智超,初雨蒙,劉 濤,崔萌萌,郭永峰*
普通煙草熱激轉錄因子家族的全基因組鑒定與分析
郭 存1,2,王 奇1,李曉旭3,張震彪1,2,文利超1,2,鄧智超1,2,初雨蒙1,劉 濤1,2,崔萌萌1,郭永峰1*
(1.中國農業科學院煙草研究所,煙草行業基因資源利用重點實驗室,青島 266101;2.中國農業科學院研究生院,北京 100081;3.湖南中煙工業有限責任公司技術中心,長沙 410007)
熱激轉錄因子(heat shock transcription factor, Hsf)家族成員廣泛參與植物生長發育和非生物脅迫的調控。本研究應用生物信息學和比較基因組學方法從普通煙草中鑒定了49個Hsf家族成員,發現這些Hsf家族成員均具有較為保守的寡聚化結構域。通過系統進化分析可將煙草和擬南芥的Hsf家族成員劃分成3個分支與14個亞家族。共線性分析顯示煙草Hsf基因與雙子葉植物(擬南芥,番茄和葡萄)基因組成共線基因對的數量要大于與單子葉植物(水稻和玉米)基因組成共線基因對的數量。全基因組復制預測共有12個基因可組成7條復制基因對。啟動子分析發現基因啟動子區域存在著多個與非生物脅迫相關的順式作用元件。表達模式分析證實家族成員在不同組織的表達具有差異,并且大多數家族成員可響應熱和干旱脅迫。研究結果可為煙草Hsf轉錄因子家族成員的功能研究奠定生物信息學基礎。
普通煙草;熱激轉錄因子;共線性;全基因組復制;表達模式
高溫是影響植物生長的一種非生物脅迫,高溫環境下作物產量和品質都會受到嚴重影響。熱激轉錄因子(Heat shock transcription factor, Hsf)是植物體內一類能夠調節高溫反應的蛋白[1-2]。有研究表明,在高溫環境下,Hsf家族成員通過與下游相關基因的熱激元件結合進而激活其轉錄,從而提高植物對高溫的抗性[3-4]。Hsf家族成員含有較為保守的寡聚化結構域,根據寡聚化結構的特征可將Hsf家族劃分成3個亞支(A、B和C)[5-7]。
目前,熱激轉錄因子已在多種植物基因組中發現,在擬南芥[5]、水稻[8]、馬鈴薯[9]、番茄[10]、小麥[11]、玉米[12]和葡萄[13]中分別鑒定出了21、25、27、24、82、25和19個Hsf成員。研究發現,擬南芥大部分與高溫脅迫相關的基因均受到A分支Hsf家族成員的調控,其中包括、、和。在三重突變體中,高溫響應基因的轉錄水平顯著降低[4,14]。此外,與野生型相比,四重突變體表現出生長遲緩且對溫度更敏感的表型。在番茄和擬南芥中分別過表達和基因可以增強植物耐旱性和耐熱性[15-16]。此外在煙草中異源過表達或基因都能夠提高煙草的耐熱性[17-18]。據報道,和除了在植物的耐熱性方面起作用外,還參與調控植物耐缺氧性和氧化應激耐受性反應[19-20]。在高鹽和干旱環境下,基因的表達量顯著上調,并且在ABA介導的鹽、干旱和高溫脅迫中起正向調節作用[21]。
煙草是一種重要的經濟作物,高溫、干旱等非生物脅迫影響其正常生長發育,并誘導多種根莖類病害發生,進而影響煙葉的產量和品質。Hsf轉錄因子參與多種非生物脅迫的響應,而煙草中Hsf家族成員信息還未見報道。本研究首次從煙草基因組序列中鑒定出49個Hsf轉錄因子家族成員,并對其進行了系統進化、保守基序、共線性、復制事件、順式作用元件和表達模式等分析,為進一步解析基因在煙草非生物脅迫響應過程中的功能奠定基礎。
1 材料與方法
1.1 材料與儀器試劑
本試驗所用的材料為普通煙草品種K326,保存于煙草行業煙草基因資源利用重點實驗室。K326種子經過滅菌后均勻地撒在MS培養基中,在光照培養箱中培養8 周(光周期:16 h光照/8 h黑暗;25 ℃)。挑選長勢一致的煙苗,一部分轉移到37 ℃培養箱中進行高溫處理,另一部分吸干根部水分后轉移到濾紙上做干旱處理。在處理0、3、6 h時對煙苗整株取樣,并于液氮中保存。所有樣品取3次生物學重復。
本研究用的RNA提取試劑盒購自北京康為世紀公司,反轉錄試劑盒與SYBR?Green Premix Pro Taq HS qPCR Kit熒光定量試劑盒購自湖南艾科瑞生物工程有限公司。熒光定量儀器Roche LightCycler 480購自美國羅氏公司。
1.2 方法和條件
1.2.1 NtHsf轉錄因子家族成員的鑒定和序列分析 從擬南芥基因組數據庫(https://www.arabidopsis.org/)中下載已報道的AtHsf家族成員的蛋白序列[22],并利用MAFFT工具在默認參數下進行序列比對,利用HMMER工具基于比對結果建立HMM文件。普通煙草K326蛋白序列數據庫從茄科基因組數據庫(https://solgenomics.net/organism,Sol Genomics Network)中下載得到,并用建立的HMM文件在默認參數下對數據庫進行檢索,初步得到煙草NtHsf序列。Pfam數據庫(https://pfam.xfam.org/)被用來分析所鑒定的蛋白序列,并去除不含有寡聚化結構域的序列。利用ProtParam工具(https://web.expasy.org/protparam/)對新鑒定的NtHsf序列進行理化性質分析。
1.2.2 NtHsf轉錄因子家族成員的系統進化分析 利用MAFFT在默認參數下對已報道的擬南芥AtHsf成員和新鑒定的煙草NtHsf成員進行蛋白全長序列的比對[23]。基于比對結果,利用MGEA X進行鄰接樹的構建。構建鄰接樹的參數設置如下:采用Poisson Model和Pairwise Deletion,Bootstrap檢驗設為1000次[24]。
1.2.3的共線性及復制事件分析 按照先前的研究,分別下載普通煙草和其他5個物種(擬南芥、番茄、普通、水稻和玉米)的基因組注釋文件,利用McScanX在默認參數下進行共線性分析,使用TBtools將結果進行可視化展示[25]。使用ColinearScan軟件對煙草NtHsf基因的復制事件進行預測和可視化[26]。
1.2.4 NtHsf的寡聚化結構域序列分析 手動提取NtHsf的寡聚化結構域的序列,并利用MAFFT在默認參數對其進行比對與可視化[23]。
1.2.5 啟動子分析 選取基因上游2000 bp區域[27],并提交到PlantCare網站(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)進行基因啟動子順式作用元件的鑒定[28]。
1.2.6 轉錄組數據分析及可視化 從NCBI的GEO數據庫中下載得到普通煙草K326的根、莖和莖尖組織的轉錄組數據(https://www.ncbi.nlm.nih.gov,登錄號為GSE95717)[29]。從下載的數據中提取煙草基因的表達情況,利用TBtools軟件中的HeatMap程序對基因的表達量進行標準化分析和可視化展示[25]。
1.2.7 煙草樣品RNA的提取和反轉錄 利用康為世紀Ultrapure RNA Kit進行樣品總RNA的提取,cDNA第一條鏈的合成在說明書參照下進行,并將反轉錄的cDNA保存在?20 ℃待用。
1.2.8 熒光定量PCR 熒光定量PCR在Roche LightCycler 480上進行,試驗體系設置如下:2× SYBR?Green Pro Taq HS Premix 10mL, cDNA 1mL, Primer-F 0.4mL,Primer-R 0.4mL,無菌水8.2mL。PCR程序設置如下:95 ℃ 30s;95 ℃ 5s,60℃ 30s,40個循環。煙草基因(GenBank No. L18908)為內參基因[30],每個樣品進行3次技術重復。試驗數據采用2-△△Ct法進行分析[31]。本試驗所用到的引物如表1所示。
2 結 果
2.1 煙草NtHsf家族成員的鑒定和理化分析
本研究在煙草基因組中鑒定了49個家族成員(表2),并按照基因在染色體上的物理位置將新鑒定的煙草基因進行命名(~)。新鑒定的49個基因中有24個成員錨定在15條煙草染色體上。利用ProtParam工具對NtHsf成員進行理化性質分析發現,49個NtHsf成員在理化性質上存在較大差異,例如其氨基酸數目在146~504之間,相對分子量大小在16?521.7~55?842.1 Da之間,等電點范圍為4.45~10.12。
2.2 煙草NtHsf家族成員的進化分析
為了進一步研究煙草中NtHsf轉錄因子成員的進化歷程,將新鑒定的49個煙草NtHsf成員和已報道的21個擬南芥AtHsf成員進行系統進化分析。如圖1所示,這些Hsf家族成員可劃分為A、B和C三個分支,其中A和B分支又可劃分為A1-A9和B1-B3亞家族。值得注意的是,每個亞家族中均含有來自擬南芥和煙草的Hsf家族成員,說明擬南芥和煙草的物種分化時間要晚于Hsf轉錄因子的分化時間。并且在各個亞家族中,煙草NtHsf家族成員要顯著多于擬南芥Hsf家族成員,例如A1、A4、A6、B1、B2、B3等亞家族,暗示在這兩個物種分化之后,各個亞家族的NtHsf成員可能又經歷了復制事件,導致了基因的擴張。

表1 熒光定量PCR引物信息
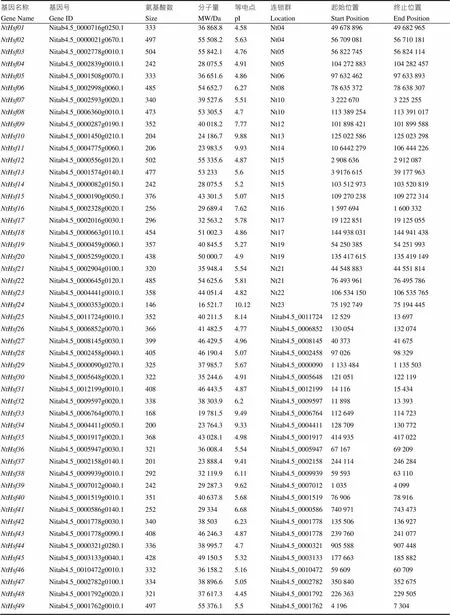
表2 普通煙草NtHsf轉錄因子家族成員鑒定及理化性質分析
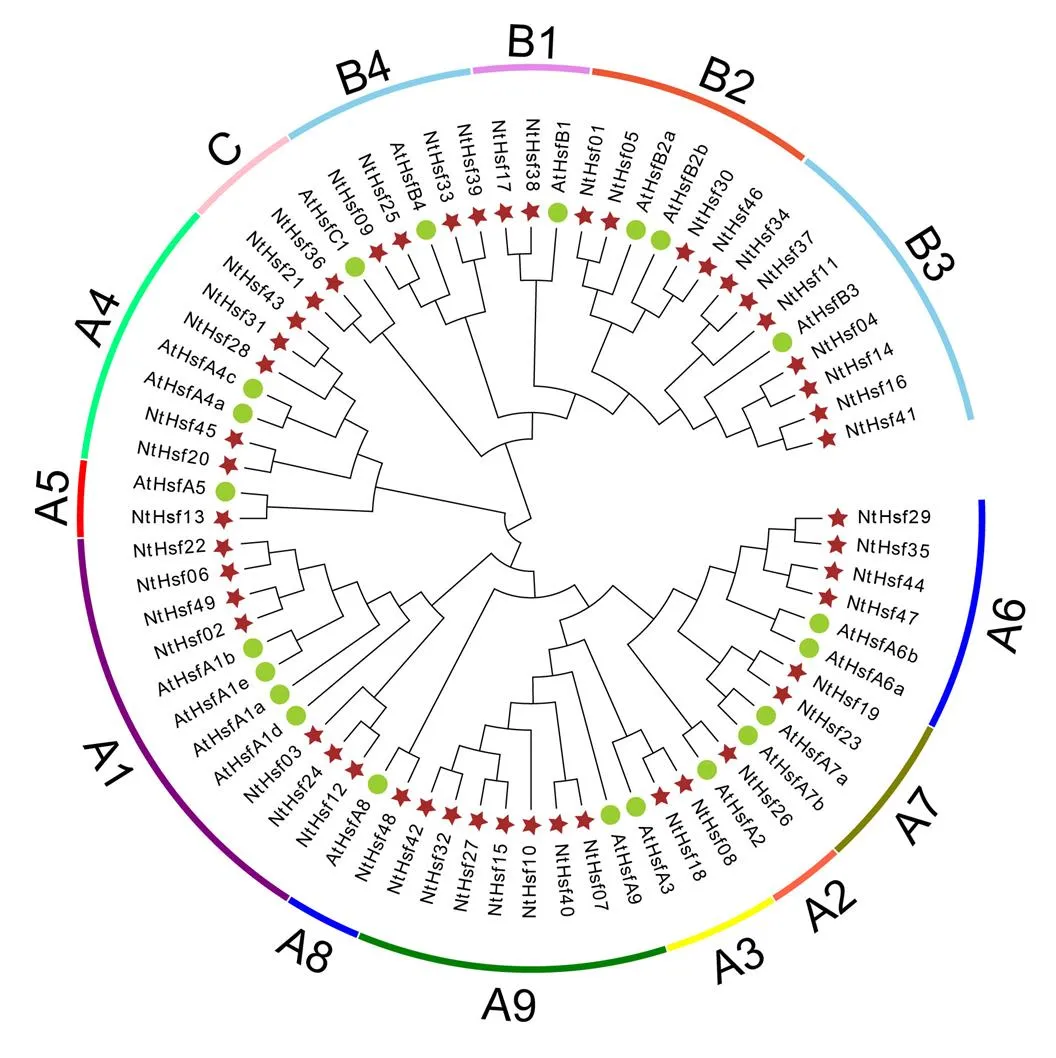
圖1 煙草和擬南芥Hsf家族成員的系統進化分析
2.3 煙草NtHsf家族成員的寡聚化結構域分析
Hsf家族成員的寡聚化結構域由螺旋卷曲結構和α螺旋結構組成,其包含了大量的疏水氨基酸殘基[1]。為了驗證煙草NtHsf家族成員的保守結構域,選取NtHsf家族成員的寡聚化結構域進行多序列比對并進行可視化。如圖2所示,新鑒定的NtHsf家族成員均具有較為保守的寡聚化結構域,由HR-A和HR-B組成,其中A類Hsf成員有30個,C類2個,B類17個。
2.4 煙草NtHsf基因的共線性分析
為進一步了解煙草家族成員的系統進化關系,分析了煙草與其他5個物種(擬南芥、番茄、葡萄、水稻和玉米)的共線性關系。如圖3所示,灰色線條代表所有具有共線關系的基因,基因則用紅色線條標記。其中,煙草中共22個基因與番茄基因成共線性關系,其次是葡萄(16個基因)、擬南芥(9個基因)、水稻(4個基因)和玉米(3個基因)。此外煙草基因與番茄、葡萄、擬南芥、水稻和玉米組成共線基因對的數量分別為30、19、11、11和7。值得注意的是,煙草中共有2個基因(和)與這5個物種的基因組成共線對,表明這2個基因可能在這些物種分化之前就已經存在。

圖2 煙草NtHsf家族成員的寡聚化結構域序列比對圖
2.5 煙草NtHsf基因家族的全基因組復制事件
系統進化分析暗示煙草基因家族可能發生了全基因組復制事件,隨后利用McScan X對煙草家族成員進行復制分析。如圖4所示,灰色線條代表煙草基因組中所有的復制基因對,而紅色線條表示基因。結果顯示,共有12個基因組成7條復制基因對,暗示這些基因可能由全基因組復制事件產生,也說明全基因組復制事件在煙草基因家族擴張過程中有一定的貢獻。
2.6 煙草NtHsf家族成員的啟動子分析
利用PlantCARE網站對的啟動子元件進行探究,并選取12個順式作用元件進行分析。如圖5所示,大多數基因啟動子中存在激素響應元件,例如響應脫落酸的元件ABRE、響應乙烯的ERE元件、響應水楊酸的元件TCA-element和響應茉莉酸甲酯的CGTCA-motif元件。值得注意的是,響應生長素的AuxRR-core元件僅在個別基因的啟動子區域中檢測到,例如、、、、、和。此外,厭氧誘導元件(ARE)、損傷響應元件(WUN-motif)、脅迫響應元件(TC-rich repeats)、WRKY轉錄因子結合位點(W-box)、MYB轉錄因子結合位點(MBS)和低溫響應元件(LTR)等6種脅迫響應元件也被檢測到廣泛地存在啟動子區域中,暗示這些基因可能參與到不同脅迫的響應過程中。同時,在一些基因的啟動子區域檢測出與分生組織發育相關的順式作用元件(CAT-box)。
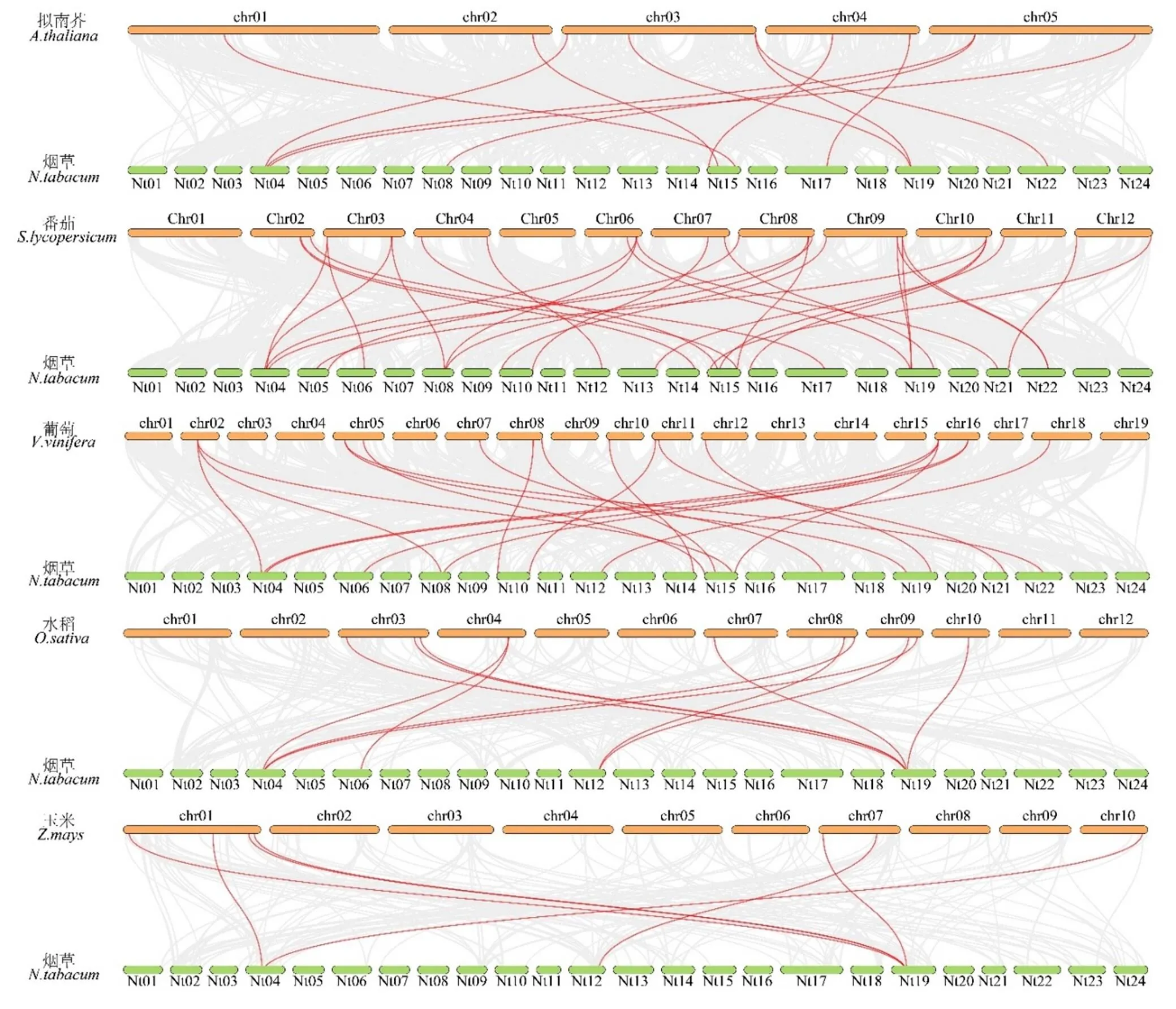
圖3 煙草和其他5個物種Hsf基因的共線性分析
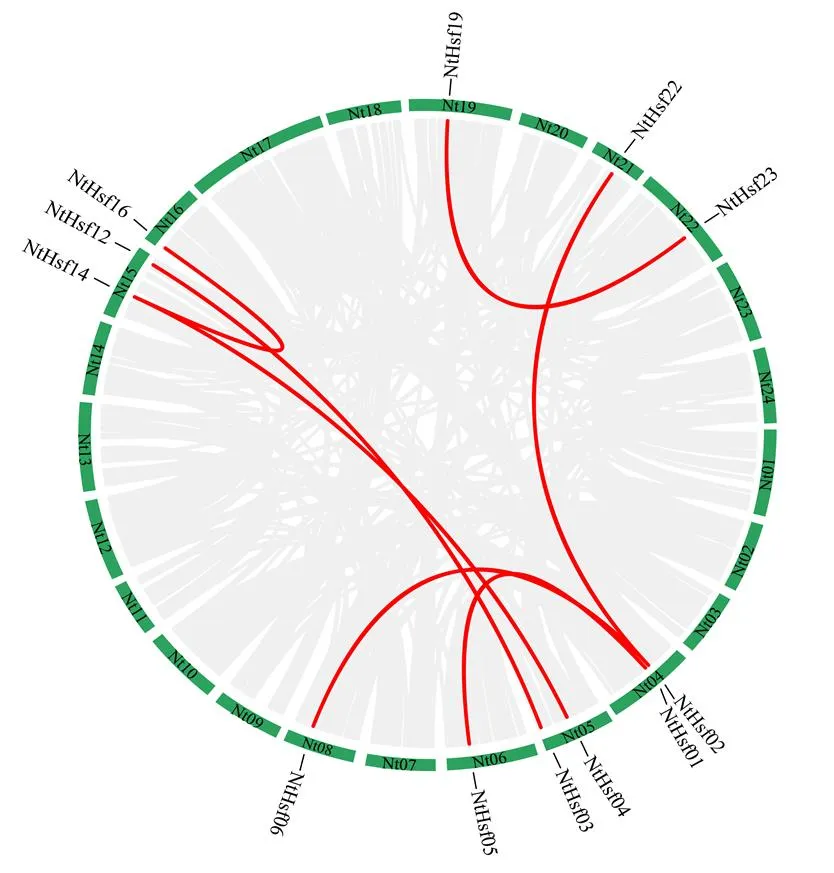
圖4 煙草NtHsf基因家族的全基因組復制分析
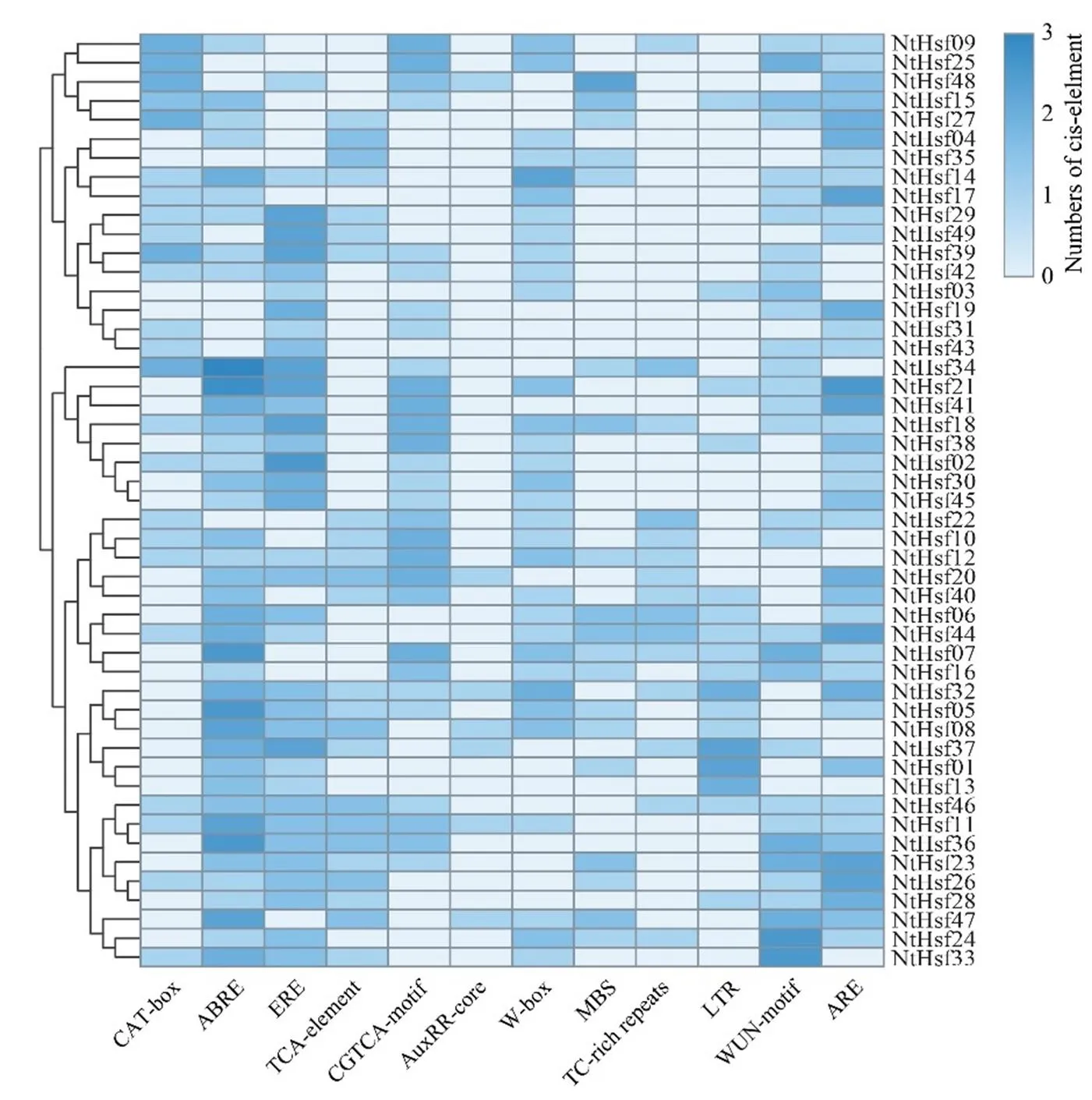
圖5 煙草NtHsf基因啟動子區順式作用元件分析
2.7 煙草NtHsf家族成員在不同組織中的表達分析
利用已發表的普通煙草轉錄組數據庫(https://www.ncbi.nlm.nih.gov,登錄號為GSE95717)對基因的表達模式進行分析。如圖6所示,家族成員在煙草根、莖和莖尖組織中的表達差異較大。多數基因在3個煙草組織中均檢測到表達,如、和等,然而一些基因并未在這3個組織中檢測到表達量,如、、和。進一步研究發現個別基因的表達模式具有組織特異性,例如、和在煙草的根和莖組織中特異高表達,在根和莖中檢測出表達量,而在莖尖中卻不表達。
2.8 煙草NtHsf家族成員在熱和干旱脅迫下的表達模式
利用熒光定量PCR的方法對煙草NtHsf家族成員在熱和干旱脅迫下的表達模式進行分析。如圖7所示,一些基因可以被高溫誘導表達,尤其是、、、和基因的表達量在高溫處理3 h后顯著增加(>10倍),而這些基因的表達水平在處理6 h后均下調,說明這些基因在高溫脅迫下的表達具有時間特異性。其余基因在高溫脅迫下的表達量均呈現出不同程度的上調表達,例如、和等基因。
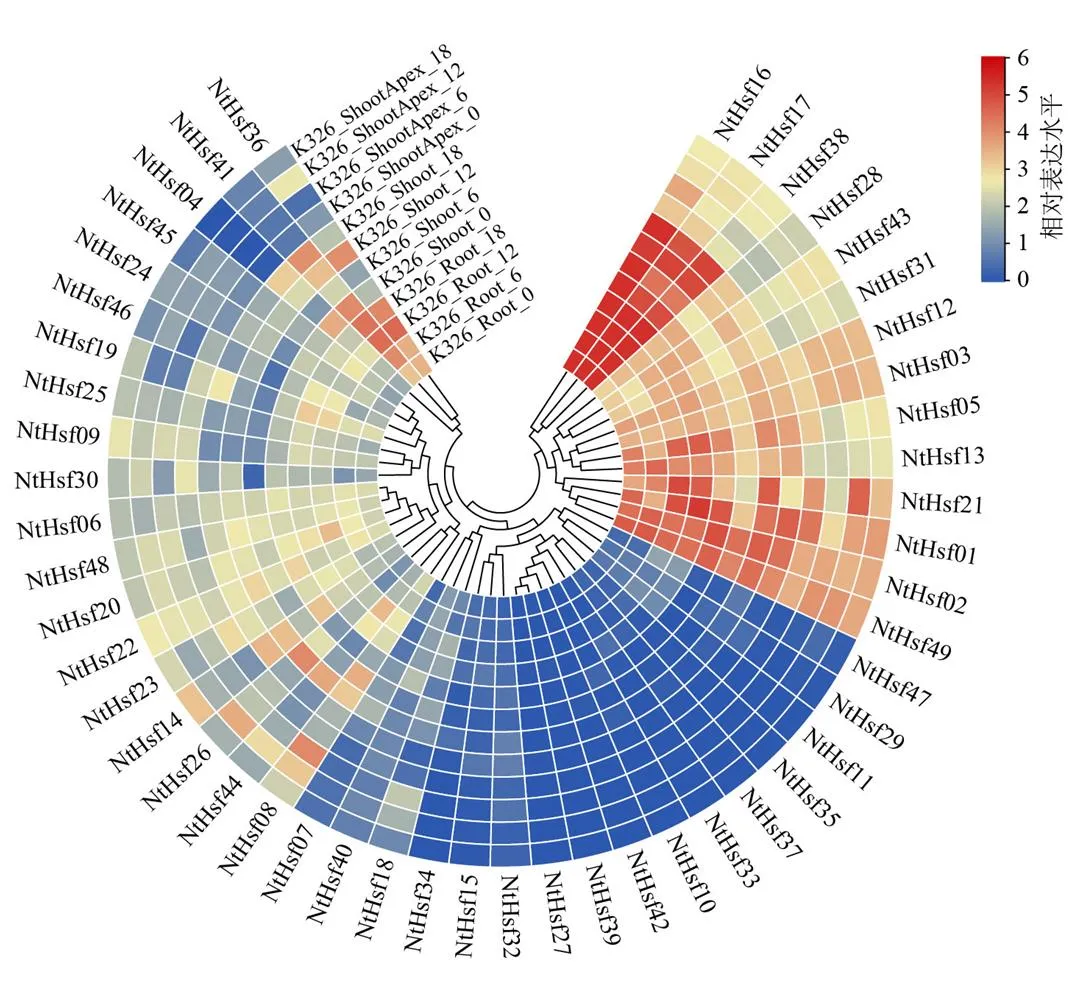
圖6 煙草NtHsf家族成員的組織表達模式分析
圖8所示的為基因在干旱處理下的表達模式,結果發現和在干旱處理3 h后的表達量達到最高峰,分別為對照的6.5倍和8.4倍。且這兩個基因在干旱處理下的表達模式具有時間特異性,其表達量在處理3 h后達到頂峰,然后在處理6 h后下降,、、和等基因的表達模式也呈現出類似的趨勢。
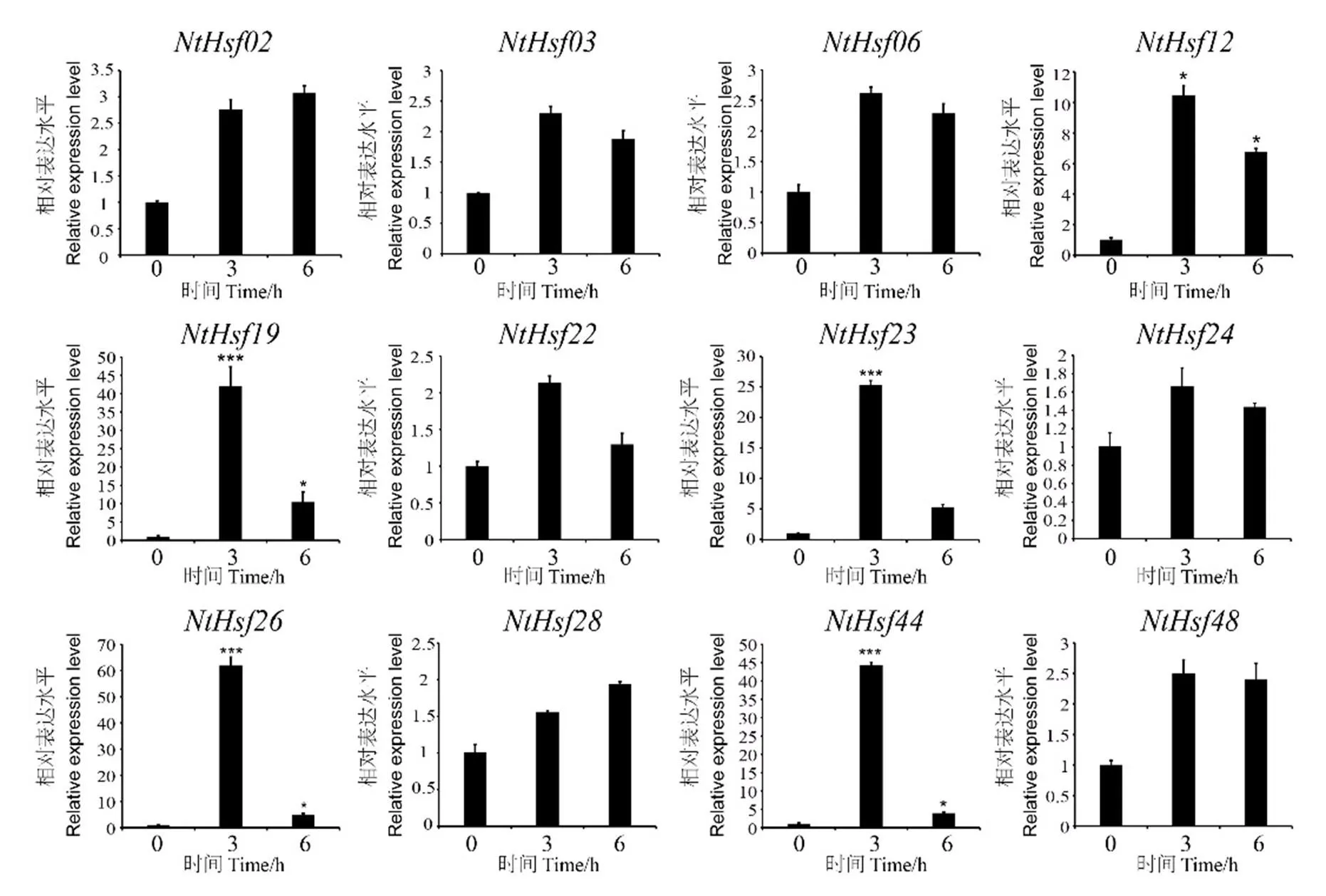
圖7 普通煙草NtHsf家族成員在高溫處理下的表達模式分析
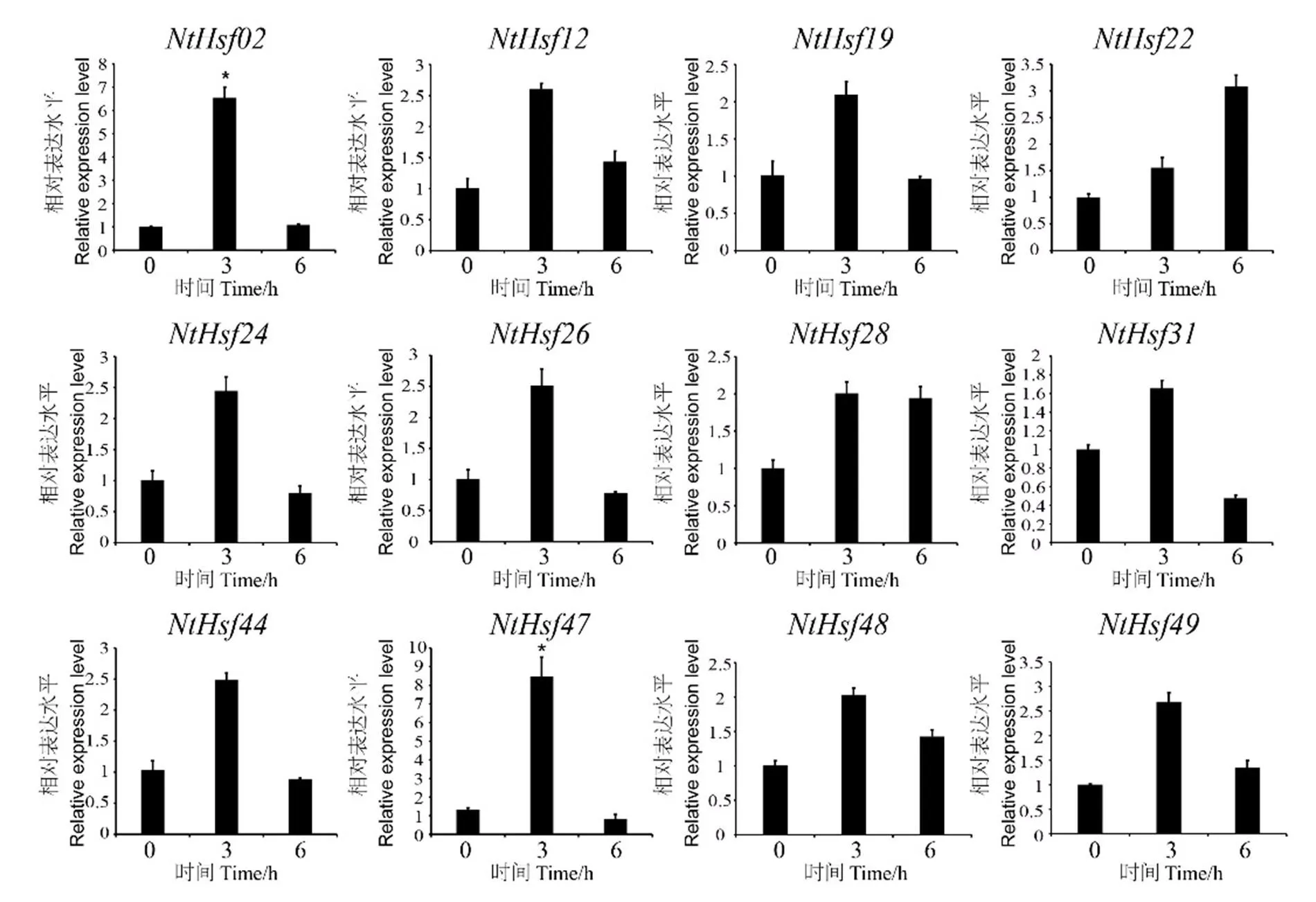
圖8 普通煙草NtHsf家族成員在干旱處理下的表達模式分析
3 討 論
煙草作為我國重要的經濟作物,時常會受到高溫等環境因素的脅迫,對煙葉的品質和生產造成負面影響。熱激轉錄因子被報道與植物的耐熱性和抗旱性有緊密的聯系,本研究采用比較基因組學的方法對煙草基因組中的NtHsf熱激轉錄因子進行了鑒定。并從系統進化、共線性、復制事件、順式作用元件和表達模式等方面對新鑒定的NtHsf轉錄因子進行分析,為之后解析煙草NtHsf家族成員的生物學功能和調控機制提供基礎。
本研究共鑒定得到49個煙草NtHsf轉錄因子,進一步分析發現其均含有較為保守的寡聚化結構域。系統進化分析表明煙草和擬南芥的Hsf家族成員可劃分為A、B和C三個分支和14個亞家族。有意思的是,每個亞家族中的NtHsf成員均比AtHsf成員多,暗示煙草中的NtHsf成員可能進行一次或多次復制事件。隨后的全基因組復制事件顯示共有12個基因經歷了復制過程,暗示這12個基因可能具有類似功能,也從側面證明復制事件在煙草NtHsf成員的擴張過程中起著重要作用。
系統進化和共線性的綜合分析是探索同源基因保守功能的有效手段之一,本研究通過分析煙草與擬南芥Hsf成員之間的系統進化和共線性關系,初步推測和探討NtHsf成員的功能。在亞家族A1中,和與聚在一起,并且分別與這兩個煙草Hsf成員組成共線基因對,說明和與基因直系同源。進一步分析發現和組成一對復制基因對,暗示其功能可能相似。此外,在番茄中過表達可增強番茄幼苗的耐熱性[15],而且表達模式發現在高溫脅迫下,和基因表達上調,暗示這兩個煙草基因可能參與到煙草的高溫脅迫響應中。值得注意的是,基因在干旱脅迫下的表達量也顯著上調,暗示該基因可能參與到煙草多脅迫響應調控中。
此外,被報道通過調控熱激反應相關基因的表達來提高擬南芥耐熱性[16]。在本研究中,與聚在同一亞家族中,并且其編碼基因預測具有共線性關系,說明其直系同源。有趣的是,在的啟動子上發現了多個與脅迫相關的順式作用元件,例如ABRE、G-box、W-box和MYB,且其轉錄本在高溫脅迫下明顯增加,暗示可能參與到煙草的高溫脅迫過程。此外,也與聚在同一亞家族中,且其編碼基因來自同一復制事件。在高溫脅迫下的表達量也有所增加,暗示可能與在煙草響應高溫脅迫方面具有相似的功能。
4 結 論
本研究利用生物信息學等方法在普通煙草基因組中鑒定到49個熱激轉錄因子基因,它們編碼的蛋白均含有較為保守的寡聚化結構域。此外,煙草NtHsf成員和擬南芥AtHsf成員被劃分成3個亞支和14個亞家族。共線性分析表明,基因和基因形成11對同源基因對,暗示這些共線基因可能具有保守的功能。啟動子分析發現大量脅迫響應相關的順式作用元件存在NtHsf成員的啟動子上,并且多數基因能夠響應高溫和干旱脅迫,說明家族成員可能在煙草的高溫和干旱脅迫過程中具有重要的功能。
[1] SCHARF K D, BERBERICH T, EBERSBERGER I, et al. The plant heat stress transcription factor (Hsf) family: structure, function and evolution[J]. Biochimica et Biophysica Acta (BBA)-Gene Regulatory Mechanisms, 2012, 1819(2): 104-119.
[2] VON KOSKULL-D?RING P, SCHARF K D, NOVER L. The diversity of plant heat stress transcription factors[J]. Trends in Plant Science, 2007, 12(10): 452-457.
[3] IKEDA M, MITSUDA N, OHME-TAKAGI M.HsfB1 and HsfB2b act as repressors of the expression of heat-inducible Hsfs but positively regulate the acquired thermotolerance[J]. Plant Physiology, 2011, 157(3): 1243-1254.
[4] YOSHIDA T, OHAMA N, NAKAJIMA J, et al.HsfA1 transcription factors function as the main positive regulators in heat shock-responsive gene expression[J]. Molecular Genetics and Genomics, 2011, 286(5): 321-332.
[5] GUO M, LIU JH, MA X, et al. The plant heat stress transcription factors (HSFs): structure, regulation, and function in response to abiotic stresses[J]. Frontiers in Plant Science, 2016, 7: 114-121.
[6] KOTAK S, PORT M, GANGULI A, et al. Characterization of C-terminal domains ofheat stress transcription factors (Hsfs) and identification of a new signature combination of plant class A Hsfs with AHA and NES motifs essential for activator function and intracellular localization[J]. The Plant Journal, 2004, 39(1): 98-112.
[7] YE J, YANG X, HU G, et al. Genome-wide investigation of heat shock transcription factor family in wheat (L.) and possible roles in anther development[J]. International Journal of Molecular Sciences, 2020, 21(2): 608-619.
[8] WANG C, ZHANG Q, SHOU H X. Identification and expression analysis ofin rice[J]. Journal of Zhejiang University Science B, 2009, 10(4): 291-300.
[9] TANG R, ZHU W, SONG X, et al. Genome-wide identification and function analyses of heat shock transcription factors in potato[J]. Frontiers in Plant Science, 2016, 7: 490-508.
[10] YANG X, ZHU W, ZHANG H, et al. Heat shock factors in tomatoes: genome-wide identification, phylogenetic analysis and expression profiling under development and heat stress[J]. PeerJ, 2016, 4: e1961.
[11] DUAN S, LIU B, ZHANG Y, et al. Genome-wide identification and abiotic stress-responsive pattern of heat shock transcription factor family inL.[J]. BMC Genomics, 2019, 20(1): 1-20.
[12] LIN Y X, JIANG H Y, CHU Z X, et al. Genome-wide identification, classification and analysis of heat shock transcription factor family in maize[J]. BMC Genomics, 2011, 12(1): 1-14.
[13] GUOTIAN L, FENGMEI C, YI W, et al. Genome-wide identification and classification of HSF family in grape, and their transcriptional analysis under heat acclimation and heat stress[J]. Horticultural Plant Journal, 2018, 4(4): 133-143.
[14] LIU HC, LIAO HT, CHARNG YY. The role of class A1 heat shock factors (HSFA1s) in response to heat and other stresses in[J]. Plant, Cell & Environment, 2011, 34(5): 738-751.
[15] LI H Y, CHANG C S, LU L S, et al. Over-expression ofheat shock factor gene () enhances chilling tolerance in transgenic tomato[J]. Botanical Bulletin of Academia Sinica, 2003, 44: 129-140.
[16] HIGASHI Y, OHAMA N, ISHIKAWA T, et al. HsfA1d, a protein identified via FOX hunting usingcDNAs improves heat tolerance by regulating heat-stress-responsive gene expression[J]. Molecular Plant, 2013, 6(2): 411-422.
[17] ZHU X, WANG Y, LIU Y, et al. Overexpression oftranscription factor fromimproved heat tolerance of transgenic tobacco[J]. PLoS One, 2018; 13(11): e0207277.
[18] XU L, GAO J, GUO L, et al. Heat shock protein 70 (HmHsp70) fromconfers thermotolerance to tobacco[J]. AMB Express, 2020; 10(1): 1-10.
[19] LI C, CHEN Q, GAO X, et al.modulates expression of stress responsive genes and enhances tolerance to heat and oxidative stress in[J]. Science in China Series C: Life Sciences, 2005, 48(6): 540-550.
[20] SCHRAMM F, LARKINDALE J, KIEHLMANN E, et al. A cascade of transcription factor DREB2A and heat stress transcription factor HsfA3 regulates the heat stress response of[J]. The Plant Journal, 2008, 53(2): 264-274.
[21] HUANG Y C, NIU C Y, YANG C R, et al. The heat stress factor HSFA6b connects ABA signaling and ABA-mediated heat responses[J]. Plant Physiology, 2016, 172(2): 1182-1199.
[22] 李曉旭,郭存,蒲文宣,等. 普通煙草WOX轉錄因子家族的全基因組鑒定及分析[J]. 中國煙草學報,2021,27(1):90-100.
LI X X, GUO C, PU W X, et al. Genome-wide identification and systemic analysis of WOX family genes in tobacco[J]. Acta Tabacaria Sinica, 2021, 27 (1): 90-100.
[23] KATOH K, STANDLEY D M. MAFFT multiple sequence alignment software version 7: improvements in performance and usability[J]. Molecular Biology and Evolution, 2013, 30(4): 772-780.
[24] KUMAR S, STECHER G, LI M, et al. MEGA X: molecular evolutionary genetics analysis across computing platforms[J]. Molecular Biology and Evolution, 2018, 35(6): 1547-1549.
[25] CHEN C, CHEN H, ZHANG Y, et al. TBtools: an integrative toolkit developed for interactive analyses of big biological data[J]. Molecular Plant, 2020, 13(8): 1194-1202.
[26] 李志遠,宋青松,劉江,等. 普通煙草脂氧合酶基因家族鑒定及表達模式分析[J]. 中國煙草科學,2021,42(5):86-94.
LI Z Y, SONG Q S, LIU J, et al. Identification and expression analysis of the lipoxygenase gene family in tobacco (L.) [J]. Chinese Tobacco Science, 2021, 42(5): 86-94.
[27] GUO C, WANG Q, LI Z Y, et al. Bioinformatics and expression analysis ofgenes reveal their potential functions in flower abscission and stress response in tobacco (L.)[J]. Frontiers in Genetics, 2021, 12: 670794.
[28] LESCOT M, DéHAIS P, THIJS G, et al. PlantCARE, a database of plant-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences[J]. Nucleic Acids Research, 2002, 30(1): 325-327.
[29] EDWARDS K, FERNANDEZ-POZO N, DRAKE-STOWE K, et al. A reference genome forenables map-based cloning of homeologous loci implicated in nitrogen utilization efficiency[J]. BMC Genomics, 2017, 18(1): 1-14.
[30] LI Z, CHAO J, LI X, et al. Systematic analysis of the bZIP family in tobacco and functional characterization ofinvolvement in salt stress[J]. Agronomy, 2021, 11(1): 148-165.
[31] LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2?ΔΔCTmethod[J]. Methods, 2001, 25(4): 402-408.
Genome-wide Identification and Systemic Analysis of the Hsf Gene Family inL.
GUO Cun1,2, WANG Qi1, LI Xiaoxu3, ZHANG Zhenbiao1,2, WEN Lichao1,2, DENG Zhichao1,2, CHU Yumeng1, LIU Tao1,2, CUI Mengmeng1, GUO Yongfeng1*
(1. Key Laboratory of Tobacco Gene Resources, Tobacco Research Institute of CAAS, Qingdao 266101, China; 2. Graduate School of Chinese Academy of Agricultural Science, Beijing 100081, China; 3. Technology Center, China Tobacco Hunan Industrial Co., Ltd., Changsha 410007, China)
Heat shock transcription factor (Hsf) family members are widely involved in the regulation of plant development and abiotic stress responses. In the current study, 49 Hsf members were identified from the tobacco genome by bioinformatics and comparative genomics methods, all of which contained conserved oligomerization domains. The Hsf members from tobacco andwere categorized into 3 clades and 14 subfamilies according to the phylogenetic analysis. The number of collinear gene pairs betweenandgenes from dicots (, tomato and grape) was more than the collinear gene pairs with thegenes from monocots (rice and maize). Besides, a total of 12genes were predicted to have arisen from duplication events. Promoter analysis showed that there were a variety of-elements related to abiotic stress responses on thepromoters. The various expression patterns ofin different tissues were detected, and mostgenes were induced by heat and drought stress. The results presented by this study might provide insights for the further studies on NtHsf members.
tobacco; heat shock transcription factor; collinearity; duplication events; expression pattern
S572.01
A
1007-5119(2022)03-0047-10
10.13496/j.issn.1007-5119.2022.03.008
中國農業科學院科技創新工程(ASTIP-TRIC02);湖南中煙工業有限責任公司科技項目(KY2022YC0010);中央級公益性科研院所基本科研業務費專項(1610232022009)
郭 存(1994-),男,在讀博士研究生,主要從事煙草育種研究。E-mail:82101191076@caas.cn。
,E-mail:guoyongfeng@caas.cn
2021-12-07
2022-04-15

