飼料玉米品質分析的化學計量學方法
劉亞 史勇 師旭明 孫美樂 宋魚 仙鶴 姚國民



摘要:以飼料玉米為研究對象,采用化學計量學方法,利用全波段光譜數據建立玉米粗蛋白預測的簡單快速精準預測模型。結果表明:原始光譜經去趨勢算法預處理后,Rank-KS算法選擇校正集和預測集,使用偏最小二乘(Partial least square,PLS)方法進行建模,校正集和預測集的相關系數分別為0.991 5和0.981 3,校正集和預測集的均方根誤差分別為0.063 4和0.113 8。預測集的相對分析誤差RPD為5.02,大于評估閾值3.0。所建模型精度和穩定性較為理想,可滿足在線生成檢測的要求。
關鍵詞:化學計量學;飼料玉米;近紅外光譜;Rank-KS算法
中圖分類號:S816? ? 文獻標識碼:A? ? 文章編號:1674-1161(2022)03-0057-04
玉米是用途最為廣泛的飼料作物,可為家禽和牲畜提供多種生長所需的營養成分,被稱為“飼料之王”。粗蛋白含量是評價飼料玉米品質最常見和最重要的指標,傳統的粗蛋白含量檢測主要采用實驗室濕化學方法,存在檢測周期長、試劑種類多且用量大、操作繁瑣等缺點。近紅外光譜分析技術具有無損、快速、樣品前處理簡單、多組分同時在線檢測等優點,目前已被大中型飼料企業用于飼料玉米原材料驗收和成品出廠檢驗。近年來,近紅外光譜技術結合化學計量學方法所建立的光譜與待測組分屬性值關系模型技術不斷發展,簡單快速的分析預測模型可以顯著降低近紅外光譜分析儀器的造價,成為推廣近紅外光譜技術應用的一個重要方向。以飼料玉米為研究對象,結合化學計量學方法,利用全波段光譜數據建立玉米粗蛋白預測模型,以期為近紅外光譜技術在飼料品質的在線快速檢測提供依據。
1 實驗數據
實驗數據選用EVRI網站(http://eigenvector.com/data/Corn/index.html)公開的玉米近紅外光譜數據集。該數據集包含80個玉米籽粒樣本的近紅外光譜信息,以及相應的水分、脂肪、粗蛋白和淡粉含量的營養指標數據。本研究使用的玉米光譜數據和粗蛋白含量數據,光譜儀波長范圍均為1 100~2 498 nm,波長間隔為2 nm,共有700個波長點。玉米粗蛋白含量變幅7.65%~9.71%,平均值8.67%,標準差0.50,變異系數5.75%。
2 化學計量學方法
2.1 光譜數據預處理
為剔除光譜曲線中的噪聲,突出原始光譜中的有效信息,本研究分別采用歸一化變換、變量標準化變換(Standard normal variable,SNV)、多元散射校正(Multiplicative scattering correction,MSC)、導數處理、去趨勢算法對玉米原始近紅外光譜進行預處理,并比較預處理的效果,篩選出最佳的預處理方法。
2.2 樣本集選擇
Rank-KS算法是一種對校正集和預測集樣品的空間分布優選的新方法,分為兩個過程。一是“Rank”過程,即利用濃度梯度排序法的思想,將理化參量按數值大小順序排列,然后將整個數據區間平均分為p份;二是“KS”過程,即在每個小數據區間分別使用KS方法,校正集選自光譜空間中差異性顯著的樣本,dx可以為歐氏距離,也可以為馬氏距離。計算時,需要首先預定選出的校正集樣本個數為m,然后每個小數據區間需要選出的樣本個數為m/p,其中,p是均分區間個數。假如有一個小數據區間中樣本個數小于m/p,則將此小數據區間內樣本全部選為校正集。若最終選出的實際樣本個數m_real小于m,則用余留下的樣本再經KS法挑選出m-m_real個樣本進行補充。預測集選擇時,將理化參量按數值大小順序排列,隨后把整個性質區間均分為n個小區間,逐個在性質小區間內隨機抽取出一個樣本填入驗證集,即可得到由n個樣本組成的驗證集。該方法綜合考慮光譜空間和理化參量空間對樣本進行選擇,可以明顯改善樣本集隨理化參量變化的分布均勻性。
2.3 偏最小二乘模型
偏最小二乘(Partial least square,PLS)方法集中了多元線性回歸、典型相關性分析和主成分回歸分析三種分析方法的優點,能夠最大程度地利用光譜信息,提取出表征光譜數據變異的最大信息,對模型自變量具有良好的解釋性以及良好的預測功能。因此,本研究中使用PLS方法構建原始光譜及預處理后的光譜與玉米粗蛋白含量的預測模型,并用獨立的樣本進行驗證。模型評價指標有校正集相關系數(Rc)、校正集均方根誤差(RMSEC)、預測集相關系數(Rp)和預測集均方根誤差(RMSEP)、預測集標準偏差與標準誤差的比值RPD。
3 結果與分析
3.1 光譜預處理分析
近紅外光譜法存在吸收峰強度較弱、多組分信號重疊、背景干擾嚴重等問題,如何從復雜的光譜中提取待測組分的定量信息,消除背景和噪聲等無關信息的干擾,一直是研究的重要內容。玉米光譜數據在采集過中,可能受到玉米籽粒形狀的不規則性和光譜儀器本身溫濕度響應特性的影響,導致玉米光譜數據除含有玉米自身的化學信息外,還含有其他無關信息和噪聲,如電子噪聲、樣品背景雜散光、基線漂移等。為消除這些因素對玉米本身光譜的影響,需要對原始光譜進行預處理,預處理效果如圖1所示。
由圖1可以看出,歸一化預處理、SNV預處理和MSC預處理均改變了光譜曲線的變幅范圍,數據波動性變小;導數處理后的光譜吸收峰的個數明顯增多,尤其是二階導數預處理,同時光譜曲線的平滑連續性有所下降;去趨勢算法預處理的光譜曲線更加平滑,吸收峰的位置更加明顯,吸收峰的寬度明顯增加,較好地克服了近紅外光譜原始信息存在的缺陷。
3.2 偏最小二乘模型建立
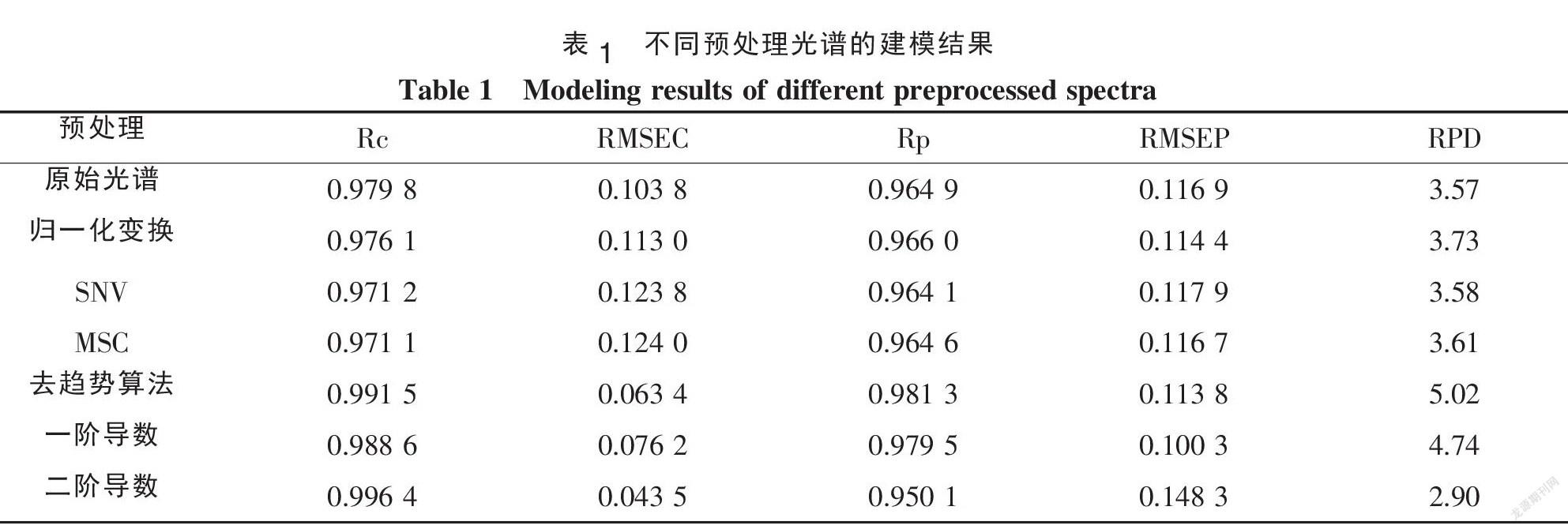
對經過預處理后的光譜采用Rank-KS算法選擇校正集樣本60個、獨立驗證集樣本20個,采用偏最小二乘法建立玉米粗蛋白的定量模型,結果如表1所示。
從表1可以看出,原始光譜所建立的PLS模型校正集和預測集相關系數分別為0.979 8和0.964 9,均方根誤差分別為0.103 8%和0.116 9%。與原始光譜相比,經去趨勢算法預處理后的光譜所建立的PLS模型效果最佳,這說明Rank-KS算法通過光譜空間和理化性質空間上樣本的空間距離來選擇校正集樣本,并考慮校正集在理化性質空間分布的均勻性,選出的校正集和驗證集可以明顯改善樣本數隨理化性質分布的均勻性。預測模型的校正集和預測集相關系數均明顯提高,校正集和預測集相關系數分別為0.991 5和0.981 3,均方根誤差分別為0.063 4%和0.113 8%,預測集的RPD為5.023 1,大于評價閾值3.0。所建模型的預測值與實測值得的相關性見圖2(a)和(b)。
4 結論
本研究以飼料玉米為研究對象,結合化學計量學方法,利用全波段光譜數據建立玉米粗蛋白預測的簡單快速精準預測模型,結果表明:先對原始光譜進行去趨勢算法預處理,再選用Rank-KS算法選擇校正集和預測集,使用全光譜數據進行PLS建模,校正集相關系數和均方根誤差分別為0.991 5和0.063 4,預測集相關系數和均方根誤差RMSEC分別為0.981 3和0.113 8,預測集的相對分析誤差RPD為5.02,大于評估閾值3.0,比陳素彬利用此數據所建立粗蛋白最佳預測模型的RPD值4.22還大,這說明該模型的精度和穩定性較為理想,可以滿足在線生成快速檢測的要求。
參考文獻
[1] 陳素彬.飼用玉米質量檢測的近紅外光譜法與經典方法比較[J].黑龍江畜牧獸醫,2021(7):114-118.
[2] 劉偉,趙眾,袁洪福,等.光譜多元分析校正集和驗證集樣本分布優化研究[J].光譜學與光譜分析,2014,34(4):947-951.
[3] 李江波,郭志明,趙春江,等.應用CARS和SPA算法對草莓SSC含量NIR光譜預測模型中變量及樣本篩選[J].光譜學與光譜分析,2014,34(4):947-951.
Chemometrics Method for Quality Analysis of Feed Corn
LIU Ya1,SHI Yong2*,SHI Xuming1,SUN Meile1,SONG Yu1,XIAN He1,YAO Guomin1
(1. Comprehensive Testing Ground,Xinjiang Academy of Agricultural Science, Urumqi Xinjiang 830013, China; 2. College of Electrical and Mechanical Engineering, Xinjiang Agricultural University, Urumqi Xinjiang 830052, China)
Abstract: Feed corn taken as the research object, stoichiometry was used to establish a simple and fast and accurate prediction model for maize crude protein prediction using whole-band spectral data. The results show that: After the original spectrum was preprocessed by detrending algorithm, the Rank-KS algorithm selected the correction set and prediction set, and used Partial least square (PLS) method for modeling. The correlation coefficients of the correction set and prediction set were 0.991 5 and 0.981 3, respectively. The root mean square errors of the correction set and the prediction set are 0.063 4 and 0.113 8, respectively. The relative analysis error (RPD) of the prediction set was 5.02, which was larger than the evaluation threshold of 3.0. The precision and stability of the model are satisfactory and can meet the requirements of on-line generation and detection.
Key words: chemometrics; feed corn; NIR; Rank-KS algorithm

