茶樹葉綠體基因組SNP分子標記的初步研究
羅祥宗,胡云飛,吳淋慧,趙雅琦,鄭偉銘,黎巷汝,李力*
1. 武夷學院茶與食品學院,福建 南平 354300;2. 安徽農業大學茶與食品科技學院,安徽 合肥 230036;3. 福建農林大學園藝學院,福建 福州 350002
茶作為世界上最受歡迎的飲品之一,在醫藥、健康、文化、貿易等領域都具有重要意義,在生活中也發揮著重要的作用[1]。茶樹在我國許多地區都有栽種,形成各地區獨特的茶葉品類。一些在植物學特征特性上十分相似的茶樹品種在基因的遺傳組成上很可能存在著較大差異,這給茶樹樣品的確定帶來一定困難[2],不僅會對茶樹品種的選育、種植及經營等方面造成損失,也給茶葉市場的監管帶來困擾。傳統農藝性狀的鑒別易受外界因素和鑒別時間影響,且費時費力,DNA分子標記有助于輔助茶樹品種鑒別與雜交育種的親本選擇[3]。
DNA分子標記具有多態性強,數量多,且不受環境限制等優點,在茶樹的馴化起源、遺傳育種和資源鑒定方面已被廣泛應用[4]。早期的第一代分子標記技術,例如相關序列擴增多態性(Sequence-related amplified polymorphism,SRAP)、限制片段長度多態性(Restriction fragment length polymorphism,RELP)、隨機擴增的多態性 DNA(Random amplified polymorphic DNA,RAPD)等[5-6],雖各具優點,但由于分型不穩定、基因型過多無法辨認、非共顯性等缺陷沒有被納入植物品種鑒定標準。目前,簡單重復序列(Simple sequence repeat,SSR)、單核苷酸多態性(Single nucleotide polymorphism,SNP)技術在國際植物新品種保護聯盟(International Union for the Protection of New Varieties of Plants,UPOV)的生化和分子標記技術(Biochemical and molecular labeling techniques,BMT)測試指南草案中被確定為構建DNA指紋數據庫的標記方法[7]。第二代SSR標記法雖具有多態性高和共顯性的優點,但SSR是多等位基因系統,內部存在復雜變異如Indel/SNP,而且忽視了一代測序金標準的驗證,容易導致鑒定結果存在不真實現象。相對于SSR標記存在的問題,第三代SNP標記為二等位基因系統,主要反映單個堿基的遺傳變異情況,并且易實現平臺之間數據的整合比較,在許多作物的品種鑒別中得以廣泛應用[8]。在茶樹研究方面,樊曉靜等[9]從茶樹的表達序列標簽數據庫中篩選出多態性高的核基因 SNP位點24個,構建的DNA指紋圖譜可對103個茶樹品種資源進行區分。盡管如此,茶樹SNP分子數據庫的研究仍處于起步階段,需要進一步補充完善。
相比核基因,母系遺傳的葉綠體基因在追蹤茶樹的母系親本、重建系統發育等方面能更好地提供有價值的信息。核基因組結構復雜且龐大,容易產生高突變,而葉綠體基因組小,進化速度緩慢,保守性強。此外,葉綠體基因非雙親遺傳的特性也可有效地避免分子內重組對遺傳進化研究所造成的干擾[10-11]。盡管進化速度相對緩慢,葉綠體基因組中基因的變異卻始終伴隨著物種的進化而發生,非重組和單親本遺傳使葉綠體DNA標記成為母系遺傳的良好指標,在缺乏親本信息的假定雜種后代中,無論過去幾代,都可以識別出其母系遺傳親本[12-13],這為探索植物的進化關系提供了寶貴的分子信息[14-16]。因此,一些傳統的葉綠體基因片段標記,如rpl16、psbA-trnH和rpl32-trnL基因間隔區等在植物遺傳多樣性分析中得到了廣泛應用[17]。然而,傳統的葉綠體基因分子標記在山茶屬植物種內較低階單元分類研究中的應用價值相對有限。已有研究顯示,在用葉綠體基因片段進行物種低階單元劃分時,應首先在葉綠體全基因組中篩選出適用的高度可變區[18]。
隨著作物品種資源數目的增加,傳統的作物形態標記鑒定方法已經難以對現有品種進行準確鑒定[19]。利用分子標記鑒定技術來構建作物種質資源的身份證已成為作物品種鑒定的趨勢。目前,葉綠體基因組 SNP標記在茶樹種質資源的鑒定方面鮮有報道,本研究篩選出多態性高的葉綠體SNP位點組成DNA指紋圖譜對茶樹品種進行區分及母系溯源,并結合品種資源基本信息構建品種條形碼身份證,旨在為茶樹品種保護和母本溯源等方面研究提供新的思路與方法。
1 材料與方法
1.1 試驗材料
茶樹樣品采自武夷學院茶樹種質資源圃(118°0'14.40"E,27°43'42.46"N),所采摘 169份茶樹種質樣品見表1。

表1 169份供試茶樹種質資源Table 1 169 tea germplasm resources used in this study
1.2 試驗方法
1.2.1 茶樹田間取樣與茶樹DNA的提取
采摘新鮮的茶樹嫩葉,采摘標準為一芽二葉。取0.2 g嫩葉用70%乙醇消毒后,用蒸餾水沖洗,加入液氮迅速研磨成粉末,利用植物基因組DNA提取試劑盒(康為世紀生物科技股份有限公司CW0553s)提取茶樹DNA。
1.2.2 茶樹DNA質量檢測方法
用1.0%瓊脂糖凝膠和Nano Drop分光光度計分別檢測 DNA的質量與純度。以 DNA凝膠電泳結果呈單條帶,OD260/280值在1.60~1.80,OD260/230值大于2.00為合格標準。
1.2.3 多變區篩選及通用引物設計
NCBI數據庫下載已報道的 18個茶組植物葉綠體全基因組序列(表2),使用MAFFT 7.0軟件進行序列同源比對。使用DnaSP軟件分析基因組多變區及多樣性(Pi),在多變區兩側保守區域開發茶樹 PCR通用引物用于基因組序列擴增,引物由生工生物工程(上海)股份有限公司合成。
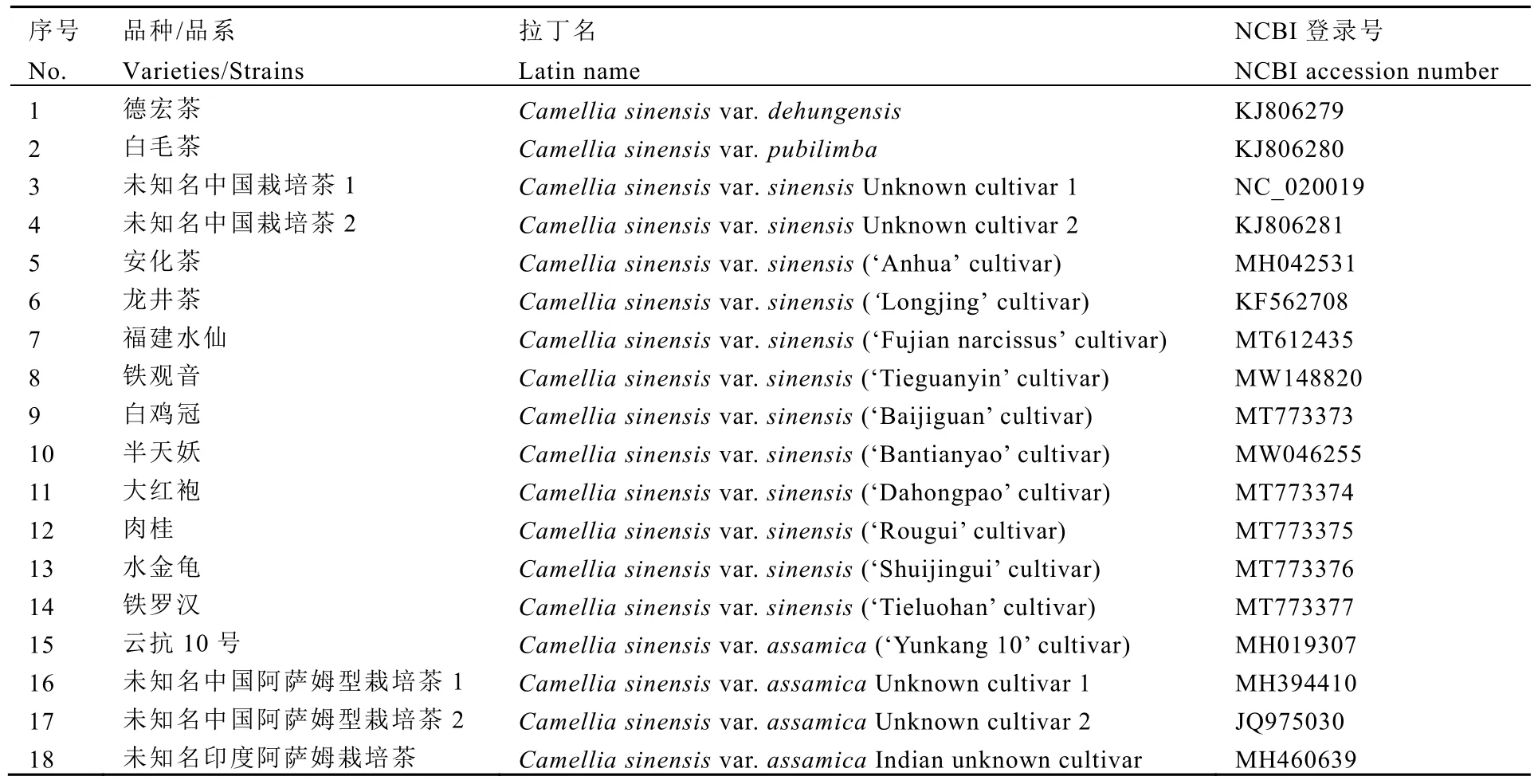
表2 NCBI下載的18個茶組植物葉綠體全基因組Table 2 The information of 18 complete chloroplast genomes of tea plants downloaded from NCBI
1.2.4 PCR擴增
30 μL反應體系:DNA模板50 ng,正、反向引物(10 μmol·L-1)各 1 μL,5×FastPfu Buffer 6 μL,2.5 mmol·L-1dNTPs 2 μL,補充ddH2O至30 μL。PCR擴增反應程序:95℃預變性 5 min;94℃變性 30 s,55℃退火 30 s,72℃延伸30 s,共30個循環;72℃延伸7 min,4℃保存。PCR產物以 1%瓊脂糖凝膠電泳為清晰單條帶為合格標準,合格產物委托生工生物工程(上海)股份有限公司進行正、反向一代測序。
1.3 數據處理
選擇測序峰圖SNP位點清晰的分子標記,利用PowerMaker 3.25軟件計算分子標記多態性信息含量(PIC)值,選擇PIC>0.05的分子標記用于DNA指紋圖譜構建。利用MEGA 5.1軟件使用最大似然法(Maximum likelihood)對茶樹樣品的基因序列進行聚類分析,利用條碼在線生成器(http://barcode.tec-it.com/ barcodegenerator.aspx)對茶樹品種身份證進行條碼轉換。
2 結果與分析
2.1 設計多變區通用引物及分子標記篩選
使用DnaSP軟件對18個已知茶組植物葉綠體全基因組序列進行多變區分析(表2),選擇多樣性指數(Pi)值大于 0.01的區域進行下一步分析(圖1-A)。在多變區兩側的保守區域設計17對通用引物在169個茶樹品種中進行PCR擴增,擴增產物大小在300~800 bp。其中瓊脂糖凝膠電泳為清晰單一條帶(圖1-B)且測序峰型清晰(圖1-C)的引物被考慮作為分子標記。經測序,有16對引物(表3)擴增出多態性,PIC 值分布范圍為0.057~0.670,平均值為0.321。16對分子標記中含測序峰圖清晰的SNP位點25個。
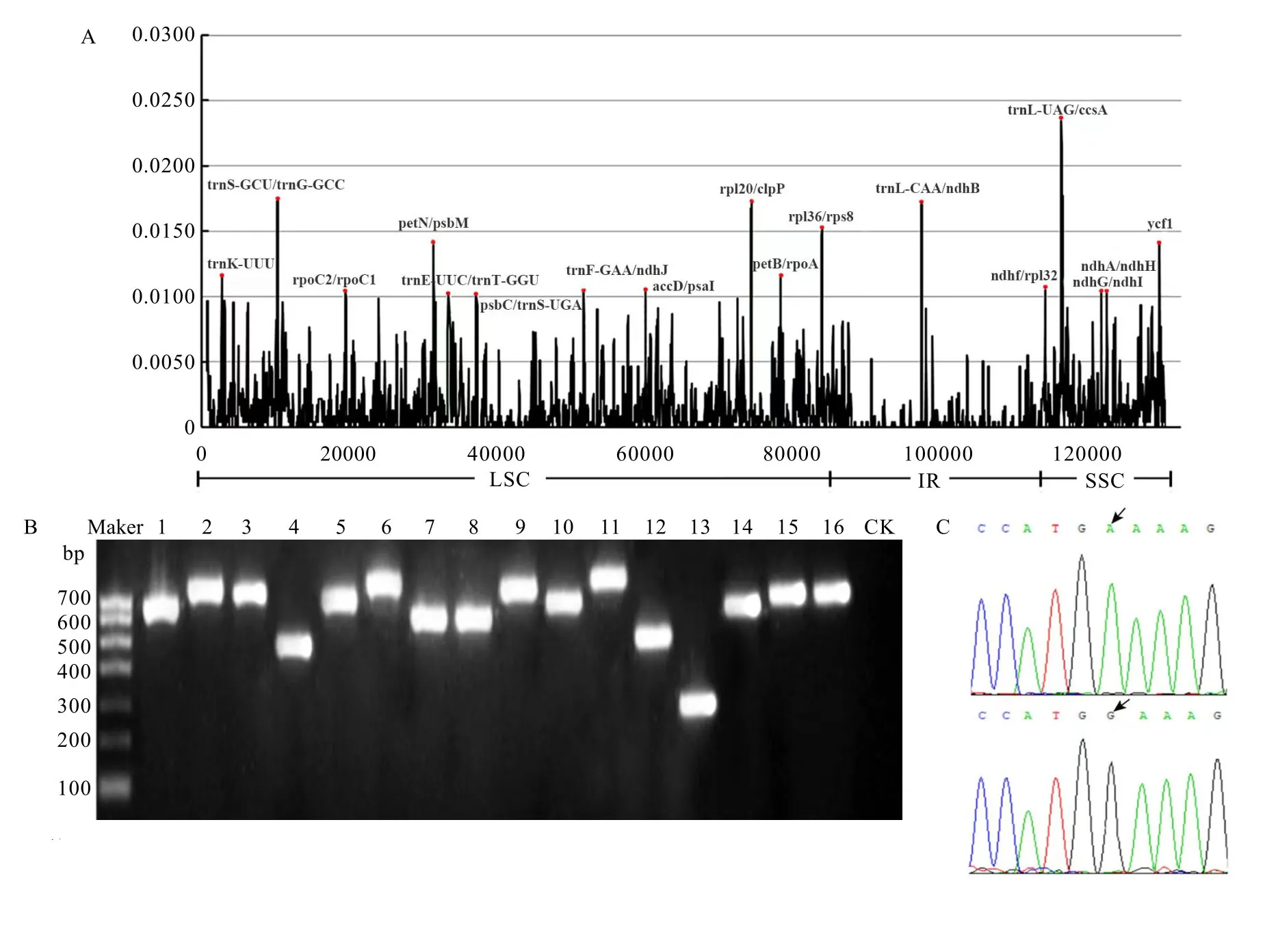
圖1 茶樹葉綠體基因組序列多變區SNP分子標記篩選Fig. 1 Screening of SNP molecular markers in the variable regions of tea plant chloroplast genome sequence
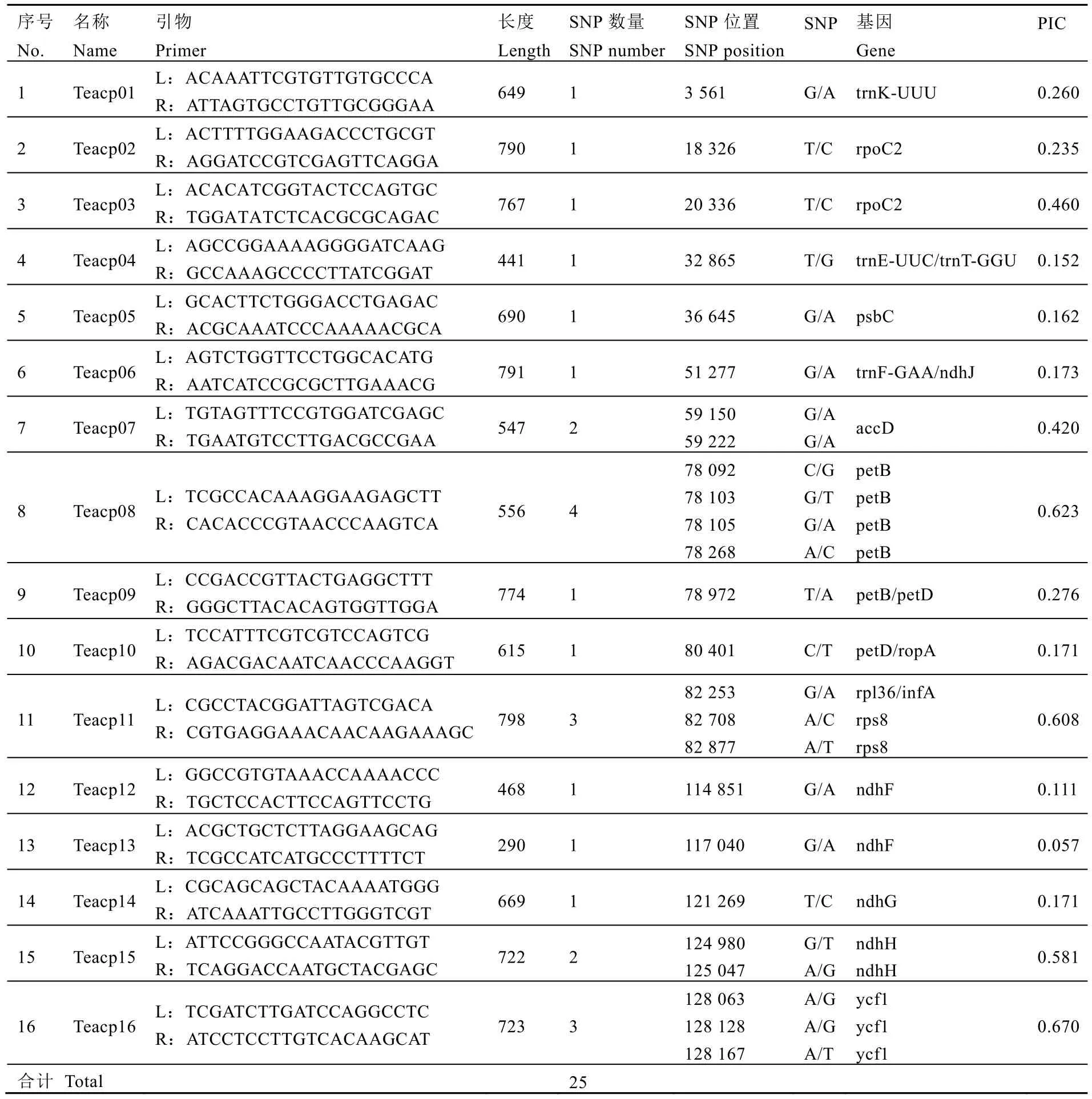
表3 16對葉綠體基因組SNP分子標記表Table 3 The information of 16 SNP molecular markers in chloroplast genome
2.2 單倍型及母系溯源初步分析
使用25個SNP可將169份茶樹種質形成118個單倍型(圖1和圖2),基于這套分子標記構建系統聚類樹,并根據已知的物種選育信息進行母系溯源分析,結果表明這套分子標記可應用于茶樹母系溯源初步鑒定。例如,丹桂是以肉桂為母本雜交而來;福云6號和福云10號是以福鼎大白茶為母本雜交而來。這些同一母系來源的品種在進化樹上的親緣關系非常接近,有的甚至共享單倍型。因此,運用這套分子標記對目前尚有疑問的 4個大紅袍品種的母系來源進行初步判斷,結果顯示,4株大紅袍的母系來源不同,大紅袍2與大紅袍4的母本親緣關系較近,但是它們與大紅袍1和大紅袍3之間的關系較遠(圖3)。
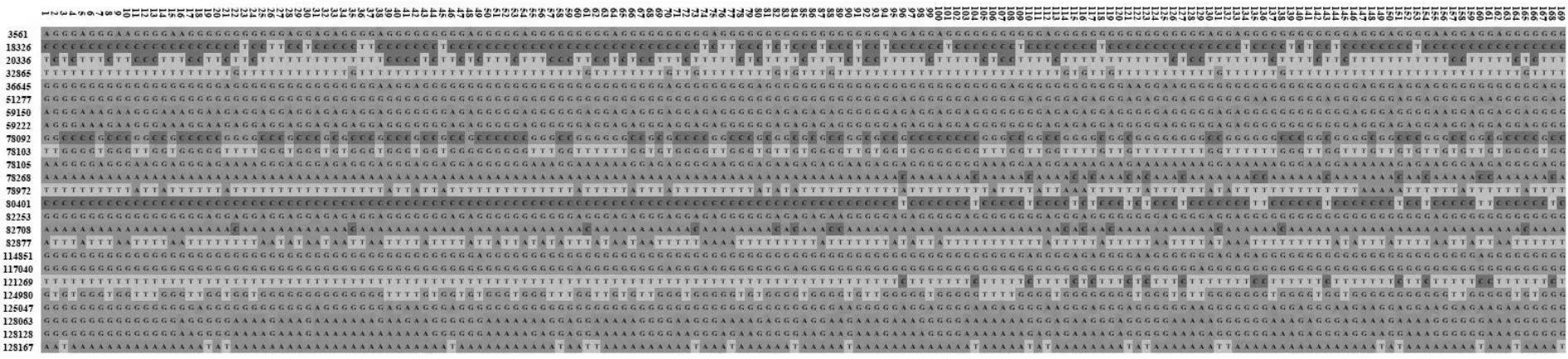
圖2 25個SNP位點串聯單倍型可視化圖Fig. 2 Visualization of 25 SNP tandem haplotypes
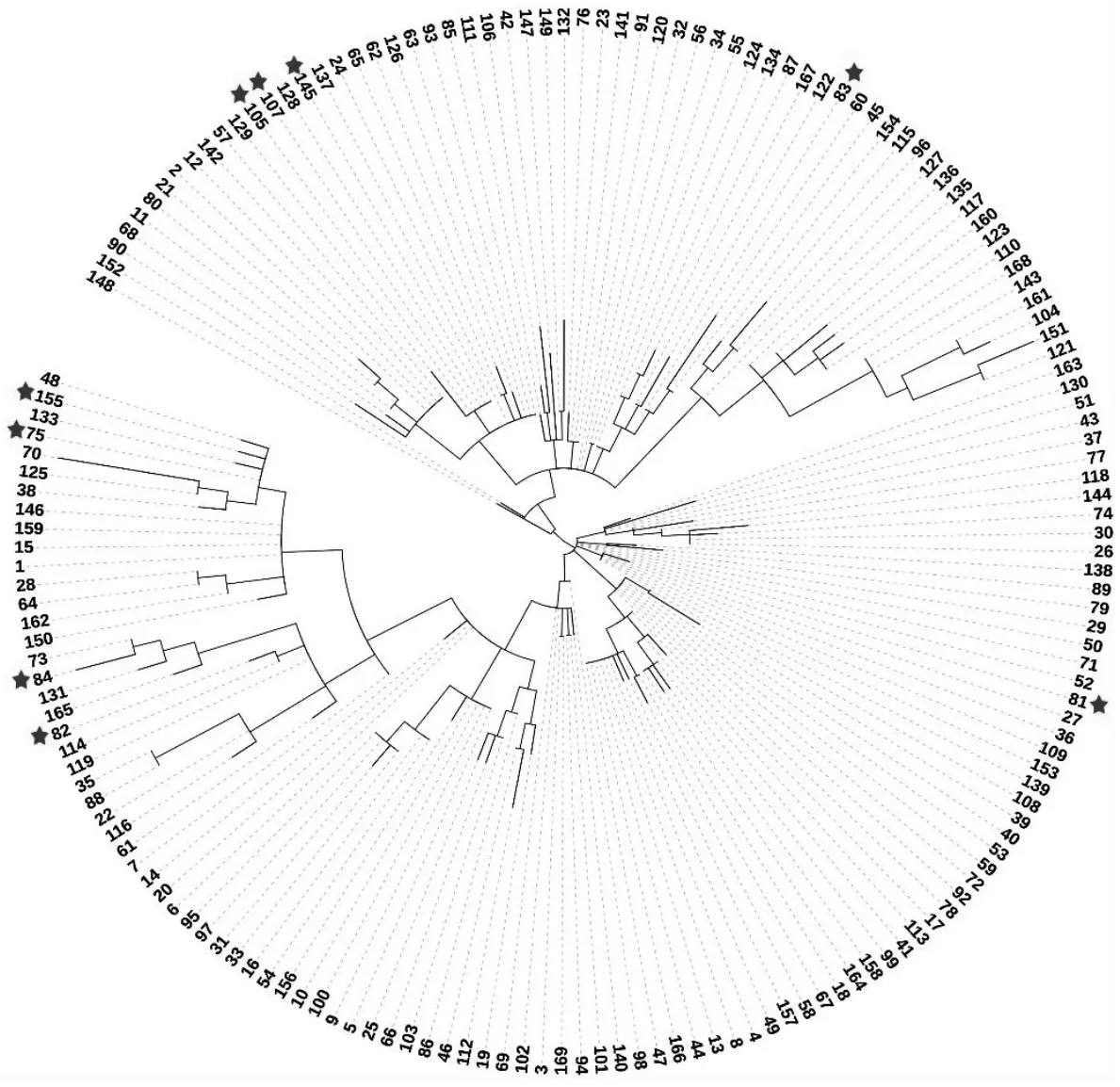
圖3 基于SNP集的聚類分析Fig. 3 Cluster analysis based on SNP set
2.3 茶樹葉綠體DNA指紋圖譜編碼
將 25個SNP位點進行數字編碼用于品種的DNA指紋圖譜信息:分別將堿基A、T、C、G轉換成數字1、2、3、4。除SNP指紋編碼外,也對品種的基本信息編碼,參考陳亮等[20]方法,將基本信息分為4個部分:(1)茶組植物物種,茶(Camellia sinensis)為1,大廠茶(Camellia tachangensis)為2,厚軸茶(Camellia crassicolumna)為 3,大理茶(Camellia taliensis)為4,禿房茶(Camellia gymnogyna)為5;(2)區域代碼,由2位數字組成,以各省、自治區、直轄市、特別行政區的行政區劃代碼組成,如福建為35,國外記為00等;(3)資源類型,野生茶樹為1,地方品種、地方種質為2,優良品種(含育成品種)為3,新選品系、株系為4,國外品種為5;(4)共享單倍型區分,相同母系的不同品種中若出現共享單倍型的分別暫以1,2,3……不同數字區分,其余特有單倍型的品種均標注為0。將品種基本信息和DNA指紋圖譜共同組成由30位數字編碼的分子身份證(表4)。例如,福鼎大白茶的品種資源基本信息為:茶(C.sinensis),產于福建,優良品種,存在共享單倍型,轉換成數字碼分別為13531。25個SNP分子標記分別為GCCTGGGGGTAAT CGATGGTTAAAA,轉換成 25位數字碼為4332444442112341244221111,則福鼎大白茶的分子身份證為1353143324444421123412442211 11。福云6號、福云10號以福鼎大白茶為母本雜交而來,三者共享SNP單倍型。分子身份證分別為 135324332444442112341244221111和135334332444442112341244221111(圖4)。
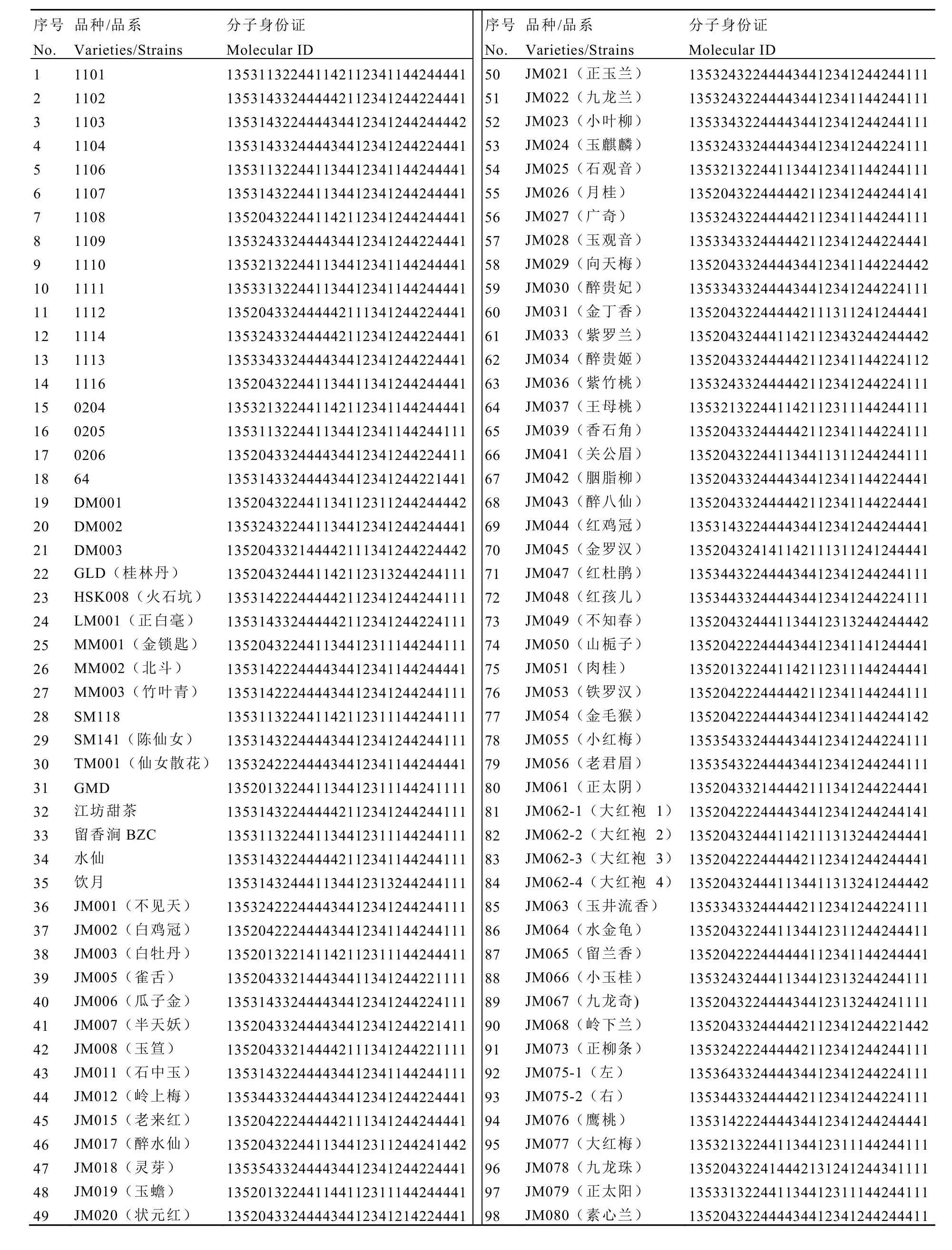
表4 169份茶樹品種資源的葉綠體分子身份證Table 4 Chloroplast molecular ID of 169 tea plant species resources

續表4
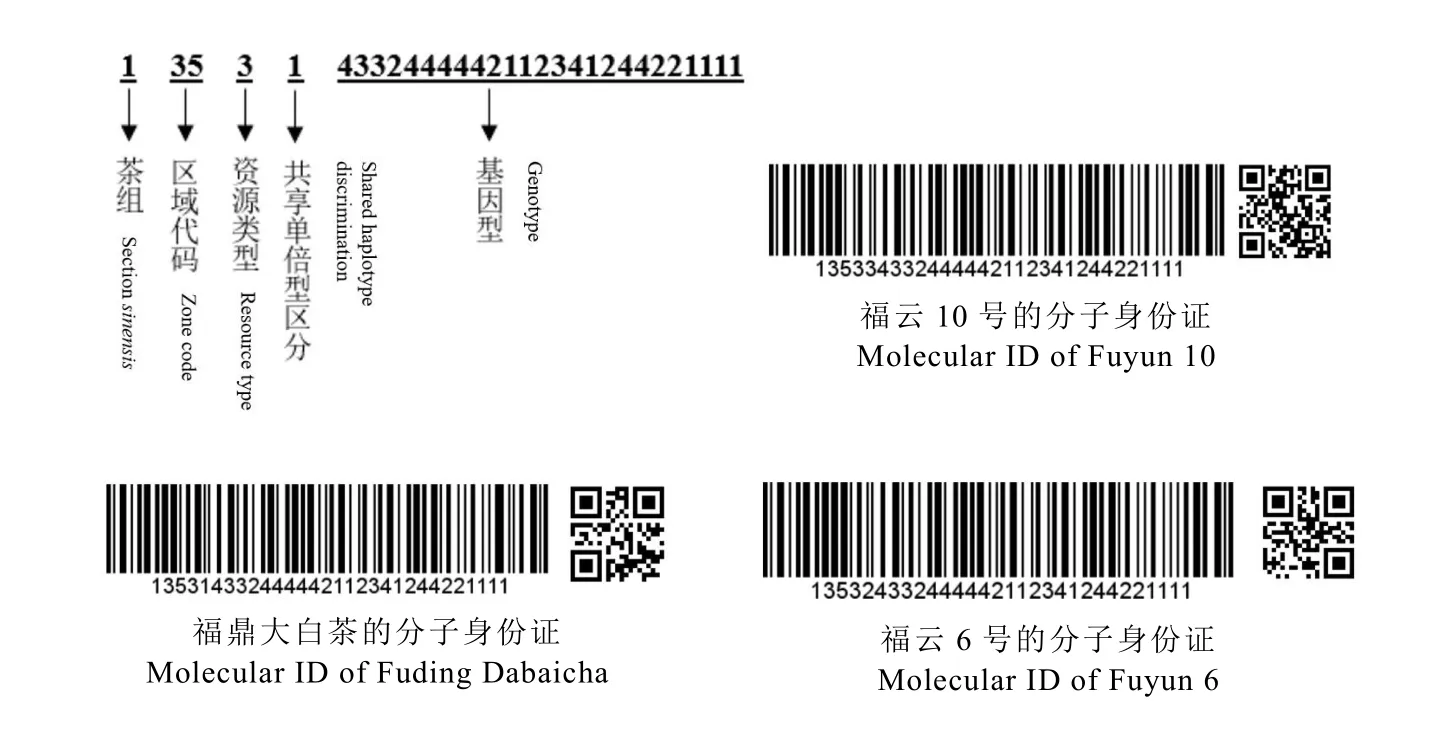
圖4 條形碼與二維碼示意圖Fig. 4 Diagram of bar code and two-dimensional code
3 討論
SNP單核苷酸多態性標記由于其正確性與高重復性已廣泛應用于作物的遺傳背景選擇和分子輔助育種[21-22]。葉綠體基因組結構保守,其單系遺傳以及非重組的特性使葉綠體基因SNP標記具有更好的群體分辨率[23],在植物不同種類的種質鑒別、物種分化及母系溯源等研究方面已得到了廣泛的應用[24-26],然而茶樹的葉綠體 SNP標記鮮見報道。本研究通過對茶樹葉綠體基因組進行 SNP分析,篩選出適用于茶樹種質鑒別與母系追溯的25個SNP位點,在169個樣本中共形成118個單倍型,結合已知的物種選育信息,發現同一母系來源的茶樹品種共享單倍型,或者親緣關系較近,這表明本研究中所使用的分子標記可應用于母系溯源初步鑒定。基于此,利用這套分子標記對武夷山 4株大紅袍茶樹關系進行分析判斷。據歷史資料記載與先前的研究結果,原本武夷山九龍窠上種植的大紅袍為三株,第二株(大紅袍2)為正本大紅袍,第一株(大紅袍1)和第三株(大紅袍3)為副本,目前的第四株(大紅袍4)來源尚存疑問[27]。本研究結果顯示,4株大紅袍的母系來源不同,大紅袍2與大紅袍4的母本親緣關系較近,而與大紅袍 1、大紅袍 3的關系較遠,這表明大紅袍 2與大紅袍4雖然直接的母系來源不同,但有更近的共同母系祖先。但是,僅以葉綠體基因組部分信息并不能完全代表物種親緣關系的情況,有性雜交繁殖過程中還會出現細胞器基因與核基因水平轉移、漸滲等情況,呈現復雜的網狀進化[28-29]。因此,這套 SNP標記只能作為母系溯源的初步判斷,闡明4株大紅袍具體的關系還需進一步結合更多的葉綠體基因組位點、線粒體基因組與核基因組信息。此外,在品種鑒別方面,由于葉綠體基因組的保守性,這套SNP標記在一些母系相同的品種中,因其存在共享單倍型,仍未能得以有效區分,需要進一步補充完善更多的有效SNP位點。盡管如此,這套葉綠體SNP標記可在169個茶樹種內實現118個單倍型的區分(70%),體現出較高的區分能力,有一定的應用價值。與基于DNA大小片段分離的 SSR分析相比,SNP分析可通過高通量的形式自動化檢測。因此,這些茶樹葉綠體 SNP分子標記有望結合競爭性等位基因特異性 PCR(Kompetitive allele specific PCR,KASP)、DNA芯片法等實現高通量自動化檢測茶樹種質。同時,該數據在基因組水平上拓展了茶樹可用分子標記的范圍,也為茶樹品種的種質資源鑒定、親緣關系評價、細胞質遺傳特性等研究提供了新的思路和方法。

