一株抗革蘭陽性菌的戈登氏菌WA4-43 全基因組測序與分析
和夢穎 劉文彬 林震鳴 黎爾彤 汪潔 金小寶
(廣東藥科大學生命科學與生物制藥學院 廣東省生物活性藥物研究重點實驗室,廣州 510006)
目前使用的大多數抗生素均來源于鏈霉菌,但是鏈霉菌分離得到的次級代謝產物大多是已知重復的[1]。目前關于稀有放線菌產生生物活性物質的相關研究較少[2]。但已有研究從Mycobacteriumspecies分 離 獲 得asukamycin 和 apramycin[3], 從Amycolatopsis species分離獲得rifamorpholines 等抗生素[4]。因此,稀有放線菌的抗生素生產潛力未得到充分利用,值得進一步深入研究[5]。
戈登氏菌是一種稀有放線菌。戈登氏菌常見應用于產生類胡蘿卜素[6],降解含油廢水[7],以及吸附重金屬等[8],而有關戈登氏菌抗菌活性和物質的研究較少。課題組前期從蜚蠊腸道中分離得到15 株戈登氏放線菌,并從WA8-44 菌株中分離出Actinomycin D、Actinomycin X2、Collismycin A 等 對真菌和細菌都有活性的化合物[9];從菌株WA4-31中分離出Actinomycin X2、Mojavensin A 等有一定抗真菌、抗腫瘤活性的化合物等[10]。
自天藍色鏈霉菌完成全基因組測序以來[11],基因組測序技術與生物學信息分析技術越來越成熟,利用基因組挖掘可以幫助我們更好地預測編碼基因和基因功能,預測出次級生物合成基因簇數量,具體基因功能以及與已知化合物同源性等。由于大多數基因在實驗室常規條件下處于沉默狀態,不表達或表達量低。除了常規的改變培養基參數,如添加化學誘導劑,共培養等方法可以激活沉默基因簇外[12],在全基因組測序的基礎上,以基因組為導向的天然產物挖掘技術,基因組挖掘技術避免了傳統挖掘方法的繁瑣和隨機,大大增加了新型天然產物的發現概率。采用基因組挖掘分析其生物合成基因簇,添加啟動子、異源表達等方法能更好地發現新天然產物,避免不必要的浪費[13]。
本文在前期研究基礎上,選取一株具抗革蘭陽性菌的菌株WA4-43 進行全基因組測序,并分析其基因組數據和生物合成基因簇,為后續研究提供基礎。
1 材料與方法
1.1 材料
1.1.1 實驗菌株 菌株WA4-43 為本課題組前期從蜚蠊腸道中分離獲得并保存于本室。白色念珠菌(ATCC 10231)、耐甲氧西林金黃色葡萄球菌(ATCC 43300)、金黃色葡萄球菌(ATCC 25923)、枯草芽孢桿菌(ATCC 6633)、表皮葡萄球菌(ATCC 25989)、大腸埃希氏菌(ATCC 25922)、肺炎克雷伯氏菌(ATCC 13883)和銅綠假單胞菌(ATCC 25924)均購置至于廣東省微生物研究所。
1.1.2 主要試劑及儀器 2×Taq PCR Master Mix,TaKaRa 公司;瓊脂糖凝膠,biowest 公司;EZUP 柱式細菌基因組DNA 抽提試劑盒,上海生工生物工程股份有限公司;T100TM PCR 儀,Bio-Rad 公司; LB培養基、PDA 培養基、PDB 培養基、高氏一號瓊脂培養基、ISP-1 和ISP-2 培養基均購于廣東環凱微生物科技有限公司。
1.2 方法
1.2.1 菌株WA4-43 培養及培養特征 將菌株WA4-43 接種于高氏一號固體平板上,28℃恒溫培養箱培養 5-8 d,觀察菌落形態、色素和產孢情況等特征,接種ISP-1,于28℃、180 r/min 搖菌 2 d。
1.2.2 16S rRNA 基因序列測定及系統發育分析 根據EZUP 柱式細菌基因組DNA 抽提試劑盒步驟提取菌株基因組DNA,使用16S rRNA 基因的通用引物27F(5′-AGAGTTTGATCMTGGCTCAG-3′)和1492R(5′-TACGGYTACCTTGTTAGGACTT-3′)[14]PCR 擴增16S rRNA。PCR 反 應 體 系(50 μL): DNA 模 板2 μL,2×Taq PCR Master Mix 25 μL, 引 物27F(10 μmol/L)和引物1492R(10 μmol/L)各 1 μL,ddH2O 21 μL。PCR 產物用1.0%瓊脂糖凝膠電泳檢測是否有相應大小條帶,送至北京六合華大基因科技有限公司廣州分公司測序。測序結果進行Blast 同源序列比對,下載NCBI 數據庫中親緣較近的40 株不同種類的戈登氏菌16S rRNA 基因核心序列,利用MEGA5.0[15]中的最大似然值法構建系統發育樹。
1.2.3 菌株WA4-43 粗提物抗菌活性測定 接種ISP-1,于 28℃、180 r/min 搖菌 2 d,以5%的接種量接種ISP-2 發酵液,于28℃、180 r/min 發酵14 d。發酵液離心得到上清液,上清液分別使用乙酸乙酯和正丁醇萃取3 次,減壓濃縮。粗提物用甲醇配置成10 mg/mL 樣品備用。采用管碟法[16]測定樣品對耐甲氧西林金黃色葡萄球菌、金黃色葡萄球菌、枯草芽孢桿菌、表皮葡萄球菌、大腸埃希氏菌、肺炎克雷伯氏菌、銅綠假單胞菌和白色念珠菌的抑菌活性。
1.2.4 菌株WA4-43 全基因組測序與組裝 全基因組測序由北京百邁客生物科技有限公司完成。首先使用Canu v1.5[17]軟件組合裝配過濾后的subreads,再通過Racon v3.4.3 軟件對組裝的結果進行矯正,后通過Circlator v1.5.5 軟件進行環化和調整起始位點,最后用Pilon v1.22 軟件進一步進行糾錯,以此來得到準確度更高的基因組序列。
1.2.5 菌株WA4-43 的基因組組分分析與基因組功能注釋 使用Rfam[18]數據庫、tRNAscan-SE v2.0[19]軟 件、Nr[20]數 據 庫、GO[21]數 據 庫、eggNOG[22]數據庫、Pfam[23]數據庫、COG 數據庫[24]和KEGG[25]等數據庫對基因組序列進行比對分析,獲得注釋信息以及功能;通過軟件Prodigal v2.6.3[26]動態編程算法較準確預測基因組中的編碼基因;使用antiSMASH v6.0.1[27]進行菌株WA4-43 生物合成基因簇(biosynthetic gene clusters, BGCs)分析;利用PRISM 4[28]完成生物合成基因簇化合物的預測。
1.2.6 菌株WA4-43 的比較基因組分析 GenBank中下載2022年3月之前所有Gorclonia terrae菌屬全基因組數據,進行生物信息學分析。
2 結果
2.1 菌株WA4-43培養特征
菌株WA4-43 在高氏一號固體培養基上生長良好,生長周期7 d 左右。菌落呈粉橘色,表面干燥不透明,呈不規則小圓型。經革蘭染色可見,菌株WA4-43 為短小棒狀革蘭陽性菌,掃描電鏡顯示菌絲呈短桿狀,無縱膈,如圖1 所示。
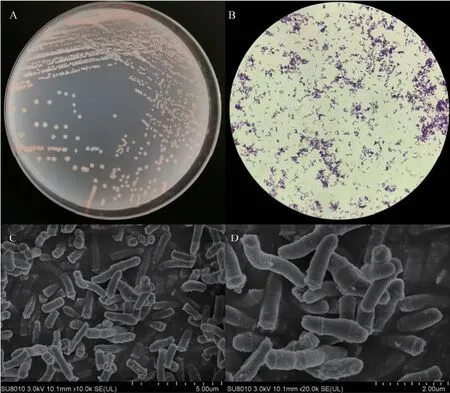
圖1 菌株WA4-43 菌株特征Fig. 1 Characteristics of strain WA4-43
2.2 菌株WA4-43分子生物學鑒定
2.2.1 菌株WA4-43 歸屬及系統發育樹的構建 通過PCR 擴增16S rRNA 基因并測序用于菌株WA4-43鑒定,PCR 擴增產物經測序為1 372 bp 的16S rRNA序列。將序列上傳至GenBank 數據庫中進行Blast同源性比對,選取較相似的40 個戈登氏菌代表性菌株多序列比對,利用MEGA 5.0 中的最大似然值法構建系統發育樹結果,如圖2 所示。結果表示該菌株與G. terraestrain DSM 43249 處于同一分支,其16S rRNA 基因相似度為100%,與G. lacunaestrain BS2 基因相似度為98.91%,結合菌株生理生化特征[29-30](表1)和16S rRNA 基因分析,初步鑒定菌株WA4-43 為Gordonia terrae。
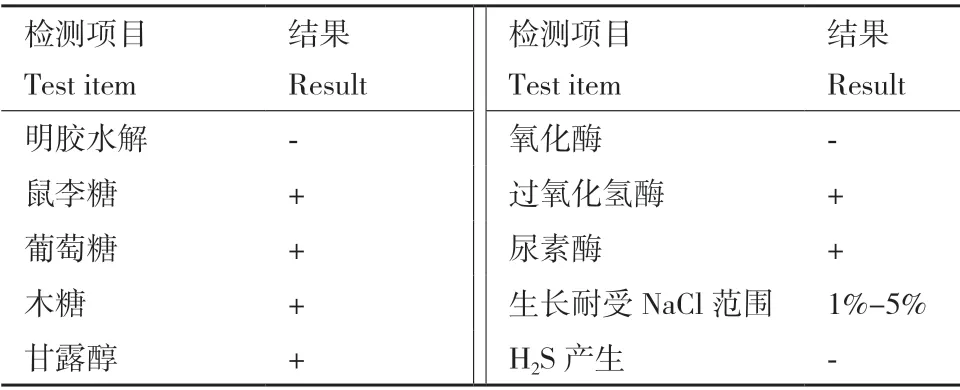
表1 菌株WA4-43 生理生化特征Table 1 Physiological and biochemical characteristics of strain WA4-43
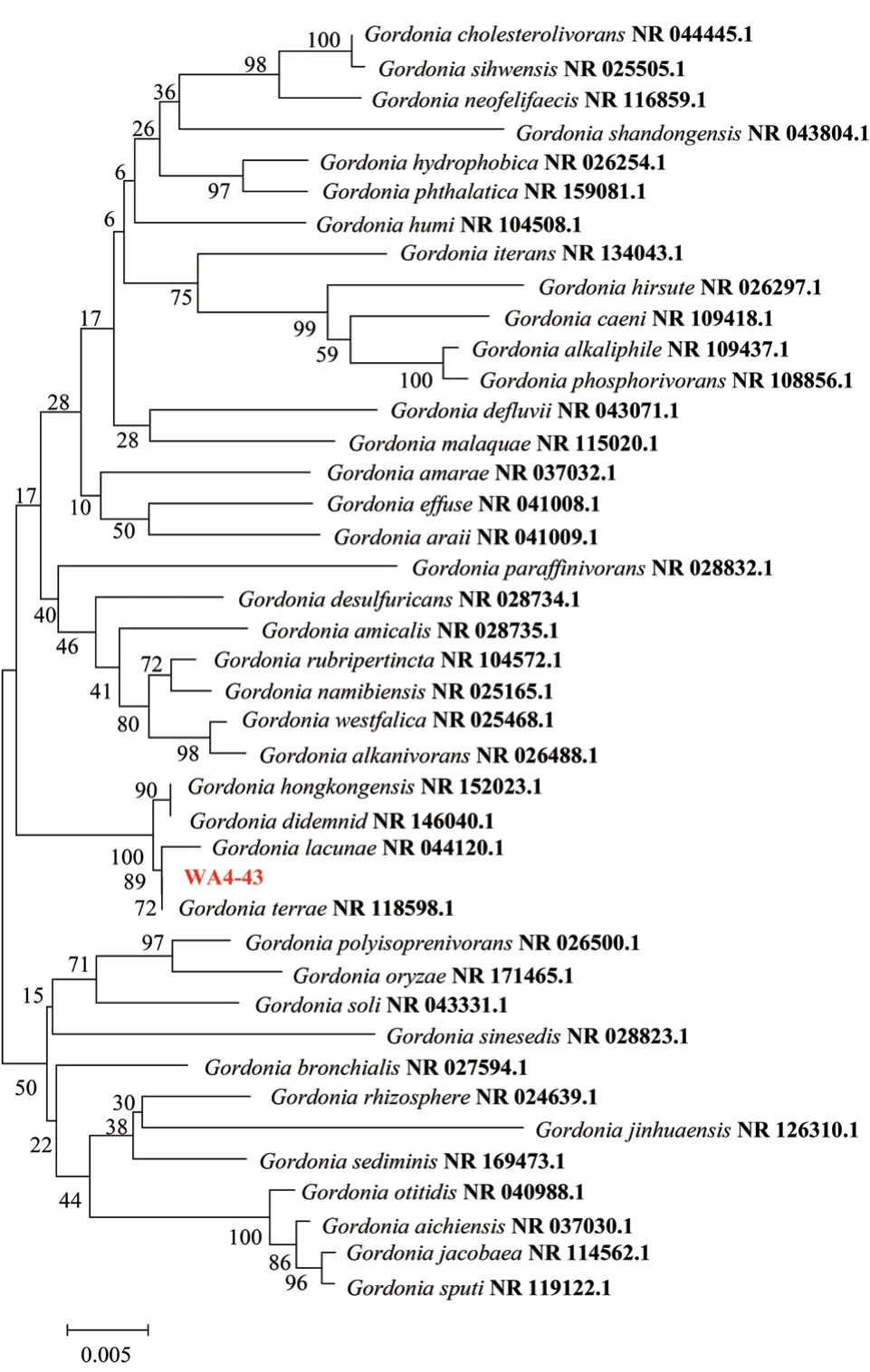
圖2 菌株WA4-43 系統發育樹Fig. 2 Phylogenetic tree of strain WA4-43
2.2.2 菌株WA4-43 次級代謝產物抗菌活性初步測定 菌株WA4-43 乙酸乙酯部位對4 種革蘭陽性菌:耐甲氧西林金黃色葡萄球菌、金黃色葡萄球菌、枯草芽孢桿菌和表皮葡萄球菌均有抑制作用(圖3),抑菌圈分別為(19.0±0.47)cm、(19.7±1.25)cm、(21.0±0.82)cm、(15.0±0.47)cm。菌株乙酸乙酯粗提物對大腸埃希氏菌、肺炎克雷伯氏菌、銅綠假單胞菌和白色念珠菌無活性。此外菌株正丁醇部位對上述菌株均無活性。
2.2.3 菌株WA4-43 全基因組測序與分析 將菌株WA4-43 全基因序列上傳至GenBank 獲得登錄號:CP084736.1。菌株WA4-43 基因全長5 438 735 bp,含有Contig 數量1 個,Contig N50 為5 438 735 bp,Contig N90 為5 438 735 bp,GC 含量為67.76%,基因組中包含4 963 個蛋白編碼基因,其余數據庫注釋基因如表2 所示。非編碼RNA 常具有特殊的二級結構,其中rRNA 9 個,包括23S rRNA 3 個,5S rRNA 3 個,16S rRNA 3 個;tRNA49 個;other ncRNA 40 個。
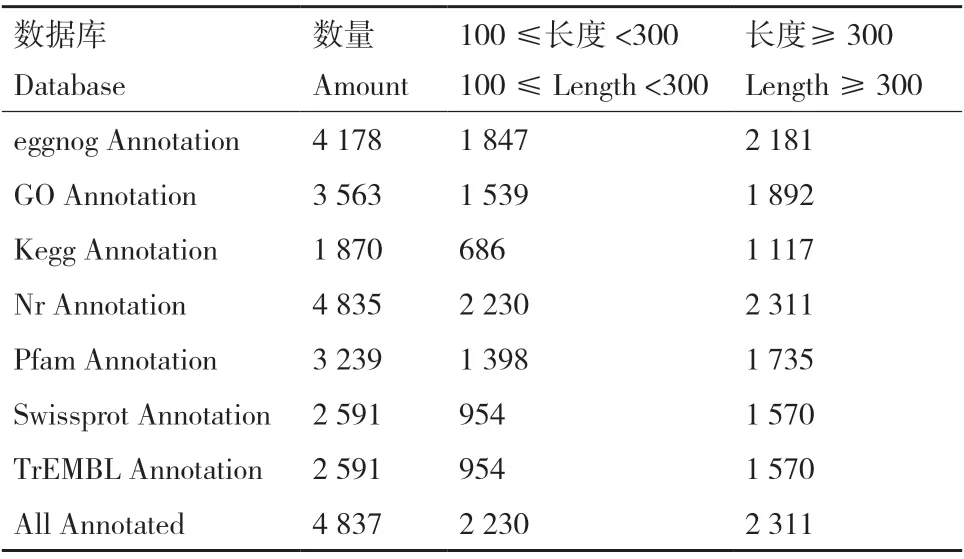
表2 菌株WA4-43 功能注釋的基因數量和大小Table 2 Number and size of functionally annotated genes in strain WA4-43
利用菌株WA4-43 全基因序列預測得到的tRNA、rRNA、重復序列、GC 含量等信息,制作基因組圈圖如圖4 所示,可視化地看出基因組各組分在全基因組上的各種位置關系。
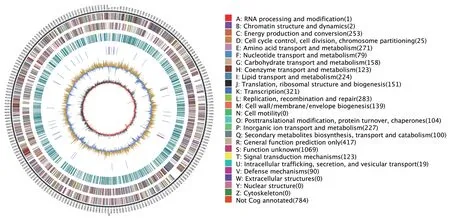
圖4 菌株WA4-43 基因組圈圖Fig. 4 Genome circle map of strain WA4-43
2.3.1 GO 功能注釋 在菌株WA4-43 中,有3 563個基因在GO數據庫得到注釋,注釋結果包含三大類,細胞組分(cellular component)、分子功能(molecular function)、生物過程(biological process),分析基因組各種功能注釋信息,如圖5 所示。藍色: 細胞組分;紅色: 分子功能;綠色: 生物過程。在細胞組分分類中共有9 類功能基因得到注釋,其中基因比例較大的為membrane 和membrane part。在分子功能
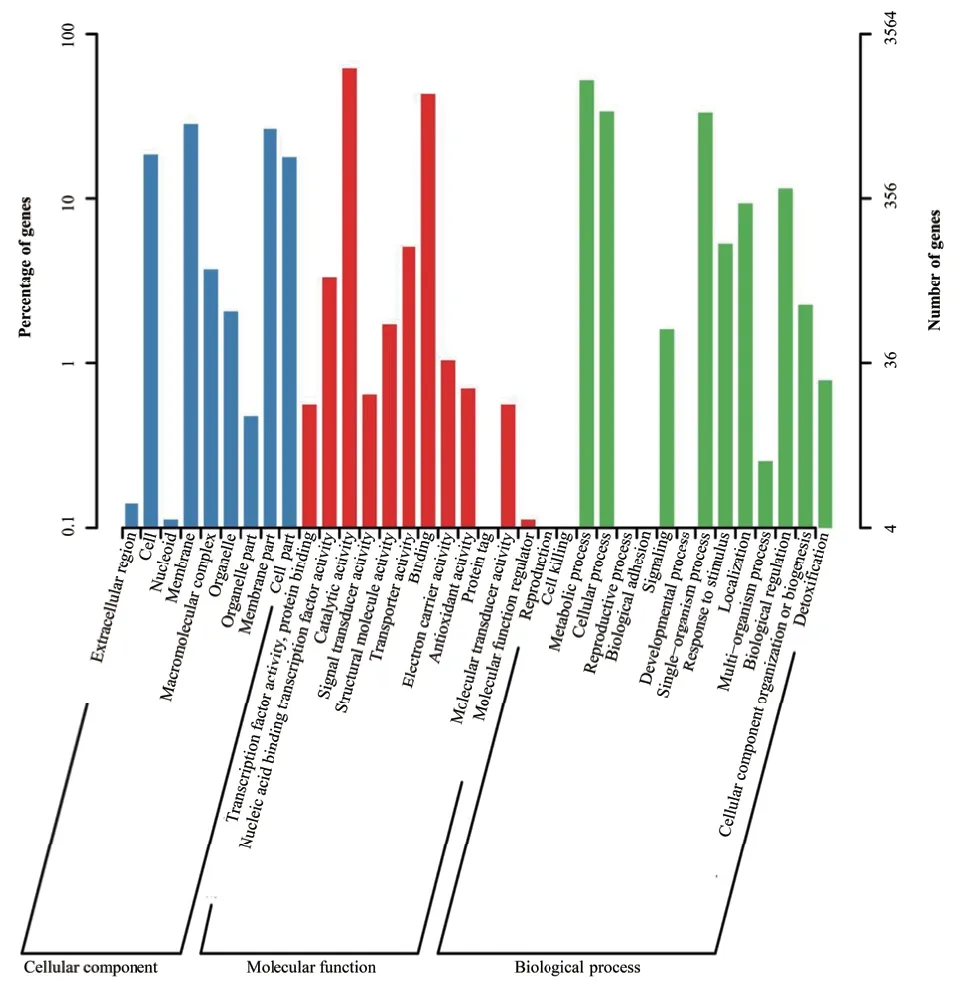
圖5 菌株WA4-43 基因組GO 功能注釋Fig. 5 GO functional annotation on the genome of strain WA4-43
2.3 菌株WA4-43基因組注釋
分類類別中共有11 類基因得到注釋,參與catalytic activity 和binding 的獨立基因比較多。在生物過程這個分子功能中,有10 類基因得到注釋,其中參與metabolic process、cellular process 和single-organism process 過程的獨立基因較多。
2.3.2 KEGG 功能注釋 菌株WA4-43 共有1 870 個基因在KEGG 數據庫中得到注釋,如圖6 所示,紅色,生物遺傳信息;紫色,環境因素;綠色,新陳代謝。共有3 大類代謝通路分別為genetic information processing、environmental information processing 和metabolism,分別有5、2、41 個小類。主要參與ribosme(59 kos),abc transporters(106 kos),biosynthesis of amino acids(129 kos)等代謝過程。
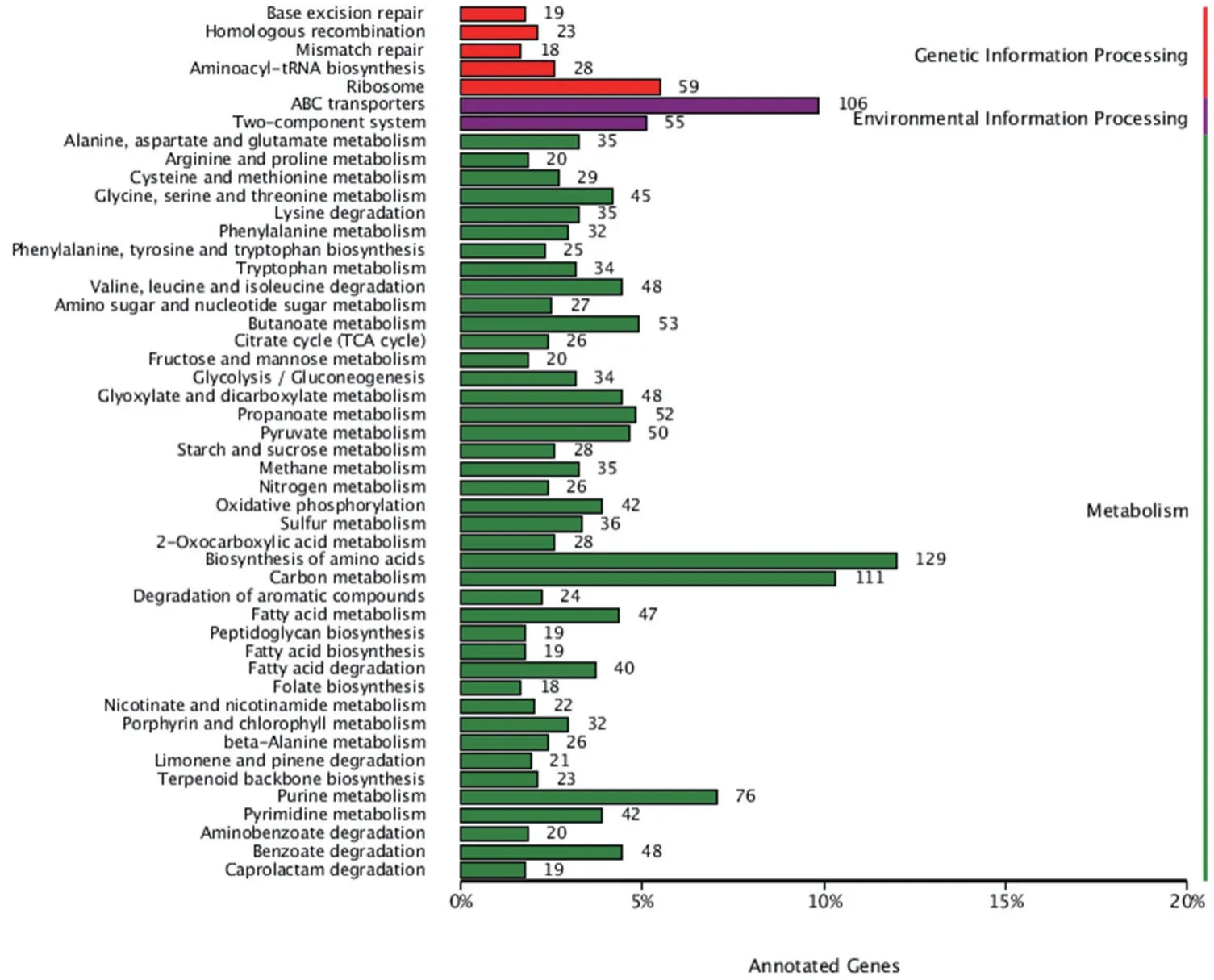
圖6 菌株WA4-43 基因組KEGG 的功能注釋Fig. 6 KEGG functional annotation of strain WA4-43 genome
2.3.3 COG 功能注釋 通過將菌株WA4-43 基因信息于COG 數據庫進行比對,共有4 178 個基因獲得注釋,共有22 類,如圖7 所示,其中function unknown 和general function prediction only 類 別 基 因數量最多分別為1 069 個和417 個,占注釋基因的25.22%和9.84%。其余功能基因占比較高的、大于5% 的 分 類 有energy production and conversion(253個,占比5.97%)、amino acid transport and metabolism(271 個,占比6.39%)、lipid transport and metabolism(224 個,占比5.29%)、transcription(321 個,占比7.57%)、replication,recombination and repair(283 個,占 比6.68%)、inorganic ion transport and metabolism(232 個,占比5.47%)。占比較少的類別為RNA processing and modification(1 個,占比0.02%)、cytoskeleton(1 個,占比0.02%)。

圖7 菌株WA4-43 基因組COG 功能注釋Fig. 7 Functional annotation of COG in the genome of strain WA4-43
2.3.4 次級代謝基因簇預測 使用軟件AntiSMASH對菌株WA4-43 全基因組數據進行分析,如表3 所示,菌株WA4-43 有13 個次級代謝生物合成基因簇,包括四氫嘧啶類(ectoine)、萜烯類(terpene)、非核糖體多肽類(NRPS)、鐵載體(siderophore)、核糖體合成和翻譯后修飾肽(RiPPs)等,共預測到6 種可能的次級代謝產物,其中,同源性大于等于75%的次級代謝基因簇預測產物的有ectoine;同源性低于75%的預測產物有SF2575、ishigamide、oxalomycin B、kanglemycin A / kanglemycin V1/kanglemycin V2 和desferrioxamine。

表3 菌株WA4-43 基因組中次級代謝基因簇的預測Table 3 Prediction of secondary metabolic gene clusters in the genome of strain WA4-43
基因簇使用軟件PRISM 4 可以預測出BGC4、5、6、7、11、12 化合物分子式,如圖8 所示。
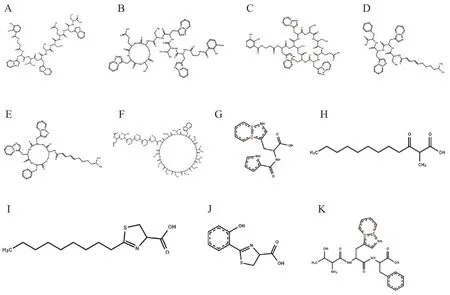
圖8 菌株WA4-43 生物合成基因簇化合物預測Fig. 8 Prediction of biosynthetic gene cluster compounds of strain WA4-43
BGC10 中包含核心生物合成基因1 個,轉運基因2 個,額外生物合成基因1 個,其他基因3 個,如表4 所示,其中核心生物合成基因GE002626 全長810 bp,通過Blast 對菌株WA4-43 蛋白序列與Nr數據庫進行比對,該基因與Nocardia farcinica中對應基因相似,GO 數據庫分析顯示其生物過程與蛋白質水解有關,也與細菌的防御反應有關;分子功能與肽酶活性有關。

表4 BGC10 基因預測及數據庫分析Table 4 BGC10 gene prediction and database analysis
2.3.5 比較基因組分析 將菌株WA4-43 與9 株G.terrae全基因序列進行比對分析,它們基因組大小范圍為:5.17-5.71 Mb,其中基因組最大的為土壤來源的G. terraeNCTC10669 和G. terraeNRRL B-16283,最小的為G. terraeC-6;編碼蛋白范圍為:4 480-5 007 個,GC 含量范圍為:67.7%-68%,均低于70%,其中GC 含量最高的為G. terraeK,最低的為G.terraeUMB0777。多數G. terrae為環狀染色體,包括G.terraeWA4-43、G. terraeNCTC10669、G. terraeNRRL B-16283 等,如表5 所示。
經antiSMASH 預 測 和 分 析,10 株G. terrae次級代謝基因簇類型較少,但同源性均非常低。有154 個次級代謝基因簇,83 個次級代謝基因簇預測有已知化合物,如表5 所示,每個菌株都有RiPPlike 類化合物分別為pimaricin 和kanglemycin A /kanglemycin V1 / kanglemycin V2,都 有NRPS 類 的ishigamide 和ectoine 類的ectoine。
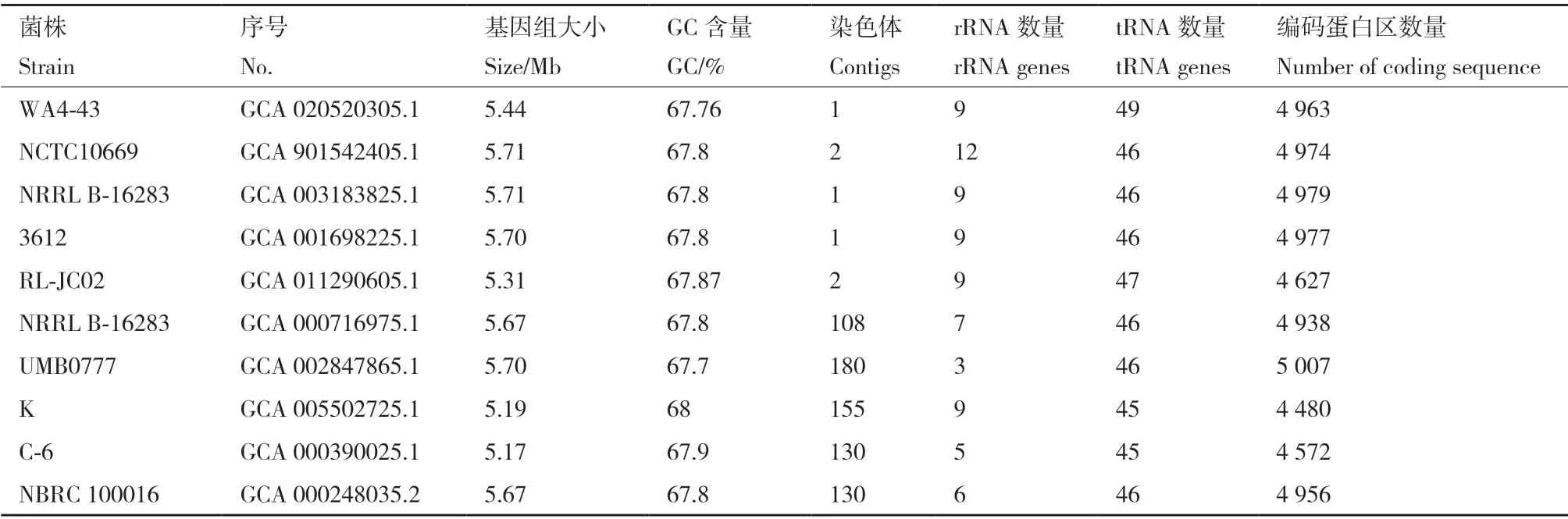
表5 10 種Gordonia terrae 基因組特征比對Table 5 Comparison of genomic characteristics of 10 Gordonia terrae species
比較G. terrae與6 種其他類型的戈登氏菌,可以看出,所有戈登氏菌均有ectoine 類的ectoine 同源性為75%,其他專有的預測化合物有,G. namibiensis有NRPS 類的atratumycin 同源性為7%,有PKS 類的GE81112 同 源 性 為7%;G. lacunae有NRPS 類的mycobactin 和glycinocin 同 源 性 分 別 為30% 和4%;G. ankookensis有NRPS 類 的pepticinnamin E 和pyxidicycline A、pyxidicycline B 同源性分別為10%和6%等,如圖9 所示。
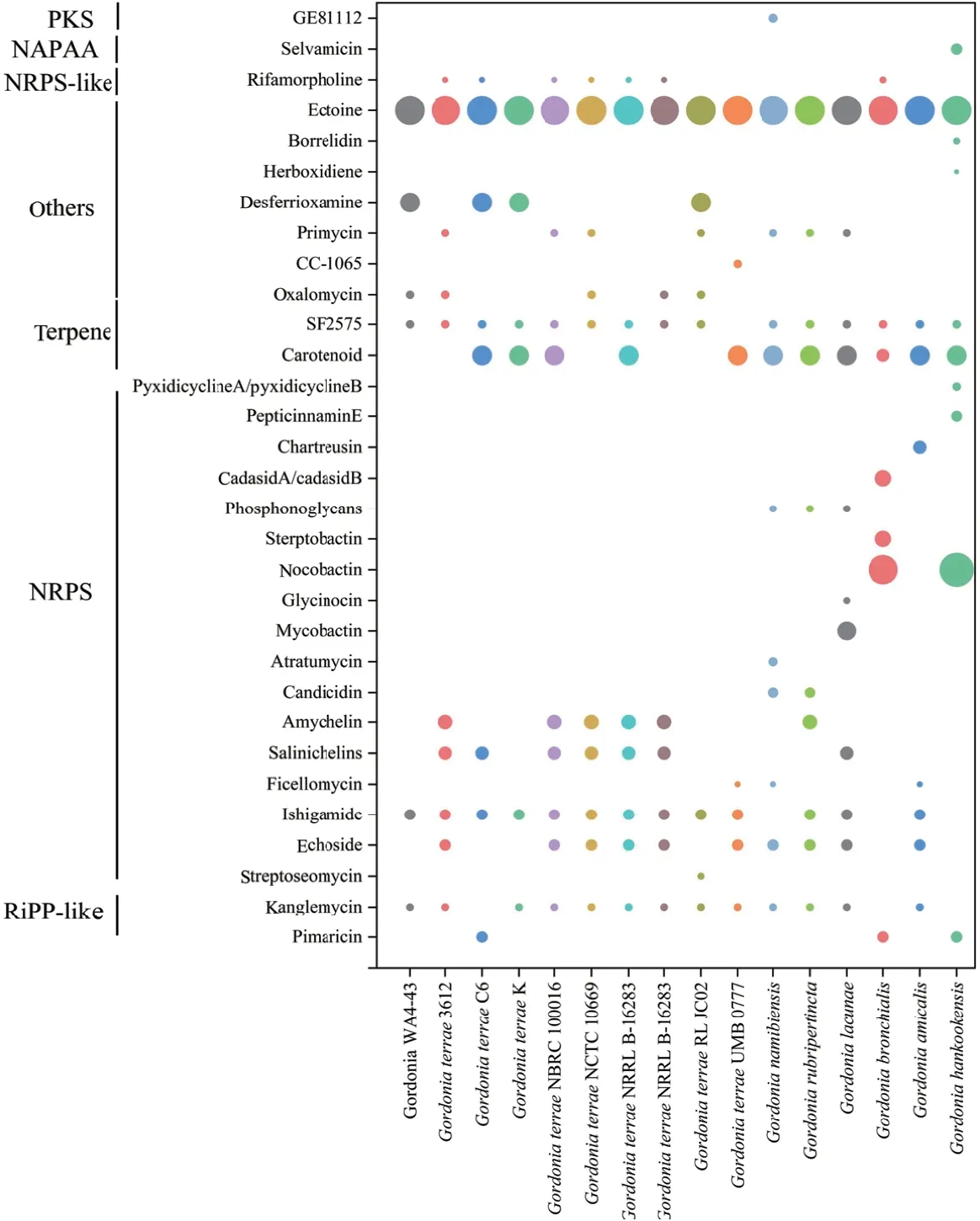
圖9 AntiSMASH 預測Gordonia 生物合成基因簇Fig. 9 Predicting the Gordonia biosynthetic gene cluster by AntiSMASH
3 討論
戈登氏菌次級代謝產物首次發現僅抗革蘭陽性菌。通過菌株WA4-43 全基因測序分析及antiSMASH次級代謝產物合成基因簇預測,可以知道菌株WA4-43 較鏈霉菌基因組較小,次級代謝產物基因簇種類數量較少[31],有7 個假定基因簇,預測基因簇與已知分離出化合物合成基因簇相似度均小于等于75%,表明菌株WA4-43 有研究意義,具有合成新穎化合物的潛力。
BGC1 與四氫嘧啶類型的ectoine 的相似性為75%,ectoine 是重要的應對外界環境變化的相容性 溶 質[32];BGC2 與萜烯類型 的SF2575相似性為6%,SF2575 對多種癌細胞系具有抗癌活性,可以抑制DNA 拓撲異構酶[33]; BGC9 與萜烯類型的oxalomycin B 相似性為6%,oxalomycin B 具有抗腫瘤,抗病毒的作用,以及關于HIV 抑制劑方面的研究[34];BGC12 與非核糖體多肽類、鐵載體類型的desferrioxamine 相似性33%等[35];BGC10 核糖體合成和翻譯后修飾肽類型的Kanglemycin A/kanglemycin V1 /kanglemycin V2[36],相似性為5%,預測化合物最早由Amycolatopsis vancoresmycinaDSM 44592 菌株產生,是利福平同系物,對金黃色葡萄球菌、表皮葡萄球菌、單核細胞增生李斯特菌等革蘭氏陽性菌有抑制活性,與菌株WA4-43 乙酸乙酯粗提物活性相似,但是同源性低,提示菌株有發現新抗革蘭陽性菌藥物的潛能。這些基因簇同源性均很低,預測到至少有3 種抗生素。
10 株G. terrae共有48 473 個蛋白編碼基因,其核心基因可能涉及菌體的基礎代謝以及適應環境等來維持其基本生命特征。如菌株WA4-43 COG 數據庫分析所示,多為未知功能,提示菌種具有新穎性,多參與轉錄、DNA 復制、結合和修復、氨基酸轉運與代謝、能源的產生交換、無機離子運輸與代謝等使菌株具有基礎的代謝和轉運功能,使細菌適應環境來維持最基本的生命特征。G. terrae菌株之間次級代謝基因簇的差異可能與它們生活環境不同有關,是它們適應不同生長環境的表現。
比較不用類型的戈登氏菌,其中RiPP-like 類型 的pimaricin、kanglemycin A /kanglemycin V1/kanglemycin V2 至少有一個,pimaricin 對霉菌、酵母菌和真菌都有極強的抑制能力,但對細菌、病毒等其他微生物沒有抑制作用[37],因為PiPP 類型天然產物是由遺傳編碼的前體肽及其同源修飾酶組成的[38],可能說明RiPP-like 類型的化合物可能決定戈登氏菌的抗菌專一性;特殊預測化合物一般是NRPS 類型,如NRPS 類 ishigamideG. terrae中均有且同源性為11%、5 株有NRPS 類oxalomycin B 而其他幾類戈登氏菌沒有,說明這兩個基因簇有區別于其他菌株的特性,有研究意義。
4 結論
菌株WA4-43 是首次發現的具有抗革蘭陽性菌戈登氏菌。該菌株生物合成基因簇新穎,具有合成獨特結構化合物的潛能。前期研究發現菌株次級代謝產物產量較低,提示我們后續可通過異源表達等手段進行天然產物的研究。