廣西三江茶樹種質資源遺傳多樣性分析
蒙容君,陳亮,許原,林緯,周歧偉,謝義林,賴定清,賴家業
廣西三江茶樹種質資源遺傳多樣性分析
蒙容君1,陳亮2,許原1,林緯3,周歧偉3,謝義林3,賴定清4*,賴家業3*
1 廣西大學林學院,廣西 南寧 530004;2. 中國農業科學院茶葉研究所,浙江 杭州 310008; 3. 廣西大學農牧產業發展研究院,廣西 南寧 530004;4. 三江縣多耶樓茶業有限公司,廣西 三江 545500
為明確廣西三江茶樹種質資源遺傳多樣性,采用簡單重復序列間擴增(ISSR)和相關序列擴增多態性(SRAP)2種標記技術,對三江地區72份地方茶樹種質資源與5個引進品種進行遺傳多樣性分析,兩種標記結果都顯示出高水平的遺傳多樣性(H=0.30,I=0.44),群體間顯示出高的遺傳分化(Gst=0.056)和基因流(Nm=8.64)。群體內觀察到的遺傳變異遠高于群體間的遺傳變異。聚類分析結果顯示,大部分三江茶樹種質資源聚為一類,與引進品種劃分成兩個亞群,聚類分析結果與主坐標分析(PCoA)的結果一致。本研究為三江茶樹種質資源的保護和利用提供了分子水平的證據。
茶樹;分子標記;遺傳多樣性;種群結構
茶葉作為最受世人喜愛的三大無酒精飲料之一,多由山茶科(Theaceae)山茶屬()茶樹[(L.) O. Kuntze]的嫩芽葉制作而成。廣西柳州三江縣位于黔桂湘三省交界處,地處茶樹起源中心的輻射范圍內,茶樹栽培歷史悠久,擁有豐富的茶樹種質資源[1]。近年來,由于一些野生茶具有品質優良、風味獨特且成本低等特點,導致古茶樹資源被過度開采,部分茶樹資源因此消失,嚴重阻礙了茶樹種質資源保存和良種選育事業進程和發展。因此開展茶樹種質資源遺傳多樣性研究,對三江縣茶樹種質資源的保護和利用、專用新品種選育以及茶產業健康發展等具有重要意義。
DNA分子標記技術是檢測種質資源多樣性的有效工具[2-3],在植物分子標記技術中簡單重復序列間擴增(ISSR)和相關序列擴增多態性(SRAP)是使用廣泛的兩種標記,具有諸多優點。ISSR分子標記以加錨簡單重復序列(SSR)寡聚核苷酸作引物,對位于反向排列的SSR之間的DNA序列進行PCR擴增,非擴增SSR本身,因其試驗重復性強,可解釋更高程度的DNA多態性等優點,在水稻[4]、玉米[5]、甘蔗[6]等作物的遺傳育種中被廣泛使用。劉青等[7]利用ISSR分子標記技術對貴州省145個古茶樹個體進行遺傳多樣性分析,對古茶樹資源的保護具有重要指導作用。孫雪梅等[8]利用ISSR對云南67份茶樹種質資源進行了遺傳多樣性分析,揭示了供試樣品之間的親緣關系,為云南省擴寬地方茶樹種質的遺傳基礎及野生茶樹遺傳潛力評估提供了理論依據。SRAP分子標記技術通過雙引物設計對基因的特定區域進行擴增,正向引物對外顯子區域進行特異擴增,反向引物對內含子區域或啟動子區域進行特異擴增[9],該標記具有穩定、產率高、便于擴增目標片段等特點[10]。林萍等[11]利用SRAP分子標記技術對同屬山茶屬的12個無性系油茶品種進行遺傳差異比對,為長林系列的無性系油茶種源的開發和利用構建理論基礎。Zhang等[12]利用EST-SSR、SCoT和SRAP 3種標記對秦巴山區50份茶樹種質資源進行遺傳多樣性以及群體結構評估,揭示了外來茶樹品種的引進對秦巴山區茶葉基因庫產生的影響。在DNA分子標記分析遺傳多樣性研究中,綜合運用多種分子標記,能最大程度優化聚類分析[13]。本研究利用12個ISSR引物和16對SRAP引物組合,對72份三江茶樹種質資源及5份外省引進的栽培品種進行遺傳多樣性、遺傳結構與分化以及聚類與遺傳距離分析比較,以期明確廣西三江地方茶樹種質資源遺傳多樣性現狀,旨在為三江茶專用新品種選育提供理論基礎。
1 材料與方法
1.1 試驗材料
2010年采集廣西柳州三江侗族自治縣域內形態上具有代表性的自然生長茶樹枝梢,采用扦插繁育后種植于三江縣多耶樓茶業有限公司本地茶樹種質資源圃,在經過對總體樣本進行形態學分析的基礎上,選出具有代表性的三江地方茶樹種質資源72份(編號為1~72),同時以5個(CK1~CK5)外省引進的茶樹優良品種作為對照,基本信息見表1。每份茶樹品種資源采集10~15片中部無害蟲的嫩葉,使用變色硅膠干燥,在–20?℃下保存。
1.2 茶樹基因組DNA的提取與檢測
取經過干燥處理的茶樹嫩葉,采用植物基因組DNA快速抽提試劑盒CW0531S(江蘇康為世紀生物科技股份有限公司)提取樣本DNA。使用0.8%的瓊脂糖凝膠電泳法檢測DNA完整性,采用超微量分光光度計(NanoDrop 1000)對DNA濃度和純度進行評估。合格DNA樣品用超純水稀釋至20?mg·L-1,置于冰箱?20?℃保存備用。
1.3 引物的篩選
本研究使用的ISSR引物來源于哥倫比亞大學公布的第9套100個ISSR通用引物序列[14];SRAP引物參考文獻[15-16]設計的234對SRAP引物組合,共包含13條上游引物(Me1~Me13)和18條下游引物(Em1~Em18)。引物的篩選分為初篩和復篩2個步驟,選取11個表型性狀差異較明顯的資源與5個參照品種作為初篩樣本,選出擴增產物主帶明顯、條帶清晰的引物;使用所有樣本進行復篩,選擇多態性高、擴增條帶清晰、重復性好的引物作為最終引物;最后以每條引物Tm值為基礎溫度,設置12個溫度梯度,根據擴增條帶的電泳結果確定每個引物的最適退火溫度。
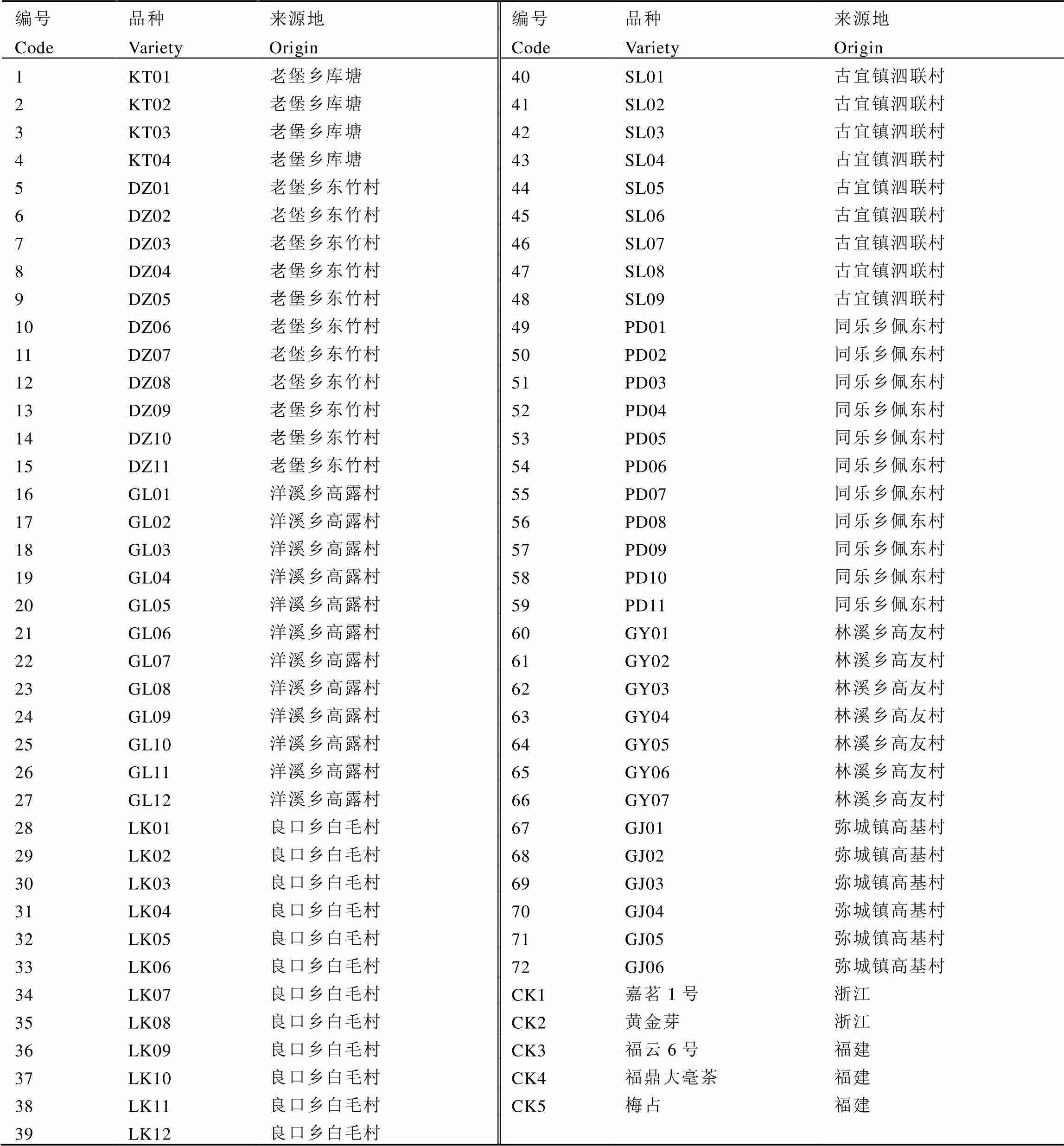
表1 茶樹種源信息
1.4 PCR擴增
ISSR-PCR擴增體系(25?μL):1?μL DNA模板、1?μL引物(20?mg·L-1,上下游引物各0.5?μL)、12.5?μL Powerpol 2X PCR Mix、10.5?μLddH2O。ISSR-PCR擴增程序:98?℃預變性45?s;98?℃變性10?s,退火(每個引物退火溫度見表2)30?s,72?℃延伸100?s,共30個循環;72?℃延伸5?min,4?℃保存。
SRAP-PCR擴增體系(25?μL):1?μL DNA模板、1?μL引物(20?mg·L-1,上下游引物各0.5?μL)、12.5?μL Powerpol 2X PCR Mix、10.5?μL ddH2O。SRAP-PCR擴增程序:98?℃預變性45?s;98?℃變性10?s,退火(每個引物退火溫度見表2)30?s,72?℃延伸100?s,共32個循環;72?℃延伸5?min,4?℃保存。
PCR擴增產物在含有Gelred核酸染料的1%瓊脂糖凝膠0.5×TAE緩沖液中,130?V電泳40?min后,通過超靈敏多功能凝膠成像儀(Amersham Imager 600)拍照保存。
1.5 數據統計及分析
人工讀取電泳結果數據,并進行統計處理。將條帶中清晰可見以及可重復的標記為“1”,同一位置上條帶缺失或者弱帶標記為“0”,形成“0、1”數據矩陣,分別得到ISSR、SRAP及綜合兩者(ISSR+SRAP)的數據。
1.5.1 遺傳多樣性分析
利用Excel統計每個引物或每對引物組合擴增的條帶以及多態性條帶數,計算多態位點百分率(PPB),利用POPGENE 1.32計算遺傳參數,如觀察到的等位基因數量(Na)、有效等位基因數(Ne)、Nei's基因多樣性指數(H)、Shannon信息指數(I)。
1.5.2 遺傳結構與分化分析
利用Structure軟件[17],對群體進行K為2~6的貝葉斯聚類,估算最佳群體組群數K和個體Q矩陣,利用Structure Harvester[18]計算lnP(K)和數據對數概率變化率(ΔK),利用Popgene 1.32軟件計算群體內多樣性(Hs)、總基因多樣性和基因流(Nm)。
1.5.3 聚類分析
利用Popgene 1.32軟件得到聚類分析圖,然后使用MEGA 7.0和Adobe Illustrator 2021構建聚類分析圖。利用GenAlEx 6.2加載宏軟件進行主坐標分析(PCoA)。
2 結果與分析
2.1 引物篩選結果與多態性分析
ISSR引物經過篩選,共獲得12個條帶清晰、多態性高且重復性好的引物,引物基本信息見表2。12個引物共擴增出124個條帶,多態性條帶有122條,PPB為98.39%。12個引物平均擴增條帶數為10.33條,平均多態性條帶數為10.17條,PPB最低的引物是UBC844,為84.62%,其余引物PPB均為100%,所有引物均表現出極高的多態性。擴增位點數最多的引物是UBC844,擴增出了13個條帶;其次是引物UBC835、UBC841和UBC845,均擴增出12個條帶;擴增條帶最少的引物是UBC873,只擴增出8條。以引物UBC835為例,其對72個樣本和5個對照的ISSR-PCR的電泳結果如圖1所示。
SRAP引物組合經過篩選,共獲得16個條帶清晰、多態性高且重復性好的引物組合,引物組合基本信息見表3。16個SRAP引物組合共擴增出196個條帶,多態性條帶有193條,PPB為98.47%。16條引物組合平均擴增條帶數為12.25條,平均多態性條帶數為12.06條,PPB最低的引物組合是Me7/Em6和Me10/Em3,為92.86%,所有引物均表現出極高的多態性。擴增位點數最多的引物組合是Me5/Em8,擴出了17條條帶;其次是引物組合Me2/Em15,擴增出16條條帶;擴增條帶最少的引物組合是Me1/Em15,只擴增出6條。以引物組合Me10/Em3為例,其對72個樣本和和5個對照的SRAP-PCR的電泳結果如圖2所示。
綜合ISSR和SRAP分析結果可知,兩個標記共擴增出320個條帶,多態性條帶有315條,PPB為98.44%,平均擴增條帶數為11.43條,平均多態性條帶數為11.25條。
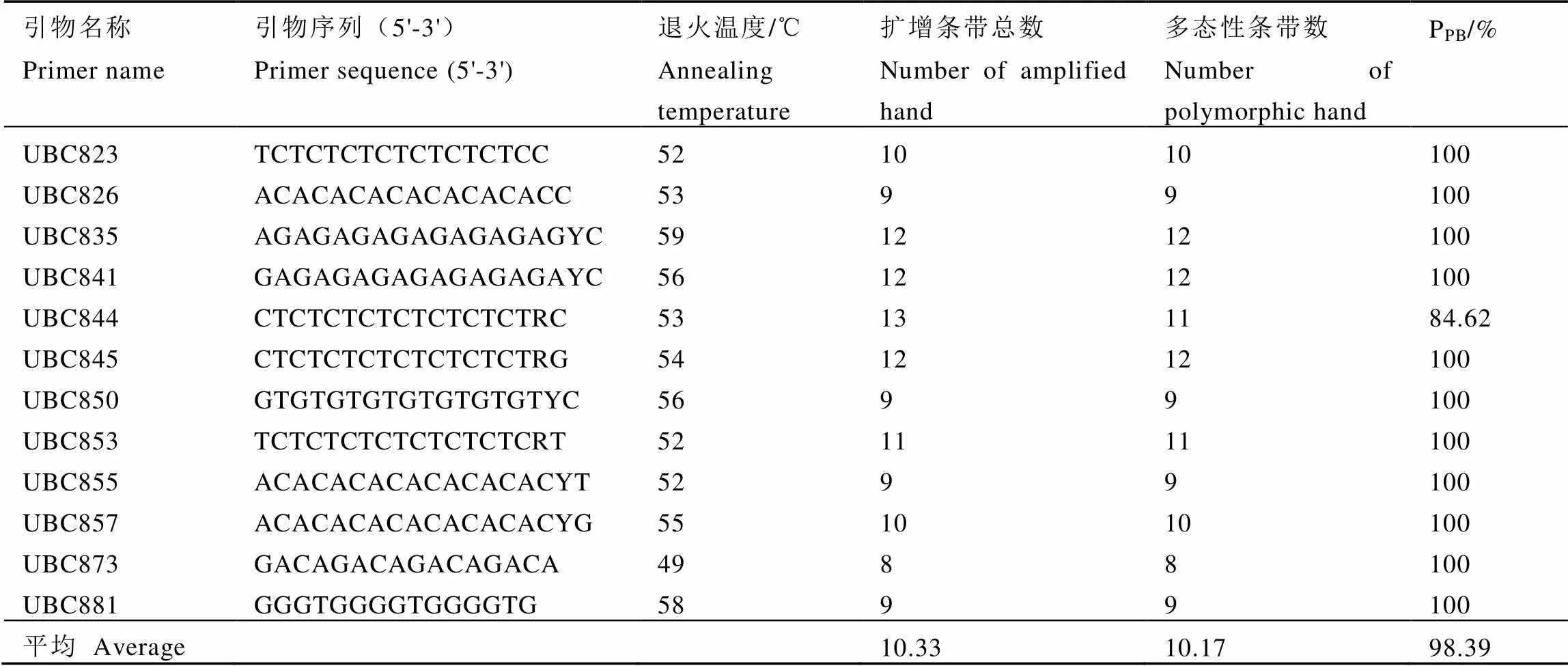
表2 ISSR引物信息及擴增結果

注:M為Marker,1~72代表72份三江縣茶樹種質資源,73~77代表CK1~CK5
2.2 遺傳多樣性分析
將所選的72份三江地方茶樹種質資源使用Popgene 1.32軟件計算等位基因數量(Na)、有效等位基因數(Ne)、Nei's基因多樣性指數(H)、Shannon信息指數(I)等相關系數,結果如表4所示。
ISSR引物檢測到的平均Na為1.99,Ne為1.47,H為0.29,I為0.44;SRAP引物組合檢測到的平均Na為1.99,Ne為1.49,H為0.30,I為0.46,兩種標記的結果趨于一致。72份三江地方茶樹種質資源的平均Na為1.99,Ne為1.48,H為0.30,I為0.45。
2.3 遺傳分化與遺傳結構分析
將參試的77份茶樹種質資源按照種源地劃為3個群體(廣西、浙江和福建),計算群體內多樣性(Hs)、總基因多樣性(Ht)和基因流(Nm)等遺傳參數。結果表明(表4),ISSR引物檢測的Ht和Hs分別為0.30和0.28;SRAP引物檢測的Ht和Hs分別為0.31和0.29,兩種標記所得到的結果趨于一致。
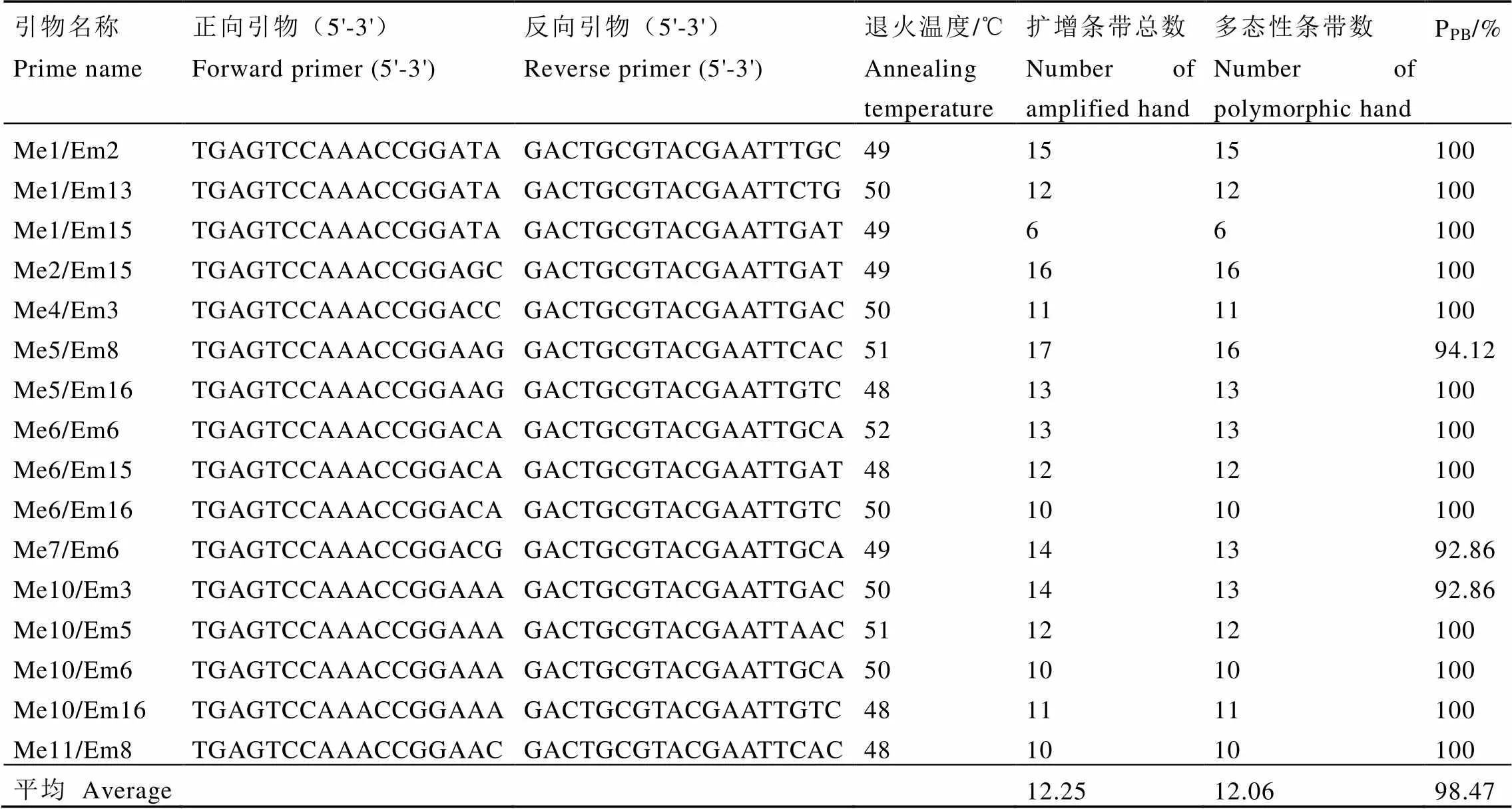
表3 SRAP引物信息及擴增結果

注:M為Marker,1~72代表72份三江縣茶樹種質資源,73~77代表CK1~CK5
結合ISSR標記和SRAP標記結果,77份茶樹種質資源的Ht為0.31,Hs為0.29;種群間分化系數(Gst)為0.056,則相應的種群內的Gst為0.944,基因流系數為8.64。通過ISSR和SRAP標記結果,單獨對2個對照群體(浙江群體與福建群體)進行遺傳結構分析,種群間Gst為0.251,Nm為1.49。
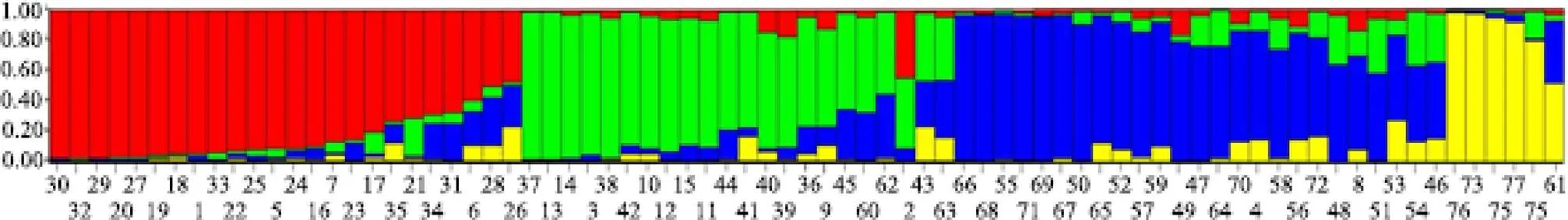
72份三江地方茶樹種質資源與5個引進品種的遺傳結構存在差異(圖3)。基于ΔK推測最佳亞群體數,K的最大值出現在K=4,ΔK=37.65。當假設供試茶樹種質資源存在4個亞群體(K=4)時,根據Q矩陣排列結果,GY02與5個引進品種聚為1個亞群,其余茶樹種質資源則分為另外3個亞群。
2.4 聚類與遺傳距離分析
分別利用ISSR標記、SRAP標記、以及ISSR+SRAP標記的基因型數據對77份茶樹種質資源進行非加權組平均法(UPGMA)聚類。基于ISSR標記的UPGMA樹狀圖顯示(圖4),樣本間遺傳相似系數為0.726~0.960,變幅為0.234。在遺傳相似系數為0.750時,可將全部樣本劃分為4個亞群。第1亞群包括除了GY02和LK12之外的70份三江茶樹種質資源,第2亞群為LK12,第3亞群為GY02,第4亞群為5個引進品種。樣本間的遺傳距離為0.041?2 ~0.777?2,其中LK12和黃金芽的遺傳距離最遠,遺傳距離最近的是GJ02和GJ03。
基于SRAP標記的UPGMA樹狀圖顯示(圖5),樣本間遺傳相似系數為0.515~0.882,變幅為0.367。其中,在遺傳相似系數為0.535時,可將全部樣本劃分為3個亞群。第1亞群包括除GY02之外的71份三江茶樹種質資源;第2亞群包含GY02、嘉茗1號、福云6號、福鼎大毫茶和梅占;第3亞群為黃金芽。樣本間的遺傳距離為0.125~0.663,其中LK06和福鼎大毫茶的遺傳距離最遠,遺傳距離最近的是GJ02和GJ03。
基于ISSR+SRAP標記的UPGMA樹狀圖顯示(圖6),樣本間遺傳相似系數為0.534~0.913,變幅為0.379。其中,在遺傳相似系數為0.572時,可將全部樣本分為3簇。第1簇包括71個三江茶樹種質資源,第2簇為GY02,第3簇為5個引進茶樹品種。樣本間的遺傳距離為0.091?6~0.626?7,其中遺傳距離最遠的為LK06和福云6號,遺傳距離最近的是GJ02和GJ03。
使用Genalex加載宏將兩種標記結果進行PCoA分析(圖7),兩個PCoA軸可以分別解釋12.65%和8.28%的遺傳變異。將主坐標分析圖分成Ⅰ、Ⅱ、Ⅲ、Ⅳ 4個區域。三江茶樹種質資源主要分布在Ⅰ、Ⅲ、Ⅳ區,小部分分布于Ⅱ區且大部分散點較為集中,而5個參照品種均分布于Ⅳ區,且與三江的種質資源距離較遠。雙標記的PCoA結果與UPGMA聚類結果基本一致。
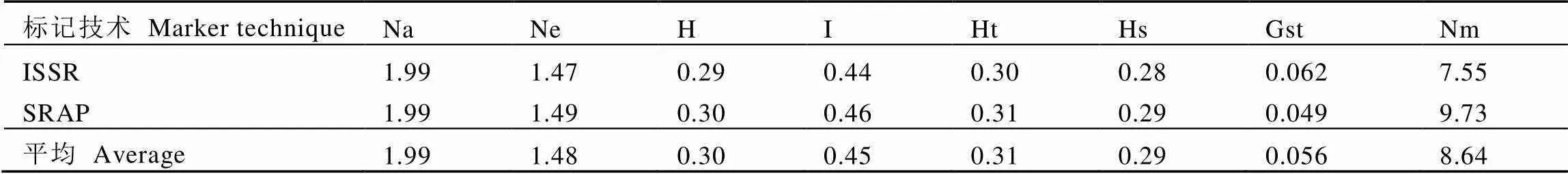
表4 遺傳多樣性與遺傳分化系數
注:1~72代表72份三江縣茶樹種質資源,73~77代表CK1~CK5
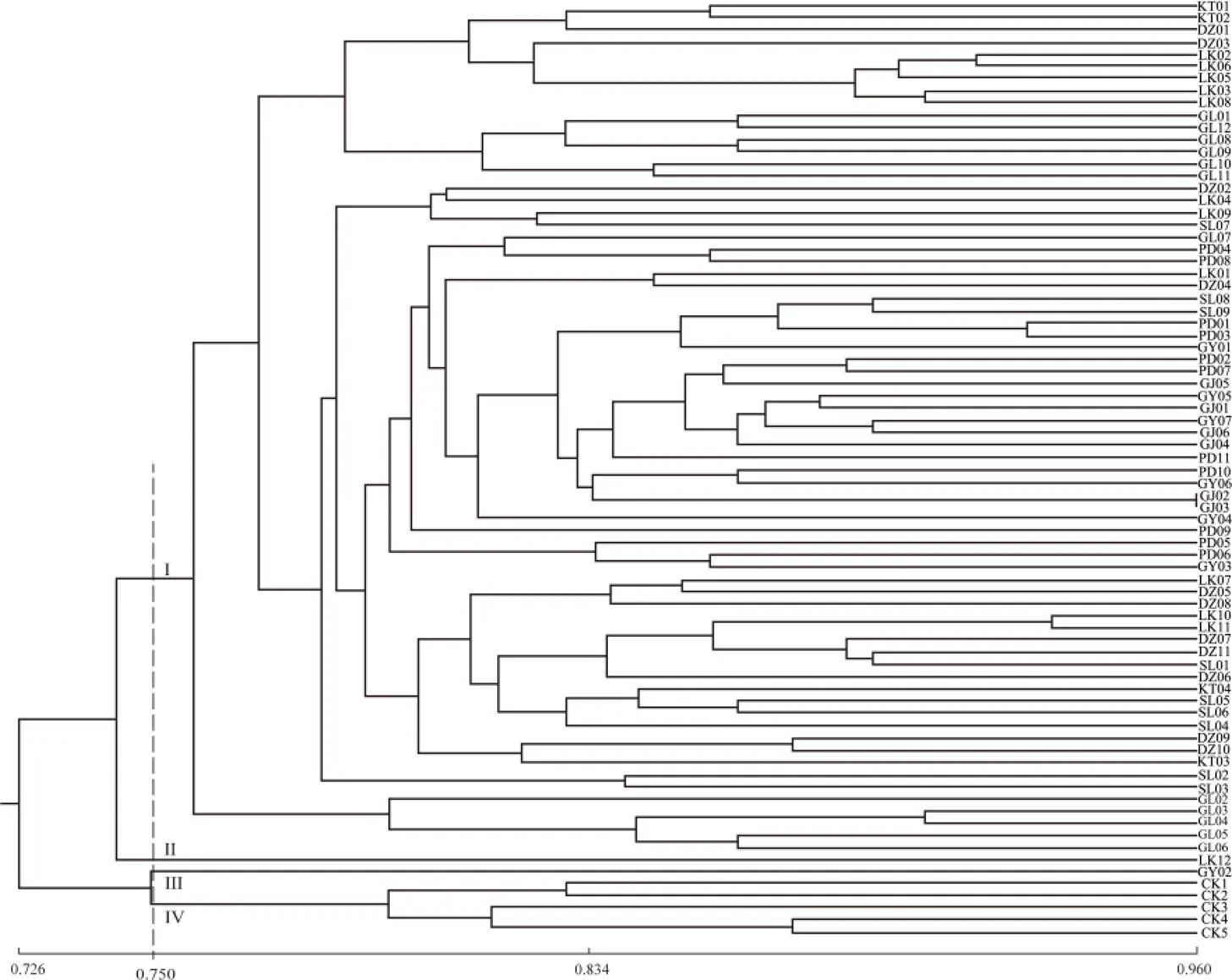
圖4 ISSR標記77份茶樹種質資源的UPGMA聚類圖

圖5 SRAP標記77份茶樹種質資源的UPGMA聚類圖

圖6 ISSR+SRAP標記77份茶樹種質資源的UPGMA聚類圖
3 討論
對茶樹種質資源進行遺傳多樣性評估,可以全面有效反映茶樹群體的品種現狀,也是品種選育和種質創新的基礎。本研究中使用12個ISSR引物和16個SRAP引物組合標記對72個三江茶樹種質資源進行遺傳多樣性分析,共檢測到320個等位基因條帶,其中多態性條帶315個,PPB平均值達到了98.44%,ISSR與SRAP標記的Na平均值均為1.99。劉彤等[19]利用ISSR分子標記分析了柳州九萬山93份茶樹種質資源,結果顯示,112個等位基因條帶平均多態比例為93.8%;鄭丹琳等[20]利用ISSR分子標記對柳州汪洞鄉87份種質資源進行分析,10條引物共擴增出67條譜帶,多態比例達95.5%。前人的研究和本研究的結果相似,標記的引物均顯示極高的多態性比例,說明柳北區域融水——三江地區茶樹種質資源在分子水平上遺傳多樣性水平高。同時也驗證了本研究中篩選出的ISSR引物和SRAP引物組合對試驗材料的遺傳多樣性檢測效率高,可有效揭示茶樹群體的遺傳多樣性。從擴增位點數來看,SRAP的檢測效率略高于ISSR的檢測效率。
雙標記結果分析顯示,Nei's基因多樣性指數(H)和香農信息指數(I)的平均值分別為0.30和0.45;與寧靜等[21]基于ISSR分子標記對湖南黃金茶群體遺傳多樣性分析結果(H=0.30,I=0.45)相似,略低于李丹等[22]基于ISSR分子標記對江華苦茶群體進行遺傳多樣性分析結果(H=0.38,I=0.56),但高于陳濤林等[23]基于ISSR分子標記對汝城白毛茶群體的分析結果(H=0.26,I=0.40)。與其他研究對比,三江茶樹種群遺傳多樣性處于較高的水平。
綜合本研究ISSR和SRAP數據,3個茶樹種群間的變異為5.6%,而種群內的變異系數為94.4%,Nm指基因在群體間或群體內的運動[24],普遍認為,當Nm<1時,造成遺傳分化的主要原因是遺傳漂變;當Nm>1,基因流阻礙了基因漂變,進而出現遺傳分化[25]。本研究中,雙標記的Nm為8.64,群體間的Nm系數均大于1,說明本研究所標記的種群間遺傳分化遠高于種群內遺傳分化,與Yao等[26]對全國450份茶樹核心種質資源遺傳結構和分化研究結果相似。茶樹進化過程中,由于自然或人工選擇、隨機漂變等原因,茶樹群體會出現遺傳分化[27],并且茶樹雌雄同株、風媒異花授粉的植物學特性[28],使得所選的種群遺傳變異的大多數來自種群內部。各群體之間的生境異質性較大,環境效應增強了三江茶樹群體與對照茶樹種質群體間的遺傳分化水平,而在三江茶樹種群內近交,使等位基因均質化,頻繁的基因交流也阻止了遺傳分化的發生。本研究所有樣品均采自三江地區,但引進品種在該地栽培年限較短,基因有效交流頻率在自然條件下較低,出現本地種源與外來品種在基因水平上分化較為明顯的結果。
聚類分析結果表明,當77份茶樹種質資源分為兩個亞群時,第一個亞群為除GY02之外的所有三江茶樹資源,而GY02與5個參照茶樹種則聚為另一個亞群。在Structure遺傳結構分析圖中,假設所選的種質資源分為4個亞群,GY02與5個參照品種聚為一類。說明GY02與三江地方茶樹種質資源在分子水平上差異較為明顯。根據初步形態學標記的結果顯示,三江茶樹群體的芽頭茸毛總體呈現出無茸毛或少茸毛特點,而GY02芽頭茸毛密,春季的生長勢強,在茶樹群體種中有較高辨識度,多項形態學指標與三江群體種存在特異性。3個聚類結果均顯示,GJ02和GJ03的遺傳一致性最高,且遺傳距離系數最小,說明GJ02和GJ03親緣關系較為接近。
ISSR聚類結果中,三江種質資源的亞群里,LK12在更高系數的遺傳相似性比較中被劃分成單獨一組,而在SRAP聚類結果中,除GY02以外的所有三江茶樹種質資源聚為一個亞群。推測針對基因組不同片段的不同標記,單個標記不能完全反映群體之間的關系。由于ISSR和SRAP標記都是顯性標記,因此本研究將兩個標記結合起來進行分析,避免單個標記的限制。兩種分子標記的遺傳距離結果都顯示三江地方茶樹種質資源與外省引進品種具有明顯的分化。
三江的茶樹種群經過長期的自然選擇和遺傳進化,出現一批適應當地地理氣候的茶樹品種,說明三江茶樹種質資源豐富,具有良好的開發前景。目前已經從中選育了多耶樓1號和多耶樓2號兩個茶樹新品種,2022年獲得了農業農村部植物新品種權授權;另外還有3個品種已經通過了植物新品種特異性、一致性和穩定性(Distinctness,uniformity,stability,DUS)現場考察。本研究中的72份茶樹種質資源的遺傳多樣性較為豐富,結合聚類和農藝、品質性狀分析結果,選用綜合性狀優異且有一定遺傳差異的材料作為育種親本,對三江茶樹種質遺傳改良和拓寬遺傳多樣性具有一定的現實意義。
[1] 楊世雄. 廣西的茶樹資源[J]. 廣西林業科學, 2021, 50(4): 414-416. Yang S X. Tea-plant () in Guangxi, China [J]. Guangxi Forestry Science, 2021, 50(4): 414-416
[2] 賈繼增, 高麗鋒, 趙光耀, 等. 作物基因組學與作物科學革命[J]. 中國農業科學, 2015, 48(17): 3316-3347. Jia J Z, Gao L F, Zhao G Y, et al. Crop genomics and crop science revolutions [J]. Scientia Agricultura Sinica, 2015, 48(17): 3316-3347.
[3] 毛娟, 江鴻鍵, 楊如兵, 等. 野生和栽培大理茶居群的遺傳多樣性與群體結構[J]. 茶葉科學, 2021, 41(4): 454-462. Mao J, Jiang H J, Yang R B, et al. Genetic diversity and population structure of wild and cultivatedpopulations [J]. Journal of Tea Science, 2021, 41(4): 454-462.
[4] 李燕平, 譚艷平, 徐鑫, 等. 水稻特異分子標記的建立及其在新品種駿優522的應用[J]. 農業生物技術學報, 2013, 21(11): 1295-1301. Li Y P, Tan Y P, Xu X, et al. Establishment of specific molecular markers and application for new variety Junyou 522 in rice () [J]. Journal of Agricultural Biotechnology, 2013, 21(11): 1295-1301.
[5] 段夢冉, 劉豐澤, 葛建镕, 等. 吉林省主推玉米品種的SSR分子標記純度鑒定[J]. 作物雜志, 2022(5): 34-41. Duan M R, Liu F Z, Ge J R, et al. Purity identification of SSR molecular markers for main maize varieties in Jilin Province [J]. Crops, 2022(5): 34-41.
[6] 徐榮, 徐永艷, 何麗蓮, 等. 甘蔗野生近緣種遺傳多樣性的ISSR分析[J]. 西南農業學報, 2022, 35(2): 264-270. Xu R, Xu Y Y, He L L, et al. Genetic diversity of wild cane species revealed by ISSR markers [J]. Southwest China Journal of Agricultural Sciences, 2022, 35(2): 264-270
[7] 劉青, 趙德剛, 趙懿琛. 古茶樹種質資源遺傳多樣性ISSR分析[J]. 種子, 2021, 40(5): 7-14. Liu Q, Zhao D G, Zhao Y C. Genetic diversity analysis by ISSR markers for germplasm resources of ancient tea tree [J]. Seed, 2021, 40(5): 7-14.
[8] 孫雪梅, 趙才美, 李友勇, 等. 云南茶樹地方品種與野生茶遺傳多樣性的ISSR分析[J]. 西南農業學報, 2018, 31(5): 901-906. Sun X M, Zhao C M, Li Y Y, et al. Genetic diversity of tea landraces and wild tea germplasmsin Yunnan revealed by ISSR analysis [J]. Southwest China Journal of Agricultural Science, 2018, 31(5): 901-906.
[9] Li G, Quiros C F. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica [J]. Theoretical and Applied Genetics, 2001, 103(2): 455-461.
[10] 李巧燕, 林瑞慶, 朱興全. SRAP分子標記及其應用概述[J]. 熱帶醫學雜志, 2006(4): 467-469, 478. Li Q Y, Lin R Q, Zhu X Q. SRAP overview of molecular markers and their applications [J]. Journal of Tropical Medicine, 2006(4): 467-469, 478.
[11] 林萍, 姚小華, 王開良, 等. 油茶長林系列優良無性系的SRAP分子鑒別及遺傳分析[J]. 農業生物技術學報, 2010, 18(2): 272-279. Lin P, Yao X H, Wang K L, et al. Identification and genetic analysis ofChanglin series superior clones by SRAP molecular marker [J]. Journal of Agricultural Biotechnology, 2010, 18(2): 272-279.
[12] Zhang Y, Zhang X, Chen X, et al. Genetic diversity and structure of tea plant in Qinba area in China by three types of molecular markers [J]. Hereditas, 2018, 155: 22. doi: 10.1186/s41065-018-0058-4.
[13] 黃錦春, 萬思琦, 陳揚, 等. 利用ISSR與SRAP分子標記分析金線蓮種質資源遺傳多樣性[J]. 浙江農林大學學報, 2023, 40(1): 22-29. Huang J C, Wan S Q, Chen Y, et al. Genetic diversity ofbased on ISSR and SRAP molecular markers [J]. Journal of Zhejiang A&F University, 2023, 40(1): 22-29.
[14] 付銓盛. 毛冬青ISSR指紋圖譜的建立及成分動態規律初探[D]. 廣州: 廣州中醫藥大學, 2016: 16-17. Fu Q S. Establish fingerprint forbased on ISSR PCR and preliminary of ingredients cumulative [D]. Guangzhou: Guangzhou University of Chinese Medicine, 2016: 16-17
[15] Ferriol M, Picó B, Nuez F. Genetic diversity of a germplasm collection ofusing SRAP and AFLP markers [J]. Theoretical and Applied Genetics, 2003, 107(2): 271-282.
[16] Budak H, Shearman R C, Parmaksiz I, et al. Molecular characterization of Buffalograss germplasm using sequence-related amplified polymorphism markers [J]. Theoretical and Applied Genetics, 2004, 108(2): 328-334.
[17] Pritchard J K, Stephens M, Donnelly P. Inference of population structure using multilocus genotype data [J]. Genetics, 2000, 155(2): 945-959.
[18] Earl D A, Vonholdt B M. STRUCTURE HARVESTER: a website and program for visualizing STRUCTURE output and implementing the Evanno method [J]. Conservation Genetics Resources, 2012, 4(2): 359-361.
[19] 劉彤, 葛智文, 陳濤林, 等. 柳州九萬山野生茶樹種質資源遺傳多樣性的ISSR分析[J]. 南方農業學報, 2015, 46(11): 1943-1948. Liu T, Ge Z W, Chen T L, et al. Genetic diversity of wild tea germplasm resources in Liuzhou Jiuwan Mountain revealed by ISSR markers [J]. Journal of Southern Agriculture, 2015, 46(11): 1943-1948.
[20] 鄭丹琳, 陳濤林, 葛智文, 等. 柳州汪洞鄉古茶樹種質資源遺傳多樣性的ISSR分析[J]. 分子植物育種, 2018, 16(11): 3629-3635. Zheng D L, Chen T L, Ge Z W, et al. ISSR analysis of the genetic diversity of ancient tea plant germplasm resources from Wangdong Village in Liuzhou [J]. Molecular Plant Breeding, 2018, 16(11): 3629-3635.
[21] 寧靜, 李健權, 董麗娟, 等. “黃金茶”特異種質資源遺傳多樣性和親緣關系的ISSR分析[J]. 茶葉科學, 2010, 30(2): 149-156. Ning J, Li J Q, Dong L J, et al. Genetic diversity and relationship of tea germplasm resources ‘Huangjincha’ () revealed by ISSR markers [J]. Journal of Tea Science, 2010, 30(2): 149-156.
[22] 李丹, 李端生, 楊春, 等. 江華苦茶種質資源遺傳多樣性和親緣關系的ISSR分析[J]. 茶葉科學, 2012, 32(2): 135-141. Li D, Li R S, Yang C, et al. Genetic diversity and relationship of tea germplasm resourcesvar.cv. Jianghua revealed by ISSR markers [J]. Journal of Tea Science, 2012, 32(2): 135-141.
[23] 陳濤林, 王漢超, 羅軍武, 等. 汝城白毛茶種質資源遺傳多樣性和親緣關系的ISSR分析[J]. 分子植物育種, 2017, 15(9): 3780-3787. Chen T L, Wang H C, Luo J W, et al. Genetic diversity and relationship of tea germplasm resourcesvar.cv. Rucheng revealed by ISSR markers [J]. Molecular Plant Breeding, 2017, 15(9): 3780-3787.
[24] Grant V. The evolutionary process. A critical study of evolutionary theory [B]. New York: Columbia University Press, 1991.
[25] Zhang Z, Yang Q, Niu Y, et al. Diversity analysis and establishment of core collection among(Thunb.) Koidz. in Qinba mountain area of China using ISSR and SRAP markers [J]. Genetic Resources and Crop Evolution, 2021, 68(3): 1-18.
[26] Yao M Z, Ma C L, Qiao T T, et al. Diversity distribution and population structure of tea germplasms in China revealed by EST-SSR markers [J]. Tree Genetics & Genomes, 2012, 8: 205-220.
[27] Wachira F N, Waugh R, Hackett C A, et al. Detection ofgenetic diversity in tea () using RAPD markers [J]. Genome, 1995, 38(2): 201-210.
[28] 束際林, 陳亮. 茶樹花粉形態的演化趨勢[J]. 茶葉科學, 1996, 16(2): 37-40. Shu J L, Chen L. Study on the evolution route of tea pollen morphology [J]. Journal of Tea Science, 1996, 16(2): 37-40.
Genetic Diversity Analysis of Tea Genetic Resources in Sanjiang, Guangxi
MENG Rongjun1, CHEN Liang2, XU Yuan1, LIN Wei3, ZHOU Qiwei3, XIE Yilin3, LAI Dingqing4*, LAI Jiaye3*
1. College of Forestry, Guangxi University, Nanning 530004, China; 2.Tea Research Institute, Chinese Academy of Agricultural Sciences, Hangzhou 310008, China; 3. Institute of Agriculture and Animal Husbandry Development, Guangxi University, Nanning 530004, China; 4. Sanjiang County Duoyelou Tea Industry Co., Ltd., Sanjiang 545500, China
In order to clarify the genetic diversity of tea genetic resources in Sanjiang, Guangxi, two marker techniques, simple inter-repeat sequence amplification (ISSR) and correlated sequence amplification polymorphism (SRAP) were used to analyze the genetic diversity of 72 local tea genetic resources and five introduced cultivars in Sanjiang region. The results of both marker techniques show high levels of genetic diversity (H=0.30, I=0.44), high genetic differentiation between populations (Gst=0.056) and gene flow (Nm=8.64). The genetic variation observed within populations was much higher than that between populations. The cluster analysis shows that most of the tea genetic resources in Sanjiang were clustered into one group and the introduced cultivars were clustered into the other, which was consistent with the results of PCoA. This study provided evidence at the molecular level for the protection and utilization of tea germplasmin Sanjiang.
, molecular marker, genetic diversity, population structure
S571.1
A
1000-369X(2023)02-147-12
2022-12-15
2023-01-19
三江縣本地茶樹產業科技創新示范基地建設項目(桂科AD19245192)
蒙容君,男,碩士研究生,主要從事林木(包括茶樹)遺傳育種研究。*通信作者:752102015@qq.com;laijiayie@126.com

