基于滇黃精轉錄組序列的SSR標記開發及其在黃精屬資源分析中的應用
錢麗華 嚴建立 吳曉疆 阮松林 尹舒雅 崔海瑞


摘要:本研究基于滇黃精轉錄組序列開發簡單重復序列(SSR)標記并將其應用于黃精屬資源分析。設計合成了45對SSR引物,經PCR擴增驗證,選擇其中20對SSR引物對75份黃精屬資源進行分析。結果表明,共在46 416個Unigene中檢出含有二核苷酸~六核苷酸重復類型的SSR位點60 238個,序列SSR發生頻率為22.78%,平均分布距離約7.07 kb;SSR位點中的主導類型是二核苷酸和三核苷酸重復,分別占50.06%和34.89%。測試的45對SSR引物中有34 對(75.56%)可擴增SSR條帶。篩選的20對引物共擴增出153個條帶,多態率為98.69%,每對引物擴增條帶4.00~14.00個,平均7.65個,不同SSR標記的多態性信息含量為0.626~0.973,平均為0.870。75份材料的等位基因數和遺傳相似系數分別為7.00~52.00個和0.531~0.941,平均值分別為24.65個和0.689,顯示出豐富的遺傳多樣性。基于SSR標記分析的聚類圖顯示,在遺傳相似系數0.666處可將供試材料分為4類,較好地反映了供試材料的分類歸屬。此外,還發現5份多花黃精材料具有特異性的SSR條帶擴增或缺失,可作為不同多花黃精材料鑒定的重要分子依據。本研究開發的SSR標記多態性較高,能夠有效揭示黃精屬種質資源的遺傳多樣性,對于豐富黃精分子標記種類、構建遺傳圖譜、促進種質資源的評價與育種應用、開展特定性狀的輔助選擇等研究都具有重要的意義。
關鍵詞:黃精;轉錄組;SSR標記開發;資源分析
中圖分類號:S567.2文獻標識碼:A文章編號:1000-4440(2023)05-1120-12
Development of simple sequence repeat (SSR) markers based on transcriptome sequences of Polygonatum kingianum and their application in analysis of Polygonatum germplasm resourcesQIAN Li-hua1,YAN Jian-li1,WU Xiao-jiang2,RUAN Song-lin1,YIN Shu-ya1,CUI Hai-rui2
(1.Hangzhou Academy of Agricultural Sciences, Hangzhou 310024, China;2.Institute of Nuclear-Agricultural Sciences, Zhejiang University, Hangzhou 310058, China)
Abstract:In this study, simple sequence repeat (SSR) markers were developed based on transcriptome sequence data of Polygonatum kingianum and were utilized in germplasm resource analysis of Polygonatum. Forty-five pairs of SSR primers were designed and synthetized, then 20 of them were selected by PCR amplification verification test to analyse 75 Polygonatum resources. The results showed that, a total of 60 238 SSR loci including dinucleotide to hexanucleotide repeats were detected from 46 416 Unigenes in P. kingianum transcriptome, with an occurring frequency of 22.78% and an average distribution distance of 7.07 kb. Dinucleotide repeat and trinucleotide repeat were the dominant types, with frequencies of 50.06% and 34.89% of all SSRs, respectively. Among 45 pairs of SSR primers, 34 of them showed amplification ability, which accounted for 75.56%. A total of 153 bands were amplified by the selected 20 pairs of primers, with a polymorphism rate of 98.69%. Each pair of primer could amplify 4.00-14.00 bands, with an average of 7.65 bands. The polymorphic information content varied from 0.626 to 0.973 for different SSR markers, with an average value of 0.870. Number of allele genes and genetic similarity coefficients of 75 materials were 7.00-52.00 and 0.531-0.941 respectively, and the average values were respectively 24.65 and 0.689, which displayed rich genetic diversity. A dendrogram constructed based on SSR markers showed that, the test materials could be classified into four cluster groups with the genetic similarity coefficient of 0.666, which presented a good reflection of affiliation in taxonomy of tested Polygonatum materials. In addition, five P. cyrtonema materials showed specific present or absent SSR bands, which could be used as important molecular basis for identification of different materials. The SSR markers developed in this study were highly polymorphic and could be used to effectively reveal the genetic diversity of the Polygonatum germplasm resources. The markers are of great significance for enriching molecular marker types, constructing genetic linkage map, promoting the evaluation and breeding application of germplasm resources, and carrying out assisted selection of specific traits in Polygonatum.
Key words:Polygonatum;transcriptome;simple sequence repeat (SSR) marker development;resource analysis
黃精是多年生草本植物,屬百合科(Liliaceae)黃精屬(Polygonatum Mill),全世界有70余種,主要分布于北半球,分布地包括中國、朝鮮半島、俄羅斯、歐洲和北美等[1]。滇黃精(Polygonatum kingianum)、黃精(P. sibiricum)和多花黃精(P. cyrtonema)的根莖統稱為黃精,含有甾體皂苷類、多糖類、黃酮類、生物堿類等多種活性成分,在抗衰老、抗腫瘤、免疫調節、抗菌、抗病毒、降血糖血脂等方面具有重要的藥用價值[1-2],作為藥食同源藥材,黃精在保健品、食品、日化領域都有廣泛應用。
種質資源的鑒定與科學評價是合理利用遺傳資源和選育新品種的基礎。中國黃精屬種質資源有30余種[3],傳統的鑒別大多依據產地和形態,但因其植物形態上具有過渡性,生藥學性狀較相似,地理分布存在重疊性,種間也常伴有雜交現象而界限模糊,導致其分類和種的識別與鑒定十分困難,且存在爭議[4-6]。與傳統鑒別方法相比,DNA分子鑒定技術可在基因組水平上揭示植物存在差異的內在遺傳本質,因其客觀性與準確性迅速成為傳統中藥鑒定的重要手段,并在黃精屬植物中得到了應用[7-8]。
簡單重復序列(SSR)是指基因組中以幾個核苷酸(一般1~6個)為基本單位的重復序列重復多次構成的一段DNA,其長度具有高度變異性,是建立多態性DNA標記的資源[9],其種屬特異性強,在品種鑒別和育種中應用很廣[10-11]。在黃精屬植物研究中,基于生物素富集法[12-13]、特異性長度擴增片段測序(SLAF-seq)[14]、基因組測序[15]、轉錄組測序[16-17]等不同策略,已開發了一些SSR引物,分析了引物在黃精屬植物間的可轉移性[12-13],并利用這些SSR標記進行了指紋圖譜構建[16]和遺傳多樣性分析[14-15]。這些研究為黃精屬植物的準確鑒別和進一步開展新品種選育奠定了基礎。
目前,黃精屬植物能夠利用的SSR 標記數量較少,尚不能滿足相關遺傳研究與育種的實際需要[14,17]。轉錄組測序技術是近年來新崛起的全面快速獲取物種特定組織或器官在某狀態下的所有轉錄本信息的研究方法,在藥用植物研究中也得到了廣泛應用[18-20]。轉錄組測序獲得的數據為SSR 標記的開發提供了良好的基礎。本研究對滇黃精轉錄組序列中的SSR位點與特征進行挖掘和分析,開發SSR 標記并將其用于75份黃精屬植物資源的分析,以期為黃精屬種質資源的合理利用與新品種選育提供科學依據。
1材料與方法
1.1供試材料
供試黃精屬植物材料75份,均種植于杭州市農業科學研究院黃精資源圃。其中多花黃精53份、黃精17份、長梗黃精4份、川黃精1份(表1)。
1.2基因組 DNA 提取
取黃精幼嫩葉片,洗凈晾干后按每份100~200 mg放入1.5 ml離心管中,加液氮用組織研磨器(上海凈信實業發展有限公司產品)在60 Hz頻率下磨至粉末狀,按DNA抽提試劑盒(上海生工生物工程股份有限公司產品)操作說明書提取DNA,采用微量分光光度計(Thermo Scientific公司產品)測定DNA濃度,經1%瓊脂糖凝膠電泳檢測DNA的質量和完整性,-15 ℃保存備用。
1.3序列來源與SSR挖掘
從美國國家生物技術信息中心(NCBI)網站(https://www.ncbi.nlm.nih.gov/bioproject/474626)下載滇黃精(Polygonatum kingianum)鞭芽轉錄組測序后組裝和拼接的Unigene序列,共203 772條,總長度約426 Mb[21]。利用SSRIT軟件對這些序列按二核苷酸、三核苷酸、四核苷酸至六核苷酸重復分別不少于6次、5次、4次的要求進行SSR查找,對查找出的SRR相關信息進行統計和分析。
1.4SSR引物設計與PCR
根據SSR相關信息統計和分析結果,參考重復基元的出現頻率和重復次數,選擇部分SSR用PRIMER 3(http://frodo.wi.mit.edu/)設計引物,引物由杭州擎科生物技術有限公司合成。
從參試樣品中隨機抽取10個樣本,將其DNA等量混合作為模板,并對引物進行不同退火溫度(50~62 ℃)的PCR 測試。SSR-PCR反應體系為:含7.5 μl的2×Taq Master Mix(北京百奧萊博科技有限公司產品)、1.0 μl DNA模板(約50 ng)、上下游引物各0.5 μl(終濃度0.4 μmol/L)和5.5 μl的超純水。PCR循環條件:94 ℃預變性5 min;35個循環,每個循環94 ℃變性45 s,適宜溫度退火45 s,72 ℃延伸45 s;72 ℃終延伸10 min。對75份黃精資源分析時PCR擴增所采用的退火溫度為58 ℃。
混合模板測試引物時采用1.5%的瓊脂糖凝膠電泳(AGE)檢測有無擴增產物。黃精資源分析時采用10%聚丙烯酰胺凝膠電泳檢測擴增產物,進樣量為1 μl,以110~120 V恒壓電泳約2.5 h,銀染檢測,掃描電泳圖譜并保存。
1.5擴增條帶的統計與分析
對所擴增產物的電泳圖譜進行條帶數量記錄(有記作1,無記作0),生成原始數據矩陣。根據 Botstein 等[22]描述的方法計算多態性信息含量(PIC值)。采用 NTSYS-pc 2.10 軟件分析數據,利用 SIMQUAL 程序計算各黃精材料間的遺傳相似系數,用非加權成對算術平均法(UPGMA)進行聚類生成聚類圖。
2結果與分析
2.1滇黃精轉錄組 SSR的分布、頻率及特征
對滇黃精203 772條Unigene序列搜索后發現46 416條序列含有SSR位點(表2)。其中36 003條序列只含1個SSR位點,其他10 413條序列含有2個及以上的SSR位點,個別序列所含SSR位點數高達9個。
在滇黃精總長度約426 Mb的轉錄組序列中共檢出60 238個SSR位點(表3),平均約7.07 kb檢測到1個SSR,序列發生頻率(含有SSR的序列數與序列總數之比)為22.78%,出現頻率(SSR總數與序列總數之比)為29.56%。
在檢出的SSR重復類型中,二核苷酸至六核苷酸重復均有出現,但頻率差異很大(表3)。其中二核苷酸和三核苷酸重復占主導地位,出現頻率分別為14.80%和10.31%,分別占總SSR的50.06%和34.89%;四核苷酸至六核苷酸重復數量較少,出現頻率分別為2.02%、0.86%和1.57%, 分別占SSR總數的6.82%、2.90%和5.33%。
滇黃精轉錄組序列中含有的SSR基元種類有262種,各基元重復次數為4~52次,基元越長,重復次數越少(表4)。盡管四核苷酸至六核苷酸SSR發生頻率較低,但其基元的種類卻較多,占重復基元總數的94.7%;而占據主導地位的二核苷酸和三核苷酸重復基元種類則較少,僅占重復基元總數的5.3%。
在二核苷酸重復基元中,出現頻率較高的依次是AG、AT、AC和CG,AG和 AT重復次數大多分布在6~12,而AC和CG則以6~9次重復為主(表5)。在三核苷酸重復基元中,出現較多的4種基元依次是AGG、AAG、AAT和ACG,占三核苷酸SSR的比例均在13.0%以上,其他6種基元出現的頻率較低,占三核苷酸SSR的比例為4.7%~8.1%。所有三核苷酸重復基元均以5~8次重復為主,重復次數在9次及以上的只占約3.2%。
2.2SSR引物篩選
根據滇黃精轉錄組序列中SSR的挖掘結果,設計合成了45對引物(表6),并進行溫度梯度PCR測試。經測試發現,11對引物(PkSSR3、PkSSR12、PkSSR15、PkSSR16、PkSSR17、PkSSR19、PkSSR20、PkSSR34、PkSSR38、PkSSR42、PkSSR45)無擴增產物,其余34對引物有擴增產物,可擴增引物約占75.56%,其中31對引物擴增出了預期大小的條帶,另外3對引物(PkSSR1、PkSSR13、PkSSR27)的擴增產物大小大于預期長度,圖1是部分引物的擴增結果。
2.3SSR引物的多態性
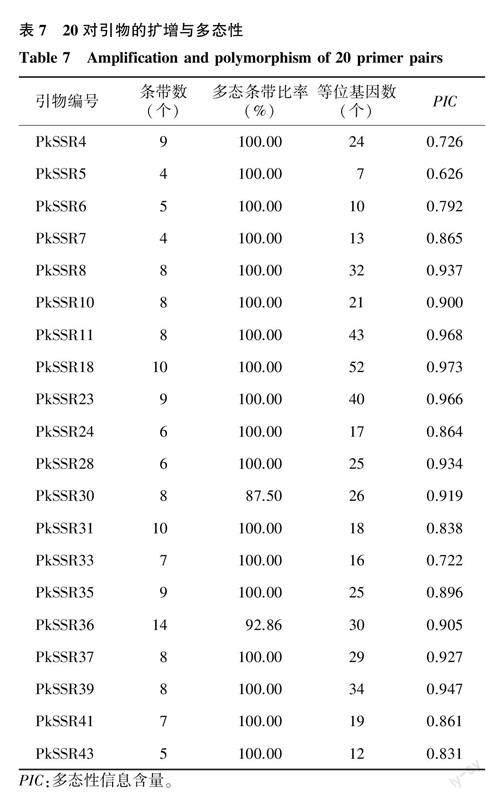
選擇其中條帶清晰的20對SSR引物(表7)對75份參試材料進行分析。20對引物共擴增出153個條帶,多態性條帶151個,多態率為98.69%。不同SSR的擴增條帶數為4.00~14.00個,平均7.65個,多態性條帶比率為87.50%~100.00%。不同SSR標記分析所揭示的75份材料等位基因數為7.00~52.00個,平均24.65個。20個SSR標記的PIC為0.626~0.973,大多在0.800以上,平均值為0.870。結果表明這些SSR標記的多態性較高,對不同材料具有較強的鑒別能力。
2.4遺傳多樣性與親緣關系聚類分析
采用20個SSR 標記對75份黃精材料進行分析,發現有5份多花黃精材料出現特異性的SSR條帶擴增或缺失(表8)。其中H-QDN-45和H-CA-1具有特異性擴增條帶,分別來自標記PkSSR31和PkSSR36,而H-QDN-49、H-QDN-43和H-HZ-4這3份材料則表現為特異性條帶缺失,缺失條帶分別來自標記PkSSR5、PkSSR11、PkSSR41。本試驗中發現的這些特異性的SSR條帶擴增和缺失可作為不同多花黃精材料鑒定的重要分子依據,也揭示了參試多花黃精材料遺傳的特異性。
經聚類分析(圖2)發現:參試的75份材料的遺傳相似系數為0.531~0.941,大部分材料的遺傳相似系數為0.60~0.80,平均值約為0.689。其中以多花黃精H-CA-4(52號)與H-CA-4-2(53號)間的遺傳相似系數最高,這2個種源都來自淳安左口鄉同一個采集點,因采集時發現兩者葉片背部顏色有一定差異而作為2個種源,實則為同一個種質,而多花黃精H-QDN-38-2(32號)與長梗黃精H-LA-3(75號)間遺傳相似系數最低。從結果來看,筆者發現多數材料之間具有一定的遺傳相似性,而少數材料之間遺傳差異較大,且遺傳相似系數變化范圍較大,說明參試材料之間具有遺傳多樣性。
在遺傳相似系數約0.666處,可將參試材料劃分為4個類群:A和C均為單一種群,分別包含11份黃精和34份多花黃精,而B和D則為不同種混聚類群。其中,聚在B類的有16份材料,包括12份多花黃精和4份長梗黃精,長梗黃精在此類群中單獨歸入同一亞類;D類有14份材料,包括7份多花黃精、6份黃精和1份川黃精,且后兩者也歸入同一亞類。這一結果表明,聚類分析結果與種類歸屬符合度較好。
3討論與結論
前人研究結果表明,不同物種中SSR位點的分布和頻率不同。造成這種差異的原因有三個:一是物種遺傳基礎特異性, 有研究認為SSR出現頻率隨著物種基因組變大而越來越低,與物種單拷貝DNA百分比之間存在高度顯著的正相關線性關系[23];二是SSR搜索標準(SSR重復類型及次數、長度等)不同,統計的SSR發生頻率則相差較大;三是分析和統計的SSR序列數和類型等數據量不同。比如搜索的序列數可以從幾千到幾十萬不等,統計的SSR類型也不盡相同,在黨參[24]、甘草[25]、黃秦艽[26]和厚樸[27]中統計的SSR數據中包含單核苷酸重復,而在杜仲[28]、多花黃精[17]以及本研究中統計的SSR則未包含單核苷酸重復。本研究共在46 416條序列中發現二核苷酸至六核苷酸SSR位點60 238個,序列發生頻率22.78%,平均分布頻率約1/7.07 kb。與模式植物水稻和擬南芥相比,滇黃精SSR發生頻率和分布頻率高于擬南芥,低于水稻[29]。與其他藥用植物相比,滇黃精序列SSR發生頻率高于杜仲(2.9%)[28]、丹參(3.79%)[30]、人參(7.3%)[31]和黨參(12.22%)[24],但低于甘草(60.10%)[25]、黃秦艽(30.73%)[26]和厚樸(52.75%)[27],平均分布頻率高于杜仲(1/26.13 kb)[28]、丹參(1/12.74 kb)[30],但低于人參(1/5.80 kb)[31]、黨參(1/4.52 kb)[24]、甘草(1/3.23 kb)[25] 、黃秦艽(1/4.11 kb)[26]和厚樸(1/3.43 kb)[27]。與黃精屬植物相比,滇黃精SSR發生頻率遠高于多花黃精(9.73%),但分布頻率則略低于多花黃精(1/5.91 kb)[17]。
不同植物中SSR優勢重復基元的出現數量不同,這可能與其編碼相應蛋白質時使用頻率有關,比如擬南芥[32]。理論上,在數量足夠且無偏倚的情況下,單核苷酸至六核苷酸重復基元可能產生的種類分別有2種、4種、10種、33種、102種、350種[33]。本研究從滇黃精轉錄組中搜索出的SSR基元種類也較為豐富,共262種。二核苷酸和三核苷酸理論上的重復類型全部出現,但四核苷酸至六核苷酸只出現了理論上的一部分,說明滇黃精轉錄組中 SSR也存在一定的偏倚性。在滇黃精轉錄組SSR中,二核苷酸和三核苷酸重復是主要類型,分別占總SSR的50.06%和34.89%,這與多花黃精及大多數植物表達序列中 SSR 以二核苷酸、三核苷酸為主要類型相符[29,34]。從滇黃精占主導地位的二核苷酸、三核苷酸重復基元種類來看,二核苷酸重復基元以AG/CT最多, GC/CG 最少,種類與多花黃精相近,但三核苷酸重復基元種類滇黃精比多花黃精多出ACG/CGT和ACC/GGT 2種基元,且主要的2種基元AGG/CCT(2 145)與AAG/CTT(2 143)數量基本相近,而多花黃精中AGG/CCT則比AAG/CTT高出5.4%[17],但均以AGG/CCT最多。而同樣是單子葉植物的小麥中卻以AAC/GTT居多,大麥、玉米、水稻和高粱則均以CCG/CGG為主要類型[29]。
基于NCBI數據庫中大量的表達或轉錄組序列可開發多種類型的分子標記[35-36],其中開發較多和應用廣泛的是SSR,也被稱為genic-SSR[10,37]。由于來自已知的表達基因序列,所以能夠更好地幫助挖掘植物重要性狀連鎖的基因,了解基因與表現型之間的聯系[38-39]。此外genic-SSR序列的兩端單拷貝序列保守性較高,具有較好的可轉移性[35-36,40],可用于種內甚至是種間的遺傳多樣性評價。本研究中有24.4%的引物無擴增產物,這與多花黃精[17]、油菜[41]及茶樹[42]中的研究結果類似,這可能是滇黃精與多花黃精、長梗黃精、川黃精對應的序列存在差異導致引物同源性低、引物與模板不匹配或者跨越內含子等原因造成的。
目前基于轉錄組測序已對多花黃精和黃精開發了genic-SSR標記[16-17],但數量有限且應用甚少。王世強等[16]根據黃精轉錄組測序數據鑒定出黃精多糖代謝合成途徑中關鍵酶基因的SSR 位點,篩選出12對多態性豐富的SSR 引物,并應用在32 份野生黃精資源的遺傳多樣性分析中, PIC平均為0.46,SSR標記聚類結果能揭示供試材料之間的親緣關系。然而關于這12對引物的研究結果反映的只是不同材料多糖代謝的遺傳差異,難以用來全面評估材料間的遺傳多樣性。陳友吾等[17]通過對多花黃精轉錄組測序數據的分析,發現SSR 位點出現頻率高,重復單元類型豐富,在測試的50對SSR引物中,有29對(58%)擴增出符合預期的產物,但這些標記尚未應用于黃精屬植物的資源分析,其多態性還有待實際研究去評判。本研究在明確滇黃精轉錄組測序數據中SSR位點信息和特征的基礎上,測試了45對SSR引物,有34對(75.6%)具有擴增產物,并利用其中20對引物對黃精、多花黃精、長梗黃精及川黃精共75份資源進行了分析,發現所測試的引物具有較高的多態性,PIC平均值達到0.870。聚類結果能夠反映參試材料的親緣關系,與分類歸屬具有較好的吻合度。SSR可擴增引物比率和PIC均高于上述2篇文獻[16-17]報道的結果,同時也證明根據滇黃精Unigene序列設計的SSR引物對黃精、多花黃精、長梗黃精及川黃精均具有可轉移性。
綜上,本研究基于滇黃精轉錄組序列開發的SSR標記,是對現有滇黃精轉錄組數據的進一步發掘和利用;所建立的標記對于豐富黃精屬植物分子標記類型、構建遺傳圖譜、促進資源的評價與育種應用、開展特定性狀的輔助選擇等都具有重要的意義。
參考文獻:
[1]ZHAO P, ZHAO C C, LI X, et al. The genus Polygonatum: a review of ethnopharmacology, phytochemistry and pharmacology [J]. Journal of Ethnopharmacology, 2018, 214:274-291.
[2]張嬌,王元忠,楊維澤,等. 黃精屬植物化學成分及藥理活性研究進展[J]. 中國中藥雜志, 2019, 44(10): 1989-2008.
[3]中國科學院《中國植物志》編輯委員會.中國植物志(第15卷) [M]. 北京: 科學出版社,1978: 58-82.
[4]王雨婷,劉婉瀅,沈舶寧 ,等. 黃精的本草考證[J]. 中醫藥學報, 2019,47(3): 81-86.
[5]田啟建,趙致. 黃精屬植物種類識別及資源分布研究[J]. 現代中藥研究與實踐,2007,21(1):18-21.
[6]魏曉楠,郝鐵成,劉慶華,等.中藥鑒別方法與技術探究[J]. 中國野生植物資源,2018,37(4):65-69.
[7]龍炳宏,蔣向輝,宋榮,等 . DNA條形碼在黃精屬藥用植物鑒定與遺傳多樣性分析中的應用[J]. 植物科學學報,2022,40(4):533-543.
[8]石乃星,文國松,趙明富.黃精屬植物 DNA 分子鑒定技術應用研究進展[J]. 植物遺傳資源學報, 2021,22(5): 1209-1218.
[9]TAUTZ D. Hyper-variability of simple sequences as a general source for polymorphic DNA markers[J]. Nucleic Acids Research, 1989, 17(16): 6463-6471.
[10]VARSHNEY R K, GRANER A, SORRELLS M E. Genic microsatellite markers in plants: features and applications[J]. Trends in Biotechnology, 2005, 23(1): 48-55.
[11]KALIA R K, RAII M K, KALIA S, et al. Microsatellite markers: an overview of the recent progress in plants[J]. Euphytica, 2011, 177(3): 309-334.
[12]LIU T,CHENG W J,ZHOU S,et al.Eleven polymorphic microsatellite loci in Polygonatum umfilipes and cross—amplification in other congeneric species[J].Conservation Genetics Resources,2010,2(S1): 77-79.
[13]CHENG W J,LIU T T,WU H L,et al.Isolation and characterization of twelve polymorphic microsatellite loci in Polygonatum cyrtonema and cross—species amplification[J].Conservation Genetics Resources ,2010,2:105-107.
[14]朱巧,鄧欣,張樹冰,等. 黃精屬 6 種植物的 SSR 遺傳差異分析[J]. 中國中藥雜志, 2018,43(14):2935-2943.
[15]WOOKJIN K,YUN-UI J,YOUNGMIN K,et al.Evaluation of genetic diversity of Polygonatum spp by the analysis of simple sequence repeat[J].Korean Herb Medicine Informations,2014,2(2): 41-47.
[16]王世強,王立儒,劉帥,等. 基于 SSR 標記的黃精品種(系)DNA 指紋圖譜庫構建[J].分子植物育種,2018,16(6):1878-1887.
[17]陳友吾,廖榮俊,葉碧歡,等. 多花黃精轉錄組 SSR 位點分析及分子標記開發[J]. 中草藥,2020,51 (1):182-189.
[18]趙振宇, 王仕玉, 郭鳳根,等. 轉錄組測序及其在藥用植物上的應用[J]. 基因組學與應用生物學, 2017, 36(2): 820-825.
[19]劉厚伯,上官艷妮,潘胤池,等. RNA-Seq在藥用植物研究中的應用[J]. 中草藥, 2019, 50(21):5346-5354.
[20]慧芳,劉秀巖,李宗諭,等. 轉錄組測序技術在藥用植物研究中的應用[J]. 中草藥, 2019, 50(24): 6149-6155.
[21]WANG Y, LIU X, SU H, et al. The regulatory mechanism of chilling-induced dormancy transition from endo-dormancy to non-dormancy in Polygonatum kingianum Coll.et Hemsl rhizome bud[J]. Plant Molecular Biology, 2019,99:205-217.
[22]BOTSTEIN D, WHITE R L, SKOLNICK M, et al. Construction of genetic linkage map in man using restriction fragment length polymorphisms[J]. The American Journal of Human Genetics, 1980, 32(3): 314-331.
[23]MORGANTE M, HANAFEY M, POWELL W. Microsatellites are preferentially associated with nonrepetitive DNA in plant genomes[J]. Nature Genetics,2000, 30:194-200.
[24]王東,曹玲亞,高建平. 黨參轉錄組中SSR 位點信息分析 [J]. 中草藥, 2014, 45(16): 2390-2394.
[25]LIU Y, ZHANG P, SONG M, et al. Transcriptome analysis and development of SSR molecular markers in Glycyrrhiza uralensis Fisch [J]. PLoS One, 2015, 10(11): e0143017.
[26]WANG L, WANG Z, CHEN J, et al. De novo transcriptome assembly and development of novel microsatellite markers for the traditional Chinese medicinal herb, Veratrilla baillonii Franch (Gentianaceae) [J]. Evolutionary Bioinformatics, 2015, 11(S1): 39-45.
[27]代嬌,時小東,顧雨熹,等. 厚樸轉錄組SSR 標記的開發及功能分析 [J]. 中草藥, 2017, 48(7): 2726-2732.
[28]黃海燕,杜紅巖,烏云塔娜,等. 基于杜仲轉錄組序列的SSR分子標記的開發[J]. 林業科學, 2013, 49(5):176-181.
[29]李永強,李宏偉,高麗鋒,等. 基于表達序列標簽的微衛星標記(EST-SSRs)研究進展[J]. 植物遺傳資源學報, 2004, 5(1):91-95.
[30]鄧科君,張勇,熊丙全,等. 藥用植物丹參EST-SSR標記的鑒定[J]. 藥學學報, 2009,44(10):1165-1172.
[31]LI C F, ZHU Y J XU G, et al. Transcriptome analysis reveals ginsenosides biosynthetic genes, microRNAs and simple sequence repeats in Panax ginseng C. A. Meyer [J]. BMC Genomics, 2013, 14(1): 204-205.
[32]范三紅,郭藹光,單麗偉,等. 擬南芥基因密碼子偏愛性分析[J].生物化學與生物物理進展, 2003, 30(2): 221-225.
[33]LA ROTA M, KANTETY R V, YU J K, et al. Nonrandom distribution and frequencies of genomic and EST-derived microsatellite markers in rice, wheat, and barley[J]. BMC Genomics, 2005, 6(23):23.
[34]姜春芽,廖嬌,徐小彪,等. 植物EST-SSR技術及其應用[J]. 分子植物育種,2009,7(1):125-129.
[35]李小白,崔海瑞,張明龍. EST分子標記開發及在比較基因組學中的應用[J].生物多樣性,2006, 14(6):541-547.
[36]李小白,向林,羅潔,等.轉錄組測序(RNA-seq)策略及其數據在分子標記開發上的應用[J].中國細胞生物學學報,2013, 35(5): 720-726.
[37]李小白,金鳳,金亮,等.利用建蘭轉錄數據開發genic-SSR標記[J].農業生物技術學報,2014, 2(8): 1046-1056.
[38]BOZHKO M, RIEGEL R, CHUBERT R, et al. A cyclophilin gene marker confirming geographical differentiation of Norway spruce populations and indicating viability response on excess soil-born salinity[J]. Molecular Ecology, 2003, 12(11): 3147-3155.
[39]SCHUBERT R, STARCK G M, RIEGEL R. Development of EST-PCR markers and monitoring their intrapopulational genetic variation in Piceaabies (L.) Karst[J]. Theoretical and Applied Genetics, 2001, 103(8): 1223-1231.
[40]忻雅, 崔海瑞, 張明龍,等. 白菜EST-SSR標記的通用性[J]. 細胞生物學雜志, 2006, 28(2): 248-252.
[41]李小白,張明龍,崔海瑞. 油菜EST-SSR標記的建立[J].分子細胞生物學報,2007, 40(2): 137-144.
[42]金基強,崔海瑞,陳文岳,等. 茶樹EST-SSR的信息分析與標記建立[J].茶葉科學,2006, 26(1): 17-23.
(責任編輯:陳海霞)

