畜禽泛基因組研究進展
李 珂,王宇龍,李 棟,史新娥,楊公社,于太永
(西北農林科技大學動物科技學院,楊凌 712100)
自1990年“人類基因組計劃”(Human Genome Project)[1]提出以來,基因組學得到了迅速發展,為生命科學的多個領域提供了重要的研究基礎。隨著越來越多物種的基因組被測序和組裝,研究人員發現,同一物種的不同個體間基因組信息具有較大差異,單一參考基因組并不能完整涵蓋其所有的遺傳信息,這可能會阻礙物種基因組變異的精準鑒定。2005年,Tettelin等[2]首次提出了泛基因組的概念,開啟了泛基因組學研究的新時代。隨后,泛基因組學逐漸應用于細菌、真菌及動植物等研究領域。本文對泛基因組學的發展歷程、構建策略及其在畜禽上的研究現狀進行綜述,以期為畜禽泛基因組的深入研究提供參考。
1 泛基因組的概念和發展
1.1 泛基因組的概念
2005年,Tettelin等[2]在研究無乳鏈球菌菌株的基因組時,首次提出了微生物泛基因組(或超基因組supragenomes)的概念[3-4]。在這項研究中,他們發現不同菌株之間的基因組存在明顯差異,平均每檢測1個新的菌株會出現33個新基因,這表明單一菌株的遺傳信息并不能完全代表該菌種的所有遺傳信息。泛基因組是指一個生物進化支(如物種)的全部個體基因序列的集合,它不但能夠更為全面的涵蓋物種的遺傳信息,同時為物種多樣性和進化適應性的研究提供了新的視野[5]。泛基因組在不同類型的物種中定義有所不同。在原核生物的研究中,由于細菌等原核生物的DNA序列大多具有功能,且基因序列中幾乎不含內含子,因此,在原核生物的研究領域,泛基因組通常代表著該物種的基因序列而非全部的基因組(genome)序列。真核生物的基因組是由編碼蛋白的序列(外顯子)、基因間區和內含子等非編碼序列組成。因此,真核生物的泛基因組定義的一般是物種所有的DNA序列總集合[6-7]。
泛基因組由核心基因組和可變基因組組成(圖1)。核心基因組是同一物種的所有個體中都存在的基因集合或序列,對生命活動和表型性狀至關重要;可變基因組則只存在于一個或多個個體中,這些基因序列并不在所有個體中出現[8-10],一般與通訊、毒性和防御反應有關[11-14]。可變基因組又可細分為單一個體的特有基因和兩個或兩個以上個體中的附屬基因[8-9]。在另一種分類方法中,可變基因組又可分為殼基因組(shell,占全部個體基因組的5%~95%)和云基因組(cloud,僅存在約少于5%的個體基因組中)[15]。在動植物的研究領域,可變基因與物種在特定環境的適應性或特有的生物學特征有關,如抗寒性[16]和抗病性[17]等。泛序列(pan-sequence)也被用來描述存在于其他個體基因組但不存在于參考基因組中的序列。概括地說,核心基因組可以反映物種的遺傳穩定性及本質,而可變基因組與個體適應環境的特征相關[18-19]。
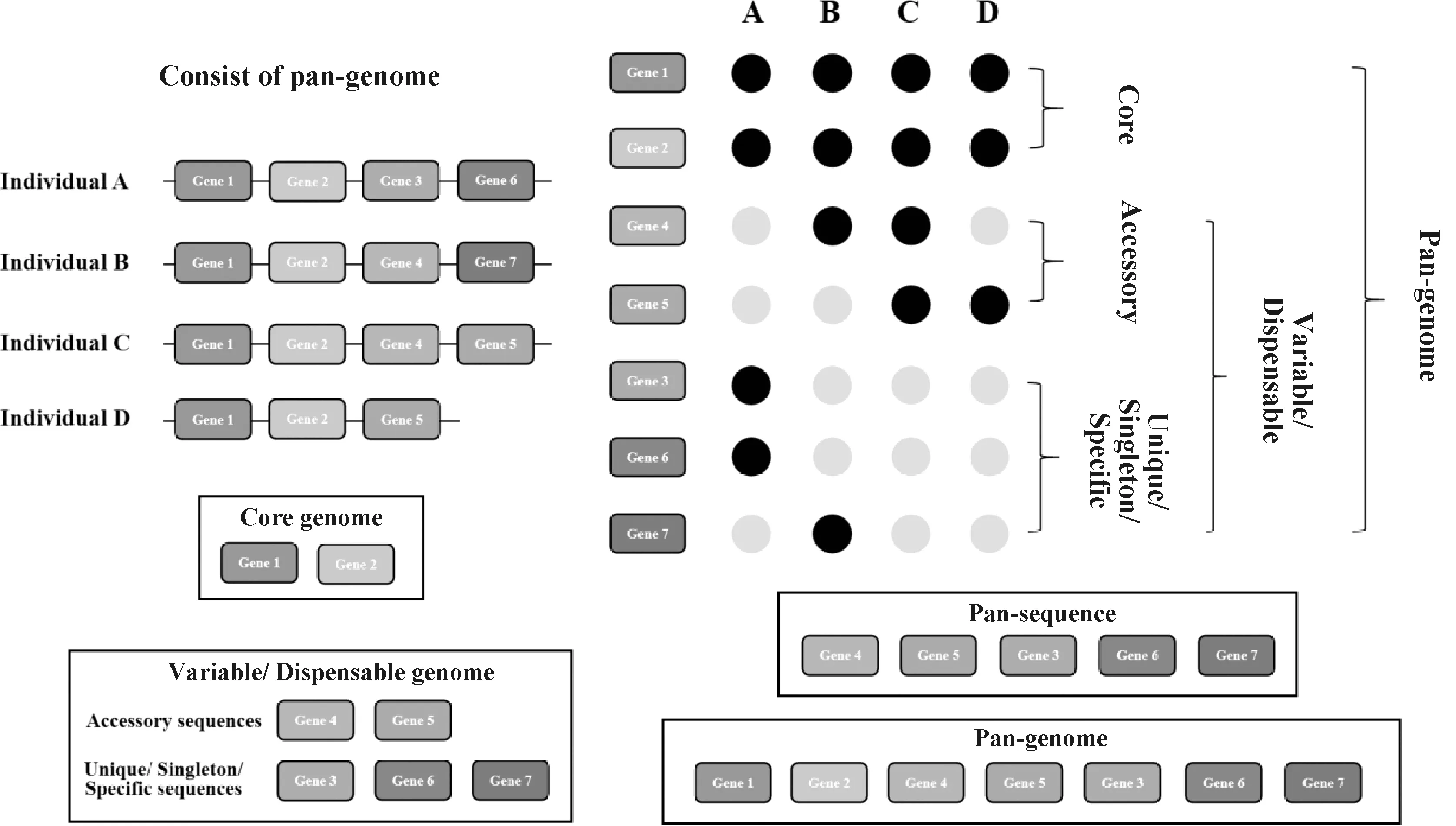
圖1 泛基因組的概念Fig.1 The concept of pan-genome
1.2 泛基因組的發展
DNA測序技術在測序速度和準確性等方面的進步,推動了泛基因組學的迅速發展[20-22]。2005年,Tettelin等[2]在細菌上首次引入“泛基因組”的概念。研究人員發現,細菌中廣泛存在基因損失和基因水平轉移[23],在不同菌株之間會發現新的可變基因。Read等[24]對遍布全球海洋的真核生物赫氏圓石藻進行基因組學研究,發現其基因組之間具有差異性,這為泛基因組在真核生物領域的發展提供了參考。2007年,Morgante等[25]首次在植物上引入泛基因組的概念,發現可變基因中的轉座子具有重要的作用。2010年,Li等[26]和Goodwin等[27]利用第二代測序技術構建了人類的首個泛基因組,該研究結果補充了人類基因組序列并首次發現了主要存在于亞洲人群內特有的基因序列。2013年,泛基因組學研究開始較為廣泛地應用于動植物研究領域[28-29]。2014年,研究人員成功構建了首個大豆的泛基因組,突破了大豆單一參考基因組研究的局限性,開啟了植物泛基因組研究歷程[30]。2019年,Chen等[31]提出園藝植物基因組計劃,構建了多個園藝植物的泛基因組,為未來園藝植物基因組遺傳變異信息的破譯提供了重要參考。
然而,由于測序技術的局限性,使用第二代全基因組測序技術得到的短序列組裝基因組仍然非常困難,這主要是由于基因組組裝需要大量的計算資源,不能很好地擴展到數十到數百個連續的基因組。隨著測序技術的成熟和測序成本的降低,光學圖譜[32]、遺傳圖譜[33]和染色體構象捕獲技術(Hi-C)[34-35]等得到了迅速的發展,增加了從千堿基大小的contigs到全染色體的序列連續性。近年來,泛基因組學的研究方法引入了第三代測序技術,不但提高了泛基因組的質量,同時縮短了泛基因組構建的時間。2020年,Song等[36]利用三代測序技術構建油菜泛基因組,為系統挖掘和揭示油菜的復雜遺傳變異提供了數據基礎。隨著多個物種高質量泛基因組的陸續發表,如蝙蝠[37]、狗尾草[38]、貽貝[39]、水稻[40]、草莓[41]、棉花[42-44]、馬鈴薯[45-46]、蠶[47]等,利用泛基因組學解析物種的復雜遺傳變異逐漸成為了基因組研究領域的熱點。
2 泛基因組的組裝策略
泛基因組的構建策略主要分為迭代組裝泛基因組、從頭組裝泛基因組以及圖形泛基因組這三種方式[48-49](圖2)。迭代組裝使用“map-to-pan”策略,用大規模的重測序數據構建泛基因組;從頭組裝泛基因組使用“assemble-to-pan”策略,對較少數量的個體進行高深度測序構建泛基因組;圖形泛基因組是在從頭組裝泛基因組基礎上,通過構建數學和計算機中被稱為圖(graph)的數據結構來展示一個物種所有的基因序列排列和結構,突破了傳統線性基因組的存儲形式,具有重要的理論意義和應用價值。
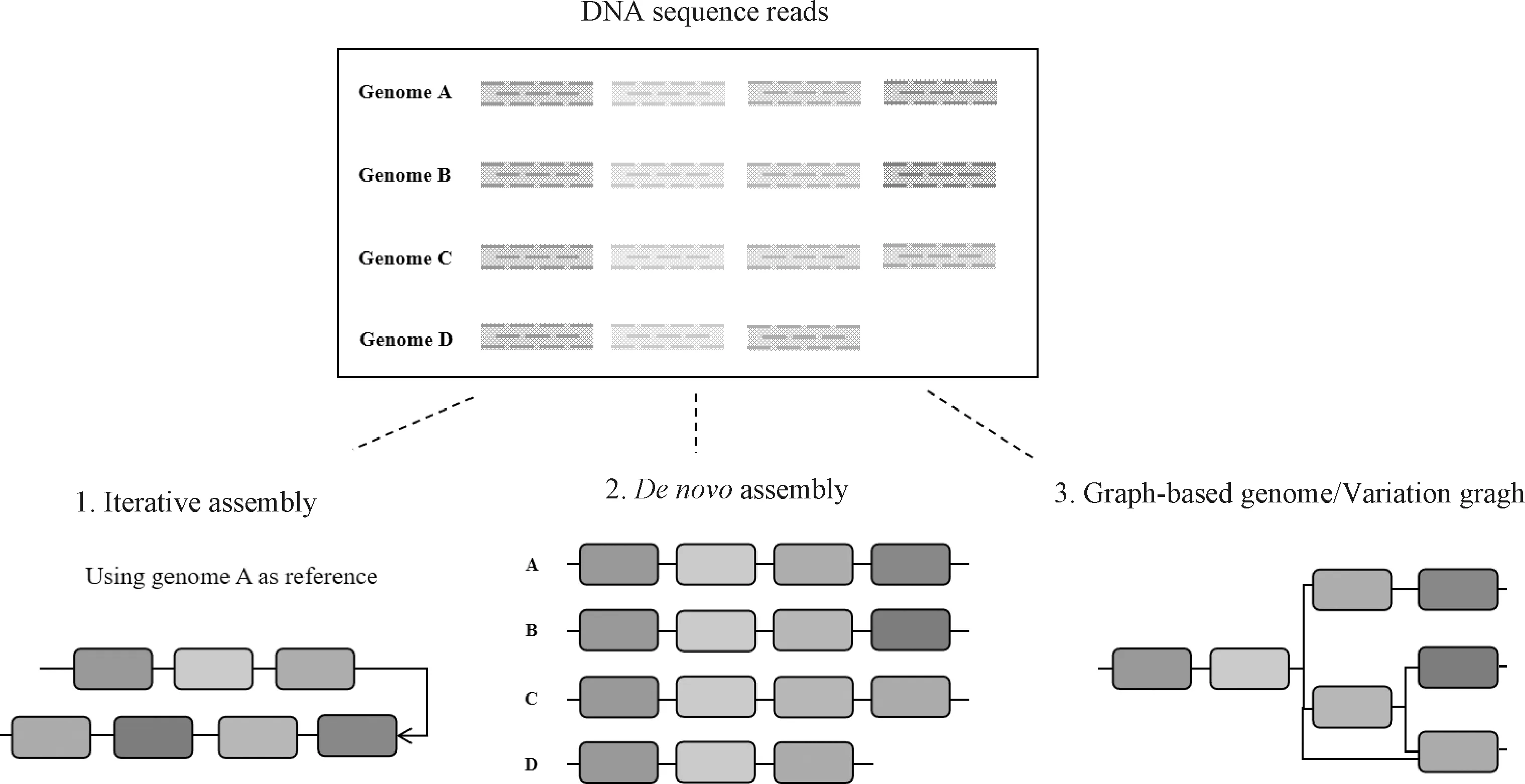
圖2 泛基因組的組裝策略Fig.2 Schematic of the pan-genome assembly approaches
2.1 迭代組裝泛基因組(iterative assembly)
迭代組裝泛基因組(iterative assembly or map-to-pan)的方法是通過對大量個體進行全基因組重測序,在從頭組裝單個基因組后再映射到參考基因組,將所有未比對上的新序列集與參考基因組合并,構建泛基因組參考序列[50-52]。2018年,Wang等[53]對3 010份水稻測序運用“map-to-pan”策略,從重測序個體中組裝出 268 Mb 的非冗余序列,補充到日本晴水稻參考基因組,為后續遺傳變異檢測和功能分析提供了數據支撐。2019年,研究人員對493份向日葵重測序數據進行分析,通過構建向日葵泛基因組,分析了基因滲入對向日葵疾病抗性的影響[54]。2020年,Alonge等[55]利用迭代組裝方式構建了100個番茄的PanSV基因組,發現sb1 loci 對于解決 QTL 背后的復雜單體型至關重要,為培育具有高產、優質、抗病蟲害等優良性狀的番茄新品種奠定了堅實的基礎。然而,迭代組裝泛基因組也存在著一定的問題。由于泛序列補充到參考基因組上順序具有不確定性,不但會導致無法真實反映新基因在泛參考基因組上的位置信息,在進行多拷貝基因的研究中也容易降低檢測效率。同時,在一些具有高度重復性和SVs普遍存在的作物基因組中,易出現組裝錯誤[56]。
2.2 從頭組裝泛基因組(de novo assembly)
從頭組裝并注釋個體基因組,通過個體基因組間的相互比較鑒定出核心與可變基因,去除冗余序列后構建泛基因組的方式被稱為從頭組裝泛基因組,多用SOAPdenovo[57]軟件進行組裝,是運用較為廣泛的方法,如人類[26]、玉米[58]、大豆[30]、大麥[59]和小麥[60]的泛基因組都是基于這種方法組裝的。其中,大豆的泛基因組研究發現,組裝大豆野生近緣種Glycinesoja能夠覆蓋94%的栽培大豆Glycinemax基因,鑒定出338個存在/缺失變異(presence/absence variation, PAV)、1 978個拷貝數變異(copy number variations, CNV)和一系列位于高度差異基因區域的單核苷酸多態性(single nucleotide polymorphisms, SNP)和小于50 bp的片段插入或缺失片段(INDELs)。基于大豆泛基因組檢測到的基因組變異信息有著重要的育種和生產價值,為大豆的研究提供了重要的數據基礎。在動物泛基因組的研究上,長頸鹿[61]、果蠅[62]和熊蜂[63]等是從頭組裝泛基因組的經典之作,為解析物種的遺傳差異和進化關系提供了重要的參考價值。從頭組裝泛基因組可以提供所有個體的基因組信息、基因和其他基因組的物理位置,能夠高效識別重復區域和拷貝數變異。然而,由于組裝基因組需要大量的測序數據和計算資源,相對來說成本較高[64]。
2.3 構建圖形泛基因組(graph-based genome/varia-tion gragh)
圖形泛基因組最早用于微生物泛基因組的構建,近年來在動植物基因組中逐漸嶄露頭角[65]。圖形基因組是一種較為理想的構建策略[66],基于從頭組裝基因組后將不同個體的基因組比對到線性參考基因組后提取變異信息。變異信息去冗余后與線性基因組整合通過多條路徑的方式來展示各種變異。這種方法不但能夠保留變異序列在染色體上的位置信息,還可以較為完整的展現物種基因組的多樣性。Vg[67]、Minigraph[68]、Cactus[69]等軟件是近年來研究人員開發的用于圖形基因組構建的工具,能夠幫助學者更好、更直觀地理解基因組的結構變異信息。2020 年發表的大豆泛基因組,是構建的首個作物高質量圖形泛基因組。研究人員使用第三代測序數據從頭組裝了26個大豆的基因組,平均 contig N50 達到了 22.6 Mb[70]。此研究挖掘到了大量利用單個參考基因組不能鑒定到的基因變異,為解析大豆種皮的亮度、顏色變化等重要農藝性狀的調控機制奠定了基礎。2021年Qin等[40]首次構建了水稻圖形泛基因組,促進了水稻功能基因組學的研究,為深度挖掘水稻基因組變異和培育突破性的水稻新品種提供了重要依據。蘿卜[71]、白菜[72]、番茄[73]、鷹嘴豆[74]、棉花[43]、馬鈴薯[46]等多個物種的泛基因組圖譜相繼被研究人員構建,為基因組功能研究和分子育種提供了重要的數據支撐。
3 畜禽泛基因組學研究
泛基因組近年來已廣泛應用于微生物、植物及動物研究中,在探究物種起源馴化以及改良遺傳育種體系等方面展現出巨大潛力。以下總結了泛基因組在畜禽領域中的最新研究成果。
3.1 泛基因組在豬上的研究
豬是農業上重要的家畜,也是生物研究和應用的重要醫學模型。隨著基因組測序組裝技術的不斷革新,豬的基因組被不斷完善。目前,豬的參考基因組是2017年公布的基于第三代測序技術組裝的杜洛克豬基因組(Sscrofa11.1),contig N50達到了48.23 Mb,與人和其他模式動物基因組組裝質量不相上下。然而,由于豬的起源馴化及其群體間表型和基因型的巨大差異[75-76],使用杜洛克豬參考基因組對世界范圍內不同豬種進行遺傳變異的研究具有很大的局限性。2017年,四川農業大學Li等[77]使用10只來自歐亞大陸的豬的基因組構建了豬的泛基因組,挖掘到了大量新的豬基因組變異信息。研究發現了豬參考基因組中不存在的137.02 Mb的缺失片段,彌補了單一參考基因組涵蓋遺傳信息有限的問題,為后續豬的基因組研究提供了新的分析思路及寶貴的遺傳資源。
2020年,西北農林科技大學Tian等[78]基于豬參考基因組(Sscrofa11.1)和11個世界范圍內具有地理和表型代表性的豬種構建了豬泛基因組,研究發現了72.5 Mb 的非冗余泛序列,其中約9 Mb的泛序列在亞洲豬基因組中的頻率顯著高于歐洲豬。轉錄組分析發現,亞洲豬中特有的TIG3基因可能是亞洲豬脂質代謝過程中的關鍵基因。同時,研究人員通過構建豬泛基因組數據庫,將泛基因組和轉錄組等數據整合,為后續研究提供重要的數據資源。
2023年,中國農業大學Jiang等[79]整合了11個豬品種構建了中西方豬的圖形泛基因組。該研究利用長讀長測序在藏豬上鑒定了7 568個特異性SVs,通過結合22頭藏豬和98頭低海拔豬的重測序數據,并與先前的研究結果比較分析得到了12個與高海拔適應性有關的候選基因。通過后續試驗鑒定出SOD1、SEMA5A、REV1、SGCD是與高海拔低氧適應性有關的候選基因,為豬遺傳育種提供了豐富的基因組變異信息。
3.2 泛基因組在牛上的研究
牛作為重要的經濟動物在許多國家都有廣泛的用途,可以為人類提供牛奶、肉類、皮革和肥料等。來自全世界的研究人員在牛泛基因組的研究領域上做出了杰出貢獻,如蘇黎世聯邦理工學院Hubert Pausch實驗室聚焦牛基因組學研究、美國農業部建立了牛泛基因組聯盟、中國研究人員結合本土黃牛構建牛泛基因組等。2020年,蘇黎世聯邦理工學院Crysnanto和Pausch[80]利用vg構建了首個牛的圖形泛基因組,顯著提高了序列比對和基因分型的準確性。隨后,該團隊結合PacBio CLR和PacBio HiFi測序技術,利用minigraph將6個高質量牛基因組整合到一個圖結構泛基因組框架中,鑒定出70 Mb的非參考序列并構建了包含新序列及變異位點的牛的圖形泛基因組,為后續多物種泛基因組的研究提供了思路和圖結構框架[81]。西北農林科技大學Gong等[82]利用12個牛屬個體從頭組裝泛基因組,發現了36.3 Mb非參考序列,為研究牛的優良遺傳性狀提供了理論基礎;愛丁堡大學Talenti等[83]利用組裝的兩頭非洲牛染色體水平基因組,并結合294頭不同牛品種的重測序數據構建了一個包含全球牛多樣性的基因組圖并鑒定出116.1 Mb的非參考序列。上述研究完善了現有牛的參考基因組(ARS-UCD1.2)并為進一步挖掘牛的遺傳多樣性提供了新的變異位點。
隨著第三代長讀長測序的迅速發展,越來越多的研究人員聚焦于研究結構變異(structural variation, SV)對復雜性狀的影響。2022年,來自瑞士、美國的牛泛基因組聯盟(Bovine Pangenome Consortium,BPC)實驗室共同構建了基于結構變異的牛泛基因組,并使用不同的測序平臺、覆蓋深度和組裝算法對泛基因組構建的潛在影響進行了探究,研究表明,20X 覆蓋度的PacBio HiFi數據可以完成牛單倍型基因組組裝,且具有較好的連續性與準確性[84]。華中農業大學Zhou等[85]構建的含57個品種898頭牛的泛基因組是目前規模最大的牛泛基因組研究,通過研究SV對基因組功能元件和QTL的影響,為牛基因組演化提供了深刻理解。
3.3 泛基因組在羊上的研究
目前,泛基因組學研究在山羊和綿羊上均有報道。2019年,Li等[86]從9個山羊denovo基因組中鑒定出38.3 Mb山羊參考基因組(ARS1)中缺失的泛序列(占基因組的1.4%),并構建了山羊泛基因組,顯著提高了山羊重測序數據的變異檢測效率。2022年,西北農林科技大學以及新疆農墾科學院研究團隊組裝了13只綿羊的單倍型基因組,構建了首個圖形化的綿羊泛基因組圖譜[87]。研究人員基于圖形化泛基因組的群體結構變異信息與綿羊尾部表型性狀進行SV-GWAS關聯分析,結果顯示,HOXB13基因與綿羊尾長性狀高度相關。結合PBS選擇信號分析,發現SVs和SNPs在基因BMP2、HAO1和PDGFD上都受到強烈的選擇信號,這為探究綿羊重要經濟性狀的內在調控機制提供了寶貴資源。
3.4 泛基因組在雞上的研究
雞是目前世界上數量最多的家養動物,在畜牧業中占有重要地位。已有多項研究表明,鳥類的基因數量和進化速度遠低于哺乳動物,且對其的馴化和育種會影響雞的基因組結構[88-89]。然而,鳥類基因數量少且進化速率低背后的機制尚未有定論,因而構建泛基因組解析雞基因組遺傳信息是研究雞重要經濟性狀的有力手段。2021年,河南農業大學和西澳大學構建了首個雞的泛基因組,發現了雞參考基因組(GRCg6a)中未被組裝的約66.5 Mb序列[90]。通過PAV-GWAS分析鑒定出許多與雞生長、胴體成分、肉質或生理特征相關的候選突變。研究人員發現,IGF2BP1的33個啟動子區域的缺失影響了雞的體型大小,這有助于未來設計具有特定性狀的雞品種。
2022年,西北農林科技大學和中國農業大學通過20只雞的全基因組測序數據構建了雞的泛基因組,并對新鑒定到的159 Mb泛序列進行了分析[91]。研究發現,泛序列中的新基因大多位于染色體亞端粒區和小染色體,具有較多的串聯重復序列;非經典的DNA二級結構導致了這些缺失序列難以被捕獲,這阻礙了雞基因組的深入研究。同時,研究人員發現新基因的替代率比已知基因高3倍,這項研究結果打破了已有的研究結論,對鳥類比較基因組學和功能基因組學的研究具有重要意義。
4 總結與展望
泛基因組學經過多年發展已逐漸成熟,是基因組學研究領域的熱點。第三代測序技術的落地極大地推動了泛基因組學的快速發展。利用泛基因組挖掘關鍵的遺傳變異信息,結合重要表型性狀和多組學數據等,是研究生物重要性狀的有力工具。未來,隨著測序技術的進一步發展和算法水平的不斷提高,實現結合端粒到端粒的基因組組裝(telomere-to-telomere, T2T)和精確檢測基因組結構變異信息等,對于泛基因組學的發展和應用具有重要意義。海量測序數據的不斷產生為構建泛基因組數據庫提供了數據基礎。通過整合多個代表性個體的基因組信息,結合多組學數據和群體變異信息為進一步挖掘調控重要性狀的基因提供基礎,也為功能基因組學的研究提供了便利。目前,泛基因組研究由于計算資源等的制約大多集中在“種”水平,但未來“屬”水平的超級泛基因組(super-pan genome)會成為泛基因組研究的新熱點和新方向。

