反芻動物Toll樣受體多基因家族的分子進化及表達模式分析
馬淑娟,徐祎潔,何 珂,馬瑞豐,朱 英*
(1.西南民族大學青藏高原研究院,成都 610041;2.浙江農林大學動物科技學院/動物醫學院,杭州 311300)
Toll樣受體(Toll-like receptors,TLRs)參與機體天然免疫識別、抵御外來微生物的入侵,在機體的先天免疫系統中起著重要作用[1-2]。TLR基因可分為非病毒性TLRs(non-viralTLRs)和病毒性TLRs(viralTLRs)。前者位于細胞膜上,主要識別微生物細菌的鞭毛蛋白、脂多糖、肽聚糖等[3-4];后者位于細胞器膜上,主要識別病毒未甲基化的CpGDNA和單鏈或雙鏈RNA[5-6]。由于TLR直接位于宿主和環境作用的交界處,其可能與病原微生物共進化,故TLR基因是研究脊椎動物體內由病原微生物驅動的自然選擇的最佳候選基因之一。
在哺乳動物中已發現13種TLRs(TLR1-13),TLR1-TLR10在陸生和水生哺乳動物中均有報道,但是TLR11-TLR13常見于小鼠、樹鼩等嚙齒類、樹鼩類動物[6-8]。根據TLR系統發生樹的相對關系,可以將TLR基因分為不同的亞家族[9-10],但哺乳動物不同類群的亞家族情況因物種數目不同而不同[11-13]。以往研究表明,所有TLR基因均受到純化選擇作用,然而不同TLR基因受到的選擇壓力不同[6,10],如非病毒性TLRs受到的選擇壓力強于病毒性TLRs。
反芻動物(偶蹄目反芻亞目)是有蹄類中物種最繁盛的動物類群,分布廣、數量大,擁有200多個現生物種[14-16],包含經過人類馴化的重要家畜資源[17-20],如家牦牛、綿羊、山羊等。目前,關于反芻動物的Toll樣受體基因家族的研究大部分都見于反芻動物單一物種,如對牦牛[18]、牛[21]、綿羊[21]、山羊[12]等個體的研究,或只針對TLRs中的某個或幾個基因成員進行TLR遺傳多樣性、分子進化分析[22-24]。在反芻動物類群中,TLR家族是否存在物種特異的膨脹、TLR亞家族的分類、反芻動物不同TLR基因和不同類群反芻動物受到的選擇壓力模式和強度,以及反芻動物非病毒性和病毒性TLR承受的選擇壓力仍不清楚。
研究表明,物種數量多且覆蓋全面的TLR研究能夠給出較真實的系統發育關系[10]。為全面探尋反芻動物TLRs基因家族的進化模式,本研究基于公共數據庫中53種反芻動物(涵蓋6個科47個屬)的全基因組,采用blast對反芻動物TLRs基因家族成員(TLR1-10)進行鑒定,對1)染色體位置和基因結構;2)亞家族分類;3)進化關系和選擇壓力;4)表達模式進行分析。研究結果有助于理解反芻動物TLR基因家族的進化,也為綿羊、牛等重要的畜牧資源和其它動物的免疫分子機制和抗病育種提供基礎數據。
1 材料與方法
1.1 研究物種
反芻動物包含鼷鹿科(Tragulidae)、麝科(Moschidae)、長頸鹿科(Giraffidae)、鹿科(Cervidae)、牛科(Bovidae)和叉角羚科(Antilocapridae)6個科,76個屬,擁有233多個現生物種[14-16]。在美國國家生物技術信息中心(National Center for Biotechnology Information,NCBI,https://www.ncbi.nlm.nih.gov/,Bethesda,MD,United States)上檢索到117個反芻動物的基因組數據(截止到2022年12月15日),涵蓋47個屬。在Bos、Cervus、Muntiacus屬中,共有8個物種具有三代參考基因組,這8個物種的參考基因組均保留在本研究的分析中,Moschus屬僅有的2個物種(二代參考基因組)也全部保留在分析中。而在其他43個屬中各選擇一個組裝質量高的二代參考基因組進行分析。最后,對53個物種進行了后續的分析,所選物種數目占反芻動物的22.7%(53/233),覆蓋了反芻動物屬的65.8%(47/76)(表1)。
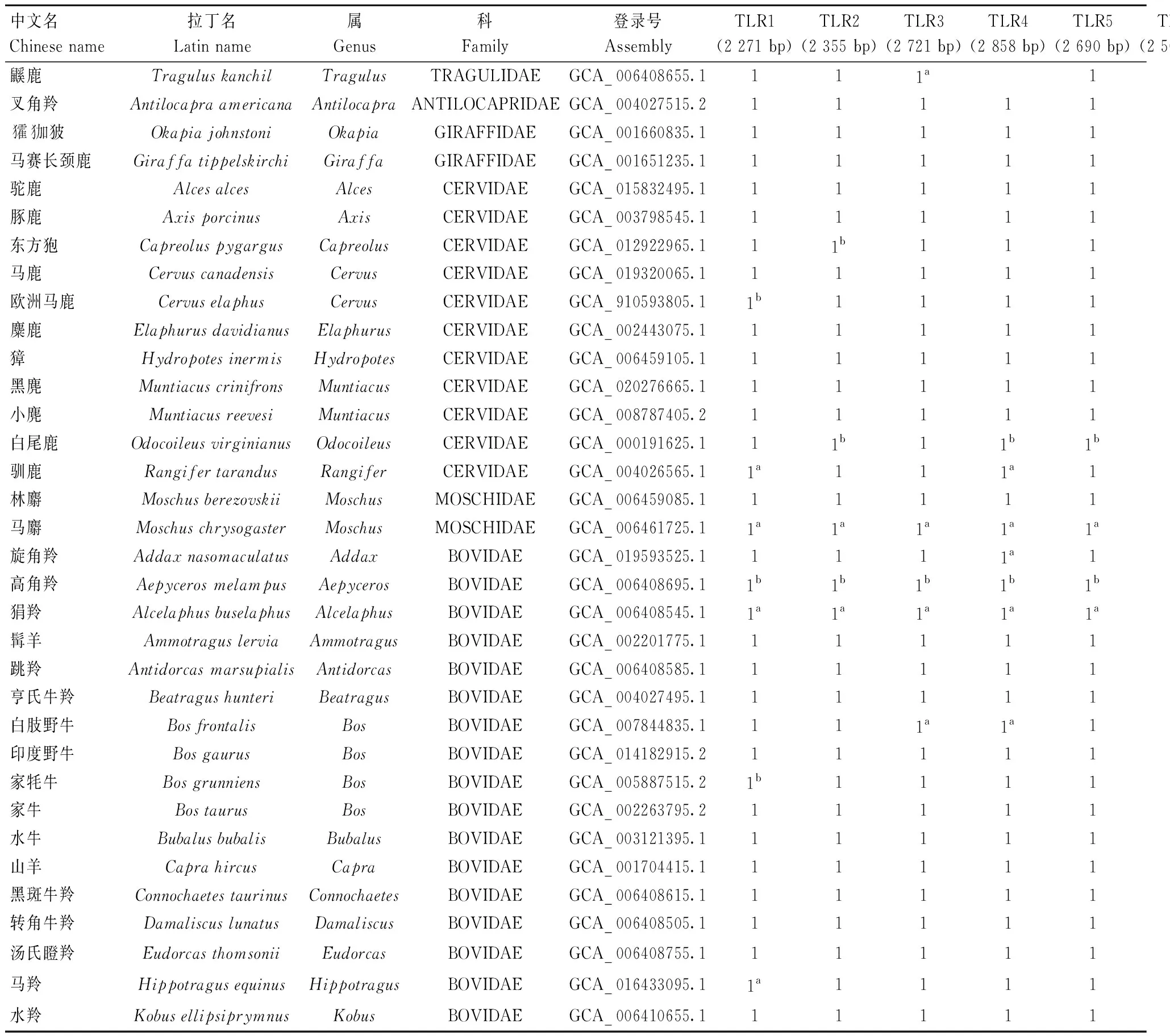
表1 反芻動物TLRs基因組數據信息Table 1 Genomic data information of ruminant TLRs
1.2 TLRs基因家族成員鑒定
在NCBI上下載反芻動物TLRs的參考基因組序列,以水牛(Bubalusbubalis)的TLR1-TLR6、TLR8-TLR10(HQ327989.1、DQ288130.1、HQ343417.1、HQ343416.1、HQ327990.1、HQ327992.1、GQ499855.1、HQ242779.1、HQ327991.1),家牛(Bostaurus)的TLR7基因(EF583900.1)作為qurey序列,用BlastN進行檢索,E-value為10-5,保留滿足于以下兩個條件的序列進行后續的分析:1)序列長度大于最長序列CDS的80%;2)不存在提前終止子。
1.3 TLRs基因家族染色體定位、基因結構分析
根據基因組數據的測序質量、完整度,選擇參考基因組組裝到染色體且具有注釋文件的物種對其TLRs基因家族成員在染色體上的分布和基因結構進行分析。本研究對滿足條件的牛科的水牛(Bovidae,Bubalusbubalis)和鹿科的馬鹿(Cervidae,Cervuscanadensis),使用TBtools[25]進行TLRs基因家族在染色體上分布和基因結構的可視化。
1.4 系統發育樹
在NCBI下載人類和藍鯨的TLRs基因家族作為外類群(Human (Hosa):TLR1 NM_003263.4、TLR2 NM_001318787.2、TLR3 NM_003265.3、TLR4 NM_003266.4、TLR5 NM_003268.6、TLR6 NM_001394553.1、TLR7 NM_016562.4、TLR8 NM_016610.4、TLR9 NM_017442.4、TLR10 NM_001017388.3; Bluewhale (Blwh):TLR1 XM_036853149.1、TLR2 XM_036853661.1、TLR3 XM_036838879.1、TLR4 XM_036856405.1、TLR5 XM_036836501.1、TLR6 XM_036853144.1、TLR7 XM_036839897.1、TLR8 XM_036839825.1、TLR9 XM_036869686.1、TLR10 XM_036853063.1),使用MEGA X[26]軟件對反芻動物、人類、藍鯨的TLRs序列進行MUSCLE多重序列比對,基于最優模型(Tamura-Nei),采用最大似然法(maximum likelihood,ML)構建TLRs基因家族的系統發育樹,Bootstrap值設為1 000,替換類型為核苷酸,其余參數為系統默認值[27-28]。
1.5 TLRs基因家族的選擇壓力分析
本研究使用非同義替換率(dN)與同義突變率(dS)的比值ω(ω=dN/dS)進行TLRs基因家族的選擇壓力分析。當ω>1時,表示受到正選擇(positive selection);ω=1時,為中性選擇(neutral selection);ω<1受到純化選擇(purifying selection)。采用MEGA X[26]計算ω。對于每個TLR基因的正選擇位點,采用在線網站(datamonkey adaptive evolution server(http://www.datamonkey.org/)中的MEME(mixed effects model of evolution)、SLAC(single-likelihood ancestor counting)、FEL(fixed effects likelihood)、FUBAR(fast unconstrained bayesian approximation)4種方法進行預測。使用MEME、SLAC、FEL方法篩選正選擇和純化選擇的標準是P-value<1,FUBAR方法則用后驗概率>0.9進行篩選。
在上述基礎上,本研究檢測牛科和鹿科,病毒性和非病毒性TLR這兩組的ω和正選擇位點數目(positively selected sites, PSS)是否存在顯著差異。利用R4.1.2的shapiro.test和var.test函數分別進行正態分布和方差齊性檢驗[29-30],對于符合條件的數據采用獨立樣本T檢驗,反之則采用kruskal檢驗[31]。
1.6 TLRs基因家族在不同組織的表達模式分析
本研究根據反芻動物基因組數據庫(http://animal.nwsuaf.edu.cn/code/index.php/Ruminantia)中的RNA seq數據分析TLRs基因家族在不同組織的表達模式。反芻動物基因組數據庫包含多個物種的RNA seq數據,由于綿羊(Ovisaries)具有豐富的組織表達數據,故本研究采用綿羊的RNA seq進行后續分析。使用TBtools軟件繪制其內分泌系統、免疫系統、神經系統、呼吸系統、消化系統共25個組織的表達模式熱圖。
2 結 果
2.1 反芻動物TLRs基因家族的數量
對53個反芻動物的全基因組進行TLR鑒定,得到53種反芻動物(鼷鹿科 1個、叉角羚科 1個、長頸鹿科2個、鹿科 11個、麝科 2個、牛科 36個,表1和圖1)共530條TLR基因序列。對于每一個TLR基因都僅鑒定出1條TLR序列,不存在多拷貝現象。通過去除短序列(67條)、假基因序列(42條),剩余421條TLR真基因序列,TLR1-TLR10的10個基因分別有39、40、45、43、44、36、43、41、44、46條序列(表1)。在科水平上,鼷鹿科共鑒定到8個TLR基因(無TLR3、TLR10),叉角羚科鑒定到9個TLR基因(無TLR8),長頸鹿科、鹿科、麝科、牛科都鑒定到10個TLR基因(表1和圖1)。在物種水平上,通過剔除短序列和假基因之后,鑒定到的TLR基因數目為0~10個。牛科的高角羚(Aepycerosmelampus)和柯氏犬羚(Madoquakirkii)鑒定到的TLR真基因數目為零。

進化樹改自文獻[14]的圖1Phylogenetic tree was modified from figure 1 in reference [14]圖1 反芻動物的TLR基因數目Fig.1 TLR genes number of ruminants
2.2 反芻動物TLRs的染色體定位、基因結構
水牛和馬鹿的TLRs家族成員在染色體上的定位不同、基因結構不同(圖2)。水牛TLR1、TLR6、TLR10連鎖,并位于7號染色體,TLR7和TLR8連鎖,位于X染色體,TLR2、TLR3、TLR4、TLR5、TLR9分別分布于17、1、3、5、21號染色體上(圖2a)。馬鹿TLR1、TLR6、TLR10位于19號染色體,TLR7和TLR8位于X染色體,TLR2、TLR3、TLR4、TLR5、TLR9分別分布于染色體1、31、30、13、22上(圖2b)。

a.水牛TLRs基因在染色體上的定位;b. 馬鹿TLRs基因在染色體上的定位;c. 水牛TLRs基因結構;d. 馬鹿TLRs基因結構a. Chromosomal localization of TLR genes in Bubalus bubalis; b. Chromosomal localization of TLR genes in Cervus canadensis; c. TLR genes structure of Bubalus bubalis; d. TLR genes structure of Cervus canadensis圖2 水牛和馬鹿TLRs基因在染色體上的定位和基因結構Fig.2 Chromosomal localization and gene structure of TLRs in Bubalus bubalis and Cervus canadensis
水牛和馬鹿的CDS全長是保守的,但基因結構分析表明,水牛和馬鹿在外顯子數目、基因長度存在差異(圖2c,圖2d)。水牛的TLR基因含有2~5個外顯子,其中TLR3基因有最多的外顯子數目,水牛的TLR基因長度位于7 828 (TLR9) ~33 883 bp (TLR10)之間(圖2c)。馬鹿TLR基因含有2~7個外顯子,其中TLR7基因具有最多的外顯子。馬鹿TLR基因基因長度在5 119(TLR9)~136 030 bp (TLR2)之間(圖2d)。不同基因的長度差異來自于內含子長度的差異上。
2.3 反芻動物的TLRs的分類
以藍鯨和人類TLRs基因作為外類群,與421條反芻動物TLRs基因序列構建的ML系統發育樹(圖3)表明,反芻動物TLR序列聚成5個基因簇,分別是TLR1 (TLR1/6/10)、TLR2、TLR4、TLR3(TLR3/5)、TLR7 (TLR7/8/9)亞家族。TLR1和TLR7亞家族擁有最多的TLR家族成員,TLR2和TLR4僅有1個直系同源基因。在每個TLR基因家族成員內部,TLR基因序列按照科聚類,叉角羚科和長頸鹿科、麝科和牛科各自形成姐妹群(圖3)。
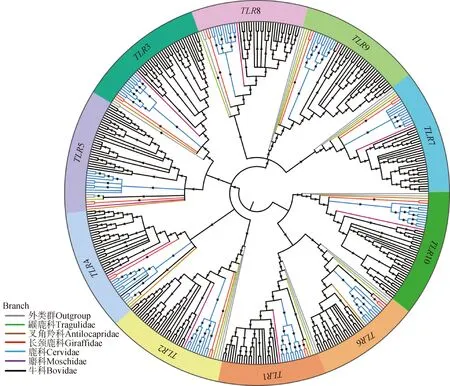
自展值大于70%的用*表示Bootstrap values larger than 70% are denoted by * 圖3 反芻動物TLR基因的最大似然樹Fig.3 ML tree of ruminant TLR genes
2.4 反芻動物TLRs自然選擇壓力
本研究檢測了53種動物10個TLR基因的自然選擇壓力模式和強度,非同義替換率(dN)和同義替換率(dS)的比值(ω)表明所有的TLR基因均受到純化選擇作用(表2),ω值從TLR8的0.121到TLR6的0.369,提示反芻動物的TLR基因受到一定的功能約束。
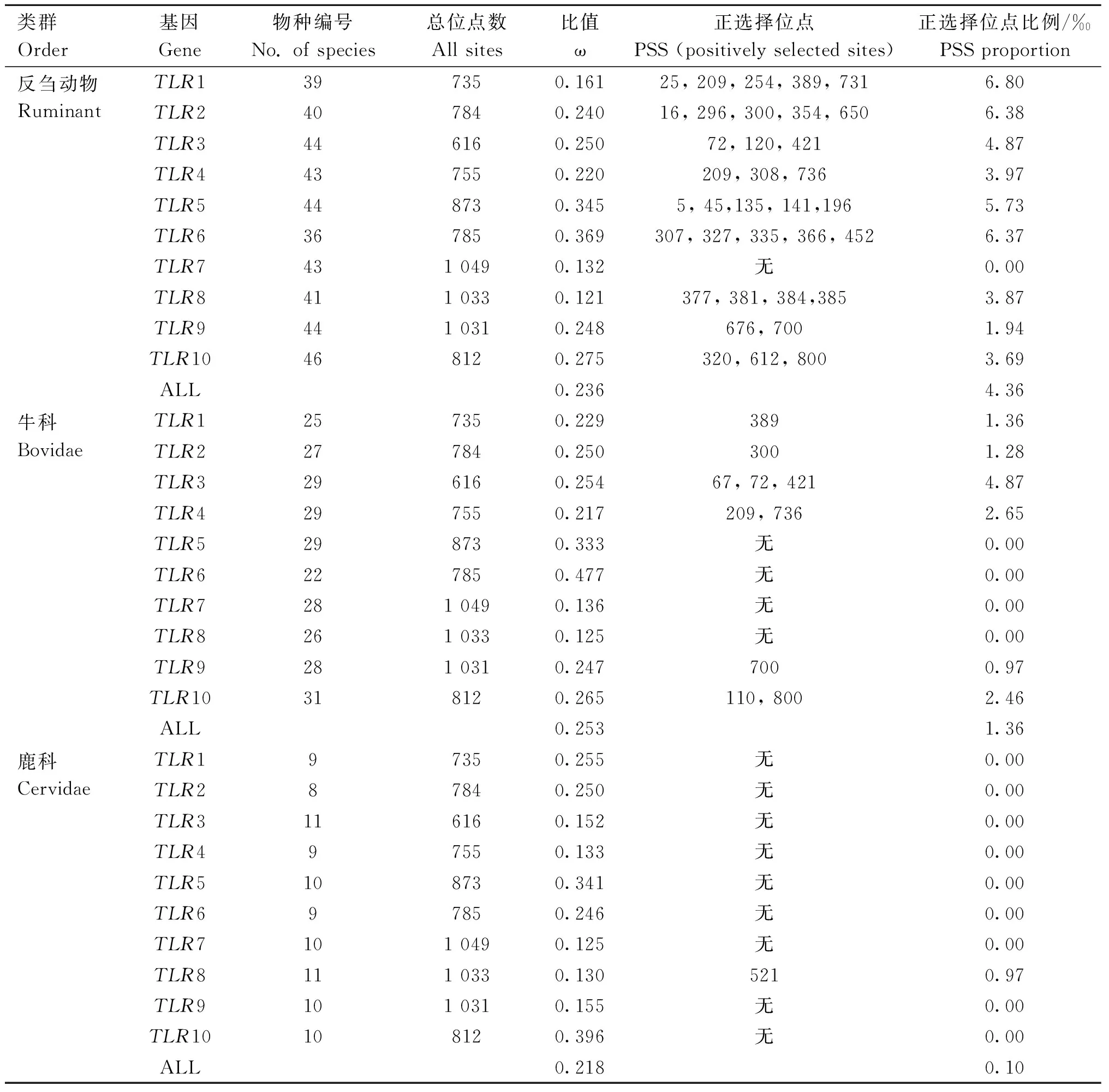
表2 反芻動物TLRs基因家族進化壓力Table 2 Evolutionary pressure of TLRs gene family in ruminants
盡管反芻動物的TLR基因整體受到明顯的純化選擇作用,但是在除了TLR7外的其他的TLR基因中均檢測到了顯著的正選擇位點(193個),其中35個是4種方法均鑒定到的(表2),提示這些位點受到強烈的正選擇作用。不同TLR基因的正選擇位點數目差異明顯,從TLR7的0到TLR1的6.80‰。在所有的直系同源TLR基因中,TLR8和TLR7分別擁有最低的ω值和最低比例的正選擇位點,提示這兩個基因受到的選擇約束高于其他直系同源TLR基因。牛科和鹿科ω均小于1,都受到強烈的純化選擇,但是相比于鹿科,牛科具有更高的ω值和更高比例的正選擇位點,提示鹿科TLR基因受到的約束力高于牛科(圖4a-b,表2;牛科ω=0.253,鹿科ω=0.218, χ2=7.303, df =1,P=0.007;牛科PSS比例為0.253‰,鹿科PSS比例為0.218‰,χ2=0, df=1,P>0.05)。在所有反芻動物的TLR基因中,非病毒性TLRs(TLR1/2/4/5/6/10)比病毒性TLRs(TLR3/7/8/9)具有更高的ω和更多的正選擇位點數目(圖4c-d,病毒性ω=0.188,非病毒性ω=0.264, t=1.650, df=8,P=0.138;病毒性PSS比例為2.67‰,非病毒性PSS比例為5.20‰, t=2.586, df=8,P=0.032)。
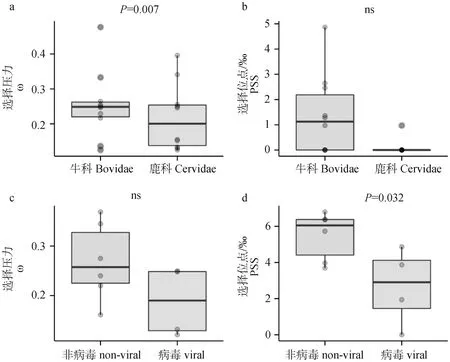
a. 牛科和鹿科的ω; b. 牛科和鹿科的正選擇位點的比例; c. 非病毒性和病毒性TLR的ω; d. 非病毒性和病毒性TLR的正選擇位點的比例。ns. 差異不顯著a. ω of Bovidae and Cervidae; b. Percentage of positively selected sites in Bovidae and Cervidae; c. ω of non-viral and viral TLRs; d. Percentage of positively selected sites in non-viral and viral TLRs. ns. Not significant圖4 反芻動物TLR基因的選擇壓力Fig.4 Selection pressure of TLR genes in ruminants
2.5 綿羊TLRs基因家族在不同組織的表達模式
本研究對綿羊10個TLRs基因家族成員在不同系統和組織中的組織表達量計算算術平均值,通過表達量熱圖發現幾乎所有TLR基因都在免疫組織器官上表達(圖5,灰色色塊顏色所示)。TLR2/9/1/4在免疫組織的表達量整體高于TLR3/7/5/8/6/10,TLR2和TLR9基因在外周血單個核細胞(peripheral blood mononuclear cell,PBMC)中存在高水平的表達。

圖5 綿羊TLR基因表達模式Fig.5 Expression patterns of TLR genes in Ovis aries
3 討 論
反芻動物對于人類社會具有重要的價值及生物學意義,對于反芻動物的TLR家族研究有助于理解反芻動物免疫系統的進化機制。在已報道文獻中,通過PCR擴增的方法確定牦牛、山羊、牛、綿羊[12,18-19,32]具有TLR1-TLR10 10個TLR基因,這些研究僅包含了13種反芻動物,約占反芻動物總數的5.6%(13/233)。本研究借助于NCBI中豐富的反芻亞目基因組數據,通過BLAST對反芻亞目的6個科,47個屬(約占65.8%),53個物種(約占物種總數的22.7%)的TLR基因家族進行了鑒定。較以往研究,本研究的物種覆蓋了反芻亞目全部6個科及一半以上的屬,首次對反芻亞目譜系的TLR基因家族的基因拷貝數目、自然選擇壓力進行了研究。
本研究發現,反芻亞目的10個TLRs基因均是單拷貝,表明TLR在反芻亞目中相對保守。在其他反芻動物的研究中,如牦牛[18]、山羊[19]、水牛[12],TLR1-10基因均為單拷貝,與本研究結果一致。在鳥類研究中發現TLR1、TLR2存在多拷貝現象[3]。除了鳥類,脊椎動物的其他類群都具有TLR1-10基因,表明TLR基因數目在脊椎動物中也是保守的。本研究采用小鼠的TLR11/12/13基因作為query序列,在反芻亞目所有物種中鑒定到的序列長度過短(低于query序列的50%),推測TLR11/12/13在反芻亞目中可能為假基因。對于TLR1-10,某些物種鑒定到的假基因,根據近源物種的情況,推測是由測序或者組裝拼接錯誤導致的。
本研究基于421條TLR序列的系統發育樹,將反芻亞目的TLR基因分為5個亞家族,分別是TLR1家族(TLR1/6/10)、TLR2家族(TLR2)、TLR3家族(TLR3/5)、TLR4家族(TLR4);TLR7家族(TLR7/8/9)。這與以往的TLR基因進化研究結果不完全一致[6,10,12]。在已有研究中,TLR1/6/10由于具有較高的序列相似性都被歸為同一個亞家族,TLR7/8/9與此類似,也聚為一簇,但TLR4與其他9個TLRs基因差異較大,單獨聚為一簇,與本研究結果一致[9-10]。
TLR1亞家族是最大的亞家族之一,包含TLR1、TLR6、TLR10。水牛、馬鹿(本研究)、家牛[33]、人類[6,10]的染色體位置表明TLR1、TLR6、TLR10位于同一條染色體,這一結果支持了3個TLRs基因進化可能是非獨立的。此外,這3個基因的高度相似性可能源于配體結合的特性,如TLR1、TLR6、TLR10與TLR配體結合作為異二聚體以傳遞細胞信號并誘導相關的免疫反應[34]。而TLR7亞家族的TLR7/8/9都在細胞內膜上表達,都具有種屬特異的配體,都屬于病毒型TLRs基因。有研究表明,TLR8和TLR9與它們的配體結合作為內體[35],表明保守的TLR在組合和傳輸細胞信號的過程中通過相似的模式在組合配體中起重要作用。此外,TLR7和TLR9在病毒衍生基序的TLR中的選擇模式與細菌PAMPs相互作用的模式有很大不同。而TLR7和TLR8位于同一條染色體。其他未組裝到染色體的基因組數據表明,超過80%的物種都顯示TLR1、TLR6和TLR10以及TLR7和TLR8都分別在同一個scaffold。以上可能是TLR1/6/10和TLR7/8/9分別被歸為同一亞家族的原因。TLR4亞家族只有TLR4基因,該基因是識別革蘭氏陰性菌LPS的一個免疫基因[34],與其他TLR基因相似性較低,表明TLR4與其他TLR基因可能在功能上存在差異。
而TLR2、3、5在以往不同的研究中聚類情況不盡相同。TLR2在有些研究中與TLR1/6/10被納入TLR1亞家族,如Liu等[10]對脊椎動物TLRs基因家族的研究。在本研究中,TLR2與TLR1/6/10聚為不同的基因簇,提示在反芻動物的進化過程中TLR2基因與其他3個基因產生了較大的分化。在脊椎動物TLR進化中,TLR3和TLR5聚為不同的簇,而反芻動物的TLR3和TLR5在序列上更為相似,故聚為一支,提示TLR3和TLR5在反芻動物外產生了分化。
ω結果表明,反芻動物TLRs基因家族成員都經歷了純化選擇,這與兩棲類[5-6]、鳥類[3,36]、魚類[37]等其它脊椎動物研究結果一致,表明TLRs基因功能在進化上是相對保守的。值得一提的是,反芻動物的TLRω(0.121~0.369)與快速進化的基因,如極光激酶(AKs)[38]、DAZ基因類似[39]。在科水平上,牛科總體選擇壓力大于鹿科,正選擇位點數量也多于鹿科反芻動物,表明鹿科在進化水平上受到更強的約束力。病毒型TLRs比非病毒型TLRs受到更強的功能選擇約束,這與前人關于靈長類、樹鼩類、兩棲類動物研究的結果一致[6,13]。這可能因為病毒TLRs在免疫應答中維持其識別病毒核酸功能,同時也要對自身損傷的結構進行識別,從而避免非同義替換的出現或者非同義突變的積累,以此來維持自身的功能[34]。此外,非病毒TLRs有些在功能上會存在冗余,會識別相同的病原微生物PAMPs,能夠接受該類病毒發生非同義突變,在種群受到正選擇時保留下來[6,34]。
綿羊的TLRs基因在免疫系統組織中高表達,該結果與以往研究結果一致[7,12,18],表明TLRs在抵抗病原體入侵中發揮重要作用。TLR2和TLR9基因在PBMC組織上高表達,PBMC包含較多的淋巴細胞(T細胞、B細胞、NK細胞),是先天免疫系統和適應性免疫系統的關鍵組成部分,可以很好的保護機體免受病毒、細菌的感染[40]。TLR3基因在綿羊內分泌系統中存在高表達現象,這可能與其具有獨特的識別雙鏈RNA病毒的功能有關,表明TLR3在綿羊內分泌系統疾病的發生中發揮重要作用,可能為綿羊生殖疾病引起的生殖能力低下和其它問題的解決提供有效幫助[7,34]。此外,本研究發現綿羊的TLR1和TLR4表達相近,TLR6和TLR10在所有組織中表達水平相似,這可能是因為它們在鑒定病原微生物和參與生物體的抗病性和免疫應答過程中相互配位且共同發揮作用[7-8]。
4 結 論
本研究闡明了反芻動物TLR基因家族的基因拷貝數目、系統發育關系、選擇壓力。結果表明,反芻動物的TLR1-10均是單拷貝基因,可歸為5個亞家族。盡管每個TLR基因在整體上都受到強烈的純化選擇作用,但是仍有氨基酸位點受到正選擇作用。相對于牛科,鹿科在進化歷程中受到更強的選擇約束。反芻動物的病毒性TLRs比非病毒性TLRs受到更強的自然選擇約束。由于本研究的局限性,無法確認在某些物種中鑒定到的TLR1-10假基因確實是由測序造成的。由于鼷鹿科、長頸鹿科和麝科所包含的物種數目均少于3個,不能滿足正選擇位點的分析條件,故這3個科在進化過程中的所受到的選擇作用仍不清楚。此外,雖然本研究利用多種方法檢測到反芻動物的多個正選擇位點,但是缺少疾病的表型數據,這些正選擇位點是否與疾病相關聯仍然是未知的,從而不能更好地反映Toll樣受體蛋白與其致病對應物之間的進化關系。在未來的研究中,可以通過正選擇位點與疾病的關聯分析并結合正選擇位點在蛋白三維結構功能區的定位研究來確認正選擇位點的功能。

