一株鼠冠狀病毒的全基因組序列特征分析
趙 靜,馬曉華,南曉偉,柴薩薩,李利利,段招軍,
冠狀病毒(Coronavirus,CoV)是一類單股正鏈RNA病毒,是RNA病毒中基因組最大的一類病毒,長度約為30 kb左右。冠狀病毒是人類和脊椎動物重要病原體,可引起呼吸道、腸道、肝臟和神經系統等多種器官的疾病[1]。冠狀病毒目前分為4個屬:Alpha冠狀病毒、Beta冠狀病毒、Gamma冠狀病毒和Delta冠狀病毒,其中Alpha冠狀病毒屬和Beta冠狀病毒屬主要感染哺乳動物,并引起呼吸系統和消化系統的癥狀;Gamma冠狀病毒屬和Delta冠狀病毒屬主要在禽類中發現[2]。Alpha冠狀病毒屬成員HCoV-229E、HCoV-OC43、HCoV-NL63和HCoV-HKU1已明確為可以感染人類的冠狀病毒成員[3];Beta冠狀病毒屬由A、B、C和D亞群組成[4],多個成員可引起嚴重疾病,如嚴重急性呼吸系統綜合征冠狀病毒(SARS-CoV)、中東呼吸綜合征冠狀病毒(MERS-CoV)和新型冠狀病毒(SARS-CoV-2)均曾經或正在引起全球重大公共衛生事件,尤其是SARS-CoV-2引起全球CoVID-19大流行對人類的影響是空前的。
鼠類(Rats)是一類自然界廣泛存在并且數量眾多的哺乳動物,屬于嚙齒目(Rodentia)鼠科(Muridae),是多種病原庫的天然儲存庫之一,也是人獸共患傳染病病原體來源之一[5],目前已基本明確嚙齒動物是沙粒病毒、漢坦病毒、呼腸孤病毒、黃病毒等多種人獸共患病病原體的自然宿主,其中許多病毒可引起嚴重的疾病[6]。鼠類攜帶病毒具有豐富的種類和多樣性,而鼠類與人類或家畜等接觸密切,這就增加了鼠類病原體跨種傳播感染人類的風險。已有研究表明,Alpha屬冠狀病毒成員人類冠狀病毒HKU-1和OC43可能是由鼠類中冠狀病毒發生跨種傳播到人類并引起傳播[6]。
冠狀病毒具有極高的基因多樣性,在世界各地的嚙齒動物、蝙蝠及其他野生動物中均廣泛分布[8-9]。已有研究表明SARS-CoV、MERS-CoV以及引起全球大流行的SARS-CoV-2均可能起源于野生動物,這提示了冠狀病毒造成人獸共患風險較高[10]。近幾年在全球尤其在我國新發現的鼠類冠狀病毒不斷增加[11-12]。在2021年有研究者系統評估了動物源性病毒的人獸共患潛力以及傳播潛力,發現了幾種與嚙齒動物有關的冠狀病毒被列在前50,其中就包括我國學者在我國浙江省鼠中發現的LAMV[13]。因此監測和發現野生動物中冠狀病毒對未來可能發生的冠狀病毒跨種傳播至關重要。本研究在我國內蒙古地區采樣的鼠類樣本進行病毒組研究的過程中發現一株新型Alpha冠狀病毒,并對全基因組的特點和系統進化進行分析,了解病毒的基因組特性和進化地位,豐富了鼠類中病毒組成的種類。
1 材料與方法
1.1 標本 于2016年在我國內蒙古自治區呼倫貝爾市莫力達瓦達斡爾族自治旗的鼠類進行樣本采集,依照不破壞種群的前提下,使用野外布鼠夾進行鼠類樣本收集,采集后送至當地生物安全二級實驗室進行解剖后收集組織樣本,最終獲得腸組織/糞便樣本61份,肝組織樣本61份以及肺組織樣本61份,干冰運送至中國疾病預防控制中心實驗室后置于-80 ℃中保存待用。
1.2 樣本前處理、核酸提取和高通量測序 在生物安全柜中取約0.3 g組織樣本置于1.5 mL離心管中,加入1 mL預冷的MEM培養基和4-6顆鋼珠(預先高壓),置于組織研磨儀中70 Hz研磨3 min。研磨液置于8 000 r/min離心10 min,將腸組織樣本分別混為4個樣本庫,肝組織樣本混為4個樣本庫,肺組織樣本混為1個樣本庫,共9個樣本庫進行下一步處理;取樣本庫上清用 0.22 μm的濾器過濾去除樣本中的細菌和組織碎片以減少樣本的背景;取過濾后的173 μL懸液中加入7 μL的DNase I和20 μL的10×DNase Buffer,37 ℃水浴1 h,使用核酸提取試劑盒根據說明書提取核酸,用Qubit2.0定量RNA濃度,將核酸送至深圳華大基因有限公司進行文庫構建和高通量測序。
1.3 鼠種類鑒定 根據文獻,鼠種類鑒定通過擴增鼠的細胞色素b基因部分片段進行鑒定[14]。通過BLAST比對確認鼠種類。引物序列如下:上游引物L7:5′-ACCAATGACATGAAAAATCATCG-TT-3′,下游引物H15915:5′-TCTCCATTTCTGGTTTACAAGAC-3′。
1.4 病毒逆轉錄及全基因組的擴增 將提取的核酸用SuperScript Ⅱ試劑盒根據說明書進行逆轉錄,得到cDNA置于-20 ℃儲存。為驗證病毒序列,根據高通量測序拼接獲得的病毒序列,使用Primer Premier 5設計覆蓋全部拼接序列的引物以驗證序列的準確性,全基因組擴增引物以cDNA作為模板,使用Premix Taq酶進行PCR擴增驗證,5′和3′末端采用RACE的方法進行擴增,用1.5%瓊脂糖凝膠電泳對上述PCR產物進行鑒定,送往北京天一輝遠公司進行測序。
1.5 序列比對拼接與系統發育構建 將測序結果用DNASTAR的SeqMan子程序進行拼接;利用MEGA11軟件進行多序列比對并采用最大似然法構建系統發育樹,Bootstrap設置為1 000;采用RDP4.0和SimPlot軟件對序列進行重組分析,SimPlot軟件Window Size設置為1 000,Step Size設置為100,參數模型設置為Kimura model。
2 結 果
2.1 基因組結構特性 我們在研究內蒙古地區的鼠類樣本病毒組成的過程中,在其中一個腸道組織樣本庫中發現了冠狀病毒的reads,組裝拼接得到一株新的Alpha屬冠狀病毒,設計引物驗證后該樣本庫的所有單個樣本進行篩查,結果顯示一只鼠的一份腸道樣本(MCX-13)為陽性,這只鼠類在樣本采集時根據解剖形態學鑒定為小家鼠,通過擴增鼠的細胞色素b基因部分片段進行鑒定結果確定該病毒來自于為小家鼠(Musmusculus)。
該病毒組全基因組長度為 27 674 bp,G+C含量為42.31%,暫命名為NMR-13其基因組長度與其最為相近的病毒株FiCoV/UMN2020接近,但較其他鼠冠狀病毒的長度較短。該病毒基因組與鼠類Alpha冠狀病毒屬的基因組結構類似,包含8個常見的開放閱讀框(Open Reading Frame,ORF)以及5′UTR和3′UTR區,依次為5′UTR-ORF1ab-S-ORF3-E-M-ORF6-N-ORF8-3′UTR,其中ORF1ab基因編碼非結構蛋白,S、E、M、N基因編碼結構蛋白,ORF3、ORF6和ORF8編碼3個輔助蛋白;此外,5′端包含286 bp的非編碼區和3′端包含266 bp非編碼區及polyA結構。NMR-13的基因組結構與我國浙江省發現的LRNV及多個相似的變異株以及英國發現的病毒株UKRn3 等鼠冠狀病毒相比,在ORF1ab和S基因之間缺少一個編碼NS2蛋白的ORF2開放閱讀框,該基因目前在多數的鼠類Alpha冠狀病毒中存在,而本研究中發現的NMR-13和與之相近的FiCoV/UMN2020 、AcCoV-JC34和Lijiang-170病毒株中未發現ORF2。此外,本研究發現的NMR-13在N基因下游3′非編碼區存在一個編碼109個氨基酸的ORF8。序列已上傳至GenBank,序列號為OQ939561.1。
冠狀病毒普遍包含保守的轉錄調控序列(Transcriptional Regulatory Sequence,TRS),NMR-13包含冠狀病毒科成員保守的轉錄所需的轉錄調控序列,與鼠類冠狀病毒LRNV 、AcCov-JC34及HKU3類似,在NMR-13基因組中5′-AACUAA-3′為主要核心轉錄調控序列,如表1所示。

表1 冠狀病毒基因組特點分析
2.2 同源性分析 同源性分析結果顯示,NMR-13與美國發現的FiCoV/UMN2020病毒株具有最高的相似性,為91.3%,各個ORF的核苷酸同源性為84.9%~92.8%,其中ORF8核苷酸和氨基酸同源性最低,僅為84.9% 和74.1%;FiCoV/UMN2020是一株實驗室小鼠體內的分離出的病毒。NMR-13其次與我國發現的多株鼠類冠狀病毒Lucheng/Lijiang-170、Lucheng/Ruian-83和Lucheng/Lijiang-71等多個病毒株同源性較高,為79.5%~81.0%,但S基因同源性低,僅為50.2%~61.4%,詳細分析見表2。根據blast比對結果顯示,相近的鼠類Alpha冠狀病毒除FiCoV/UMN2020和UKRn3病毒株,其余病毒株均來自于中國,提示了病毒傳播的區域性特點。NMR-13編碼的ORF8僅與FiCoV/UMN2020以及我國發現的鼠類冠狀病毒株和一株犬類冠狀病毒具有同源性,其中與FiCoV/UMN2020毒株的氨基酸同源性為74.1%,而與其它我國相近毒株的氨基酸同源性僅為46.4%~53.6%。
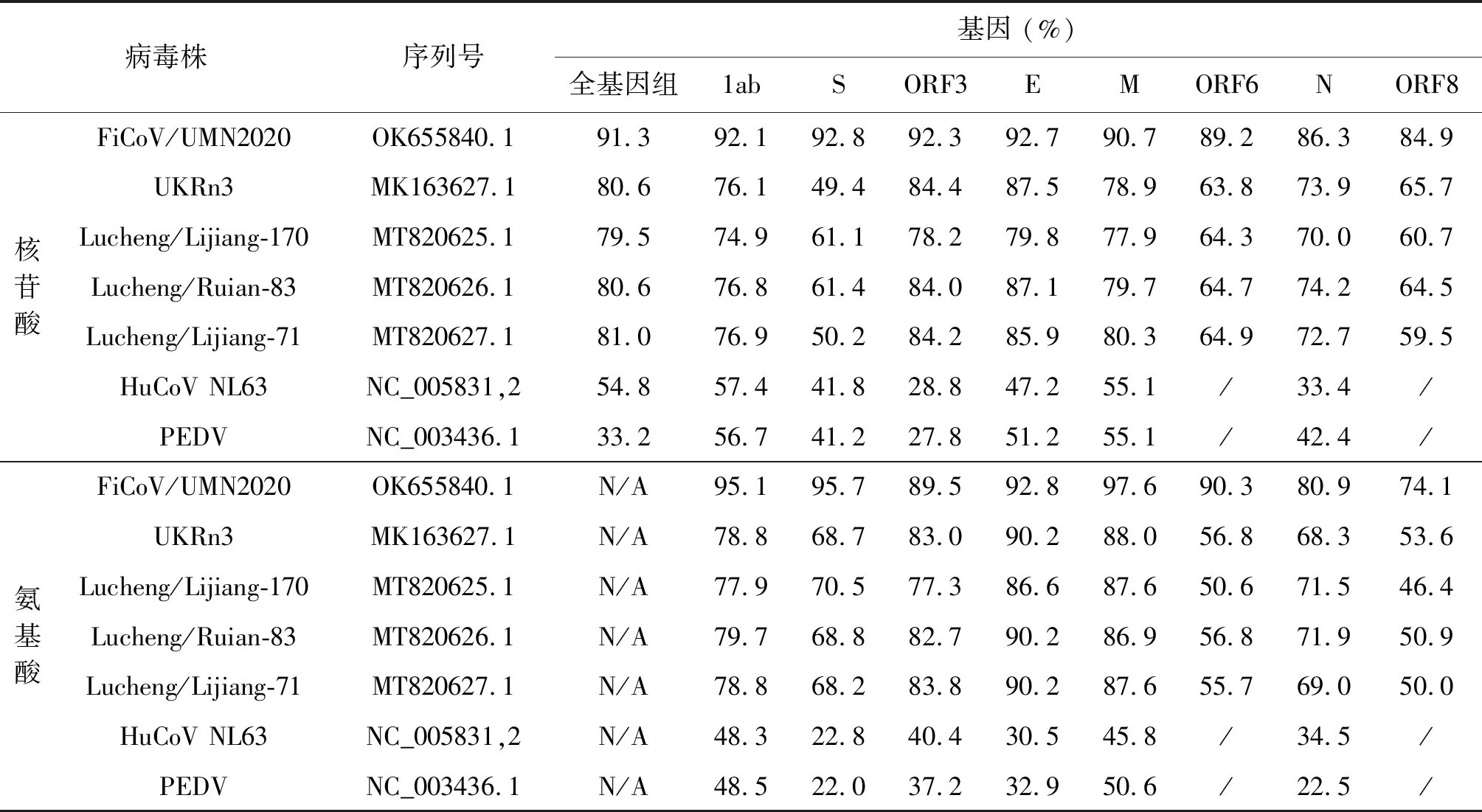
表2 NMR-13與參考株序列的相似度分析
2.3 系統發育分析 為確定NMR-13與已知冠狀病毒的進化關系,構建全基因組、RdRp區和S基因的最大似然樹。全基因組系統發育樹顯示所有的來自嚙齒動物Alpha冠狀病毒單獨聚為同一進化分支,包括來自我國云南、浙江和山東等地區發現的毒株、英國(UKRn3)和美國(FiCoV/UMN2020)的毒株。NMR-13與來自美國的FiCoV/UMN2020親緣關系最近聚在一起。基于S基因和RdRp區的構建的系統發育樹也顯示類似的拓撲結構,詳見圖1、圖2和圖3。目前發現的鼠類Alpha冠狀病毒多數發現自我國,包括云南、浙江和山東等地區。
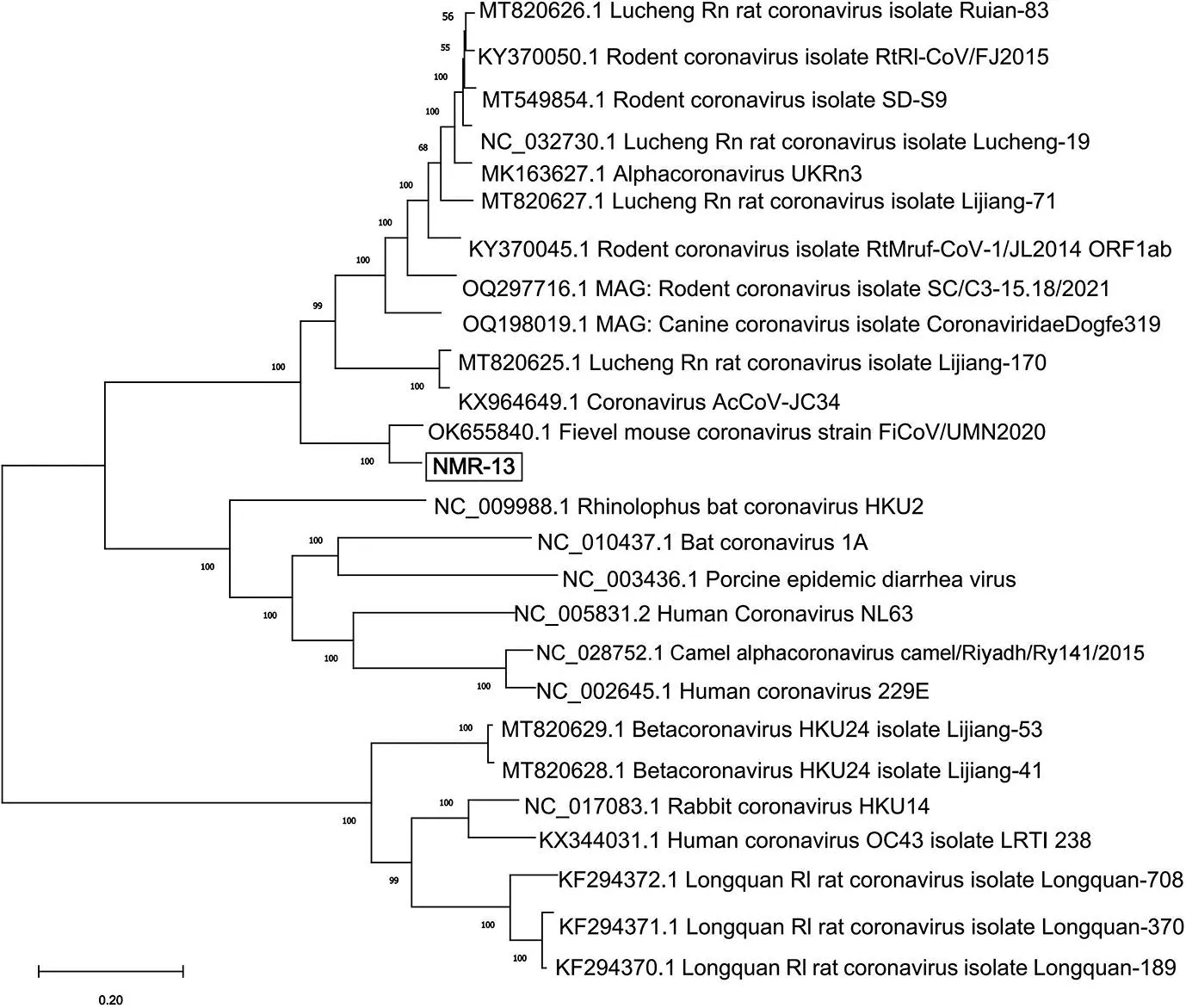
注:加粗字體代表本研究的毒株。

注:加粗字體代表本研究的毒株。
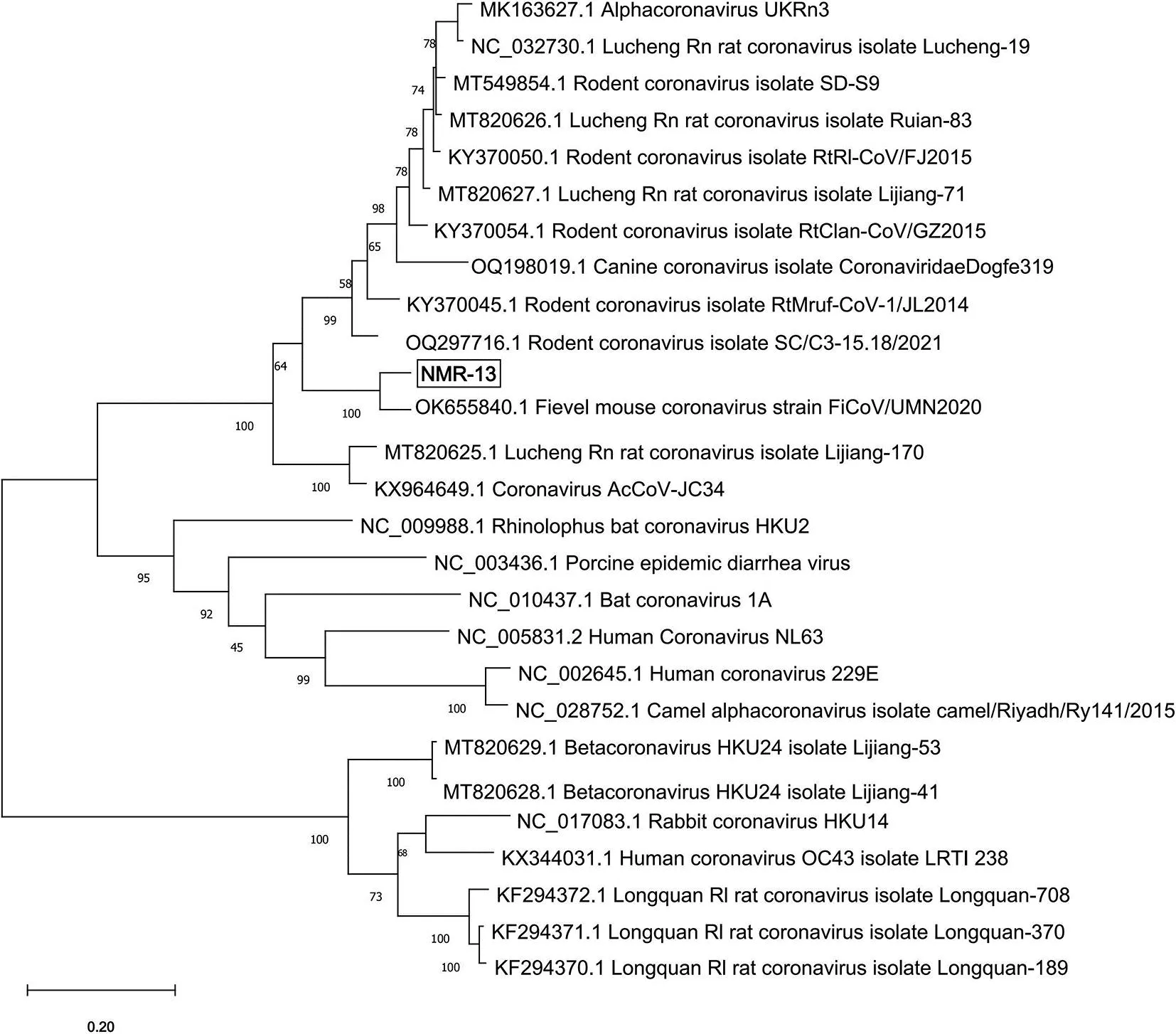
注:加粗字體代表本研究的毒株。
2.4 序列重組分析 冠狀病毒中重組情況較為常見[15],使用RDP4.0軟件和SimPlot軟件對NMR-13的全基因組和其他的冠狀病毒進行序列相似性分析和重組分析,以NMR-13作為參考序列進行重組分析,未發現該病毒株有重組現象。重組分析發現,NMR-13與我國Lucheng/Lijiang-170、Lucheng/Ruian-83和Lucheng/Lijiang-71等病毒株除S區同源性較低外,ORF1a的部分區域同源性也較低,見圖4。

圖4 NMR-13與已知冠狀病毒序列的重組分析)
3 討 論
冠狀病毒是在鼠類中常見的病毒之一,并且表現出較高的基因多樣性,具有廣泛的地理分布范圍。近年來發生多起冠狀病毒跨種傳播感染人類的事件發生,因此冠狀病毒是一種對人類具有重大威脅的人獸共患病原體。
本研究在鼠類中發現一株Alpha冠狀病毒屬成員,并獲得完整基因組,命名為NMR-13。同源性和系統發育分析表明NMR-13病毒株與發現自美國的FiCoV/UMN2020病毒株最為相近,全基因組及各ORF同源性在90%左右。NMR-13其次與我國發現的多株鼠Alpha冠狀病毒病毒株同源性僅為80%左右,根據全基因組、保守的RdRp區以及非保守的S基因構建的系統進化樹也支持NMR-13與FiCoV/UMN202最近的親緣關系而與其余我國鼠類Alpha冠狀病毒病毒株進化關系稍遠。鑒于中國與美國遙遠的地理距離,NMR-13與美國發現的毒株更近而與我國毒株更遠的原因可能與病毒在物種間的病毒傳播的的關系相關性小,而更可能與病毒在進化過程中的趨同效應有關。鼠類物種繁多,目前已在多種鼠類中發現Alpha冠狀病毒[11-12,16],本研究發現的NMR-13與最為相近的FiCoV/UMN2020病毒株均來自于小家鼠,在已有的研究對Alpha冠狀病毒是否可能與宿主具有共進化的關系研究中并未發現共進化的相關性,而更可能在鼠類不同物種間跨種傳播,形成鼠類Alpha冠狀病毒的多樣性。
NMR-13的基因組結構與我國浙江省發現的LRNV及多個相似的變異株以及英國發現的病毒株UKRn3 等鼠冠狀病毒相比,在ORF1ab和S基因之間缺少一個預測的編碼NS2蛋白的ORF2開放閱讀框。在以往發現的多個鼠類Alpha冠狀病毒,多數的病毒株在ORF1ab與S基因之間包含一個NS2基因,包括我國發現的Lijiang-71、Lucheng-19和Ruian-83以及英國發現的UKRn3,這些病毒株基因組的NS2蛋白均與Beta冠狀病毒的NS2基因具有最高同源性,約為40%左右,這可能提示該基因可能來自于未被發現的Beta冠狀病毒,且是在較為久遠的歷史進化中獲得。而在本研究中發現的NMR-13、FiCoV/UMN2020、Lijiang-170和AcCoV-JC34病毒株中未發現NS2的存在[11-12]。已發現的鼠類Alpha冠狀病毒ORF1ab和S基因之間編碼的蛋白有兩種形式,即插入NS2或插入NS2和(NS)2b兩個假設的蛋白,目前該基因的功能尚不明確[11]。Alpha冠狀病毒一般具有相似的基因組結構,包括ORF1ab-S-ORF3-E-M-N,而在一些種或毒株種,存在一些額外的ORF,例如TGEV, BatCoV、HKU2和BatCoV-512等毒株在其近3′末端包含一個ORF7。本研究中的NMR-13在近3′非編碼區包含一個額外的編碼109個氨基酸的ORF8,該ORF也僅在部分鼠冠狀病毒和一株犬冠狀病毒中發現,各毒株由于發現前后的區別,目前在GenBank中有些命名為ORF8而有些命名為ORF9,該ORF的功能目前尚不清楚。
本研究豐富了鼠類中冠狀病毒多樣性,冠狀病毒多以引起重大公共衛生事件的身份進入人們視野,Alpha屬冠狀病毒可以感染許多嚙齒動物,而嚙齒動物是地球上最多樣化的哺乳動物,他們與人類接觸密切,是人獸共患傳染病重要的病原體來源之一,未來的研究應集中于了解冠狀病毒的多樣性和宿主間跨越的可能性,因此我們需要對哺乳動物種群的冠狀病毒感染進行更廣泛的監測。
利益沖突:無

