在不同養殖背景下鱖mc1r組織表達差異和啟動子轉錄調控分析
賈曉丹 梁旭方 何 珊
(1.華中農業大學水產學院,華中農業大學鱖魚研究中心,武漢 430070;2.農業農村部淡水生物繁育重點實驗室/農業動物遺傳育種與繁殖教育部重點實驗室,武漢 430070)
體色在動物的生存繁衍、逃避敵害、信號交流和光保護等方面起著重要作用,是脊椎動物多樣性最豐富的形態之一[1]。魚體顏色不僅會隨著生命周期的改變而改變,紫外線、攝食及水體背景顏色也會影響魚體顏色[2]。例如斑馬魚(Danio rerio)會迅速通過細胞中黑色素的聚集或分散來適應環境的變化[3]。魚類之所以擁有五彩斑斕的體色,是由于魚體中存在無數的色素細胞,而黑色素細胞是目前研究最為廣泛且深入的色素細胞類型[4,5]。
黑素皮質素 1 受體基因(Melanocortin 1 receptor,mc1r)屬于黑素皮質素家族成員,最早在黑色素細胞中被發現,是皮膚黑色素合成的關鍵基因[1],mc1r無內含子且其蛋白包含7個跨膜結構域,mc1r可以與促黑素細胞激素(α-MSH)與促腎上腺皮質激素(ACTH)結合形成偶聯受體,并且α-MSH與mc1r的親和力更高,是皮膚黑色素合成主要信號通路[7,8]。目前,mc1r結構、功能及毛色表型在人、鼠、豬和牛等哺乳動物中已有較為深入研究,在大菱鲆(Scophthalmus maximus)、虹鱒(Oncorhynchus mykiss)、歐洲鱸(Dicentrarchus labrax)和斑馬魚等少數魚類中也進行了初步研究[4,9,10]。有研究顯示魚類體色的深淺與mc1r基因的表達量呈負相關[11]。斑馬魚mc1r基因敲除導致其背部和腹側皮膚色素沉著顯著減少[12]。mc1r基因的改變與穴居魚(Astyanax)色素沉積減少有關[13]。甌江彩鯉(Cyprinus carpio var.color)在敲除mc1r基因后,會改變皮膚斑塊形成模式[14]。在日本比目魚(Paralichthys olivaceus)中發現mc1r在色素沉著區域具有高的基因表達量[15]。同時mc1r可以作為魚類系統發育研究的良好分子標記。
鱖(Siniperca chuatsi)隸屬于鱸形目,真鱸科,鱖屬,是主要原產湖北的我國傳統名貴肉食性魚,肉質細嫩,營養價值高,是我國淡水養殖的理想選擇[16]。養殖過程發現,同種鱖在不同的養殖環境中其體色及色彩圖案有明顯區別。為了清楚mc1r在不同表型魚體中的表達模式,以及轉錄調控機制,本實驗擬以鱖為研究對象,從鱖基因組數據庫中獲取mc1r基因序列并進行相關的生物信息學分析,其次通過實時熒光定量PCR分析不同養殖背景下產生的深淺色鱖組織表達差異。最后通過對mc1r基因5′-側翼區啟動子序列進行克隆和轉錄活性分析,鑒定鱖mc1r基因核心啟動子區域及關鍵轉錄因子,從而為魚類mc1r基因的表達調控及后續研究提供理論依據。
1 材料與方法
1.1 實驗用魚及實驗條件
實驗所用鱖來自華中農業大學鱖研究中心,將實驗魚分別放入黑、白背景并帶有連續水過濾和通風系統的圓形魚缸中,魚缸直徑為1 m,水深1.2 m,光照為自然光。每個魚缸12尾魚(80±5) g,每組3個重復,正常飼養3個月。在養殖結束后,從每個魚缸中隨機選取6尾魚,150 mg/L MS-222麻醉后解剖以獲得肝臟、腸道、肌肉、背部皮膚、腹部皮膚、鰓、心臟、眼睛、大腦和腎臟組織,并在液氮中快速冷凍,隨后在-80℃冰箱保存,用于后續基因表達分析。
1.2 mc1r生物信息學分析
鱖mc1r基因序列來自筆者所在課題組翹嘴鱖基因組數據庫,利用NCBI (https://www.ncbi.nlm.nih.gov/) 檢索人(NP_002377.4)、鼠(NP_032585.2)、歐洲鱸(CAY39344.1)、大口黑鱸(XP_038556432.1)、尼羅羅非魚(XP_005467175.1)、青鳉(XP_01149 0066.1)、斑馬魚(NP_851301.1)、虹鱒(NP_00118 2107.1)、河豚(NP_001127927.1)、大西洋鮭(XP_013982515.1)、大菱鲆(XP_035485284.1)、日本比目魚(XP_019954259.1)、尖吻鱸(XP_01852 3508.1)、雞(NP_001026633.2)氨基酸序列。使用SMART(https://smart.embl-heidelberg.de/)分析蛋白質結構域,使用SOPMA(https://npsa-prabi.ibcp.fr/)和SWISS-MODEL(http://www.swissmodel.expasy.org/)分析蛋白質二級和三級結構,利用Clustal分析氨基酸序列一致性,并利用MEGA10.0軟件以鄰接法(neighbor-joining)構建系統發育進化樹(bootstrap=1000)。
1.3 組織表達分析
利用NCBI設計mc1r的特異性引物mc1r-F和mc1r-R,并以鱖核糖體蛋白L13a(rpl13a)作內參基因用于組織表達分析,所用引物見表1。使用RNAiso Plus (TaKaRa)提取鱖不同組織總RNA,然后用多功能酶標儀(Bio-Tek)和1.2%瓊脂糖凝膠電泳檢測提取RNA的純度和濃度。使用HiScript?ⅡReverse Transcriptase (Vazyme)反轉錄合成cDNA,置于-20℃ 冰箱保存。以cDNA為模板,使用SYBR qPCR Master Mix試劑盒(Vazyme)進行RT-qPCR,RT-qPCR反應體系為模板cDNA 1 μL,上游引物和下游引物各0.4 μL,SYBR 10 μL,用ddH2O將反應體系補足到20 μL。反應程序為95℃,預變性3min,95℃變性10s,57℃退火30s,進行39個循環,72℃延伸45s。并使用2-ΔΔCt值法測定mc1r基因的相對表達量[17]。

表1 組織表達分析所用引物Tab.1 Primers for tissue expression analysis
1.4 mc1r基因啟動子不同片段克隆和分析
通過在線軟件JASPAR(http://jaspar.genereg.net)、AliBaba2.1(http://gene regulation.com/pub/programs/alibaba2/index.html)預測潛在的轉錄因子結合位點。轉錄起始位點(TSS)和TATA盒由BDGP(http://www.fruitfly.org/seqtools/promoter.html) 和Promoter 2.0 (http://www.cbs.dtu.dk/services/Promoter/)預測。以鱖仔魚DNA為模板,選擇鱖mc1r第一外顯子之前的啟動子序列作為研究對象,設計啟動子不同長度片段的擴增引物,所設計的引物見表2,進行普通PCR擴增。PCR反應體系為20 μL: 鱖基因組DNA 1 μL,ddH2O 8 μL,高保真酶10 μL,上下游引物各0.5 μL。反應程序: 95℃預變性5min,95℃變性30s,57℃退火15s,72℃延伸(2 kb/min),35個循環;72℃延伸5min。
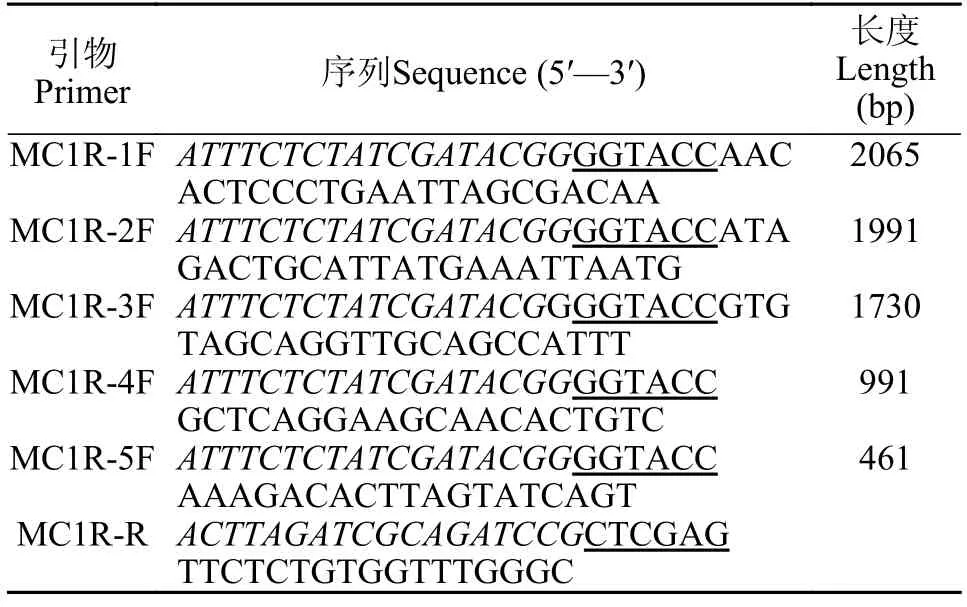
表2 鱖魚基因啟動子不同長度片段的擴增引物Tab.2 Primers used for fragments amplification of Chinese perchmc1rpromoter
1.5 雙熒光素酶質粒構建
將pGL3-Basic質粒用kpnⅠ和XhoⅠ內切酶雙酶切。將擴增正確的PCR產物膠回收后克隆到雙酶切的pGL3-Basic質粒中,選擇陽性重組質粒進行測序。測序正確的重組質粒分別命名為pGL3-2065、pGL3-1903、pGL3-1730、pGL3-991和pGL3-461。
1.6 細胞培養、瞬時轉染及雙熒光素酶活性測定
如前所述,進行了細胞培養、瞬時轉染和雙熒光素酶活性測定[18]。HEK 293T細胞在轉染前在24孔板中鋪板,并在37°C的充滿5% CO2的培養箱中過夜培養,使轉染當天的細胞總量達到75%。然后按照制造商的說明使用lip2000 (Promega) 以pGL3-basic載體作為陰性對照,轉染鱖mc1r不同啟動子片段重組質粒。每孔轉染液為opti-MEM培養基100 μL,質粒500 ng,pRL-TK 20 ng,lip2000 1.5 μg。培養24h后,將轉染后的細胞用1x DPBS洗滌兩次后用裂解緩沖液(Promega)裂解細胞,之后收集細胞,并根據制造商說明書使用雙熒光素酶檢測試劑盒(Promega)檢測mc1r啟動子相對活性。啟動子的相對活性用螢火熒光素酶的值/海腎熒光素酶的值表示。
1.7 數據分析
采用SPSS 25.0軟件進行統計學分析。以鱖rpl13a作為內參基因,采用2-ΔΔCt方法計算相對基因表達水平[17],單因素方差分析(One-way ANOVA)用來檢測樣本之間的差異性。使用GraphPad Prism 7軟件進行繪圖。統計學上的顯著性水平設置為P<0.05。
2 結果
2.1 鱖體色
鱖體色如圖1所示,在黑色和白色背景魚缸中養殖3個月后,黑色背景魚缸中養殖的鱖體色較深(圖1A),而白色背景魚缸中養殖的鱖體色較淺(圖1B)。

圖1 黑、白色養殖背景產生的深色鱖和淺色鱖Fig.1 Black and white tank backgrounds produced the dark and light-colored Chinese perch phenotype
2.2 鱖mc1r基因序列和系統發育分析
根據鱖基因組數據庫和NCBI下載的基因序列可知,mc1r基因開閱讀框為978 bp,編碼325個氨基酸且3′末端具有1 bp的Poly(A)尾巴,但不含脊椎動物典型的加尾信號(AATAAA)。通過TMHMM和Conserved Domains分析發現鱖mc1r基因的蛋白也為7跨膜結構域,其他報道物種相符。二級結構分析顯示Mc1r蛋白包含169個α螺旋(52.00%)、10個β折疊(3.08%)、98個無規卷曲(27.38%)和57個延伸結構(17.54%)。三級結構分析表明,該蛋白也主要以α螺旋和無規卷曲為主(圖2)。氨基酸序列比對和系統發育分析顯示,鱖mc1r基因與大口黑鱸(Micropterus salmoides)同源性高達95.7%,與歐洲鱸(Dicentrarchus labrax)同源性為95.4%,且聚為一支;其他魚類如斑馬魚(Danio rerio)、虹鱒(Oncorhynchus mykiss)和大西洋鮭(Salmo salar)聚為另一支;而與人類(Homo sapiens)及小鼠(Mus musculus)的同源性較低(圖3)。
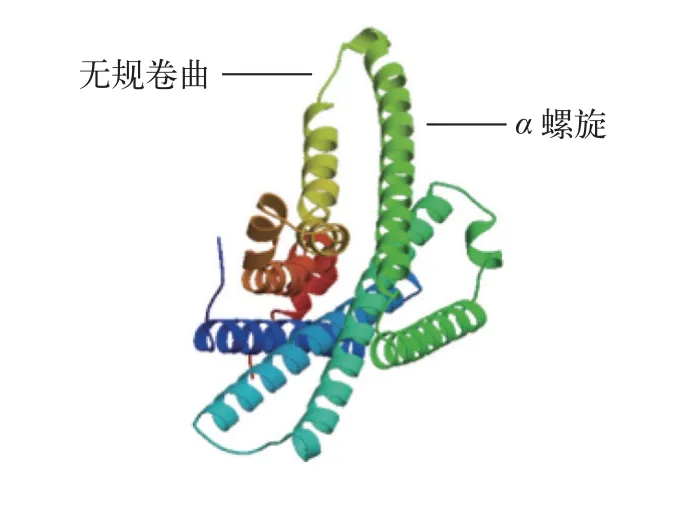
圖2 鱖Mc1r蛋白的三級結構Fig.2 Tertiary structure of Mc1r protein in Chinese perch
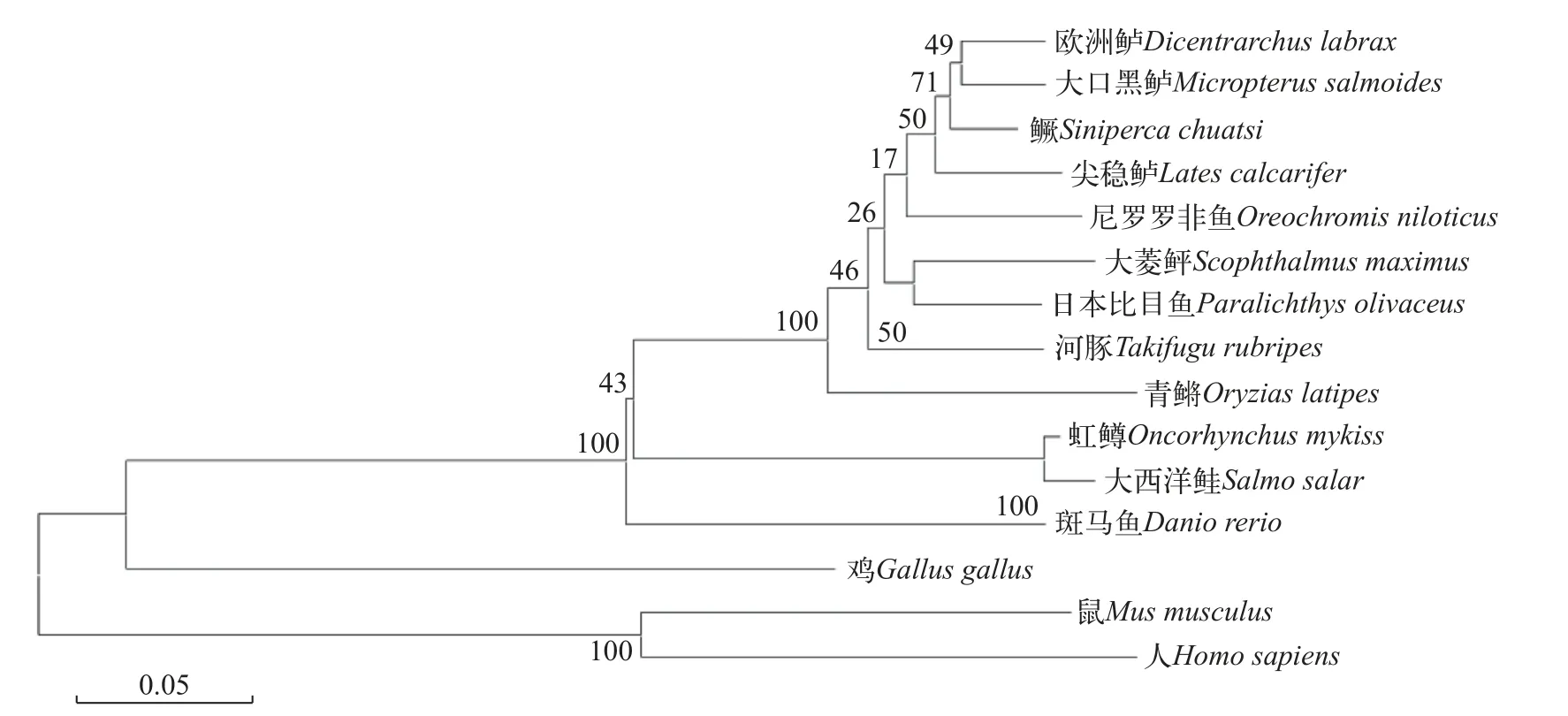
圖3 鱖與其他脊椎動物mc1r基因氨基酸序列系統進化樹Fig.3 Phylogenetic tree of amino acid sequence ofmc1rgene in andarin fish and other vertebrates
2.3 表達差異分析
如圖4所示,對于深色體表鱖,其表達量由高到低依次為背部皮膚、腹部皮膚、肝臟、眼、腦、腎臟、肌肉、心臟、鰓和腸,且背部皮膚表達量最高,顯著高于其他組(P<0.05)。在淺色體表鱖中,腦中表達量最高,其次是腹部皮膚,且顯著高于其他組(P<0.05)。
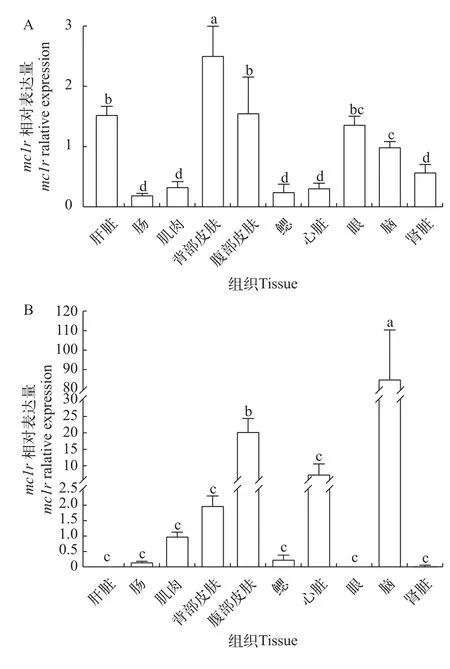
圖4 鱖mc1r在不同組織中的表達分析Fig.4 Analysis ofmc1rexpression among different tissues in Chinese perch
2.4 mc1r基因啟動子表達載體構建
將構建成功的pGL3-2065、pGL3-1903、pGL3-1730、pGL3-991和pGL3-461重組質粒電泳檢測發現,目的條帶與PCR產物條帶一致,經測序與啟動子序列一致,表明重組質粒構建成功,并可以用于啟動子活性分析。
2.5 mc1r啟動子報告基因活性檢測
將構建好的鱖mc1r重組質粒和pGL3-basic對照質粒分別與pRL-TK瞬時共轉染到HEK 293T細胞,培養24h后收集細胞,并檢測各重組質粒和對照質粒的熒光值,分析各個重組質粒的活性。結果顯示,重組質粒熒光活性與對照質粒pGL3-Basic均有顯著性差異(P<0.05),其中pGL3-461的相對熒光活性最高,提示-159∽+292為鱖mc1r基因啟動子的核心區域。隨著啟動子區域逐漸截短,pGL3-991熒光素酶活性明顯低于pGL3-461,說明-159∽+292 bp可能存在轉錄激活子結合位點或正向調控元件;-698∽-160 bp可能具有轉錄抑制因子結合位點或負調控元件(圖5)。通過在線軟件JASPAR和AliBaba 2.1預測可知預測出的核心啟動子區域存在小眼畸形相關轉錄因子(MITF)和SP-1等關鍵轉錄因子結合位點(圖6)。
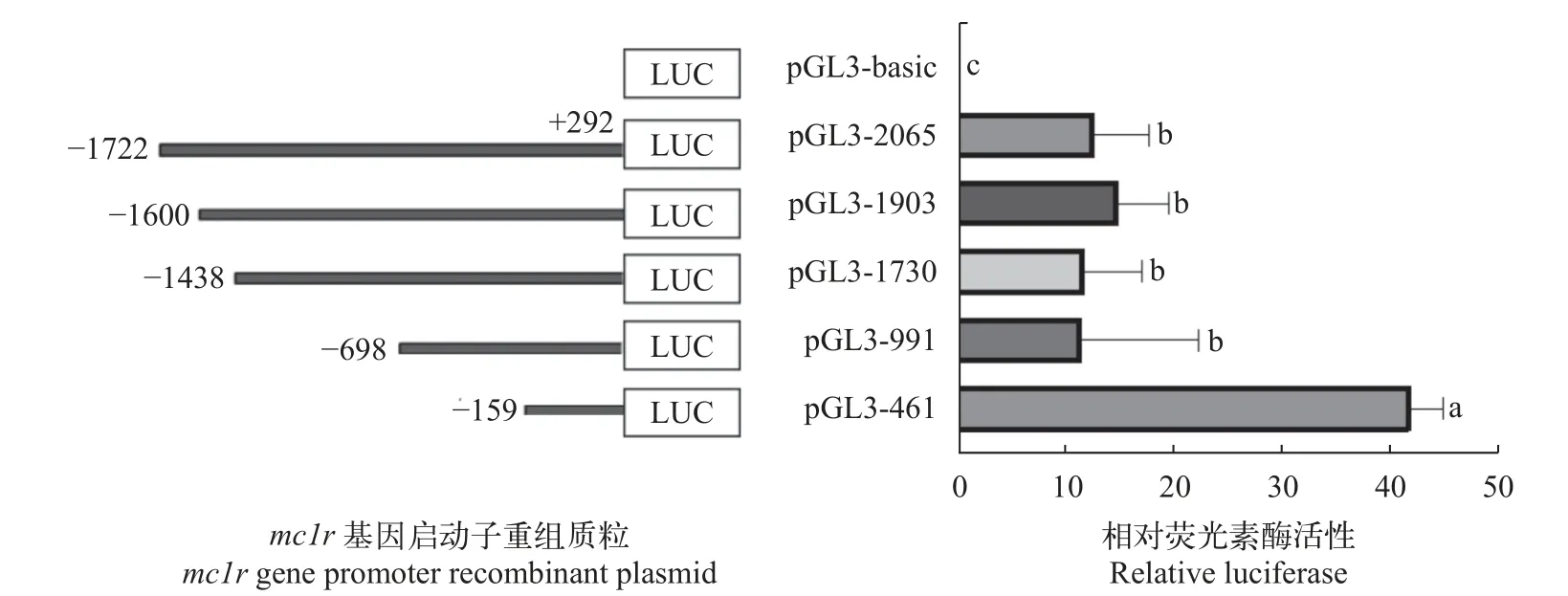
圖5 鱖mc1r基因不同啟動子重組質粒的相對熒光素酶活性Fig.5 Relative luciferase activity of recombinant plasmid ofmc1rgene in Chinese perch

圖6 轉錄因子結合位點Fig.6 Transcription factor binding sites
3 討論
3.1 鱖mc1r分子特征及系統發育
盡管已有100多個基因位點被確定與脊椎動物色素沉著有關,但黑皮質素系統始終是色素表型的關鍵決定因素[19,20]。有大量的研究表明mc1r與動物毛色、膚色等緊密相關,其在參與色素合成及色素沉積方面發揮了重要作用。哺乳動物和鳥類的皮膚和毛發顏色變化緩慢,通常需要數周時間。然而,在不同的養殖環境中,魚類可以通過黑色素顆粒的快速運動迅速改變身體顏色[21]。在本研究中,通過對鱖mc1r序列分析可知mc1r基因開閱讀框為978 bp,編碼325個氨基酸,屬于G蛋白偶聯受體超家族,在魚類及哺乳動物序列中高度保守,有7個跨膜結構域,與其他報道物種相符[21,22]。mc1r在迄今為止分析的所有魚類和四足動物物種中以單拷貝基因存在[12,24]。通過氨基酸序列同源比對和系統發育進化樹分析發現,鱖mc1r在硬骨魚類和哺乳動物間呈現不同的同源性及親緣關系。其中,鱖與歐洲鱸和大口黑鱸序列相似性最高,親緣關系更近,與哺乳動物親緣關系最遠,這與鱖隸屬于鱸形目相符合。
3.2 不同養殖背景下鱖體色及mc1r組織表達差異
本研究將鱖分別養殖在黑、白色背景魚缸中,與白色養殖背景中的淺色體表鱖相比較,在深色魚缸中鱖體色呈現黃綠色,頭部的細長條紋、軀干部縱寬的條紋、斑塊及鰭上的斑點都更清晰可見,與野生型鱖更為接近。鱖是生活在江河、湖泊、水庫中的底棲性魚類,通常白天棲息于水底或草叢茂密的地方,到晨昏或夜間才到臨岸的草叢中覓食,因此黃綠色體色及體表的斑塊或條紋更有利于隱藏自身來達到躲避敵害和順利捕食的目的[23,24]。因此,從生活習性角度考慮鱖可能更適合于養殖在黑色或深色背景中,這可能也有利于獲得健康優質鱖,促進鱖產業健康可持續發展。
本研究采用 RT-qPCR 方法檢測了鱖mc1r在不同養殖環境中的組織表達差異,結果顯示,在深色和淺色體表中鱖mc1r基因表達有很大差異。對于深色體表鱖,其背部皮膚表達量最高,其次是腹部皮膚、肝、眼、腦、腎,在心、鰓、肌肉和腸道表達量最低;對于淺色體表鱖,其在腦中表達量最高,其次是腹部皮膚和心,在背部皮膚、肌肉、鰓、腸道、腎、眼和肝中表達量最低,且腦中mc1r表達量與其他組織相比差異極顯著。這與mc1r基因參與色素沉積及與黑素皮質激素(α-MSH和ACTH)結合在魚類黑素生成途徑中發揮重要作用相一致。通過對不同背景顏色培養的東星斑 (Plectropomus leopardus)背部皮膚組織進行轉錄組分析,揭示mc1r表達量隨著背景顏色的加深而增加[25]。有研究表明,明亮的背景會導致魚體的黑色素聚集,從而導致皮膚顏色變淺;而背景顏色加深會使得黑色素分散,從而使得整個皮膚顏色加深[26]。然而,在不同魚類中,mc1r基因有著不同的表達模式。斑馬魚在大腦、眼睛、皮膚和精巢中表達,但在鰓、肝臟、肌肉和卵巢中不表達[25]。青鳉在所有組織中都有表達,包括大腦、眼睛、肌肉和皮膚等[24]。在錦鯉(Cyprinus carpio)中,其在黑色皮膚和眼中有最高的表達量,在白色皮膚、腦和肌肉表達量稍低,鰓、腎和肝臟表達量最低[26]。一些研究表明,在長期適應亮或暗背景的過程中,魚也可以通過減少或增加黑色素細胞的數量和大小來改變其顏色[21,27]。研究顯示在硬骨魚類中,背側和腹側區域顏色之間的明顯分界通過色素細胞的圖案化分布實現[20]。
3.3 鱖核心啟動子區域及關鍵轉錄因子分析
為了探究鱖mc1r啟動子區域的轉錄活性,根據在線軟件初步預測的核心啟動子區域,構建了5個5′端序列缺失報告基因載體,結果顯示不同缺失片段啟動子活性之間存在顯著差異,且-159∽+292 bp啟動子區域熒光活性最高,推測該區域為核心啟動子區域。通過在線軟件預測出核心啟動子區域存在SP-1、MITF等關鍵轉錄因子。有研究表明,在黑色素瘤細胞中,人的mc1r啟動子在-517∽-282 bp區域具有最高的熒光素酶活性,且該區域內的SP-1轉錄因子結合位點刪除后其熒光活性顯著降低[28]。在小鼠中,MITF共轉染情況下,其mc1r啟動子區域從-483 bp熒光活性顯著增加,表明MITF可以調控mc1r基因的表達[29]。MITF被認為是黑色素細胞的主要調節因子,因為它調節細胞分化、生長、存活和黑色素合成。同時,MITF也被證明參與mc1r的轉錄[30,31],一致的,有研究表明MITF可以通過黑色素細胞特異性基因(mc1r)中的E-box結合位點直接激活轉錄來調控黑色素細胞的分化[35]。因此,SP-1和MITF可能通過與mc1r啟動子區域轉錄因子結合位點結合后調控轉錄過程,促進黑色素細胞的分化與黑色素顆沉積,從而改變體色。
綜上所述,本研究中鱖mc1r基因在深淺色體表鱖組織表達存在顯著差異,深色體表鱖在背部皮膚和腹部皮膚高表達,淺色體表鱖在腦中和腹部皮膚高表達。報告基因顯示-159∽+292 bp啟動子區域熒光活性最高,推測該區域為核心啟動子區域。本研究首次克隆了鱖mc1r基因啟動子區域并進行了轉錄活性分析,并且對潛在的轉錄因子進行了預測,為魚類mc1r基因分子機制表達調控及表皮顏色可控性研究提供了理論依據。

