基于COⅠ及RAG2基因序列的我國科魚類分子系統(tǒng)研究
蘇國茂,林聰左,林貞武,黎幸有,梁日深,黃運銀
( 1.仲愷農(nóng)業(yè)工程學院 動物科技學院,廣東 廣州 510225;2.湖北生物科技職業(yè)學院,湖北 武漢 430070 )
蘇國茂1,林聰左1,林貞武1,黎幸有1,梁日深1,黃運銀2
( 1.仲愷農(nóng)業(yè)工程學院 動物科技學院,廣東 廣州 510225;2.湖北生物科技職業(yè)學院,湖北 武漢 430070 )



近年來,隨著分子生物技術的快速發(fā)展,應用合適的線粒體DNA及核DNA分子標記技術可在基因水平上有效解決魚類形態(tài)分類上懸而未決的難題[9-12]。線粒體DNA中細胞色素C氧化酶亞基Ⅰ(COⅠ)基因進化適中,已被驗證為魚類物種鑒定、分子系統(tǒng)發(fā)育研究中可靠的遺傳標記。同時,COⅠ基因5′端一段長度為648 bp的片段,能在基因水平上成功區(qū)分物種,已被廣泛應用于物種的分子鑒定[13]。重組激活基因(RAGs)是脊椎動物特異性免疫反應的關鍵基因,它由RAG1和RAG2兩個基因組成,在引起V(D)J基因重排及淋巴系細胞發(fā)育過程中起到關鍵性作用[14-15]。其中,RAG2是可靠的核基因遺傳標記,目前已經(jīng)廣泛應用于硬骨魚類系統(tǒng)進化分析的研究[16-18]。

1 材料與方法
1.1 材料來源

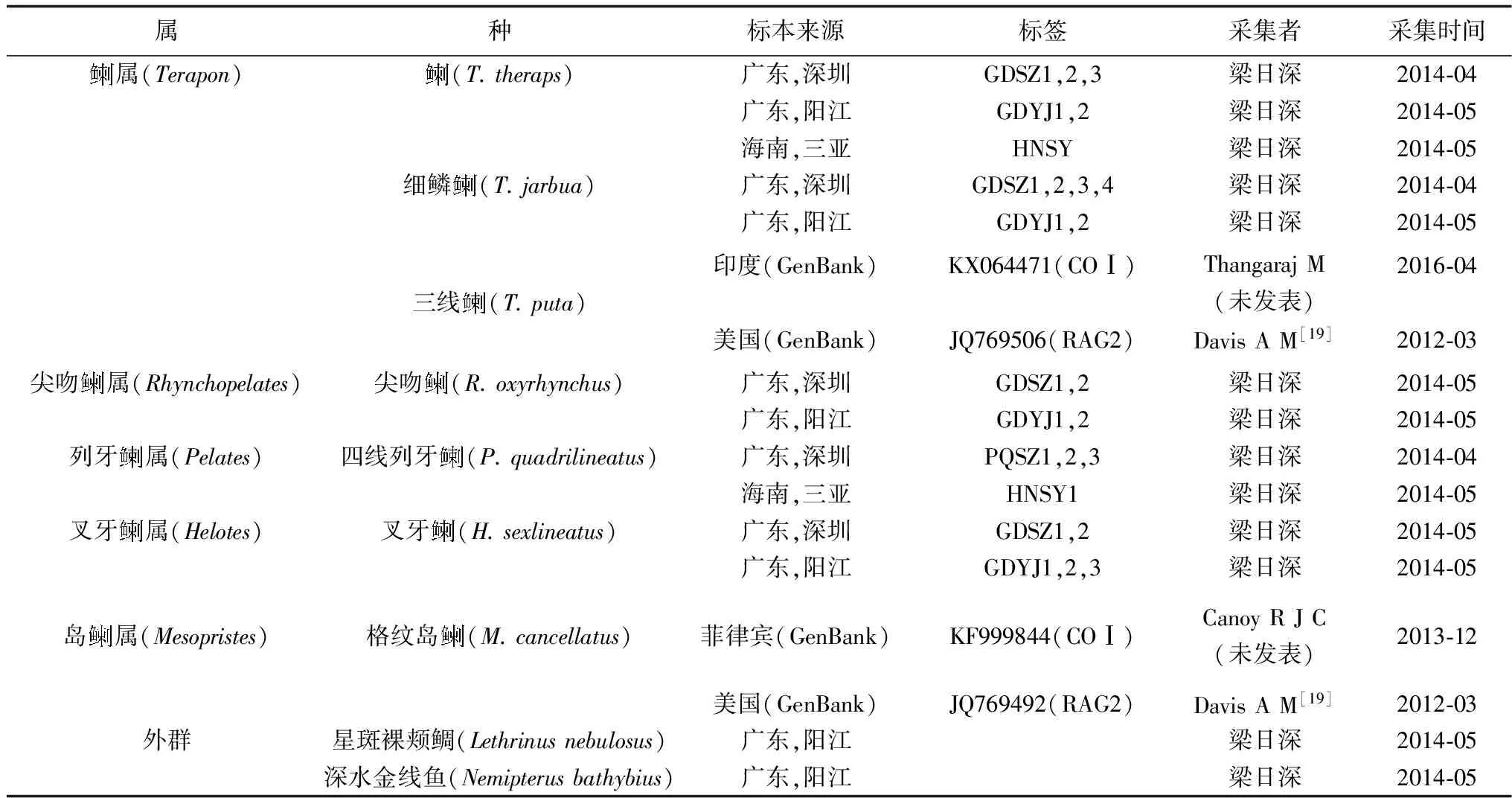
表1 試驗材料的種類和采集地
1.2 總基因組DNA的提取
基因組DNA采用動物組織DNA提取試劑盒(天根生化科技有限公司)進行提取。提取的DNA溶解于100 μL滅菌水中,1%瓊脂糖凝膠電泳檢測提取質量,-20 ℃保存?zhèn)溆谩?/p>
1.3 PCR擴增及序列測定
[16, 20],選擇用于擴增COⅠ基因及RAG2基因片段序列的引物分別為:
COⅠ-F1: 5′-TCA ACY AAT CAY AAA GAT ATY GGC AC -3′;
COⅠ-R1: 5′-ACT TCY GGG TGR CCR AAR AAT CA -3′[20];
RAG2-F1: 5′-GAG GGC CAT CTC CTT CTC CAA-3′;
RAG2-R3: 5′-GAT GGC CTT CCC TCT GTG GGT AC-3′[16]。
PCR反應體系為50 μL,其中10×buffer 5 μL,dNTPs (各2.5 mmol/L)2 μL,上下游引物各1 μL,Ex Taq酶(1 U/μL) 2 μL。PCR反應條件為:
COⅠ基因:94 ℃ 5 min,94 ℃ 30 s,60 ℃ 60 s,72 ℃ 90 s,35個循環(huán),72 ℃ 10 min;
RAG2基因:94℃ 5 min,94 ℃ 30 s,55 ℃ 60 s,72℃ 90 s,35個循環(huán),72 ℃ 10 min。
PCR產(chǎn)物經(jīng)1.5%瓊脂糖凝膠電泳分離,純化后送上海英駿生物技術有限公司進行測序。
1.4 數(shù)據(jù)分析
測序所得DNA序列利用Clustal W[21]進行比對,去除冗余序列。利用 Mega 5.0[22]計算序列的堿基組成及變異情況,基于Kimura′s 2-parameter模型計算各物種的遺傳距離。利用最大似然法分別基于COⅠ、RAG2及COⅠ+RAG2整合序列,以鱸形目裸頰鯛科的星斑裸頰鯛及金線魚科的深水金線魚作為外類群進行分子系統(tǒng)進化樹的構建,進化樹采用重復抽樣分析1000次的方法檢驗各分支的置信度。
2 結果與分析
2.1 序列分析

RAG2基因部分序列比對分析得到一致序列為729 bp,編碼243個氨基酸。堿基A、T、G、C平均含量分別為23.9%、28.2%、21.6%和26.3%,4種堿基含量相差不大,分布平均。其中A+T的含量(52.1%)高于G+C含量(47.9%)。密碼子第1、2位,4種堿基含量均相差不大,而密碼子第3位,C-3含量最高,為36.1%,A-3含量最低,為16.8%,表現(xiàn)出反A偏倚。序列保守位點613個,約占84.4%;變異位點114個,約占15.7%;簡約性信息位點85個,約11.7%。由此可知,與CO Ⅰ基因相比,RAG2基因相對保守,物種間序列差異較小。
COⅠ基因與RAG2基因序列中轉換與顛換之比值分別為2.61與3.12 (Kimura 2-parameter模型),兩個基因序列轉換明顯大于顛換,顯示其位點沒有突變飽和,可將兩基因序列所有轉換與顛換信息應用于系統(tǒng)發(fā)育樹的構建。同時,COⅠ及RAG2兩基因同質性檢驗分析得出P=1.0,說明兩基因之間不存在異質性,可將兩序列合并進行系統(tǒng)進化分析。
2.2 種間及種內(nèi)的遺傳距離

2.3 科魚類分類及分子系統(tǒng)樹


表2 基于 Kimura-2-Parameter模型計算7種科魚類COⅠ基因種間遺傳距離

表3 基于 Kimura-2-Parameter模型計算7種科魚類RAG2基因種間遺傳距離

圖2 7種科魚類基于COⅠ序列利用最大似然法構建的分子系統(tǒng)進化樹

圖3 7種科魚類基于RAG2序列利用最大似然法構建的分子系統(tǒng)進化樹
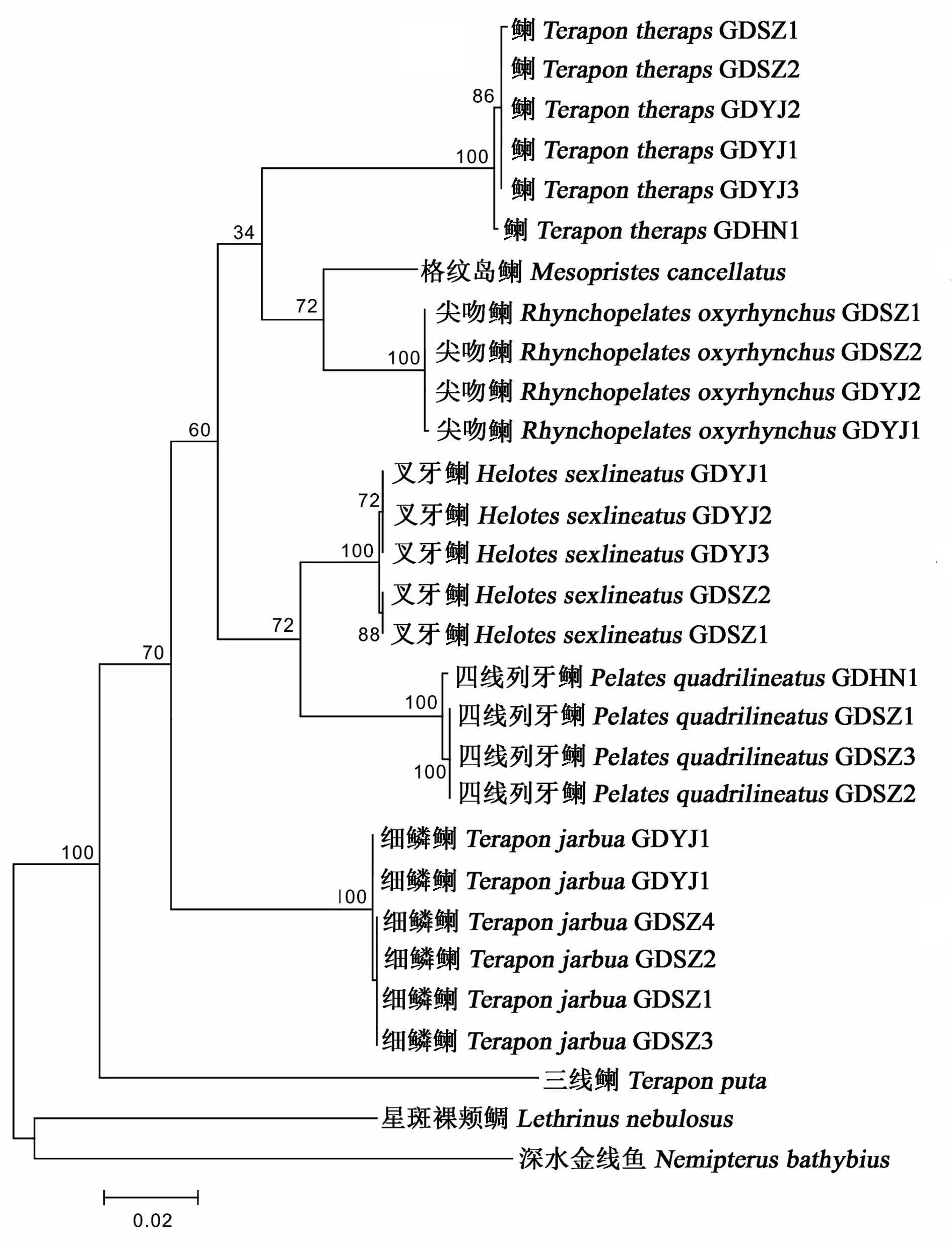
圖4 7種科魚類基于COⅠ與RAG2合并序列利用最大似然法構建的分子系統(tǒng)進化樹
3 討 論



參考文獻:
[1] 成慶泰,鄭葆珊.中國魚類系統(tǒng)檢索[M].北京:科學出版社,1987:339-341.
[2] 沈世杰.臺灣魚類志[M].臺北:臺灣大學動物學系出版,1993:360-363.
[3] 孟慶聞,蘇錦祥,繆學祖.魚類分類學[M].北京:中國農(nóng)業(yè)出版社,1995, 49(2);636-640.
[4] 劉瑞玉.中國海洋生物名錄[M].北京:科學出版社,2008:886-1066.
[5] Wu H L, Shao K T, Lai C F. Latin-Chinese dictionary of fishes names[M]. Taiwan:The Sueichan Press, 1999:1028.
[6] Nelson J S. Fishes of the world[M]. New York:John Wiley & Sons Inc, 2006:360-369.
[7] Fischer W, Whitehead P J P. FAO Species Identification Guide for Fishery Purposes. Eastern Indian Ocean(fishing area 57) and Western Central Pacific (fishing area 71). Volume 4[M]. Rome:Food and Agriculture Organization of the United Nations, 1974:1-12.
[8] Vari R P. The Terapon perches (Percoidei, Teraponidae), a cladistic analysis and taxonomic revision[J]. Bulletin of the American Museum of Natural History, 1978,159(5):175-340.
[9] Bohlen J,Slechtova V, Tan H H, et al. Phylogeny of the Southeast Asian freshwater fish genusPangio(Cypriniformes; Cobitidae)[J]. Molecular Phylogenetics and Evolution, 2011, 61(3):854-865.
[10] Eytan R I, Hastings P A, Holland B R. Reconciling molecules and morphology:molecular systematics and biogeography of Neotropical blennies (Acanthemblemaria)[J]. Molecular Phylogenetics and Evolution, 2012, 62(1):159-173.
[11] Bloom D D, Weir J T, Piller K R, et al. Do freshwater fishes diversify faster than marine fishes A test using state-dependent diversification analyses and molecular phylogenetics of new world silversides (atherinopsidae) [J]. Evolution, 2013, 67(7):2040-2057.
[12] Laakkonen H M, Strelkov P, Lajus D L, et al. Introgressive hybridization between the Atlantic and Pacific herrings (ClupeaharengusandC.pallasii) in the north of Europe[J]. Marine Biology, 2015, 162(1):39-54.
[13] Icolas H, Robert H, Louis B. Identification Canadian freshwater fishes through DNA barcodes[J]. PloS One, 2008, 3(6):1-8.
[14] Van G D,Mcblane J F, Ramsden D A, et al. Initiation of V( D) J recombination in a cell-free system[J]. Cell, 1995, 81(6):925-934.
[15] Market E,Papavasiliou F N. V (D) J recombination and the evolution of the adaptive immune system[J].PloS Biology, 2003,1(1):16.
[16] Rocha L A, Lindeman K C, Rocha C R, et al. Historical biogeography and speciation in the reef fish genusHaemulon(Teleostei:Haemulidae)[J]. Molecular Phylogenetics and Evolution, 2008, 48(3):918-928.
[17] Cramer C A,Bonatto S L, Reis R E. Molecular phylogeny of the Neoplecostominae and Hypoptopomatinae (Siluriformes:Loricariidae) using multiple genes[J]. Molecular Phylogenetics and Evolution. 2011, 59 (1):43-52.
[18] Martinez-Takeshita N, Purcell C M, Chabot C L, et al. A tale of three tails:cryptic speciation in a globally distributed marine fish of the genusSeriola[J]. Copeia, 2015, 103(2):357-368.
[19] Davis A M,Unmack P J, Pusey B J, et al. Marine-freshwater transitions are associated with the evolution of dietary diversification in terapontid grunters (Teleostei:Terapontidae) [J].Journal of Evolutionary Biology, 2012, 25(6):1163-1179.
[20] Ward R D, Zemlak T S, Innes B H, et al. DNA barcoding Australia′s fish species [J]. Philosophical Transactions of the Royal Society B,2005,360(1462):1847-1857.
[21] Thompson J D, Higgins D G, Gibson T J. Clustal W:improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position specific gap penalties and weight matrix choice[J].Nucleic Acids Research,1994,22(22):4673-4680.
[22] Tamura K, Stecher G, Peterson D, et al. Mega 6:Molecular Evolutionary Genetics Analysis version 6.0[J]. Molecular Biology and Evolution, 2013, 30(12):2725-2729.
[23] Hebert P D N, Cywinska A, Ball S L, et al. Biological identifications through DNA barcodes[J]. Proceedings of the Royal Society of London, Series B, 2003, 270(1512):313-321.
[24] 彭居俐,王緒禎,王丁,等.基于線粒體COⅠ基因序列的DNA條形碼在鯉科鲌屬魚類物種鑒定中的應用[J].水生生物學報,2009,33(2):271-276.
[25] 毛云濤,甘小妮,王緒禎.基于線粒體 COⅠ基因的沙鰍亞科魚類 DNA 條形碼及其分子系統(tǒng)發(fā)育研究[J].水生生物學報,2014,38(4):737-744.
[26] 劉璐,孫典榮,李純厚.DNA條形碼技術在鯔科魚類鑒定中的應用[J].中國海洋大學學報:自然科學版,2016,46(11):178-186.
[27] 柳淑芳,陳亮亮,戴芳群,等.基于線粒體 CO1 基因的 DNA 條形碼在石首魚科(Sciaenidae)魚類系統(tǒng)分類中的應用[J].海洋與湖沼,2016,41(2):223-232.
[28] Lee S C, Tsai M P. Molecular systematics of the thornfishes generaTeraponandPelates(Perciformes:Teraponidae) with reference to the new genusPseudoterapon[J].Zoological Studies,1999,38(3):279-286.
[30] Yearsley G K, Last P R, Hoese D F. Standard names of Australian fishes[M]. Hobart:CSIRO Marine and Atmospheric Research, 2006.
MolecularPhylogeneticAnalysisofFishesinTeraponidaefromChinaSeaBasedonMitochondrialCOⅠandNuclearRAG2GeneSequences
SU Guomao1, LIN Congzuo1, LIN Zhenwu1, LI Xingyou1, LIANG Rishen1, HUANG Yunyin2
( 1.College of Animal Technology,Zhongkai University of Agriculture and Engineering, Guangzhou 510225, China; 2.Hubei Vocational College of Bio-Technology, Wuhan 430070, China )
The 651 bp of mitochondrial DNA COⅠ sequences and 726 bp of nuclear DNA RAG2 sequences from 25 individuals of 5 Teraponidae fish collected from China Sea were determined in this study. No insertion and deletion existed in the two gene fragments. Base composition and genetic distances among and within species were calculated using Mega 5.0 and the molecular phylogenetic trees were constructed using maximum likelihood methods. The results showed that in the molecular phylogenetic tree,Teraponputawas first separated and rooted at the base of the tree, the next was speciesT.jarbua, which also formed an independent branch and located outside the other Teraponidae clusters. ThreeTeraponspecies did not formed an monophyletic group, the lastTeraponspeciesT.therapswas clustered together withRhynchopelatesoxyrhynchusandMesopristescancellatus;HelotessexlineatusandPelatesquadrilineatuswere clustered tightly as sister species, indicating that there were close phylogenetic relationships between the two species. These phylogenetic results might in accordance with the view of Lee and Tsai′s report thatT.therapsshould be removed from genusTeraponandHeloteswas combined withPelates.
Phylogeny; COⅠ gene; RAG2 gene; Teraponidae
2016-12-20;
2017-02-27.
廣東省自然科學基金資助項目(2016A030310236);廣東省教育廳青年創(chuàng)新人才類項目(2014KQNCX16);廣東省大學生創(chuàng)新創(chuàng)業(yè)訓練計劃項目(201611347067).
蘇國茂(1992-),男,碩士研究生;研究方向:水產(chǎn)動物種質資源.E-mail:1091109070@qq.com.通訊作者:黃運銀(1972-),男,工程師,碩士;研究方向:水產(chǎn)養(yǎng)殖.E-mail:379830153@qq.com.
S917
A
1003-1111(2017)05-0652-06

