基于強化學習的離散層級螢火蟲算法檢測蛋白質復合物
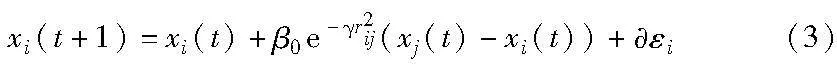







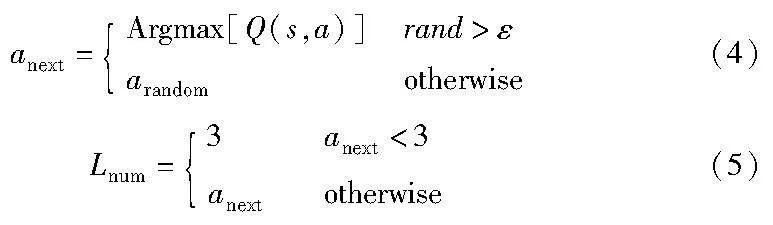



摘 要:蛋白質復合物的檢測有助于從分子水平上理解生命的活動過程。針對群智能算法檢測蛋白質復合物時假陽/陰性率高、準確率低、種群多樣性下降等問題,提出了基于強化學習的離散層級螢火蟲算法檢測蛋白質復合物(reinforcement learning-based discrete level firefly algorithm for detecting protein complexes,RLDLFA-DPC)。引入強化學習思想提出一種自適應層級劃分策略,動態調整層級結構,能有效解決迭代后期種群多樣性下降的問題。在層級學習策略中個體向兩個優秀層級學習,避免算法陷入局部最優。為了提高蛋白質復合物檢測的精度,結合個體環境信息提出自適應搜索半徑的局部搜索策略。最后,在酵母蛋白質的4個數據集上,與8種經典的蛋白質復合物檢測方法進行對比,驗證了該方法的有效性。
關鍵詞:蛋白質復合物; 螢火蟲算法; 強化學習; 層級學習策略; 局部搜索策略
中圖分類號:TP301 文獻標志碼:A 文章編號:1001-3695(2024)07-008-1977-06
doi:10.19734/j.issn.1001-3695.2023.11.0549
Reinforcement learning-based discrete level firefly algorithm fordetecting protein complexes
Abstract:Protein complexes play a crucial role in understanding life’s molecular activity process. Aiming at the problems of high false-positive/negative rate, low accuracy, and decrease in population diversity when detecting protein complexes by swarm intelligence algorithms, this paper proposed the RLDLFA-DPC. It introduced the idea of reinforcement learning to offer an adaptive level partition strategy that dynamically adjusted the level structure, solving the issue of declining population diversity in the late iteration. The algorithm also incorporated a level learning strategy where individuals learn from two excellent levels to avoid falling into a local optimum. Additionally, it utilized a local search strategy with an adaptive search radius in combination with individual and environmental information to improve the accuracy of protein complex detection. Finally, the effectiveness of the algorithm was verified by comparing it with eight classical protein complex detection methods on four datasets of saccharomyces cerevisiae proteins.
Key words:protein complex; firefly algorithm; reinforcement learning; level learning strategy; local search strategy
0 引言
蛋白質復合物是由多個蛋白質相互作用形成的大分子結構,參與了細胞的各種生物學過程,如信號傳導、代謝調控和基因表達等,在細胞內發揮著重要作用[1]。因此,準確地檢測蛋白質復合物對理解細胞內的生物學過程以及相關疾病的研究至關重要[2]。在過去的幾十年里,研究者們提出了各種各樣的計算方法檢測蛋白質復合物[3~5],然而,受蛋白質相互作用數據復雜性和噪聲的影響,它們在復雜生物系統中的應用受到了限制。
為了克服這些挑戰,研究人員利用群智能優化算法的高度自適應性和良好的優化能力,解決蛋白質復合物檢測問題。2019年,Lei等人[6]提出基于飛蛾撲火優化算法的蛋白質復合物檢測,利用逐層思想找到蛋白質復合物的核心作為火焰,讓飛蛾在火焰周圍螺旋飛行,形成復合物。同年,基于花授粉機制的蛋白質復合物檢測算法[7]被提出,通過模擬尋找最佳授粉植物花粉的過程,利用改進花授粉算法將外圍蛋白質附著在相應的核心上,生成蛋白質復合物。2022年,Wang等人[8]提出了通過自適應和聲搜索算法檢測具有多重屬性的蛋白質復合物檢測方法,利用馬爾可夫聚類算法挖掘蛋白質復合物的核心,設計蛋白質復合物形成策略來檢測附件蛋白質,并開發了一種自適應和聲搜索算法來優化算法的參數。
在眾多的群智能優化算法中,螢火蟲算法結構簡單、易于實現,自提出以來得到國內外學者的廣泛關注[9]。2016年,Lei等人[10]提出了一種新的基于螢火蟲算法的馬爾可夫聚類方法,用于從蛋白質相互作用網絡中檢測蛋白質復合物。2018年,Jenghara等人[11]將動態蛋白質相互作用網絡構造問題轉換為優化問題,通過標準復合物和基因共表達的組合定義螢火蟲算法中的吸引力函數,從而實現了蛋白質相互作用網絡的構造。同年,Zhang等人[12]提出了一種基于螢火蟲算法的蛋白質復合物挖掘新方法,定義了一種新的目標函數來尋找高內聚、低耦合的簇,最后對比了不同的蛋白質聚類方法。
雖然螢火蟲算法在蛋白質復合物的檢測過程中已經取得了重大進展,但是在這些檢測方法中,算法自身仍然存在缺陷,在檢測精度方面還有一定的提升空間。因此,提出了一種基于強化學習的離散層級螢火蟲算法,旨在解決螢火蟲算法多樣性不足,易陷入局部最優、收斂性能不高,蛋白質復合物檢測準確性低的問題。本文從以下三個方面進行改進:a)強化學習思想控制層級數,允許將種群劃分為多個層級,強化學習算法能夠根據問題的復雜性和種群的性能動態地選擇最佳的層級數,通過獎勵和懲罰機制引導個體的行為,以便更好地探索搜索空間;b)層級學習策略通過向兩個更優秀的層級移動,實現了跨層級的交流,增加了搜索空間的多樣性,避免算法過早陷入局部最優解;c)在層級內引入局部搜索策略,以便個體在同一層級內能夠更好地合作和學習,有助于改善算法的性能、加速收斂速度、提高算法的魯棒性。
1 基礎知識介紹
1.1 螢火蟲算法
螢火蟲算法(firefly algorithm,FA)是一種模擬自然界中螢火蟲發光機制的隨機優化算法[13]。為了構建FA的數學模型,使用了以下三個理想化準則:a)算法中的所有螢火蟲不區分性別;b)螢火蟲之間的吸引力和亮度成正比;c)螢火蟲的亮度與目標函數成正比。
定義1 吸引力。螢火蟲j對i的吸引力定義為
其中:β0為最大吸引力,即在光源處(r=0)螢火蟲的吸引力;γ為光吸收系數;rij為螢火蟲i到j的笛卡爾距離:
定義2 位置更新。由于螢火蟲i被j吸引,螢火蟲i向其移動并更新自己的位置,更新公式為
其中:t是算法的迭代次數;為隨機項系數;εi是由高斯分布、均勻分布得到的隨機數。
1.2 強化學習
強化學習(reinforcement learning,RL)是一種強大的機器學習方法[14,15],其核心思想在于智能體(agent)通過與環境(environment)的互動,逐漸學會在各種情境下作出決策,以最大化累積的獎勵達成特定目標。不同于傳統的監督學習,強化學習無須依賴事先標記好的訓練數據,而是通過實驗不同的行為路徑來探索并學習最優策略。這使得強化學習在許多實際問題中具有很高的適用性,尤其是在涉及復雜決策和不確定性的問題。由于強化學習可以通過學習策略實現自適應調整,研究者們將強化學習與進化算法(evolutionary algorithm,EA)結合起來,使用RL處理棘手的策略選擇或參數設置問題[16~18]。盡管EA與RL的結合在優化問題中已經取得了顯著的成果,但在FA的應用中仍然有待深入研究。螢火蟲算法通常用于解決復雜的連續優化問題,目標是尋找參數或變量的最優組合。強化學習的一個優勢是可以處理離散決策問題,這些問題通常涉及到更復雜的狀態空間和動作空間。將強化學習與螢火蟲算法結合,可以擴展其適用范圍,使其更適合解決不同類型的問題。
2 基于強化學習的離散層級螢火蟲算法
蛋白質復合物檢測是生物信息學領域的一個重要問題,旨在檢測生物系統中相互作用的蛋白質復合物。通常情況下,每個蛋白質由多個特征(如結構特征、功能特征等)表示,這樣會導致計算復雜度大大增加,容易出現維數災難問題。隨著維度的增加,搜索空間的大小以指數方式擴展和復雜化,巨大的搜索空間要求較高的搜索效率,使得標準螢火蟲算法很難在合理的時間內找到最優解。因此,為了更有效地檢測蛋白質復合物,提出了基于強化的離散層級螢火蟲算法。
2.1 編碼方式
蛋白質復合物是由n個蛋白質構成的集合,每個蛋白質都有唯一的索引,編碼方式使用n維的二進制向量來表示每個螢火蟲的位置。對于每個螢火蟲,向量的第i個元素為1,則表示第i個蛋白質是該復合物的一部分;否則,該元素為0。假設有五個蛋白質節點(P1,P2,P3,P4,P5),一個可能的編碼是“11001”,表示P1、P2和P5在復合物中,而P3和P4不在復合物中。
2.2 層級劃分
如果螢火蟲都被亮度最高的螢火蟲吸引,會導致種群的多樣性不足。為了解決上述問題,按照適應度值將螢火蟲種群分層。通常認為,適應度越好的個體,越有可能開發有希望的區域;較差適應度的個體具有更好的探索能力。這樣的分層結構可以使FA更好地平衡搜索的全局性和局部性,從而提高搜索效率和解的質量,增加種群的多樣性。
假設種群規模為N,按照適應度值將種群平均劃分為L個層級。其中,L1是最高的層級。每個層級的個體數為M=N/L,最后一個層級的個體數為N/L+N%L。
2.3 強化學習思想控制層級數
為了保持種群的多樣性,RLDLFA-DPC采用基于層級的種群結構。強化學習算法能夠根據問題的復雜性和種群的性能動態地選擇最佳的層級數,通過獎勵和懲罰機制引導個體的行為,以便更好地探索搜索空間。因此,RLDLFA-DPC采用強化學習思想控制種群的層級數。
每一次迭代結束后,層級數都會被更新。設置獎勵表和Q-table,定義狀態和動作為層級數。獎勵表初始值為0,Q-table初始值設置為隨機值。獎勵表通過適應度值更新,Q-table通過獎懲機制更新。在優化期間,當隨機數大于探索因子,即rand>ε時,根據Q-table中的Q值選擇具有最高預期獎勵的動作,以利用已有的知識;反之,以一定的探索概率隨機選擇動作,以探索新的層級數。具體公式如下:
其中:arandom是種群在當前狀態中可以采取的任何動作;Argmax[·]表示具有最高Q值的動作;Lnum是層級數,為了保持學習策略的有效性,劃分的層級至少為3。
在受到獎勵后,為了保持動作選擇策略的有效性,強化學習策略將通過以下規則更新其Q值:
Q(s,a)=Q(s,a)+α·(r+γ·maxQ(snext,anext)-Q(s,a))(6)
其中:Q(s,a)為當前狀態s采取動作a后的Q值;snext、anext為下一狀態和下一動作,r為采取當前動作后的獎勵值,如果新的適應度值大于當前的適應度值,r=1,反之,r=-1;γ為折現因子;α為學習率。圖1是獎勵表和Q-table更新示例圖,其中,行表示狀態,列表示動作。當前種群劃分的層級數為4,即L1、L2、L3、L4。α=0.1,γ=0.9。假設在狀態L2執行動作L2,r=-1,Q(L2,L2)=0.4+0.1×(-1+0.9×max(-0.1,0.4,-0.2,0.3)-0.4)=0.296。
算法1 強化學習思想控制層級數
2.4 層級學習策略
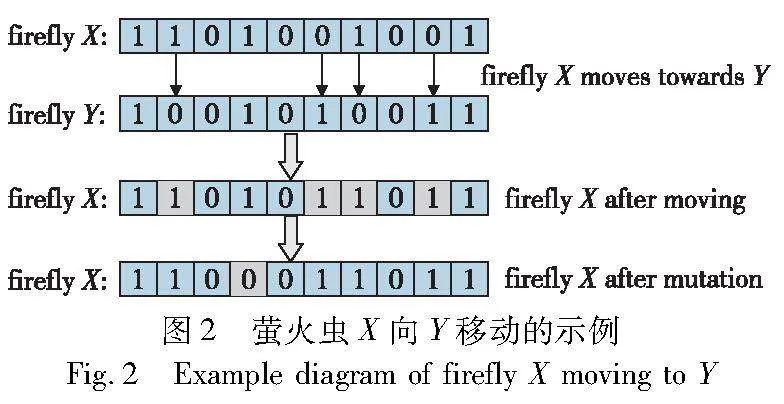
在FA中,個體之間的移動是通過光強度吸引的方式實現的,即個體向光強度較高的個體移動,以實現全局搜索和優化。然而,如果整個種群中的個體都朝向光強度最高的個體移動,可能會導致算法陷入局部最優解。因此,引入層級學習策略,通過從隨機選擇的兩個優秀層級中隨機選擇兩個個體進行學習,引入了隨機性和多樣性,有助于防止所有個體都朝向相同的方向移動,增加了算法的多樣性,從而避免算法過早陷入局部最優解。
在RLDLFA-DPC中,螢火蟲的移動由兩個動作決定:吸引力β和參數α。螢火蟲Xi向螢火蟲Xj和Xk移動的定義如式(7)所示。
2.5 局部搜索策略
層級學習策略是一種通過在不同層級的個體之間進行學習來提高算法性能的方法。雖然這種策略可以促進跨層級的信息交流,但忽略了層級內部的學習,從而限制了層級內部的搜索能力。為了克服上述缺陷,考慮在層級內引入局部搜索策略,以便個體在同一層級內能夠更好地合作和學習。層級內引入局部搜索策略可以提供更多的搜索焦點,有助于改善算法的性能、加速收斂速度、提高算法的魯棒性。
對于每個層級內的個體,引入局部搜索策略,以便更好地探索局部解空間。首先,為每個個體定義一個自適應的局部搜索半徑,該半徑決定了個體在局部搜索時應該關注的鄰域范圍,可以根據個體和環境的信息進行動態調整。以下是局部搜索半徑的定義公式:
Ri=η·max(f)fi·‖Xg-Xi‖(11)
其中:Ri是個體i的局部搜索半徑;fi是個體i的適應度值;max(f)是當前層級中適應度值的最大值;Xg是個體i當前位置Xi周圍最優解的位置;η是一個控制參數,用于調整局部搜索半徑的大小。
其中:Generation是當前迭代的代數;MaxGenerations是算法允許的最大代數。初始時,η較大,允許更廣泛的搜索,隨著代數的增加,η逐漸減小,從而限制局部搜索半徑的范圍,促使算法更加聚焦于局部搜索。這種動態調整η的方式可以使算法在早期探索解空間的廣度,在后期更專注于深度搜索。
螢火蟲個體i的位置為Xi,在局部搜索時,更新個體i的位置,使其朝向隨機選擇的個體j移動。位置更新公式如式(13)所示。
Xi=g(f(Xi,Xj,β),α)(13)
2.6 算法整體流程
綜合以上對RLDLFA-DPC各階段的討論,下面將詳細闡述整體運行流程。
a)初始化參數:種群規模為N,按照適應度值將種群平均劃分為層級數為L,當前迭代次數t,最大迭代次數Tmax。
b)創建初始種群:根據初始化參數,隨機生成初始種群,每個個體代表一個可能的蛋白質復合物結構。
c)計算適應度值:對于每個個體,利用F-measure評估函數計算其適應度值。
d)進行層級劃分:使用強化學習思想動態地控制層級數,通過獎勵和懲罰機制引導個體的行,選擇最佳的層級數,通過式(5)更新層級數,通過式(6)更新Q-table。
e)層級學習策略:根據個體的適應度,向兩個更優秀的層級學習。
f)層級內的局部搜索:在每個層級內,引入局部搜索策略,使用鄰域搜索算法對每個個體進行局部優化。
g)更新螢火蟲位置:根據層級學習策略位置更新式(7)和局部搜索策略位置更新式(13),更新個體的位置。
h)重復步驟c)~g),直到達到最大迭代次數。
i)輸出最優解:在算法停止后,根據適應度值選擇最優的蛋白質復合物解作為輸出。
3 仿真實驗及結果分析
3.1 復雜度分析
a)層級劃分:這一步涉及將種群劃分為L個層級,所以時間復雜度是O(L)。
b)獎勵表和Q-table的更新:Q-Learning涉及獎勵表和Q-table的更新,涉及狀態和動作的組合,所以其更新的時間復雜度為O(L2)。
c)層級學習策略:在隨機選擇的層級中,隨機選擇兩個個體進行學習,時間復雜度取決于隨機選擇的次數,通常可以看作是O(1)。
d)局部搜索策略:計算每個個體的局部搜索半徑涉及到個體的位置信息和環境信息,計算位置信息的復雜度為O(1),計算環境信息的復雜度可以表示為 O(N)。
綜合以上各步驟,整個算法的時間復雜度主要由Q-Learning更新部分和局部搜索策略部分決定,所以總體時間復雜度可以近似表示為O(L2)+O(N)。
3.2 參數設置
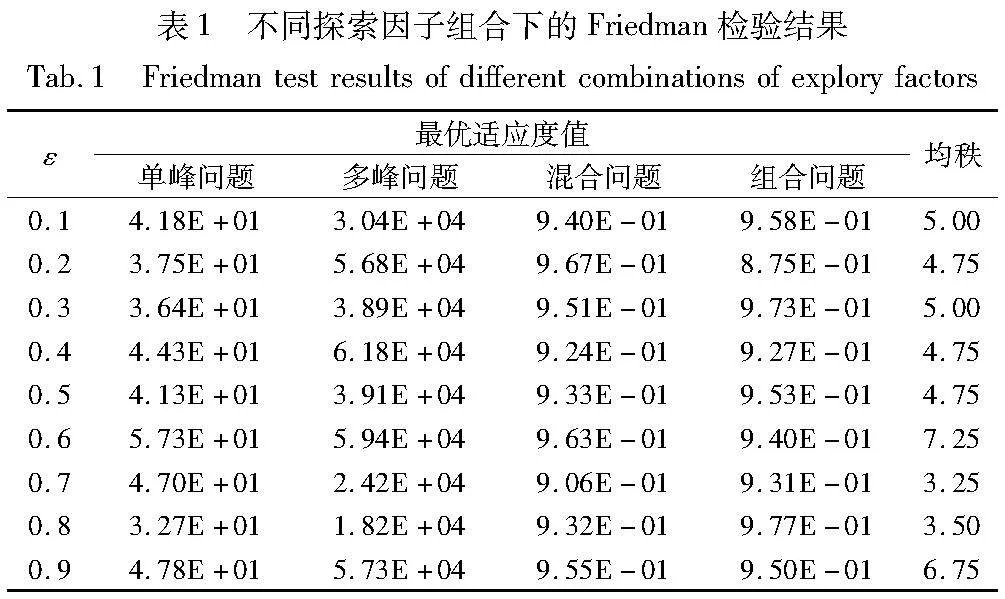
參數設置對算法性能起著至關重要的作用。根據RLDLFA-DPC的描述,算法中需要確定的關鍵參數為探索因子ε。在優化期間,當rand>ε時,根據Q-table中的Q值選擇具有最高預期獎勵的動作;反之,以一定的探索概率隨機選擇動作,以探索新的層級數。本研究使用Friedman檢驗來確定最優的探索因子,設置ε的取值為{0.1,0.2,0.3,0.4,0.5,0.6,0.7,0.8,0.9},選擇四種不同類型的基準函數進行測試,通過這些函數測試不同ε下RLDLFA-DPC的收斂性能。如表1所示,均秩值越小,說明算法的整體優化性能越好。從實驗結果可以看出,當ε=0.7時,函數的均秩最小,因此RLDLFA-DPC的探索因子ε設置為0.7。
3.3 策略有效性分析
RLDLFA-DPC采用層級劃分、強化學習控制層級數、層級學習策略和局部搜索策略改善FA。為了研究每種策略的效果,使用四種不同類型的測試函數來測試當使用不同策略組合時,算法的優化結果。策略組合如表2所示。為了公平起見,五種方法中的參數都設置為相同值。
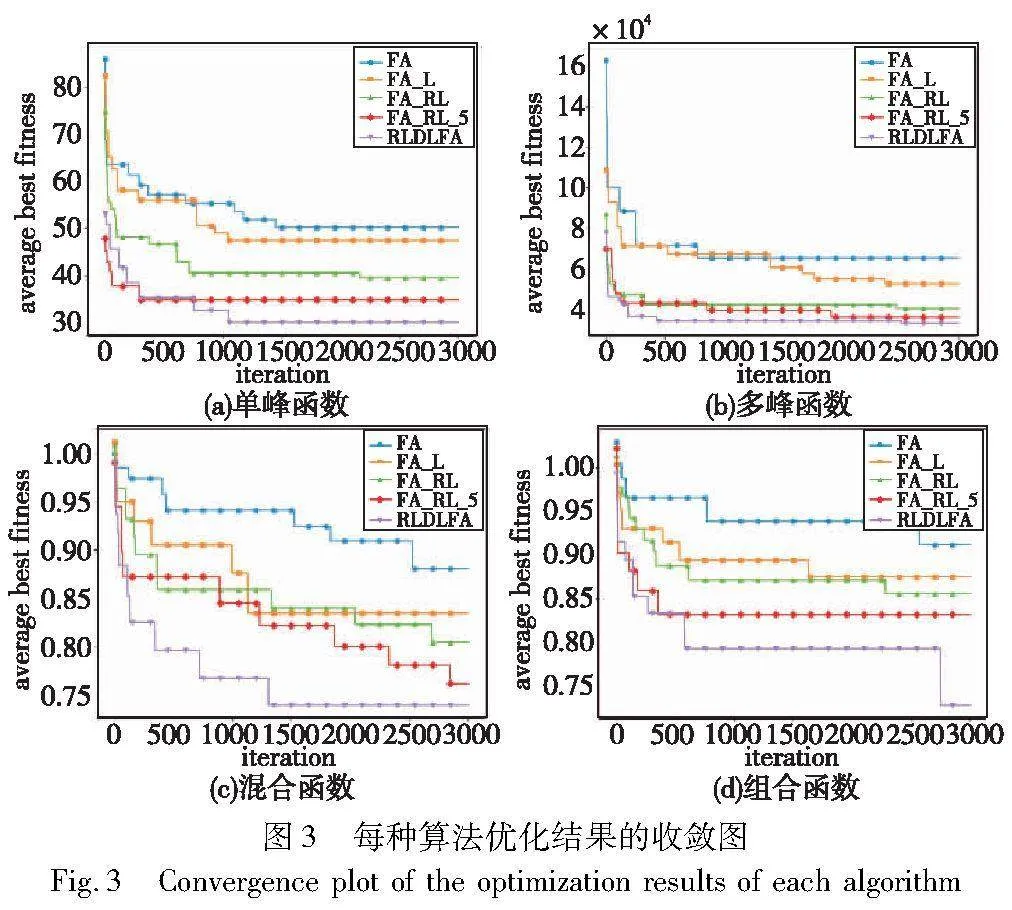
表3展示了使用不同策略組合算法的實驗對比結果。最優結果用粗體表示。從表中可以看出,在相同的參數設置下, FA、FA_L、FA_RL、FA_RL_S、RLDLFA-DPC的性能越來越好,RLDLFA-DPC在所有測試函數的性能最優, FA_L、FA_RL和FA_RL_S的性能也都優于FA。這表明采用層級劃分、強化學習思想控制層級、層級學習策略和局部搜索策略改善FA是有效的,也證明了RLDLFA-DPC方法的有效性。
為了直觀地比較每個策略組合算法的性能,表4展示了不同策略組合算法的Friedman測試結果。可以看出,測試結果與上述分析一致,RLDLFA-DPC方法的性能最好。圖3中給出了每種算法優化結果的收斂圖。從圖中可以看出,改進后的方法在收斂性上都優于FA,RLDLFA-DPC收斂性能最好。
3.4 實驗數據和評價指標
本文將改進的基于強化學習的離散層級螢火蟲算法應用到蛋白質復合物檢測的過程中,采用釀酒酵母(saccharomyces cerevisiae)的數據集DIP[19]、Gavin[20]、Krogan[21]和MIPS[22]進行測試。采用的標準數據集是CYC2008[23],該數據集包含408個蛋白質復合物。
為了評估檢測出的蛋白質復合物的性能,使用常見的三種統計評估方法,即準確率precision、查全率recall和調和平均值F-measure[24]。這些評價指標的取值都在[0.0,1.0],它們的值越高,說明檢測方法的性能越好,也從側面反映出該方法檢測蛋白質復合物的性能更優異。這三個評價指標的計算公式為
其中:TP是指算法檢測出來的蛋白質復合物和標準蛋白質復合物相匹配的個數;FP是指算法檢測出來的蛋白質復合物和標準蛋白質復合物不匹配的個數;FN是標準蛋白質復合物中沒有被檢測出的蛋白質復合物個數;F-measure是precision和recall的調和平均值。
3.5 性能對比
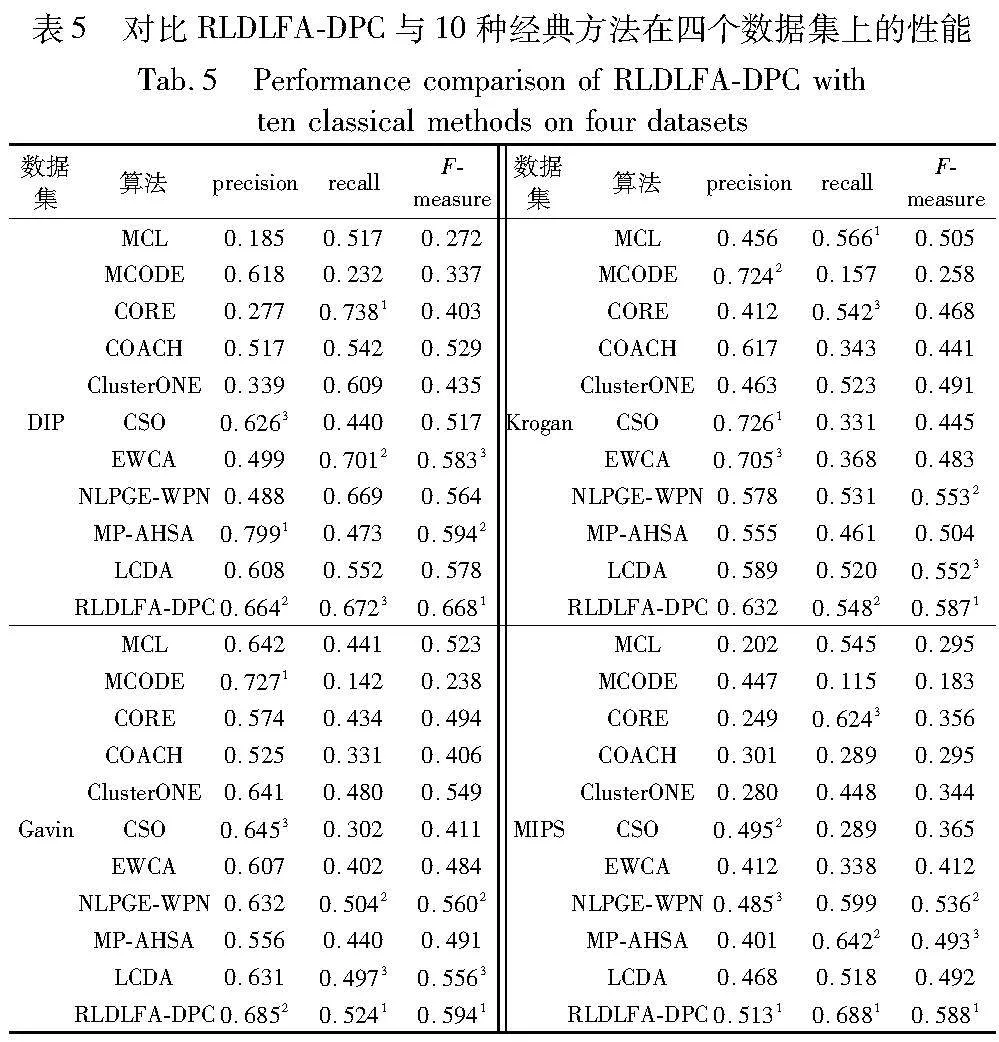
為了測試RLDLFA-DPC方法的性能,采用10種經典的蛋白質復合物檢測方法MCL[25]、MCODE[26]、ClusterONE[27]、CSO[28]、CORE[29]、COACH[30]、EWCA[31]、NLPGE-WPN[32]、MP-AHSA[8]和LCDA[33]在四個數據集上進行對比。同時為了更加清晰地對比蛋白質復合物的檢測結果,表5對比RLDLFA-DPC與10種經典方法在四個數據集上的性能。表5中,RLDLFA-DPC方法和實驗結果排名前三的上標處注明排名。實驗結果顯示,RLDLFA-DPC方法在MIPS數據集上的precision、recall和F-measure評價指標都優于其他蛋白質復合物檢測方法。在Gavin和DIP數據集上precision、recall和F-measure都處于領先地位。在Krogan數據集上,precision的值雖然略微落后于其他方法,但是F-measure優于其他方法。綜合分析,RLDLFA-DPC方法比其他蛋白質復合物檢測方法更能有效地檢測蛋白質復合物。
3.6 與已知蛋白質復合物比較
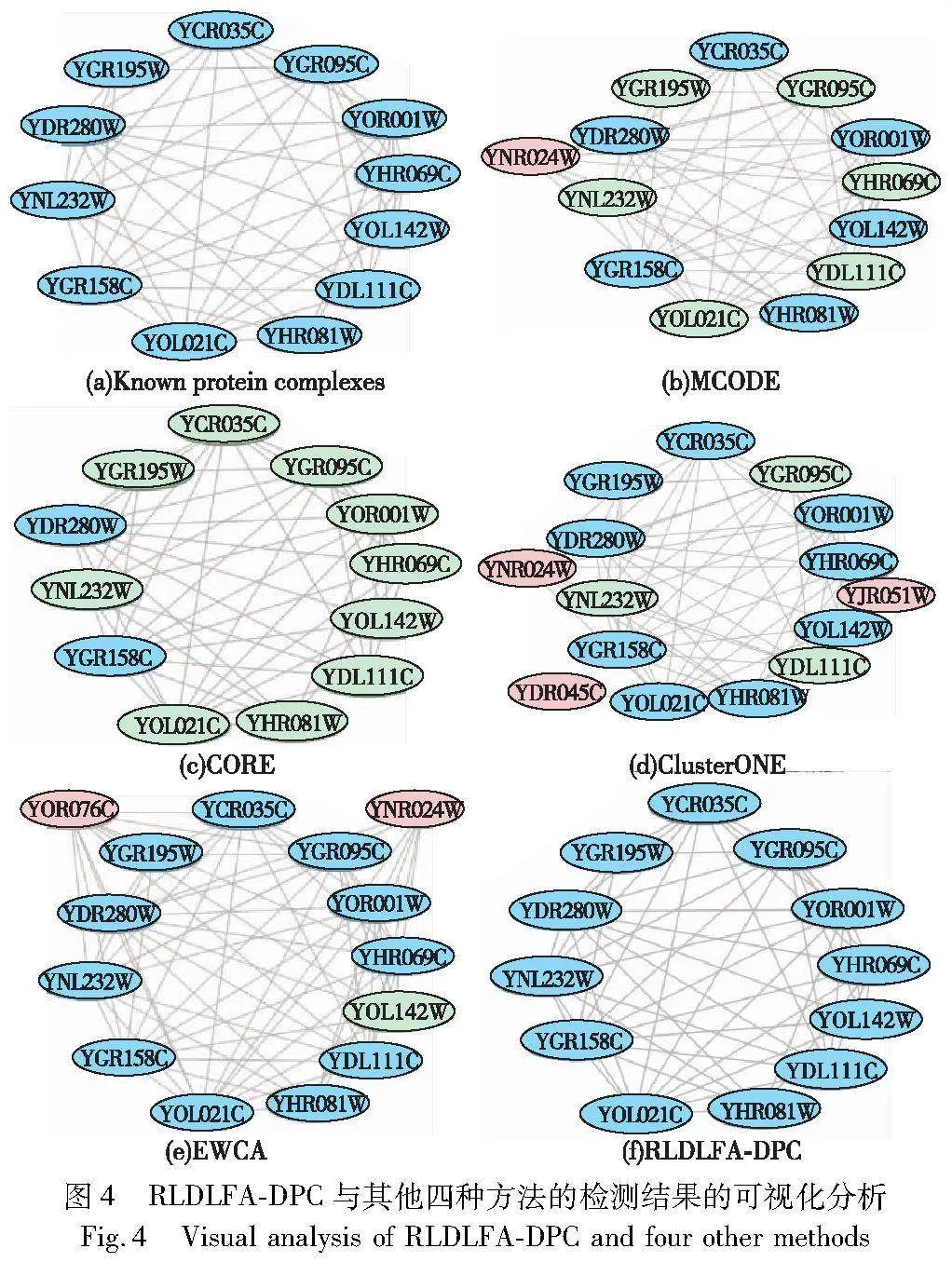
為了更好地展示算法性能的優劣,更加清楚地展示RLDLFA-DPC方法檢測結果的準確性,對比分析CYC2008標準蛋白質復合物中第265個蛋白質復合物和RLDLFA-DPC與其他四種方法在Krogan數據集上的檢測結果。該標準蛋白質復合物有YNL232W、YOL021C、YHR081W、YGR158C、YHR069C、YOL142W、YDL111C、YCR035C、YDR280W、YGR095C、YOR001W、YGR195W 12個蛋白質節點。
圖4展示了已知蛋白質復合物、RLDLFA-DPC和其他四種方法的檢測結果的可視化分析圖。藍色節點是正確檢測的蛋白質,綠色節點是未檢測出的蛋白質,粉色節點是錯誤檢測出的蛋白質。如圖4所示,CORE方法檢測效率比較低,僅正確檢測出2種標準蛋白質,MCODE和ClusterONE方法檢測效率有所提升,分別正確檢測出6種標準蛋白質和9種標準蛋白質,相較于前面幾種方法,EWCA方法的檢測結果更佳,正確檢測出了11種標準蛋白質,但也錯誤地檢測出了其他2種蛋白質。在RLDLFA-DPC方法的檢測結果中,12種標準蛋白質全部被檢測出來,并且沒有錯誤地檢測出其他的蛋白質。因此,RLDLFA-DPC方法在蛋白質復合物的檢測過程中取得了最佳性能。
4 結束語
蛋白質復合物的檢測在生物醫學中具有重要的意義,RLDLFA_DPC方法能有效提高蛋白質復合物檢測的效率和精度。該方法引入強化學習思想動態調整種群層級數量,能更好地增強種群多樣性。在迭代過程中,層級學習策略促使個體向兩個優秀層級學習,實現了跨層級學習,避免算法陷入局部最優解。通過個體和環境信息設置局部搜索半徑,自適應半徑的局部搜索策略可以對局部空間進行充分探索,實現同一層級個體的交流與協作,提高蛋白質復合物檢測精度和收斂速度。實驗結果表明,RLDLFA_DPC相較于傳統方法,能夠更有效地發現復合物結構,具有更高的檢測準確性和更快的收斂性能。該方法還具有廣泛的應用價值,未來的研究將進一步探索群體智能方法在不同領域的應用潛力,也可以結合機器學習和深度學習更有效地發現復合物結構。
參考文獻:
[1]Javad Z, Abbasali E, Samaneh B, et al. Protein complex prediction: a survey[J]. Genomics, 2020,112(1): 174-183.
[2]王金雷, 丁學明, 秦琪琪, 等. 基于協同進化信息和深度學習的蛋白質功能預測[J]. 計算機應用研究, 2023,40(12): 3572-3577. (Wang Jinlei, Ding Xueming, Qin Qiqi, et al. Protein function prediction based on coevolutionary information and deep learning[J].Application Research of Computers, 2023,40(12): 3572-3577.)
[3]Chen Bo, Xie Ziwei, Qiu Jiezhong, et al. Improved the heterodimer protein complex prediction with protein language models[J/OL]. Briefings in Bioinformatics, 2023,24(4). https://doi.org/10.1093/bib/bbad221.
[4]Liu Guangming, Liu Bo, Aimin Li, et al. Identifying protein complexes with clear module structure using pairwise constraints in protein interaction networks[J]. Frontiers in Genetics, 2021, 12: 664786.
[5]Wang Jie, Jia Ying, Sangaiah A K, et al. A network clustering algorithm for protein complex detection fused with power-law distribution characteristic[J]. Electronics, 2023,12(14): 3007.
[6]Lei Xiujuan, Fang Ming, Fujita H. Moth-flame optimization-based algorithm with synthetic dynamic PPI networks for discovering protein complexes[J]. Knowledge-Based Systems, 2019,172: 76-85.
[7]Lei Xiujuan, Fang Ming, Guo Ling, et al. Protein complex detection based on flower pollination mechanism in multi-relation reconstructed dynamic protein networks[J]. BMC Bioinformatics, 2019,20(3): 131.
[8]Wang Rongquan, Wang Caixia, Ma Huimin. Detecting protein complexes with multiple properties by an adaptive harmony search algorithm[J]. BMC Bioinformatics, 2022,23(1): 414.
[9]Cheng Zhiwen, Song Haohao, Zheng Debin, et al. Hybrid firefly algorithm with a new mechanism of gender distinguishing for global optimization[J]. Expert Systems with Applications, 2023,224: 120027.
[10]Lei Xiujuan, Wang Fei, Wu Fangxiang, et al. Protein complex identification through Markov clustering with firefly algorithm on dynamic protein-protein interaction networks[J]. Information Sciences, 2016, 329: 303-316.
[11]Jenghara M M, Ebrahimpour-Komleh H, Parvin H. Dynamic protein-protein interaction networks construction using firefly algorithm[J]. Pattern Analysis and Applications, 2018, 21: 1067-1081.
[12]Zhang Yuchen, Lei Xiujuan, Tan Ying. Firefly clustering method for mining protein complexes[C]//Proc of the 8th International Confe-rence on Swarm Intelligence. Cham:Springer, 2017: 601-610.
[13]Yang Xinshe. Nature-inspired metaheuristic algorithms[M].[S.l.]:Luniver Press, 2010.
[14]Wang Ling, Pan Zixiao, Wang Jingjing. A review of reinforcement learning based intelligent optimization for manufacturing scheduling[J]. Complex System Modeling and Simulation, 2021,1(4): 257-270.
[15]Meng Xiaoding,Li Hecheng,Chen Anshan. Multi-strategy self-learning particle swarm optimization algorithm based on reinforcement learning[J]. Mathematical Biosciences and Engineering, 2023,20(5): 8498-8530.
[16]Wu Di, Wang Shuang, Liu Qingxin, et al. An improved teaching-learning-based optimization algorithm with reinforcement learning strategy for solving optimization problems[J]. Computational Intelligence and Neuroscience, 2022, 2022: article ID 1535957.
[17]Wang Feng,Wang Xujie,Wang Shilei. A reinforcement learning level-based particle swarm optimization algorithm for large-scale optimization[J]. Information Sciences, 2022, 602: 298-312.
[18]Wang Zijia, Zhan Zhihui, Yu Weijie, et al. Dynamic group learning distributed particle swarm optimization for large-scale optimization and its application in cloud workflow scheduling[J]. IEEE Trans on Cybernetics, 2019,50(6): 2715-2729.
[19]Salwínski L, Miller C S, Smith A J, et al. The database of interacting proteins: 2004 update[J]. Nucleic acids research, 2004, 32(S1): D449-D451.
[20]Gavin A C, Aloy P, Grandi P, et al. Proteome survey reveals modularity of the yeast cell machinery[J]. Nature, 2006,440(7084): 631-636.
[21]Krogan N J, Cagney G, Yu Haiyuan, et al. Global landscape of protein complexes in the yeast Saccharomyces cerevisiae[J]. Nature, 2006, 440(7084): 637-643.
[22]Güldener U, Münsterktter M, Oesterheld M,et al. MPact: the MIPS protein interaction resource on yeast[J]. Nucleic Acids Research, 2006, 34(S1): D436-D441.
[23]Pu Shuye, Wong J, Turner B, et al. Up-to-date catalogues of yeast protein complexes[J]. Nucleic Acids Research, 2009, 37(3): 825-831.
[24]Younis H, Anwar M W, Khan M U G, et al. A new sequential forward feature selection(SFFS) algorithm for mining best topological and biological features to predict protein complexes from protein-protein interaction networks(PPINs)[J]. Interdisciplinary Sciences: Computational Life Sciences, 2021,13(3): 371-388.
[25]Enright A J, Van Dongen S, Ouzounis C A. An efficient algorithm for large-scale detection of protein families[J]. Nucleic Acids Research, 2002, 30(7): 1575-1584.
[26]Bader G D, Hogue C W V. An automated method for finding molecular complexes in large protein interaction networks[J]. BMC Bioinformatics, 2003,4(1): article No.2.
[27]Wang Jianxin, Li Min, Chen Jian’er, et al. A fast hierarchical clustering algorithm for functional modules discovery in protein interaction networks[J]. IEEE/ACM Trans on Computational Biology and Bioinformatics, 2010,8(3): 607-620.
[28]Zhang Yijia, Lin Hongfei, Yang Zhihao, et al. Protein complex prediction in large ontology attributed protein-protein interaction networks[J]. IEEE/ACM Trans on Computational Biology & Bioinformatics, 2013,10(3): 729-741.
[29]Leung H C M, Xiang Qian, Yiu S M, et al. Predicting protein complexes from PPI data a core-attachment approach[J]. Journal of Computational Biology, 2009,16(2):133-144.
[30]Wu Min, Li Xiaoli, Kwoh C K, et al. A core-attachment based method to detect protein complexes in PPI networks[J]. BMC Bioinforma-tics, 2009, 10(1): article No.169.
[31]Wang Rongquan, Liu Guixia, Wang Caixia. Identifying protein complexes based on an edge weight algorithm and core-attachment structure[J]. BMC Bioinformatics, 2019,20(1): article No.471.
[32]Yu Yang, Kong Dezhou. Protein complexes detection based on node local properties and gene expression on PPI weighted networks[J]. BMC Bioinformatics, 2022,23: article No.24.
[33]Dilmaghani S, Brust M R, Ribeiro C H C, et al. From communities to protein complexes: a local community detection algorithm on PPI networks[J]. PLoS One, 2022,17(1): e0260484.

