基于葉綠體基因組解析廣西裂果薯與裂果薯的遺傳差異及親緣關系





摘 要: 蒟蒻薯屬(Tacca)植物的分類一直存在很大的爭議,其中廣西裂果薯(Schizocapsa guangxiensis)被認為應與裂果薯(Tacca plantanginea)歸并為同一物種,但也有學者根據其形態差異劃分為不同的物種。為了明確廣西裂果薯與裂果薯的遺傳差異和系統發育關系,該研究對廣西裂果薯進行DNA高通量測序,利用生物信息軟件組裝了完整的葉綠體基因組,并與已發表的裂果薯葉綠體基因組進行比較及系統發育分析。結果表明:(1)廣西裂果薯和裂果薯的葉綠體基因組大小分別為162 149 bp和160 749 bp,GC含量都是36.90%,兩者注釋后得到的基因種類和基因數目完全一致,包含89個蛋白質編碼基因、37個tRNA基因、6個rRNA基因。(2)密碼子的偏好性分析顯示,兩物種使用的密碼子頻率存在一定差異,但是都偏好以A/T(U)結尾的密碼子。(3)與裂果薯相比,廣西裂果薯的SSC邊界發生明顯的擴張現象,是導致兩者葉綠體基因組長度差異的主要因素。(4)廣西裂果薯與裂果薯在LSC和SSC區域內存在一些序列分歧,尤其是基因間隔區,可用于開發物種特異性分子標記。(5)系統發育結果顯示,廣西裂果薯與裂果薯的親緣關系較遠,盡管廣西裂果薯寄定在蒟蒻薯屬內,但是它們屬于兩個不同的物種。該研究豐富了廣西裂果薯葉綠體基因組遺傳信息,為廣西裂果薯物種分類、遺傳多樣性分析和物種保護提供了理論依據。
關鍵詞: 廣西裂果薯, 蒟蒻薯屬, 葉綠體基因組, 遺傳差異, 系統發育分析
中圖分類號: Q943; Q949
文獻標識碼: A
文章編號: 1000-3142(2024)09-1707-14
Characterization of genetic difference and phylogenetic
relationship between Schizocapsa guangxiensis and
Tacca plantaginea based on chloroplast genome
Abstract: The taxonomy of Tacca remains is controversial all the time. Schizocapsa guangxiensis is considered to be the same species as Tacca plantanginea, but some taxonomists classify them into different species based on their morphological differences. In order to clarify the genetic differences and phylogenetic relationship between Schizocapsa guangxiensis and Tacca plantanginea, this study conducted high-throughput DNA sequencing of Schizocapsa guangxiensis, assembled a complete chloroplast genome using bioinformatics software, and compared it with the published Tacca plantanginea chloroplast genome. The results were as follows: (1) The chloroplast genome size of Schizocapsa guangxiensis and Tacca plantanginea was 162 149 bp and 160 749 bp, respectively, and they had the same GC content (36.90%). Notably, the gene types and gene amount were exactly the same in these two species, including 89 protein-coding genes and 37 tRNA genes, and 6 rRNA genes. (2) Codon preference analysis showed that there were certain differences in the codon frequencies used by the two species, but they both prefered codons ending in A/T(U). (3) Compared with T. plantanginea, the SSC boundary of Schizocapsa guangxiensis had obvious expansion, which was the main factor leading to the length variant in chloroplast genome between the two species. (4) There were some sequence divergences between S. guangxiensis and Tacca plantanginea in the LSC and SSC regions, especially the intergenic region, which can be exploited as species-specific molecular marker. (5) Phylogenetic results showed that Schizocapsa guangxiensis and Tacca plantanginea had a rather distant genetic relationship. Although Schizocapsa guangxiensis was placed in Tacca, they belong to two different species. This study enriches the genetic information of the chloroplast genome of S. guangxiensis and provides a theoretical basis for species classification, genetic diversity analysis and species protection of S. guangxiensis.
Key words: Schizocapsa guangxiensis, Tacca, chloroplast genome, genetic difference, phylogenetic analysis
蒟蒻薯屬(Tacca)是薯蕷科(Dioscoreaceae)中一個較小的屬,包含約15種植物,廣泛分布在熱帶和亞熱帶地區。這些植物因含有豐富的天然活性成分,被廣泛用于治療各種疾病(賈敏如和李星煒,2005)。如裂果薯的根部含有豐富的箭根酮內酯、皂苷和黃酮等化學成分,具有抗腫瘤、清熱解毒、理氣止痛和涼血散瘀的功效(孫悅文,2013)。此外,常被用于治療跌打損傷、急性腸胃炎和瘡瘍腫毒等疾病(云南省藥材公司,1993)。此外,該屬植物的觀賞價值也很高,聚傘形的花序大而美麗,在園藝植物中廣受青睞。近年來,由于生境破壞、過度開采和氣候變化等因素,許多種類面臨著瀕危的風險(傅立國和金鑒明,1992)。因此,加強對其保護和可持續利用至關重要。然而,當前對蒟蒻薯屬的研究主要集中在顯微鑒定、活性物質分離、藥理藥效和組織培養等方面(王慧明等,2004; Liu et al., 2006; Jiang et al., 2014; 秦燕萍等,2023),關于其系統發育關系、物種遺傳多樣性和種質資源保護的相關研究仍然相對較少(Zhang et al., 2011)。值得注意的是,蒟蒻薯屬植物的分類標準一直存在爭議,物種界定的差異很大。自1881年建立裂果薯屬(Schizocapsa)以來,學者圍繞著是否將裂果薯屬歸并到蒟蒻薯屬的問題爭論不休。Drenth(1972)認為裂果薯屬不能另立一屬,因為開裂的果實特征不足以界定一個新屬。在國際植物學名錄中,裂果薯已被歸類到蒟蒻薯屬下,并且將廣西裂果薯(S. guangxiensis)與裂果薯(T. plantanginea)歸并為同一物種。然而,也有學者認為廣西裂果薯和裂果薯是兩個不同的物種:葉薄紙質 vs. 葉紙質;外輪花被片寬圓形,內輪花被片比外輪花被裂片小l/2左右 vs. 外輪花被片披針形,內輪花被片卵圓形,內輪花被片比外輪花被裂片稍短而寬;蒴果3瓣裂至中部 vs. 蒴果3瓣裂至基部(Zhu, 2000)。此外,根據兩屬植物的染色體數目和葉表皮氣孔類型的差異,認為裂果薯屬不能歸并到蒟蒻薯屬,而應獨立出來為宜(凌萍萍和丁志遵, 1982; 凌萍萍,1985; 中國科學院生物多樣性委員會,2023)。隨著DNA條形碼技術的發展和日趨成熟,Zhang等(2011)使用ITS、atpA、rbcL、 trnH-psbA和trnL-F條形碼重構蒟蒻薯屬物種的系統發育樹來推斷屬內物種的系統發育關系,然而該研究缺乏廣西裂果薯樣本,未涉及解釋屬和種的歸并問題。
葉綠體是植物體內具有獨立遺傳物質的多功能細胞器,其基因組結構高度保守,為典型的雙鏈環狀四分體結構,即由一個大單拷貝區(large single copy,LSC)、一個小單拷貝區(small single copy,SSC)和兩個反向重復序列(inverted repeats,IRa和IRb)組成(Dobrogojski et al., 2020)。不同植物葉綠體基因組之間的結構差異主要體現在IR區的長度和方向上的變化,這是導致基因組大小變化和單拷貝基因轉化為重復基因的主要因素(Mehmood et al., 2020)。葉綠體基因組的基因按功能可以分為與光合作用相關基因、自身編碼基因和未知的開放閱讀框(Dobrogojski et al., 2020)。由于生境條件各異,不同植物使用的密碼子偏好性也存在一定的差異(Zhou et al., 2008)。與核基因組和線粒體相比,葉綠體基因組在結構、基因數量和基因組成方面呈現出更高的保守性,其相對適中的進化速率介于核基因組和線粒體基因組之間(CBOL Plant Working Group, 2009)。目前,已有多個葉綠體基因片段被開發用于物種鑒定、系統發育和群體遺傳多樣性的研究(CBOL Plant Working Group, 2009)。近年來,借助高通量測序技術對葉綠體全基因組進行分析,可以更加清晰地了解物種間的系統發育關系、種群的遺傳結構以及準確鑒定近緣物種(Daniell et al., 2021)。
本研究利用高通量測序技術對廣西裂果薯進行葉綠體全基因組測序,結合公共數據庫中已公開發表的蒟蒻薯屬植物葉綠體基因組,擬探討以下問題:(1)通過葉綠體基因組比較揭示廣西裂果薯和裂果薯的遺傳差異程度;(2)通過構建葉綠體基因組系統發育樹來推斷廣西裂果薯和裂果薯的親緣關系,為廣西裂果薯的歸并問題提供新的分子證據。
1 材料與方法
1.1 實驗材料和數據來源
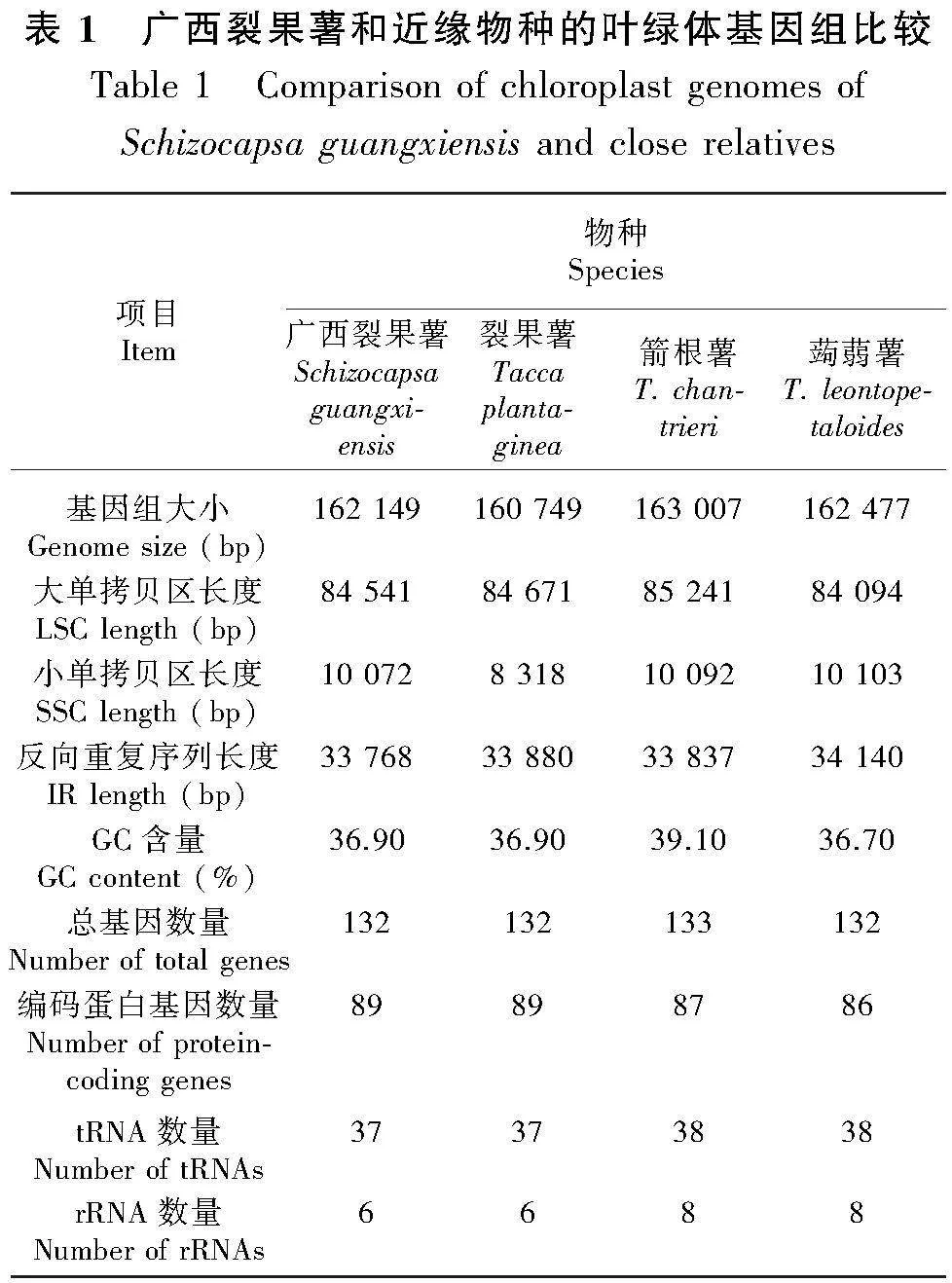
廣西裂果薯植物采集自廣西壯族自治區中國科學院廣西植物研究所。物種身份已經過該單位陸昭岑助理研究員的準確鑒定(圖1)。采集3片幼嫩葉片,用自來水沖洗干凈后保存于硅膠中干燥,用其提取總DNA。此外,我們通過NCBI數據庫(The National Center for Biotechnology Information)檢索并下載已公開發表的22個物種葉綠體基因組全長序列,包括21個薯蕷科物種,分別是參薯 [Dioscorea alata(OP 787126)]、異葉薯蕷 [D. biformifolia(OQ 526002)]、D. brevipetiolata(NC 062811)、黃獨 [D. bulbifera(NC 039708)]、D. cayenensis(MZ 848368)、薯莨 [D. cirrhosa(NC 065059)]、三角葉薯蕷 [D. deltoidea(OQ 525993)]、D. depauperata(NC 062812)、龜甲龍 [D. elephantipes(NC 009601)]、福州薯蕷 [D. futschauensis(NC 039808)]、褐苞薯蕷 [D. persimilis(NC 057257)]、D. praehensilis(NC 039837)、昆氏薯蕷 [D. quinquelobata(NC 057067)]、D. sagittifolia(NC 039854)、善司芭潤斯薯蕷 [D. sansibarensis(NC 039838)]、綿萆薢 [D. spongiosa(OQ 525998)]、山萆薢 [D. tokoro(OQ 525999)]、盾葉薯蕷 [D. zingiberensis(NC 027090)]、箭根薯 [Tacca chantrieri(KX 171420)]、蒟蒻薯 [T. leontopetaloides(NC 036658)]、裂果薯 [T. plantanginea(MT 419421.1)]和1個外類群石蒜科物種蔥蓮 [Zephyranthes bifida(NC 064147.1)]。
1.2 DNA提取和測序
采用十六烷基三甲基溴化銨(CTAB)法提取廣西裂果薯總DNA(Doyle & Doyle, 1987),分別使用瓊脂糖凝膠電泳和NanoDrop-2000紫外分光光度計(Thermo Fisher Scientific, USA)檢測DNA的質量和濃度。利用Illumina HiSeq 2000平臺進行雙端測序,測序讀長為150 bp。利用FastaQ軟件過濾原始數據,除去多余的N序列、特異性接頭序列和過短序列,獲得總數據量為5 200.34 Mb的高質量測序數據(clean data)用于后續的葉綠體基因組組裝。
1.3 葉綠體基因組組裝和注釋
將上述獲得的clean data序列導入GetOrganelle v1.7.5.0軟件(Jin et al., 2020)從頭進行葉綠體基因組組裝,參數值設置為默認值。將組裝結果文件.gfa導入Bandage 軟件中,對組裝完整度進行可視化分析(Wick et al., 2015)。使用在線葉綠體注釋平臺CPGAVAS2(http://47.96.249.172:16019/analyzer/home)以裂果薯(MT 419421.1)為參照基因組,對葉綠體的蛋白質編碼基因、tRNA和rRNA進行注釋(Shi et al., 2019)。使用GB2sequin檢查基因序列(Lehwark & Greiner, 2019),尤其是具有短外顯子和RNA編輯的起始密碼子基因,并手動校正錯誤的注釋。通過在線可視化工具OGDRAW(https://chlorobox.mpimp-golm.mpg.de/OGDraw.html)繪制葉綠體基因組圈圖(Greiner et al., 2019)。完整的廣西裂果薯葉綠體基因組信息已上傳至NCBI數據庫,登錄號為OR 805469。
1.4 密碼子使用偏好性分析
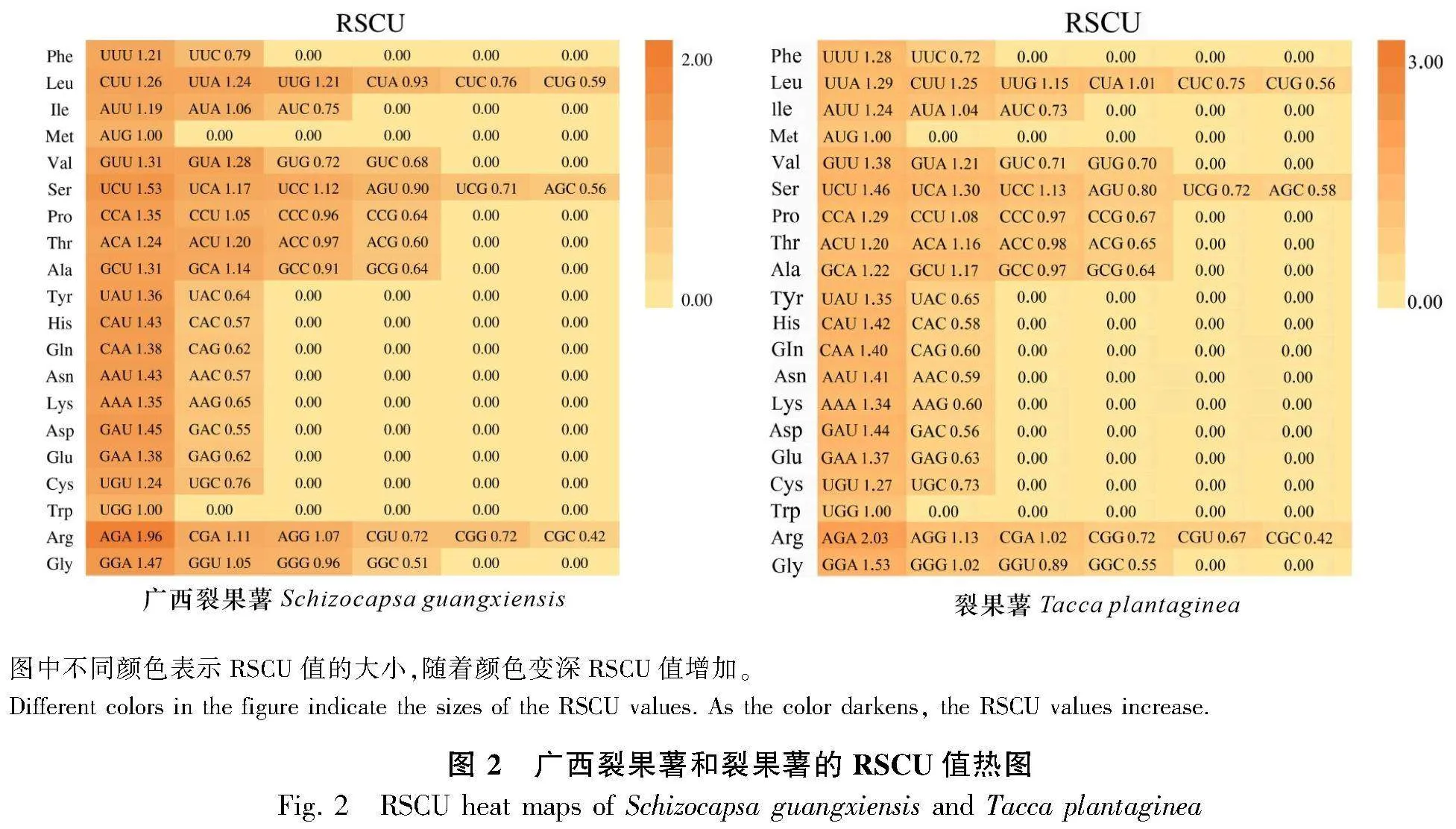
密碼子使用偏好性是物種遺傳變異和自然選擇而共同產生的結果。本研究利用CodonW v1.4.4軟件對葉綠體基因組的蛋白編碼基因進行偏好性分析,并對相對同義密碼子使用度(relative synonymous codon usage, RSCU)進行計算(Gupta et al., 2004)。為了減少誤差,選擇蛋白質編碼區長度≥300 bp。通過R語言heatmap包對RSCU進行可視化。當RSCU值>1時,表明該密碼子的使用頻率高于其他同義密碼子;當RSCU值=1時,表明該密碼子無使用偏好性;當RSCU值<1時,表明該密碼子的使用頻率低于其他同義密碼子(王婧等,2019)。
1.5 重復序列分析
利用MISA在線分析網站(https://webblast.ipk-gatersleben.de/misa)統計葉綠體基因組中的簡單重復序列(simple sequence repeats, SSR)(Beier et al., 2017),參數采用默認值,即單核苷酸、二核苷酸、三核苷酸、四核苷酸、五核苷酸和六核苷酸的最小重復次數分別為10、8、4、3、3、4,SSR之間的最小距離設為100 bp。對于其他類型的散在重復序列,我們利用REPuter在線分析網站(https://bibiserv.cebitec.uni-bielefeld.de/reputer/manual.html)進行鑒定,參數設置為最小重復序列 30 bp,漢明距離(Hamming distance)設置為3(Kurtz et al., 2001)。
1.6 葉綠體基因組IR邊界比較和分歧區域鑒定
不同植物的葉綠體基因組在IR邊界區有不同程度的收縮和擴張,是反映葉綠體基因組進化特征的主要信息,其中反向重復區在穩定葉綠體結構方面發揮著重要作用。為了更清晰地展示廣西裂果薯和裂果薯葉綠體基因組的IR邊界差異信息,我們采用IRscope在線分析網站(https://irscope.shinyapps.io/irapp/)對2個物種的葉綠體基因組邊界進行可視化分析(Amiryousefi et al., 2018)。對于序列分歧區的鑒定,我們利用mVISTA在線分析軟件(https://genome.lbl.gov/vista/index.shtml)中的Shuffle-LAGAN模型,以裂果薯為參考物種,對廣西裂果薯和蒟蒻薯屬3個物種的葉綠體基因組進行同源性比對(Frazer et al., 2004)。
1.7 系統發育分析
本研究從NCBI數據庫下載已發表的22個葉綠體基因組全長序列,包括21個薯蕷科物種和1個外類群物種(石蒜科蔥蓮)。使用PhyloSuite(V1.2.3)軟件中的MAFFTv7插件進行序列比對和矩陣構建,利用IQ-TREE 2以最大似然法(maximum likelihood, ML)構建系統發育樹,參數設置為默認(Minh et al., 2021)。將得到的樹文件導入到FigTree軟件中進行進化系統樹優化。
2 結果與分析
2.1 密碼子使用偏好性
植物在分化之前所使用的遺傳密碼子是相似的,在演化過程中由于基因突變和自然選擇而導致不同生境下的個體具有特定偏好的密碼子,最后分化成不同的物種(Romero et al., 2000)。本研究利用生信云在線網站工具分析了廣西裂果薯和裂果薯葉綠體基因組的蛋白質編碼序列相對同義密碼子使用度(RSCU)。結果顯示,廣西裂果薯葉綠體的蛋白質編碼區總長85 844 bp,共編碼54 049個密碼子,64種密碼子中,共編碼20種氨基酸,其中UAA、UAG、UGA為終止密碼子。廣西裂果薯和裂果薯編碼率最高的密碼子都是AGA(Arg,精氨酸),但裂果薯編碼率(2.03)比廣西裂果薯(1.96)的高(圖2)。廣西裂果薯和裂果薯使用密碼子偏好性最低的是CGC(Arg,精氨酸),使用度RSCU都為0.42。此外,二者使用的AUG(Met,蛋氨酸)和UGG(Trp,色氨酸)的RSCU都為1.00,表明沒有密碼子偏好性,但是其他大多數氨基酸都顯示出密碼子偏向。廣西裂果薯密碼子RSCU>1共有30個,而裂果薯密碼子RSCU>1共有31個,其中GGG(Gly,甘氨酸)在裂果薯中RSCU高于廣西裂果薯。二者密碼子RSCU > 1并且第三位以A/T(U)結尾的密碼子同為28個,廣西裂果薯以G堿基結尾的只有1個(AGG),而裂果薯以G堿基結尾的有2個(AGG、GGG),二者以C堿基結尾的都為1個(UCC)。廣西裂果薯密碼子RSCU<1共有29個,而裂果薯密碼子RSCU<1共有28個,二者大多數密碼子都以C或G結尾,RSCU偏低(圖2)。以上分析結果表明,廣西裂果薯與裂果薯葉綠體基因組密碼子偏好使用都以A/T(U)結尾的密碼子,但是二者在密碼子使用度上存在差異。
2.2 重復序列分析
SSR是廣泛存在于葉綠體基因組中的重復序列,由于其重復類型和數量豐富、共顯性遺傳、實驗結果重復性高等優點,常用于近緣物種鑒定和物種群體的遺傳多樣性分析(Bayliss et al., 2004)。由圖3:A可知,從廣西裂果薯葉綠體基因組中鑒定了66個SSR位點,裂果薯中鑒定了58個。兩者的SSR類型有單核苷酸、二核苷酸、三核苷酸、四核苷酸和五核苷酸(圖3:B)。此外,單核苷酸A/T重復單元的比例最高,在廣西裂果薯中占總SSR種類的72.73%,在裂果薯中占總SSR種類的68.96%。五核苷酸重復最少,廣西裂果薯有2個,占總SSR種類的3.03%,裂果薯只有1個,占總SSR種類的1.72%(圖3:B)。二者都沒有檢測到六核苷酸。廣西裂果薯和裂果薯中A/T、AT/AT、AAT/ATT、AAAT/ATTT、AATT/AATT和AATAT/ATATT重復基序分別占總SSR的84.85%和86.21%(圖3:A),表明兩物種都偏好使用A和T堿基。
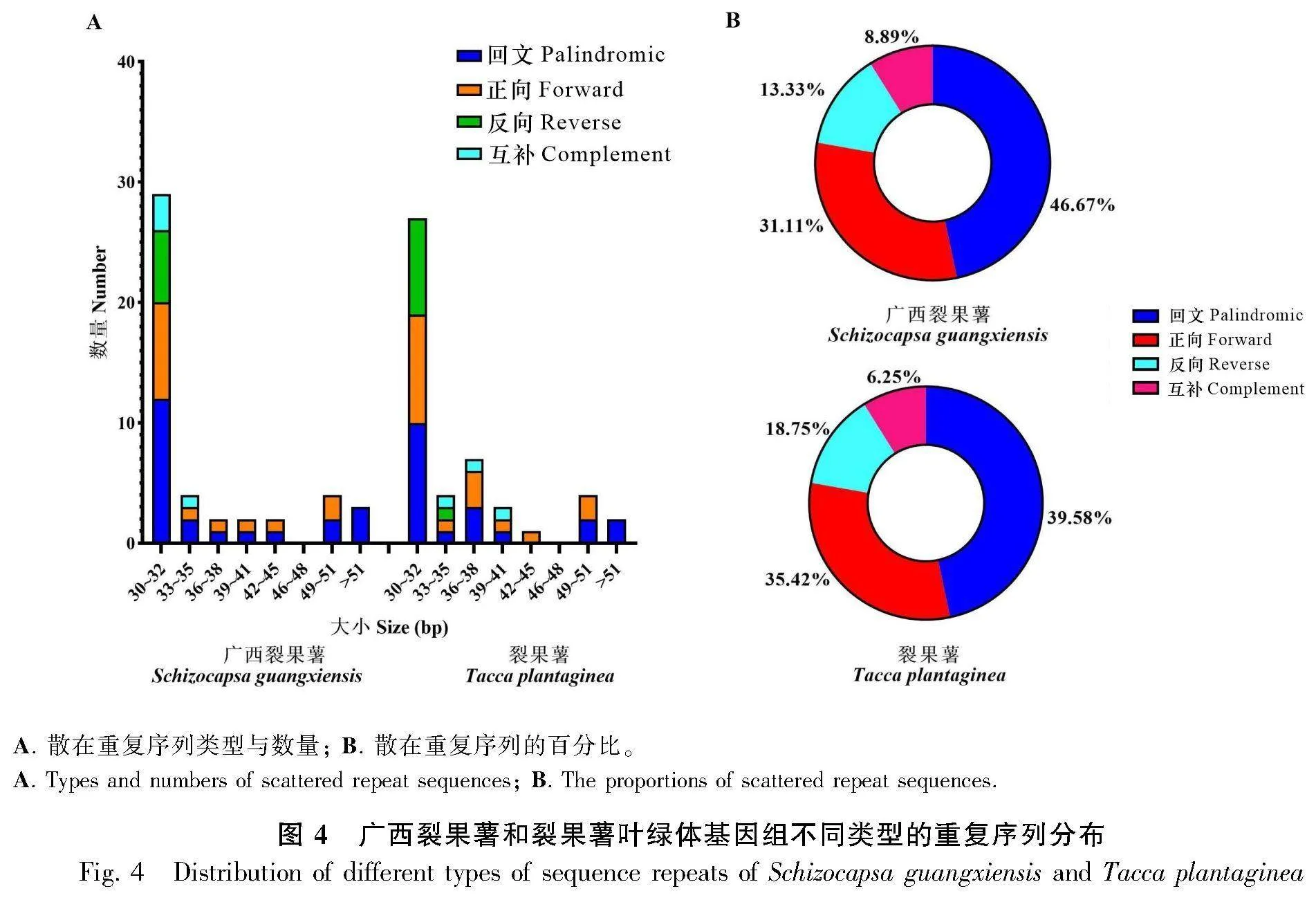
散在長重復序列的鑒定結果顯示,廣西裂果薯的葉綠體中含有45個長重復序列,包含21個回文重復序列、14個正向重復序列、6個反向重復序列、4個互補重復序列(圖4:A)。裂果薯具有48個重復,包含19個回文重復序列、17個正向重復序列、9個反向重復序列、3個互補重復序列(圖4:A)。這些重復序列主要位于蛋白質編碼區。兩者的回文重復序列的長度集中在30~60 bp之間(圖4:A),占長重復序列種類的46.67%和39.58%(圖4:B)。
2.4 葉綠體基因組IR邊界分析
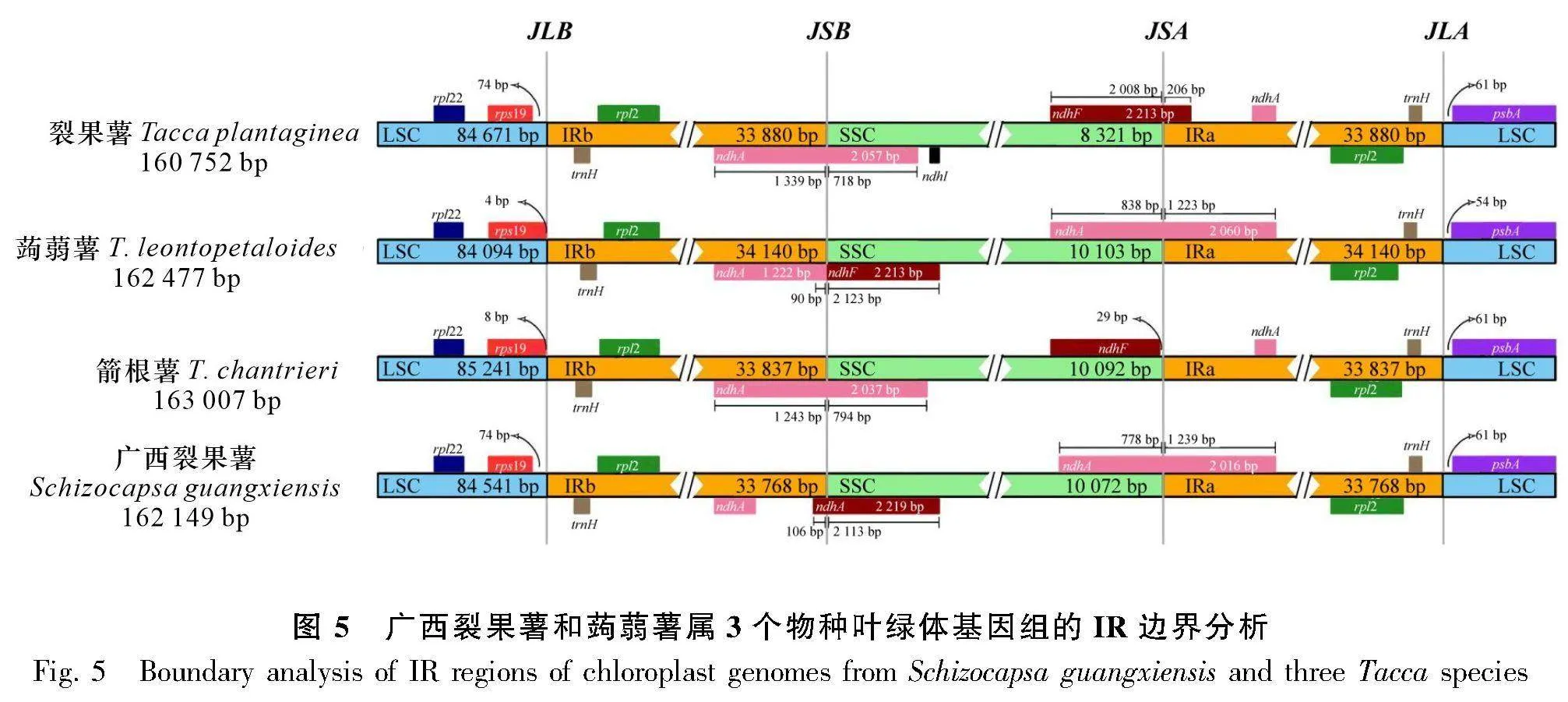
葉綠體基因組邊界的收縮與擴增現象能夠揭示葉綠體基因組結構變化和進化過程(Yang et al., 2023)。為了觀察廣西裂果薯IR區是否發生收縮或擴張,我們對廣西裂果薯和已發表的3個蒟蒻薯屬物種箭根薯、蒟蒻薯和裂果薯進行可視化分析(圖5)。4種植物葉綠體基因組均為環形四分體結構,在LSC、IRb、IRa和SSC區域中可劃分為4個邊界,即LSC/IRb邊界、IRb/SSC邊界、SSC/IRa邊界和IRa/LSC邊界。rpl22、rps19、trnH、rpl2、ycf1和psbA等基因存在于LSC/IRb、IRb/SSC、SSC/IRa和IRa/LSC邊界的交界處。我們發現不同蒟蒻薯植物的葉綠體基因組邊界存在明顯的差異,不同長度的rps19基因在4個物種中均位于LSC區,廣西裂果薯和裂果薯的rps19基因距離IRb/LSC邊界都為74 bp,而在箭根薯和蒟蒻薯中分別為8 bp和4 bp。在裂果薯和箭根薯中,ndhA基因橫跨IRb/SSC邊界,并向SSC區域分別延伸718 bp和794 bp,而廣西裂果薯和蒟蒻薯的ndhA基因橫跨IRa/LSC邊界,并分別向SSC區域延伸778 bp和838 bp。在廣西裂果薯和蒟蒻薯中,ndhF基因橫跨IRb/SSC邊界,并向IRb區分別延伸106 bp和90 bp,而裂果薯的ndhF基因橫跨IRa/SSC邊界并向IRa區延伸206 bp,只有箭根薯ndhF基因均在SSC區并距離IRa/SSC邊界29 bp。4個物種psbA基因均位于LSC區,并距離IRa/SSC邊界54~61 bp。以上結果表明,廣西裂果薯和裂果薯的LSC/IRb邊界和IRa/LSC邊界信息一致,主要的變異發生在IRb/SSC邊界和SSC/IRa邊界上,也是葉綠體基因組長度存在差異的主要原因。
2.5 葉綠體基因組序列分歧鑒定
本研究通過mVISTA在線工具分析了廣西裂果薯和蒟蒻薯屬3個物種的葉綠體基因組序列的分歧位點。圖6結果顯示,廣西裂果薯與裂果薯、蒟蒻薯、箭根薯序列相似度較高。編碼區變異程度較低且在IRa和IRb 區無明顯變異,如ycf2、rrn23、ndhB、rps12、ndhH、trnV-GAC基因具有較高的保守性。但是,廣西裂果薯與其他3個物種相比,在非基因編碼區存在明顯的差異,其中LSC區和SSC區的多樣性和變異程度較高,如trnK-UUU-trnQ-UUG、trnS-GCU-trnG-UCC、atpF、atpH-atpI、rpoB-trnC-GCA、trnE-UUC-trnT-GGU、ndhC-trnV-UAC、rbcL-accD、ycf4、petA-psbJ、rpl14-rpl16、ndhA、ccsA和ndhD序列差異較大(圖6)。這些分歧程度高的位點可以開發為屬內物種鑒定的分子標記。
2.5 廣西裂果薯葉綠體基因組特征
廣西裂果薯葉綠體基因組物理譜圖為典型的環狀雙鏈四分體結構(圖7),其中包括1個LSC區、1個SSC區和2個IR區。廣西裂果薯和裂果薯的葉綠體基因組全長分別為162 149 bp和160 749 bp,兩者序列長度差異為1 400 bp,總的GC含量都是36.90%。兩種植物的LSC長度分別為84 541 bp和84 671 bp,SSC長度分別為10 072 bp和8 318 bp,IR長度分別為33 768 bp和33 880 bp(表1)。廣西裂果薯和裂果薯葉綠體基因組的基因種類和數目完全一致,注釋到132個基因,包括89個蛋白質編碼基因,37個tRNA基因和6個rRNA編碼基因(表1)。廣西裂果薯與光合作用相關的基因44個、自我復制相關的基因54個、其他基因功能相關的基因6個和未知功能相關基因5個(表2)。在這些基因中,ndhA、ndhB、atpF、rpoC1、rps12、rpl16、rpl2、trnA-UGC、trnE-UUC、trnK-UUU、trnL-UAA、trnT-CGU各含有1個內含子,而基因ycf3、clpP各含有2個內含子(表2)。
2.6 廣西裂果薯的系統進化分析
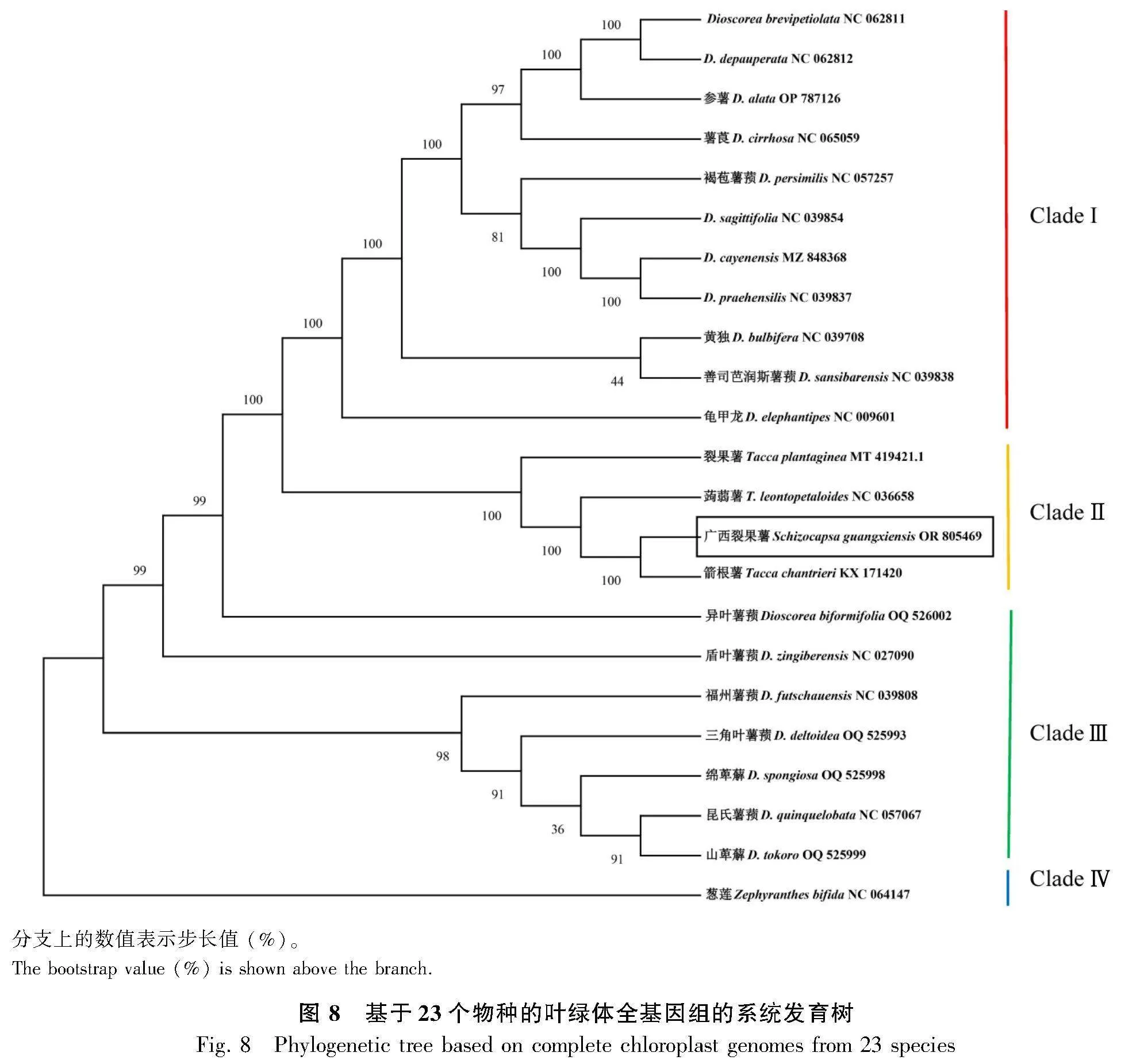
通過分析物種間的系統發育關系,可以幫助我們理解物種間親緣關系和演化途徑。本研究將石蒜科蔥蓮屬蔥蓮作為外類群,結合廣西裂果薯和其他21個薯蕷科的物種,運用最大似然法(ML)構建了系統發育樹,得到了一致的拓撲結構。結果表明,22個薯蕷科物種主要分為3個分支:分支I(Clade I)和分支Ⅲ(Clade Ⅲ)為薯蕷科薯蕷屬(Dioscorea)、分支Ⅱ(Clade Ⅱ)為薯蕷科蒟蒻薯屬(Tacca)。廣西裂果薯與箭根薯以100%的支持率聚為了一個單支,表明這兩個物種的親緣關系最近,其次是100%支持率的蒟蒻薯和裂果薯(圖8),表明廣西裂果薯和裂果薯的親緣關系較遠。
3 討論與結論
在本研究中,我們組裝了廣西裂果薯的完整葉綠體基因組。廣西裂果薯葉綠體基因組具有典型的四分體結構,與同科其他物種類似。通過比較已發布的蒟蒻薯屬物種葉綠體基因組,我們發現它們之間的基因種類和數量、順序、結構和其他特征高度保守。[JP+2]廣西裂果薯和裂果薯偏好使用的密碼子頻率不一致。在非編碼區也存在一些分歧度較高的序列,可開發為屬內物種鑒定的分子標記。現有的葉綠體基因組數據的系統發育分析證明廣西裂果薯與箭根薯關系最為密切。
通常高等植物的葉綠體基因組長度在120~160 kb范圍內,而廣西裂果薯和裂果薯的葉綠體基因組全長分別為162 149 bp和160 749 bp,兩者序列長度差異為1 400 bp。與其他被子植物類似,兩物種葉綠體基因組的IR區檢測到高GC含量,這可能是由于rRNA序列富含GC的結果。內含子在基因選擇性剪接中起著至關重要的作用,然而也有一些物種在進化過程中丟失了內含子(Qian et al., 2013)。本研究發現薯蕷科植物的葉綠體基因組在進化過程中沒有丟失任何內含子,暗示該科植物葉綠體基因組的蛋白質編碼基因高度保守。密碼子使用偏好性能反映物種基因的起源、進化和突變模式,對物種進化和遺傳研究都有重要意義(Quax et al., 2015)。通過對廣西裂果薯和裂果薯葉綠體密碼子統計發現,盡管精氨酸(Arg)是兩者使用最多的氨基酸,但是兩物種所使用的密碼子頻率不一樣,甚至其他密碼子的使用頻率也存在一定的差異,此結果對于探索它們在進化過程中所經受的選擇壓力以及物種分化提供了理論依據。在優選密碼子(RSCU>1)中,我們發現除了UUG之外,大多數密碼子以A或U結尾,但這并不是廣西裂果薯葉綠體基因組所獨有的,在單子葉植物水稻和玉米、雙子葉植物丹參和煙草中也觀察到了相似的現象(Qian et al., 2013;Asaf et al., 2016)。盡管同源基因在不同物種中具有相似的功能,但是不同物種在進化過程中由于所處的生存條件差異很大,自然會偏好選擇適應該環境的遺傳變異并固定下來,最終導致基因亞功能化而發生物種分化。根據RSCU的結果,我們認為廣西裂果薯和裂果薯之間可能已經發生物種分化。
SSR已被廣泛應用于群體遺傳學、系統發育評估和近緣物種或品種的鑒定,是區分較低分類水平物種的重要分子標記(Torokeldiev et al., 2019)。我們發現葉綠體基因組的大多數SSR都位于SSC和LSC區域的非編碼區,占SSR總數的80%以上。在廣西裂果薯葉綠體基因組中檢測到66個SSR,但裂果薯只有58個。兩者都含有相同的重復類型,但在不同類型中的數量有所差異。在許多植物葉綠體基因組中,Poly (A/T)的SSR通常比其他SSR重復類型更常見,這類SSR主要位于非編碼區,通常代表種內變異的重復次數,而二核苷酸及以上重復的SSR主要位于基因序列中,是基因功能發生變化最終形成新物種的動力之一(Tuler et al., 2015)。因此,本研究中檢測到的SSR位點可為廣西裂果薯遺傳多樣性水平分析和近緣物種鑒定提供新的分子標記。散在長重復序列也在基因組結構變異和物種分化中發揮著重要作用(Cavalier, 2002)。大多數重復序列位于基因間隔區域,其次是編碼區域。本研究中,廣西裂果薯有21個回文重復序列、14個正向重復序列、6個反向重復序列、4個互補重復序列;而裂果薯具有19個回文重復序列、17個正向重復序列、9個反向重復序列、3個互補重復序列。由此可知,二者在散在長重復序列的數目上有明顯的差異性,是兩物種遺傳物質變異的分子證據。
雖然被子植物的葉綠體基因組在結構和大小上相對保守,但是由于進化事件引起的IR區域的擴張和收縮會導致基因組的IR邊界和基因組大小發生微量變化,從而增加了被子植物葉綠體的遺傳多樣性(He et al., 2017)。我們發現廣西裂果薯和蒟蒻薯屬物種的葉綠體基因組IR區長度基本相似,尤其是IRb/LSC和IRa/LSC的邊界大致相同,主要差異發生在IRb/SSC和SSC/IRa邊界上,這是導致SSC區長度發生變化的主要原因。在許多被子植物的葉綠體基因組中IRb區的ycf1基因經常形成假基因化。然而,廣西裂果薯的IR區向SSC區發生明顯的擴張現象,形成了完整的ycf1基因。IR區的擴張也是導致廣西裂果薯葉綠體基因組變大的主要因素。此外,我們在蒟蒻薯屬其他物種中也觀察到以上現象。基于IR區邊界分析結果,我們推測廣西裂果薯可能歸屬于蒟蒻薯屬。由于形態上的相似性,利用傳統的方法對近期分化的物種進行準確區分具有非常高的挑戰性。對于某些特殊的物種,即使是使用標準DNA植物條形碼也無法區分,必要時,需針對特定物種開發物種特異性的分子標記(Zhao & Woeste, 2011)。基于mVISTA比較分析,我們發現IR區域比LSC和SSC區域更加高度保守,并且編碼區域比非編碼區域保守性高,這與其他被子植物類似(Qian et al., 2013)。廣西裂果薯葉綠體基因組中的LSC和SSC區域具有一些高水平變異序列,多數位于基因間隔區上。這些高度變異的區域可能具有解決該屬物種鑒定和構建高分辨率系統發育樹的能力,可以作為候選的蒟蒻薯屬特異性DNA條形碼。
葉綠體基因組含有豐富的變異位點,作為植物鑒定的“超級條形碼”能通過基因的缺失和排列順序等方面來鑒定物種,在物種系統發育分析中具有獨特優勢。大量研究表明,葉綠體基因組在近緣物種、居群及個體間均能實現高效鑒定,與傳統分子系統學研究相比,基于葉綠體全基因組序列構建系統發育樹具有更高的支持率和分辨率(Daniell et al., 2021)。根據傳統的形態分類,裂果薯屬已被歸入蒟蒻薯屬,并且《中國植物志》將廣西裂果薯歸并為裂果薯(中國科學院中國植物志編輯委員會,1985)。然而,2023版中國生物物種名錄中仍然將裂果薯屬作為獨立的一屬。在本研究中,我們檢索公共數據庫中可用的薯蕷科葉綠體基因組,并基于ML方法推斷廣西裂果薯和裂果薯的系統發育關系,結果顯示廣西裂果薯和箭根薯聚為一支(支持率100%),而裂果薯以100%的支持率位于蒟蒻薯屬的基部,表明廣西裂果薯和裂果薯是兩個完全不同的物種。然而,有限的類群信息會導致不同的拓撲系統樹,為了可靠地推斷物種系統發育關系,我們有必要對蒟蒻薯進行全面采樣。總體而言,廣西裂果薯完整的葉綠體基因組可為該屬和種的分類修訂提供新的分子依據。
參考文獻:
AMIRYOUSEFI A, HYVNEN J, POCZAI P, 2018. IRscope: an online program to visualize the junction sites of chloroplast genomes [J]. Bioinformatics, 34(17): 3030-3031.
ASAF S, KHAN AL, KHAN AR, et al., 2016. Complete chloroplast genome of Nicotiana otophora and its comparison with related species [J]. Front Plant Sci, 7: 843.
BAYLISS CD, DIXON KM, MOXON ER,2004. Simple sequence repeats (microsatellites): mutational mechanisms and contributions to bacterial pathogenesis. A meeting review [J]. FEMS Immunol Med Microbiol, 40(1): 11-19.
BEIER S, THIEL T, MNCH T, et al., 2017. MISA-web: a web server for microsatellite prediction [J]. Bioinformatics, 33(16): 2583-2585.
Biodiversity Committee, Chinese Academy of Sciences, 2023. Catalogue of Life China 2023 Annual Checklist [M]. Beijing: Science Press. [中國科學院生物多樣性委員會, 2023. 中國生物物種名錄(2023版) [M]. 北京: 科學出版社.]
CAVALIER T, 2002. Chloroplast evolution: secondary symbiogenesis and multiple losses [J]. Curr Biol, 12(2): R62-R64.
CBOL Plant Working Group, 2009. A DNA barcode for land plants [J]. Proc Natl Acad Sci USA, 106(31): 12794-12797.
Chinese Academy of Sciences Editorial Committee for Flora of China, 1985. Flora Reipublicae Popularis Sinicae: Volume 16. Part 1 [M]. Beijing: Science Press. [中國科學院中國植物志編輯委員會, 1985. 中國植物志: 第十六卷第一分冊 [M]. 北京: 科學出版社.]
DANIELL H, JIN S, ZHU XG, et al., 2021. Green giant-a tiny chloroplast genome with mighty power to produce high-value proteins: history and phylogeny [J]. Plant Biotechnol J, 19(3): 430-447.
DOBROGOJSKI J, ADAMIEC M, LUCINSKI R, 2020. The chloroplast genome: a review [J]. Acta Physiol Plantarum, 42: 1-13.
DOYLE JJ, DOYLE JL, 1987. A rapid DNA isolation procedure for small quantities of fresh leaf tissue [J]. Phytochemical Bull, 19: 11-15.
DRENTH E, 1972. A revision of the family Taccaceae [J]. Blumea, 20: 367-406.
FRAZER KA, PACHTER L, POLIAKOV A, et al., 2004. VISTA: computational tools for comparative genomics [J]. Nucl Acids Res, 32(2): W273-W279.
FU LK, JIN JM, 1992. China plant red data book: rare and endangerad plants: Part 1 [M]. Beijing: Science Press: 642-643. [傅立國, 金鑒明, 1992. 中國植物紅皮書-稀有瀕危植物: 第1冊 [M]. 北京: 科學出版社: 642-643.]
GREINER S, LEHWARK P, BOCK R, 2019. Organellar Genome DRAW (OGDRAW) version 1.3.1: expanded toolkit for the graphical visualization of organellar genomes [J]. Nucl Acids Res, 47(W1): W59-W64.
GUPTA SK, BHATTACHARYYA TK, GHOSH TC, 2004. Synonymous codon usage in Lactococcus lactis: mutational bias versus translational selection [J]. J Biomol Struct Dyn, 21(4): 527-536.
HE L, QIAN J, LI X, et al., 2017. Complete chloroplast genome of medicinal plant Lonicera japonica: genome rearrangement, intron gain and loss, and implications for phylogenetic studies [J]. Molecules, 22(2): 249.
JIANG J, YANG H, WANG Y, et al., 2014. Phytochemical and pharmacological studies of the genus Tacca: a review [J]. Trop J Pharm Res, 13(4): 635-648.
JIA MR, LI XW, 2005. Zhongguo Minzu Yaozhi Yao [M]. Beijing: China Medical Science and Technology Press. [賈敏如, 李星煒, 2005. 中國民族藥志要 [M]. 北京: 中國醫藥科技出版社.]
JIN JJ, YU WB, YANG JB, et al., 2020. GetOrganelle: a fast and versatile toolkit for accurate de novo assembly of organelle genomes [J]. Genome Biol, 21(1): 241.
KURTZ S, CHOUDHURI JV, OHLEBUSCH E, et al., 2001. REPuter: the manifold applications of repeat analysis on a genomic scale [J]. Nucl Acids Res, 29(22): 4633-4642.
LEHWARK P, GREINER S, 2019. GB2sequin — A file converter preparing custom GenBank files for database submission [J]. Genomics, 111(4): 759-761.
LING PP, DING ZZ, 1982. Two new species of Taccaceae from China [J]. Acta Phytotaxon Sin, 20(2): 201-204. [凌萍萍, 丁志遵, 1982. 中國蒟蒻薯科二新種 [J]. 植物分類學報, 20(2): 201-204.]
LING PP, 1985. Flora Reipublicae Popularis Sinicae: Volume 16. Part 1 [M]. Beijing: Science Press: 42-44. [凌萍萍, 1985. 中國植物志: 第十六卷第一分冊 [M]. 北京: 科學出版社: 42-44.]
LIU HY, NI W, XIE BB, et al., 2006. Five new withanolides from Tacca plantaginea [J]. Chem Pharm Bull, 54(7): 992-995.
MEHMOOD F, ABDULLA H, SHAHZADI I, et al., 2020. Characterization of Withania somnifera chloroplast genome and its comparison with other selected species of Solanaceae [J]. Genomics, 112(2): 1522-1530.
MINH BQ, SCHMIDT HA, CHERNOMOR O, et al., 2021. IQ-TREE 2: new models and efficient methods for phylogenetic inference in the genomic Era [J]. Mol Biol Evol, 37(5): 1530-1534.
QIAN J, SONG J, GAO H,et al., 2013. The complete chloroplast genome sequence of the medicinal plant Salvia miltiorrhiza [J]. PLoS ONE, 8(2): e57607.
QIN YP, ZHANG XX, HE NT,et al., 2023. Mechanism of anti-tumor effect of Saponin I of Schizocapsa plantaginea Hance on human pancreatic cancer cell PANC-1 transplanted in nude mice [J]. J Guangxi Med Univ, 40(6): 938-944. [秦燕萍, 張璇璇, 何能婷, 等, 2023. 裂果薯皂苷Ⅰ抗人胰腺癌細胞PANC-1裸鼠移植瘤的作用機制研究 [J]. 廣西醫科大學學報, 40(6): 938-944.]
QUAX TE, CLAASSENS NJ, SLL D, et al., 2015. Codon bias as a means to fine-tune gene expression [J]. Mol Cell, 59(2): 149-161.
ROMERO H, ZAVALA A, MUSTO H, 2000. Codon usage in Chlamydia trachomatis is the result of strand-specific mutational biases and a complex pattern of selective forces [J]. Nucl Acids Res, 28(10): 2084-2090.
SHI L, CHEN H, JIANG M, et al., 2019. CPGAVAS2, an integrated plastome sequence annotator and analyzer [J]. Nucl Acids Res, 47(W1): W65-W73.
SUN YW, 2013. Targeted screening anti-cancer from herbal medicine in Guangxi and the isolation and anti-caner research of steroidal saponins from Schizocapsa plantaginea Hance [D]. Nanning: Guangxi Medical University. [孫悅文, 2013. 廣西特色中草藥靶向抗腫瘤篩選及裂果薯皂苷的分離與抗腫瘤作用研究 [D]. 南寧: 廣西醫科大學.]
TOROKELDIEV N, ZIEHE M, GAILING O, et al., 2019. Genetic diversity and structure of natural Juglans regia L. populations in the southern Kyrgyz Republic revealed by nuclear SSR and EST-SSR markers [J]. Tree Genet Genomes, 15(1): 501-512.
TULER AC, CARRIJO TT, NIA LR, et al., 2015. SSR markers: a tool for species identification in Psidium (Myrtaceae) [J]. Mol Biol Rep, 42: 1501-1513.
WANG HM, WEI JF, QIU MM, et al., 2004. Characteristics and microscopic identification of Tacca plantaginea Hance [J]. China Med Mat (10): 724-725. [王慧明, 韋家福, 丘明明, 等, 2004. 水田七的性狀與顯微鑒定 [J]. 中藥材 (10): 724-725.]
WANG J, WANG TY, WANG LY, et al., 2019. Assembling and analysis of the whole chloroplast genome sequence of Elaeagnus angustifolia and its codon usage bias [J]. Acta Bot Boreal-Occident Sin, 39(9): 1559-1572. [王婧, 王天翼, 王羅云, 等, 2019. 沙棗葉綠體全基因組序列及其使用密碼子偏性分析 [J]. 西北植物學報, 39(9): 1559-1572.]
WICK RR, SCHULTZ MB, ZOBEL J, et al., 2015. Bandage: interactive visualization of de novo genome assemblies [J]. Bioinformatics, 31(20): 3350-3352.
YANG C, WANG K, ZHANG H, et al., 2023. Analysis of the chloroplast genome and phylogenetic evolution of three species of Syringa [J]. Mol Biol Rep, 50(1): 665-677.
Yunnan Medicinal Herbs Company, 1993. Directory of traditional Chinese medicinal resources in Yunnan Province [M]. Beijing: Science Press. [云南省藥材公司, 1993. 云南中藥資源名錄 [M]. 北京: 科學出版社.]
ZHANG L, LI HT, GAO LM, et al., 2011. Phylogeny and evolution of bracts and bracteoles in Tacca (Dioscoreaceae) [J]. J Integr Plant Biol, 53(11): 901-911.
ZHAO P, WOESTE KE, 2011. DNA markers identify hybrids between butternut (Juglans cinerea L.) and Japanese walnut (Juglans ailantifolia Carr.) [J]. Tree Genet Genomes, 7: 511-533.
ZHOU M, LONG W, LI X, 2008. Patterns of synonymous codon usage bias in chloroplast genomes of seed plants [J]. For Stud China, 10: 235-242.
ZHU WZ, 2000. Flora of China: Vol. 24 [M]. Beijing: Science Press; St. Louis: Missouri Botanical Garden Press: 274-275.

