基于糞便DNA宏條形碼的人工草地放牧綿羊采食組分研究


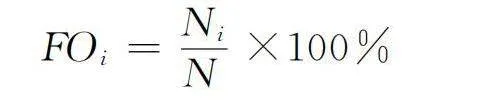
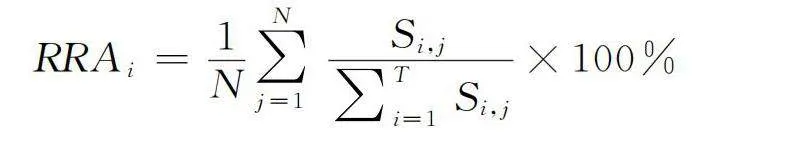
收稿日期:2024-04-10;修回日期:2024-05-15
基金項目:寧夏回族自治區重點研發項目(2017BY082);中國博士后科學基金面上項目(2019M660877);國家自然科學基金重大項目(32192463)資助
作者簡介:郭艷萍(1986-),女,漢族,河北邯鄲人,博士,助研,主要從事草地管理、草種質資源研究,E-mail:guoyp@caf.ac.cn;*通信作者Author for correspondence,E-mail:luohailing@cau.edu.cn
摘要:準確獲取食草動物食性數據有助于了解草地放牧家畜的營養狀況,同時也對掌握草地植物群落的動態變化具有重要意義。本研究通過從灘羊糞便樣本中提取DNA,利用ITS2條形碼和糞便DNA宏條形碼技術(fDNA metabarcoding),基于出現頻率(Frequency of occurrence,FO)和相對序列豐度(Relative read abundance,RRA)兩個指標快速分析獲得糞便中的物種組成。分析獲得的灘羊食物組成包含28個植物類群,主要有藜屬(Chenopodium sp.)、苜蓿屬(Medicago sp.)、蒿屬(Artemisia sp.)等隸屬于13科23屬,其中27個鑒定到種水平,物種鑒定分辨率高達96%。藜屬FO和RRA均最高,分別為100.0%和31.5%,其次為紫花苜蓿(Medicago sativa),分別為100.0%和26.9%。基于兩種算法分析得到的食物比例具有差異性和一致性,其中紫花苜蓿是放牧灘羊的主要采食來源,但攝入比例需要校正。研究證明了基于ITS2片段的糞便DNA宏條形碼技術在食草家畜快速準確食性分析中的可行性,為此類研究提供了有力借鑒。建議采用該技術時應完善潛在攝食植物DNA條形碼數據庫,準確分析目標家畜在特定區域內的采食種類。
關鍵詞:綿羊;食性;DNA宏條形碼;ITS2;物種鑒定
中圖分類號:S541 文獻標識碼:A 文章編號:1007-0435(2024)10-3043-09
Fecal DNA Metabarcoding-based Diet Composition Research of Grazing Lambs on Artificial Pasture
GUO Yan-ping1,2, WANG Yue-jun1, PENG Si-jia1, ZUO Shu-xian1, ZHANG Ying-jun3, LUO Hai-ling1*
(1. State Key Laboratory of Animal Nutrition and Feeding, College of Animal Science and Technology, China Agricultural University, Beijing 100193, China; 2. Research Institute of Forestry, Chinese Academy of Forestry, Beijing 100091, China; 3. College of Grassland Science and Technology, China Agricultural University, Beijing 100193, China)
Abstract:Acquiring accurate dietary composition information of herbivores is a valuable tactic in understanding their nutritional level and the function of the grassland ecosystem. This study collected Tan lambs fecal samples from three treatment groups:artificial pasture grazing(G),artificial pasture grazing with the indoor feeding group (GF),and the indoor feeding group (F). DNA was then extracted from the samples and the species were identified based on the ITS2 gene and DNA mebarcoding technique. Frequency of occurrence (FO) and Relative read abundance (RRA) were used to determine the diet composition. Using fecal DNA metabarcoding,we conducted diet analysis for the lambs and found 28 different plant operational taxonomics unites (OTUs) belonging to 13 families and 23 genera,of which 27 were identified to the species level,and the species identification resolution was 96%. The predominant categories were Chenopodium sp.,Medicago sp.,and Artemisia sp.. Chenopodium sp. had the highest relative FO (100.0%) and RRA (31.5%) among all the ingested foods,followed by Medicago sativa (100.0% and 26.9%,respectively). Therefore,to some extent the proportion obtained by frequency of occurrence and relative read abundance showed consistent tendencies but also generated differences. The results revealed that M.sativa was the main diet component of lambs,however its accurately proportion needs further correction. In addition,we suggested that the usage of fecal DNA barcode analysis approach should consider feeding range,targeted herbivores,and improved barcode database,which favorable to accurately determination of species and ingestion proportion. Our study highlights the utilization of fecal DNA metabarcoding based ITS2 gene in fast and high-throughput diet analysis,and provides a technical guidance for future herbivore livestock nutrition surveys and food-web studies.
Key words:Sheep;Diet analysis;DNA metabarcoding;ITS2;Species identification
食草家畜食性研究是掌握草地放牧家畜營養狀況的基礎,也是監測草地植物群落動態變化的重要載體,對于開展草地精細化管理、家畜高效飼養策略、畜產品提質增效等具有重要意義[1-2]。準確獲取放牧家畜的采食組分及采食量,是探究放牧家畜生長水平及其差異性形成原因的關鍵,同時也能反映出其選擇性采食對草原生態系統結構與功能的影響[3]。然而放牧家畜的牧食行為較為復雜,采食數據獲取難度大[4]。傳統的草食家畜食性研究方法有很多,主要包括模擬采食法、牧草采食法、食道瘺管法、瘤胃內容物法、糞便顯微分析法、植物蠟層指示劑法和近紅外光譜法等[5-7]。
自90年代以來,以飽和鏈烷為代表的植物蠟層指示劑法使用范圍最廣[8-14]。牧草中的鏈烷、長鏈脂肪酸和長鏈醇以及其他碳同位素組分可以作為“植物指紋”來直接估測放牧家畜的0uv8M8yxVYMJVCErQ9WqF2Jz8NXVU7ANwUAd0VzadhU=采食成分及采食量,對日糧組成簡單的家畜食譜的估測結果準確可靠[15-16]。盡管該方法具有準確度高、低成本、易操作、非損傷性采樣等優點,但如果飼草中的鏈烷濃度較低,或者物種間鏈烷模式相似時,隨著自由采食家畜的食物組分復雜化,準確性會明顯下降,而且植物飽和鏈烷的濃度模式會因季節波動差異較大,需要在不同季節反復取樣監測,過程繁瑣,這也是植物蠟層成員的共有缺陷。
伴隨著分子生物學研究的逐步深入和高通量測序(High-throughput sequencing,HTS)技術的快速發展,糞便DNA宏條形碼技術(fDNA metabarcoding)的出現為動物食性研究提供了一種更高效、更準確的手段[2,17-19]。DNA宏條形碼是利用高通量測序手段獲得樣本中多個物種DNA條形碼片段,通過生物信息學手段實現物種鑒定的技術[20]。該技術不受物種的個體形態及發育時期限制,對于高度降解的食物殘渣敏感性高,能夠快速獲取更高通量的物種信息,同時也避免了傳統方法的主觀誤差[21-22]。目前,該技術已廣泛應用于食草動物和食肉動物的食性研究領域中,如軟體動物[23]、節肢動物[24]、爬行動物[25]、鳥類[26]和哺乳動物[27-28]等。
灘羊作為寧夏優質綿羊品種,因其肉質鮮嫩、膻味小、風味獨特而深受消費者喜愛。隨著灘羊市場需求的加大和緩解天然草場放牧壓力的客觀需求,養殖模式逐漸由傳統的天然草場放牧向規模化舍飼或人工草場放牧轉變。前期研究發現不同飼養方式會影響羊肉的色澤、口感及營養成分[29],然而采食組分攝入與灘羊肉品質形成之間的關系尚不清晰。本研究以灘羊糞便為實驗材料,利用DNA宏條形碼技術探討其食物組成,為開展不同飼養方式下灘羊采食組分差異研究提供方法基礎,進而為灘羊精準營養和實現健康養殖提供理論依據。
1 材料與方法
1.1 試驗點概況
試驗樣地為人工混播草地,位于寧夏回族自治區固原市原州區頭營鎮馬園村(106°26′E,36°14′N,平均海拔1600 m)。樣地設置4個放牧小區,面積為516 m2。混播草地種植紫花苜蓿(Medicago sativa)、草地早熟禾(Poa pratensis)、草地雀麥(Bromus riparius)的組合比例為15%,35%及50%,畝播量為4.5 kg。
1.2 試驗動物及飼養管理
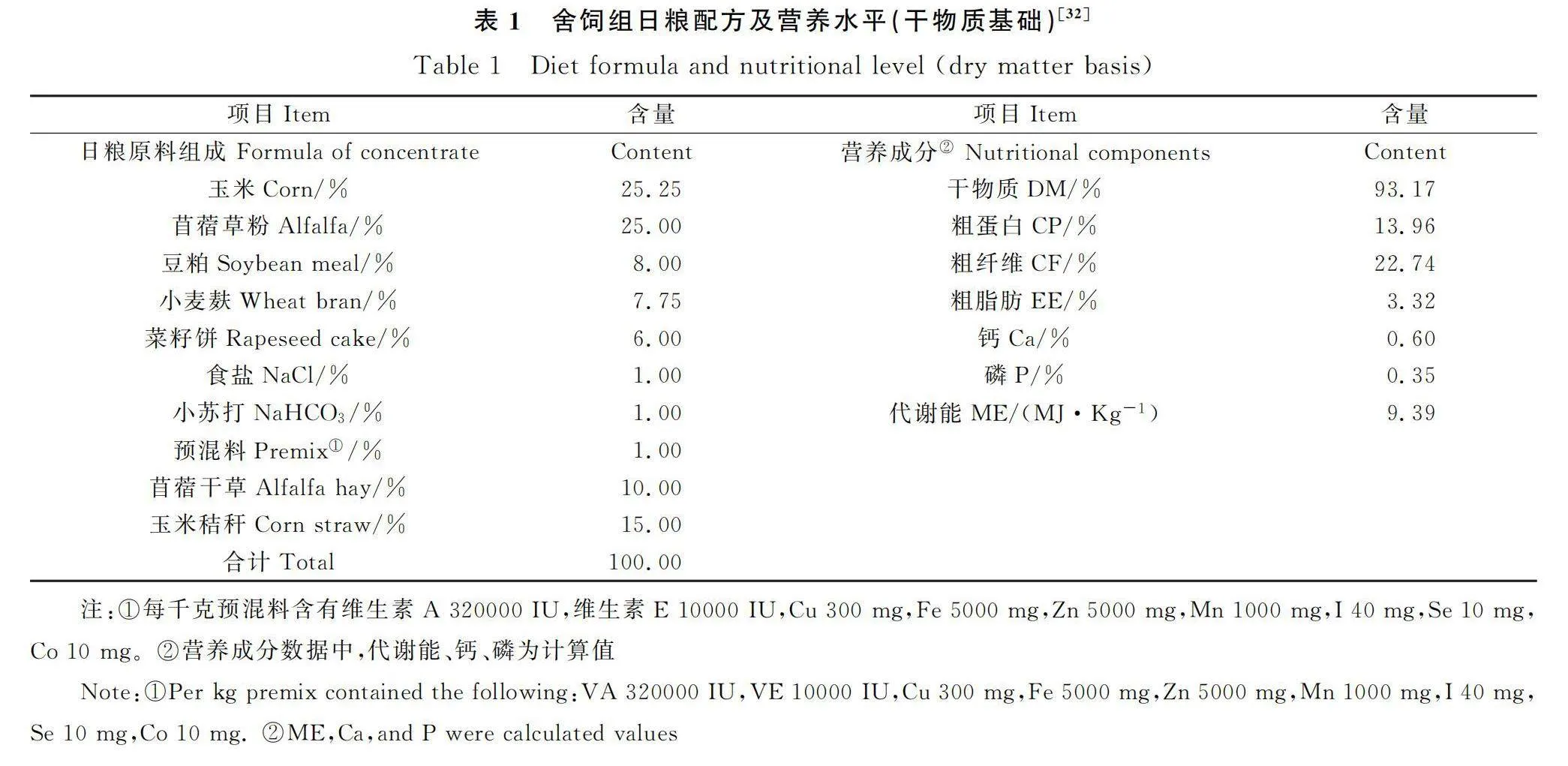
飼養試驗于2019年7—10月進行。選擇體重(19.76±0.29)kg相近的3月齡斷奶灘羊公羔33只,隨機分為3組,每組11只。試驗設置3個處理組,分別為舍飼組(F)、人工草場限時放牧補飼組(GF)和人工草場放牧組(G)。F組每天飼喂兩次,時間分別為7:30和17:30,并保證活動時間1~2 h;GF組每天7:00—11:00進行放牧,歸牧后進行補飼,試驗日糧同舍飼組;G組每天7:00—19:00進行放牧,不補飼。G組和GF組的灘羊采取輪牧形式,放牧試驗結束后,所有羊只歸圈。飼養試驗期間自由飲水。試驗日糧依據NRC(2007)肉用綿羊的營養需求,按照體重30 kg,日增重200 g的標準進行配制,日糧配方及營養水平詳見表1所示。
1.3 樣品采集和保存
分別從3個處理組中隨機挑選5只羔羊用于采集新鮮糞便樣品。具體采集方法為:采樣期間,灘羊在歸牧后單獨置于圍欄內,并在欄底鋪上干凈塑料布,在次日清晨飼喂或放牧之前收集糞樣,并迅速置于-20℃保存。將連續收集三天的新鮮糞便樣本均勻混合為15份,置于-20℃冰箱低溫保存,用于后續分析采食組分。
1.4 糞便DNA提取和PCR擴增
將采集的糞便樣品迅速粉碎,稱取200 mg放入2 mL微量離心管內,將離心管置于冰上。使用糞便基因組DNA提取試劑盒(Omega Biotek,Norcross,GA)提取灘羊糞便總DNA,提取方法參考試劑盒詳細說明。將所得DNA溶液收集到離心管后,-20℃保存。
以上述DNA為模板,基于文獻報道[30]和前期研究[31],選用引物對rD5-ITS2(5′-barcode-TCCTCCGCTTATTGATATGC-3′)和rb1-ITS2f (5′-CGATACTTGGTGTGAATTGCAG-3′)進行糞便中植物DNA核基因ITS2片段的擴增,擴增片段長度約為350 bp。在引物5′端增加由8個特異性堿基序列組合而成的“barcode”標簽,擴增同一樣品的一對引物使用相同標簽,以區分后續混合樣品高通量測序結果中的序列來源。
PCR擴增體系為20 μL,包括樣品DNA 10 ng dNTPs (2.5 mmol·L-1) 2.0 μL,5×FastPfu buffer 4.0 μL、5×FastPfu聚合酶0.4 μL,正反引物各0.8 μL (5 μmol·L-1),牛血清蛋白(BSA)0.2 μL,ddH2O補充至20 μL。PCR反應程序為94℃預變性5 min;94℃變性30 s,59℃退火60 s,72℃延伸60 s,循環45次;72℃延伸10 min。
1.5 高通量測序和數據處理
PCR產物用2%瓊脂糖凝膠電泳檢測擴增條帶大小,用AxyPrep DNA Gel Extraction Kit進行純化,應用QuantiFluor-ST (Promega,U.S.)進行定量,將擴增子等量混合,構建文庫,使用Illumina MiSeq PE300平臺完成雙端測序(Paired-end sequencing),由上海美吉生物醫藥科技有限公司完成。
測序完成后,用QIIME(version1.17)軟件對原始fastq文件進行嚴格的過濾和質控處理,并形成一個新的帶有標簽的序列文件。使用Usearch軟件,按照97%相似性對序列進行操作分類單元(operational taxonomic units,OTU)聚類,過程中使用UCHIME軟件識別并去除嵌合體序列,得到OTU代表序列。將所有優化序列匹配至OTU代表序列,選出與OTU代表序列相似性在97%以上的序列,生成OTU表格。
對OTU進行多樣性分析,以評估測序結果。利用MOTHUR(version1.30.1)軟件進行物種覆蓋程度和Alpha多樣性分析,包括Good′s coverage指數、物種豐富度估計(Chao,ACE),物種多樣性統計(香農指數Shannon index、辛普森指數Simpson index)。利用QIIME程序進行主成分分析(Principal-component analysis,PCA),用R語言(version3.2.1)中的VEGAN程序包繪圖。
利用Blastn工具,將上述OTU代表序列與NCBI中的NT數據庫進行比對,獲得每個OTU的物種注釋。按照序列同源性最高原則,對每一個代表性OTU序列進行分類學注釋。同時需要綜合考慮其他因素,結合本地物種分布信息,如匹配物種所在特定地域的多樣性,對物種進行最終注釋,確保結果可靠[33]。物種注釋的原則為:在種水平上,相似性閾值設定為不低于95%;最匹配序列只對應到單一植物物種,若該物種在當地有分布,則認定序列屬于該物種;最匹配序列對應到多個物種時,首先考慮排除在當地無分布的物種,若仍對應兩種以上,則在更高分類單元水平上(屬或科水平)對該序列進行分類注釋。
1.6 采食數據分析
進一步對注釋后的植物類群進行篩選與整理,從OTU表格中剔除真菌及動物自身的干擾序列,去除占比小于0.1%的序列,統計每份樣品中食物種類及其對應的序列條數,然后進行采食組分分析。用出現頻率(Frequency of occurrence,FO)和相對序列豐度(Relative read abundance,RRA)兩個指標估計灘羊羔羊的食性構成[34]。
FO是含某一植物類別的樣品數占總樣品數的百分比,計算公式如下:
式中,N表示出現i類食物的樣品數,N是有效樣品總數。當一份樣品中存在一種以上食物時,全部食物類別的FO之和大于100%。
RRA是某一植物類別的序列數在該樣品總植物序列的百分比,反映了相對生物量,計算公式如下:
式中,T是植物類別數,N是有效樣品總數,S是植物類別i在樣品j中的序列數。由公式可知,全部食物類別的RRA之和為100%。
2 結果與分析
2.1 原始測序數據的數據量和測序質量
本研究從人工草場放牧組、限時放牧補飼組、舍飼組三個處理組中共采集灘羊糞便樣品15份,ITS2目標區域全部擴增成功。15份寧夏灘羊糞便樣品PCR產物高通量測序結果表明,所有樣品均擴增到有效的食性數據。通過對原始測序數據進行過濾、質控之后,共收集到1 401 086條有效ITS2基因序列(valid sequences),每份樣品中含有的平均序列數為93 406條(70 046~121 613條),序列平均長度為315 bp。
本試驗中,在97%相似度水平進行聚類分析共獲得835個OTUs。采用RDP classifier貝葉斯算法對OTU有效代表序列進行分類學統計分析,然后在各個水平分析灘羊糞便中物種多樣性和豐度指數等主要指標。通過對Good’s coverage指數分析,數據顯示各個樣本的測序覆蓋率均超過了99%以上,說明本次研究測序的ITS2有效序列代表了各個樣本99.9%以上的物種種類,足以覆蓋灘羊糞便中飼糧情況。
2.2 基于OTU水平的多樣性分析
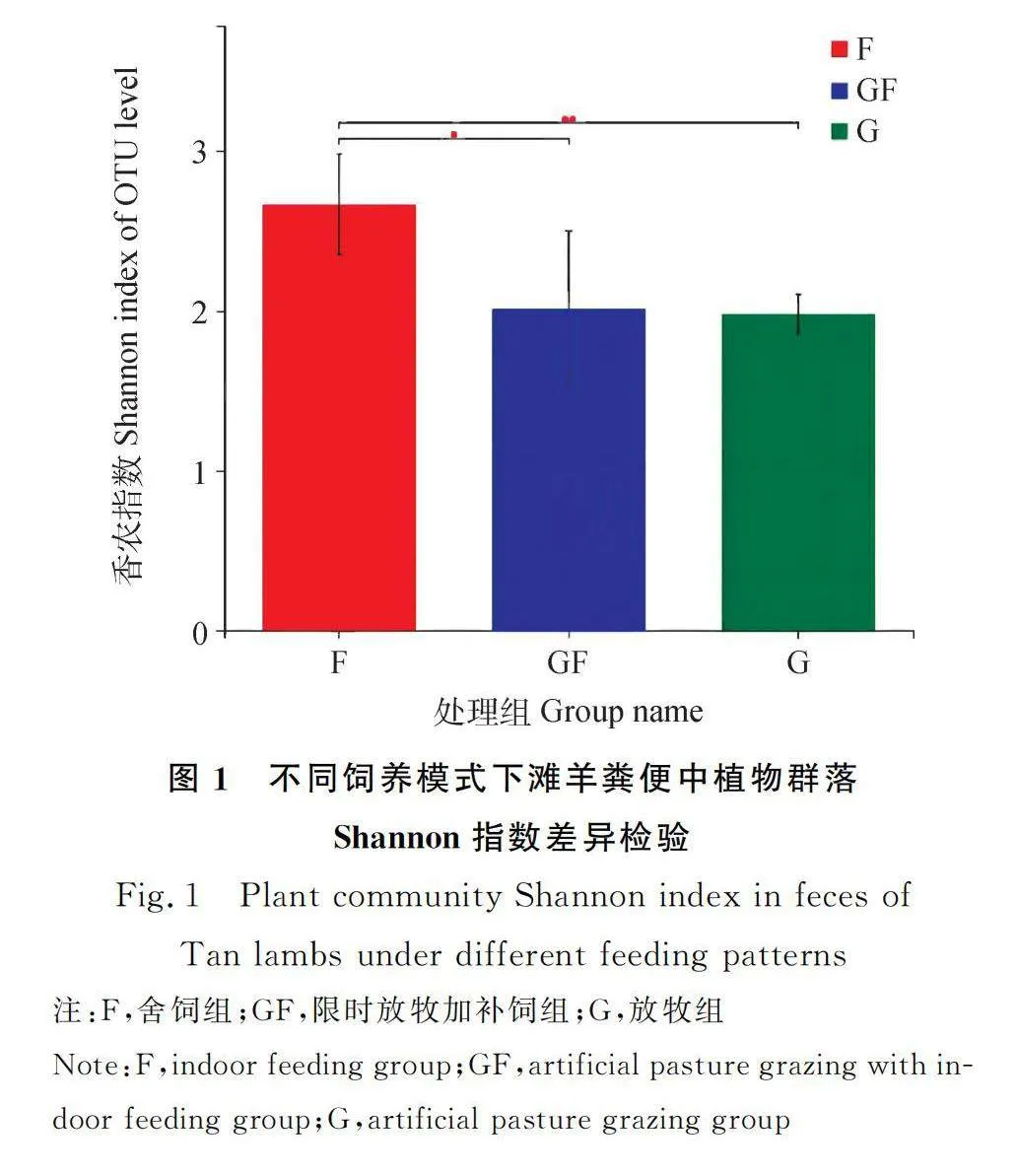
對舍飼對照組(F)、限時放牧補飼組(GF)與放牧組(G)糞便樣本中的植物群落豐富度及多樣性指數進行了比較分析(表2)。結果表明,三組樣本檢測到的OTU豐富度(Chao指數)沒有顯著差異。然而,三組糞便樣本之間的群落多樣性包括Shannon指數和Simpson指數具有顯著差異(P<0.01)。如圖1所示,通過Shannon指數組間差異分析發現,灘羊改變日糧組成后,糞便植物種群多樣性降低,放牧比舍飼的物種多樣性明顯減少(P<0.05)。
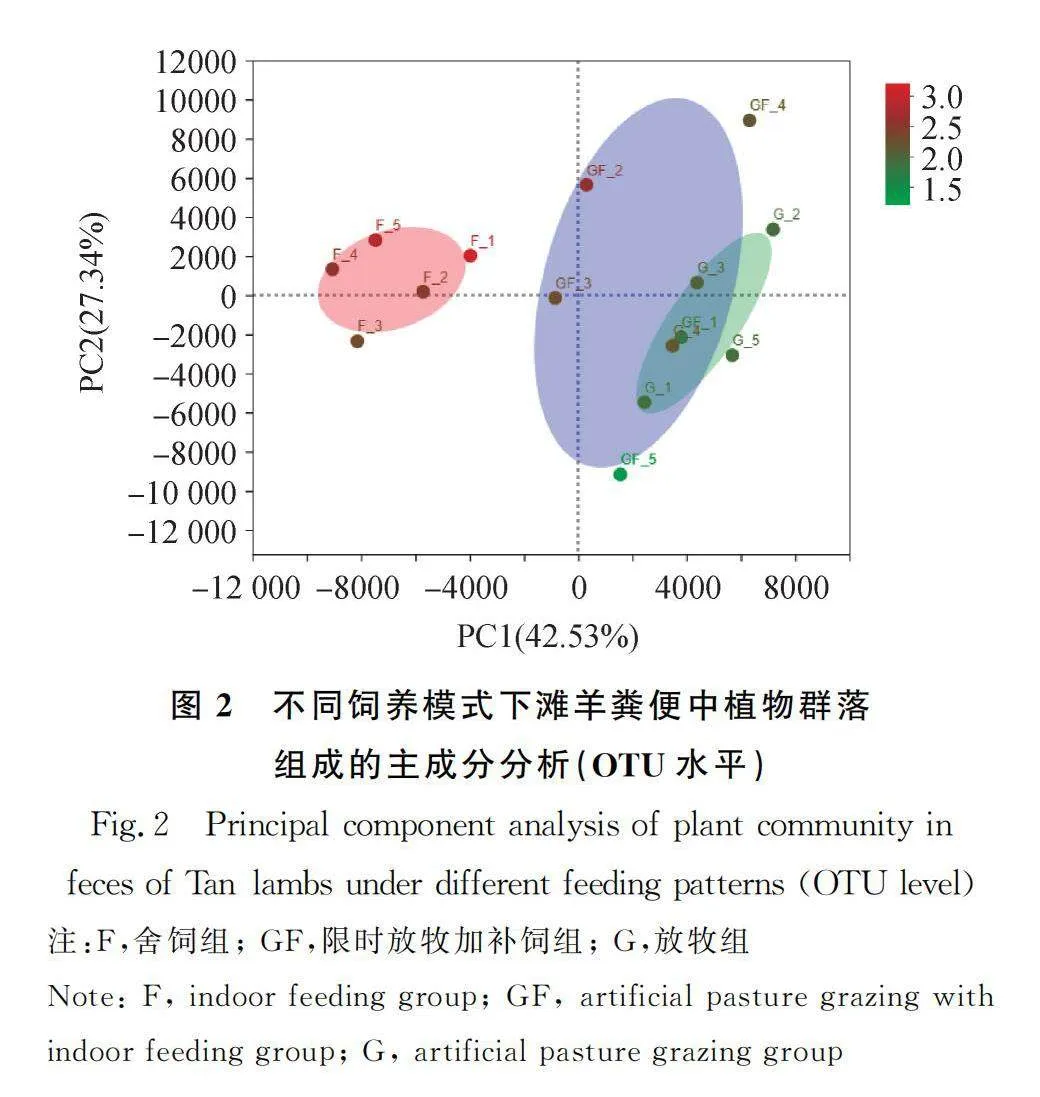
圖2不同顏色圖例分別代表不同飼養模式灘羊的糞便樣本。通過PCA分析發現,三個不同飼養模式灘羊的糞便樣本植物群落組成具有明顯差異,說明植物群落隨灘羊日糧組成的變化而呈明顯的梯度變化,舍飼組(F)和放牧組(GF和G)的群落組成具有明顯差異。PC1軸和PC2軸對結果的解釋度分別為42.53%和27.34%。
2.3 灘羊食物組成
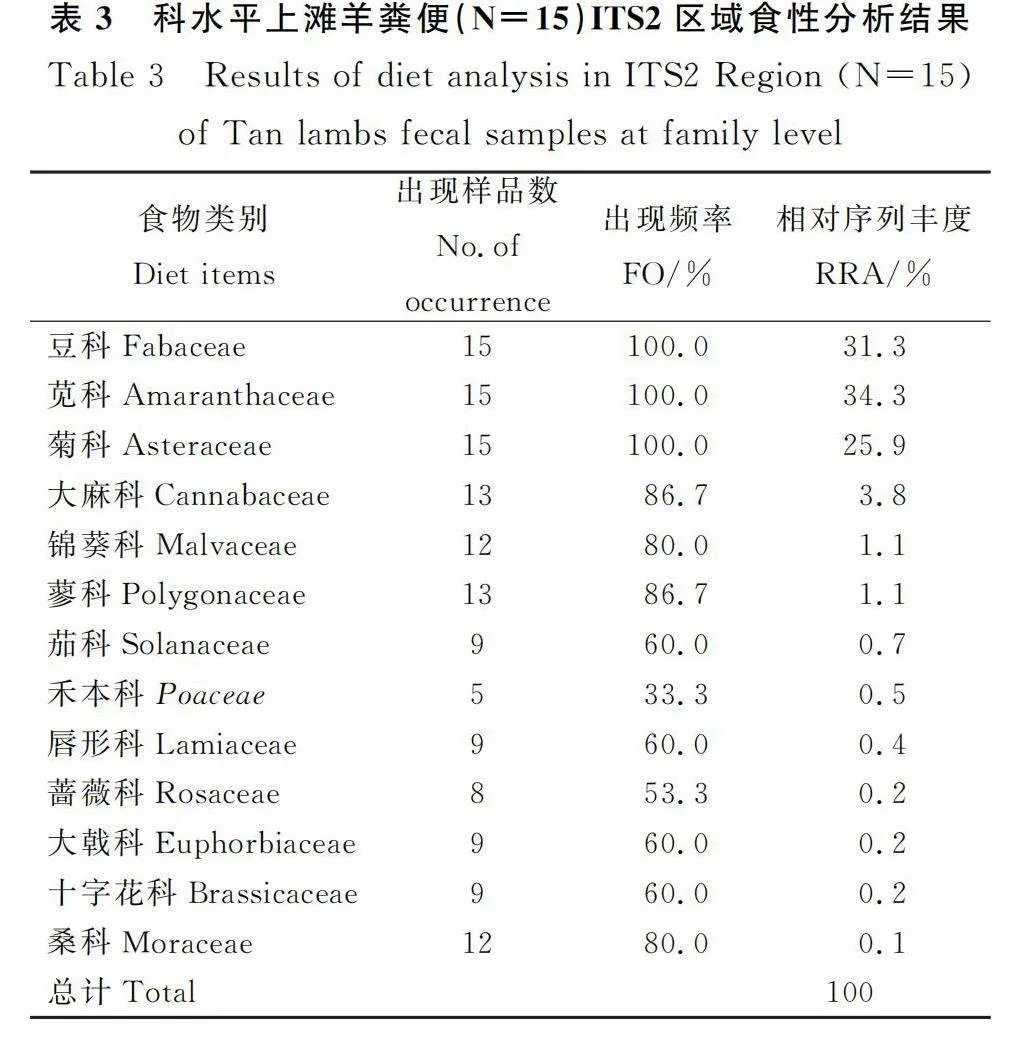
對15份糞便樣品中的ITS2有效食性數據進一步人工篩選和處理,去除序列占比小于0.1%的物種,并剔除真菌和動物自身的干擾序列,剩余有效序列注釋后共鑒定出的牧草隸屬于13科23屬28種不同的物種類別,其中27種鑒定到種水平,1種鑒定到屬水平(表3和表4)。13種科分別是豆科(Fabaceae)、莧科(Amaranthaceae)、菊科(Asteraceae)、大麻科(Cannabaceae)、錦葵科(Malvaceae)、蓼科(Polygonaceae)、茄科(Solanaceae)、禾本科(Poaceae)、唇形科(Lamiaceae)、薔薇科(Rosaceae)、大戟科(Euphorbiaceae)、十字花科(Brassicaceae)、桑科(Moraceae)。23種屬分別是藜屬(Chenopodium L.)、苜蓿屬(Medicago L.)、蒿屬(Artemisia L.)、向日葵屬(Helianthus L.)、驢食豆屬(Onobrychis L.)、大麻屬(Cannabis L.)、莧屬(Amaranthus L.)、棉屬(Gossypium L.)、薊屬(Cirsium L.)、萹蓄屬(Polygonum L.)、茄屬(Solanum L.)、玉蜀黍屬(Zea L.)、甘草屬(Glycyrrhiza L.)、百里香屬(Thymus L.)、酸模屬(Rumex L.)、毛莓草屬(Sibbaldianthe L.)、大戟屬(Euphorbia L.)、天仙子屬(Hyoscyamus L.)、錦葵屬(Malva L.)、車軸草屬(Trifolium L.)、蕓薹屬(Brassica L.)、桑屬(Morus L.)、漏蘆屬(Rhaponticum L.)。它們在所有樣品中總序列數為350,542條,占全部序列的74.99%,在NCBI GenBank數據庫中的最高一致度(Identity)均為99%以上。
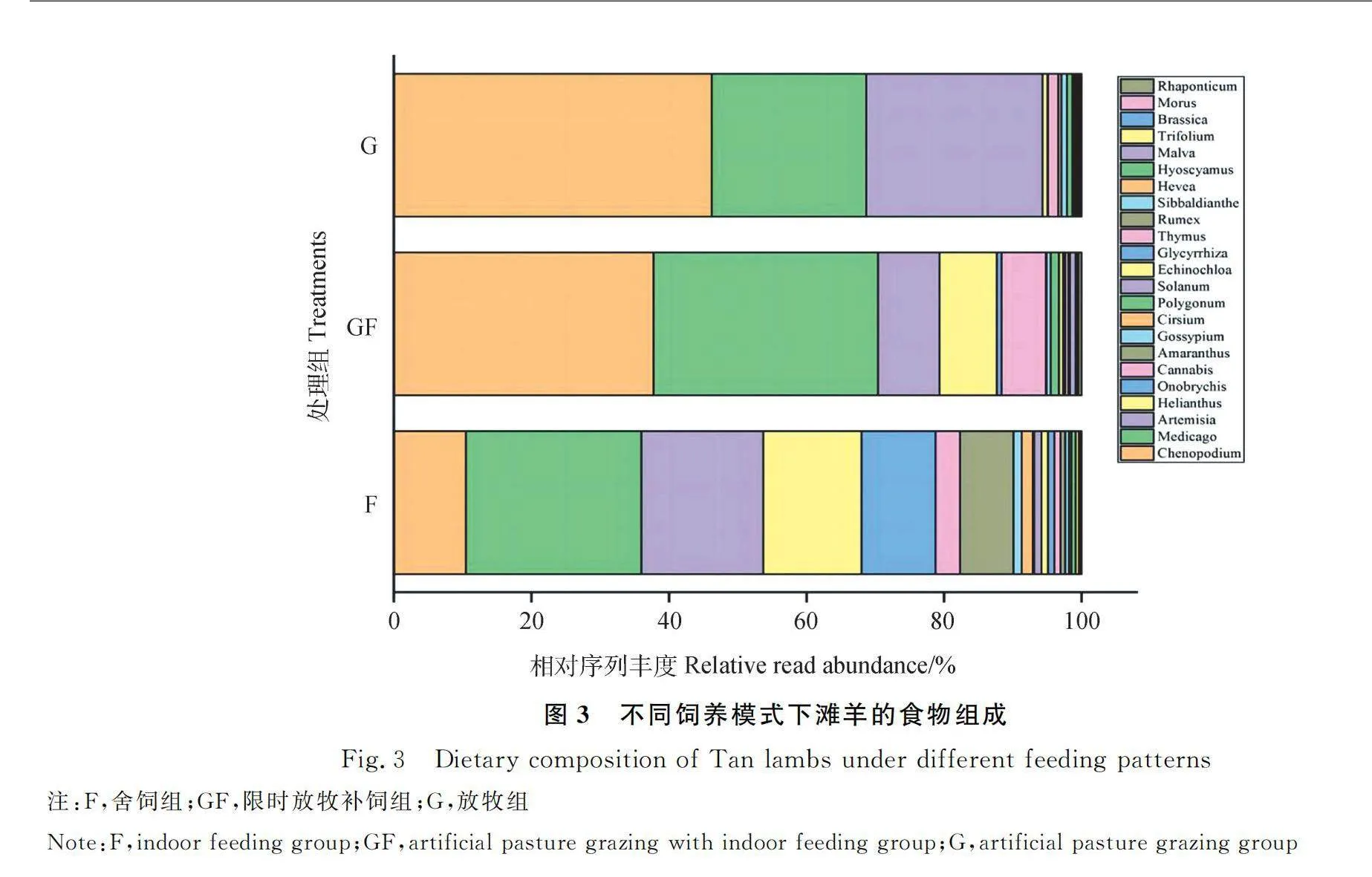
測序結果發現,藜屬和苜蓿屬是源于灘羊糞便中最主要的牧草類別,RRA分別為31.5%和26.9%。此外,蒿屬也在灘羊糞便中占有較高的比例,RRA為17.4%。同時,向日葵屬、驢食豆屬、大麻屬、莧屬等在灘羊糞便中占了較小的比例。各食物類別的FO和RRA存在顯著差異。圖3是舍飼組(F)、限時放牧補飼組(GF)和放牧組(G)灘羊糞便中的植物組成結果。可以看出,與舍飼組相較而言,放牧組的植物物種構成更為簡單。
3 討論與結論
3.1 ITS2片段用于物種鑒定
得益于分子生物學研究的進步,目前基于糞便DNA獲悉食物組成已成為食性研究領域的主流方法[35-36]。本研究使用植物ITS2基因作為識別植物物種的條形碼,共鑒定出28個植物類群(0.1%以上)。其中27個鑒定到物種水平,種水平鑒定成功率達到96%。僅鑒定到屬的1個類別為蒿屬,猜測該條形碼在蒿屬中的種間差異較小,物種分辨度有限,與在我國典型草原開展的蒙古綿羊食性鑒定研究結果類似[31]。說明本研究所用的ITS2區域通用引物rD5-ITS2/rb1-ITS2f并不是對所有植物類群都有效,因此需要對引物是否充分覆蓋研究區域潛在攝食物種進行深入研究。
豆禾混播草地主要組分為紫花苜蓿、草地雀麥和草地早熟禾。紫花苜蓿序列在灘羊糞便中的相對豐度為26.9%,位居第二,然而兩種禾本科牧草并沒有成功鑒定出來。原因可能是存在基因優先擴增現象,非禾本科植物更容易被優先擴增[30]。本次研究中禾本科鑒定成功率較低的另一個原因可能是糞便樣品保存不當引起的DNA降解。目前最新的基于糞便DNA的動物食性研究中,多采用兩步法進行樣品保存,即采集的新鮮糞便先經無水乙醇浸泡24 h,再加入硅膠置于-20℃保存[37-38]。經兩步法處理后,能從糞便中獲取更高濃度的DNA,PCR擴增實驗中的成功率也更高[39]。本研究采集的新鮮糞便樣品直接置于-20℃低溫保存,可能一定程度上會影響DNA提取和目標條帶的擴增效果,導致鑒定成功率降低。綜合目前研究,如需對家畜所采食種類進行更精確的判定,一方面通過優化保存方法避免樣品降解,另一方面需要考慮引入其他引物進行后續擴增及測序分析。此外,條形碼數據庫內物種序列信息的完備程度也將影響鑒定結果的準確性。
3.2 采食組成
以往關于動物尤其食肉動物的食性研究報道中,通常用應用食物出現頻率FO這一指標來定性描述動物的食性構成。本研究結果顯示,利用食物出現頻率FO和相對序列豐度RRA得到的食性結果差異較為明顯(表3和表4)。比如,桑屬植物其FO高達80.0%,而RRA僅為0.1%。這是由于FO反映的是某一食物組分出現的頻次,因此會增加生物量貢獻較低的食物類別所占的比例。而RRA利用食物擴增序列數與原始生物量組成相關的關系,反映了相對生物量,更加接近食物的實際生物量,對高通量食性分析提供了更多信息,潛在應用價值更大。
然而,從我們的研究結果中也發現,利用RRA描述家畜的食物組成存在一定誤差。就舍飼組而言,根據其日糧配方可知,苜蓿(苜蓿干草和苜蓿草粉)所占比例為35.0%,而通過高通量測序獲得的相對比例為25.5%,低估了9.5%。玉米(玉米秸稈和玉米顆粒)所占比例為40.25%,而測序后的相對比例僅為0.5%,低估了39.75%。分析原因,除了動物本身對不同屬種牧草的消化率存在差異外,還有一個誤差可能是因為ITS2條形碼的通用引物對于不同類群的牧草擴增效率不同。本研究團隊之前的動物實驗也發現,ITS2基因的通用引物更傾向于擴增非禾本科植物,從而會導致禾本科植物的相對比例被低估。建議可以從兩個方面繼續開展研究:首先,通過飼喂控制試驗構建定量校正模型,對目標牧草類群的估測比例進行二次校正,對采食組分估測結果加以修正完善。另外有研究證明,相比于單基因片段,復合條形碼組合能夠明顯提高物種的鑒別效果。因此,可以通過引入組合條形碼,例如ITS2 + trnH-psbA,matK + rbcL,提高對禾本科植物的鑒別效率。但是要面臨測序成本增加、測序數據更加復雜的挑戰。
綜上,本研究初步利用基于核基因ITS2片段的糞便DNA宏條形碼技術,對放牧灘羊的糞便進行高通量分子食性分析,表明該條形碼的物種分辨率高達96%,且驗證紫花苜蓿是灘羊的主要食物來源之一。未來可結合飽和烷烴法等經典方法,對測序結果加以分析與校正,為糞便DNA宏條形碼技術在家畜食性鑒定中的應用提供依據。
參考文獻
[1]SEVERUD W J,WINDELS S K,BELANT J L,et al. The role of forage availability on diet choice and body condition in American beavers (Castor canadensis)[J]. Mammalian Biology,2013,78(2):87-93
[2]KARTZINEL T R,CHEN P A,COVERDALE T C,et al. DNA metabarcoding illuminates dietary niche partitioning by African large herbivores[J]. Proceedings of the National Academy of Sciences of the United States of America,2015,112(26):8019-8024
[3]劉玉禎,趙新全,董全民,等. 放牧對草地生態系統結構與功能影響的研究進展[J]. 草地學報,2023,31(8):2253-2262
[4]丁路明,龍瑞軍,郭旭生,等. 放牧生態系統家畜牧食行為研究進展[J]. 家畜生態學報,2009,30(5):4-9
[5]汪詩平. 幾種草食動物日糧植物組成研究技術和方法的比較[J]. 草業學報,1995(3):8-16
[6]NORBURY G L,SANSON G D. Problems with measuring diet selection of terrestrial,mammalian herbivores[J]. Australian Journal of Ecology,1992,17(1):1-7
[7]崔占鴻. 放牧家畜采食量和擇食性測定方法的研究進展[J]. 家畜生態學報,2011,32(5):1-4
[8]ALI H A M,MAYES R W,HECTOR B L,et al. Assessment of n-alkanes,long-chain fatty alcohols and long-chain fatty acids as diet composition markers:The concentrations of these compounds in rangeland species from Sudan[J]. Animal Feed Science and Technology,2005,121(3-4):257-271
[9]LIN L J,ZHU X Y,JIANG C,et al. The potential use of n-alkanes,long-chain alcohols and long-chain fatty acids as diet composition markers:Indoor validation with sheep and herbage species from the rangeland of Inner Mongolia of China[J]. Animal,2012,6(3):449-458
[10]DOVE H,CHARMLEY E. Using the alkanes and long-chain alcohols of plant cuticular wax to estimate diet composition and the intakes of mixed forages in sheep consuming a known amount of alkane-labelled supplement[J]. Animal,2008,2(10):1474-1485
[11]LIN L J,LUO H L,ZHANG Y J,et al. The potential use of long-chain alcohols and fatty acids as diet composition markers:Factors influencing faecal recovery rates and diet composition estimates in sheep[J]. Animal,2009,3(11):1605-1612
[12]FERREIRA L M M,CELAYA R,SANTOS A S,et al. Evaluation of long-chain alcohols as diet composition markers in goats grazing heathland areas[J]. Animal,2012,6(4):683-692
[13]DOVE H,MAYES R W. Plant wax components:A new approach to estimating intake and diet composition in herbivores[J]. Journal of Nutrition,1996,126(1):13-26
[14]DOVE H,MAYES R W. Using n-alkanes and other plant wax components to estimate intake,digestibility and diet composition of grazing/browsing sheep and goats[J]. Small Ruminant Research,2005,59(2-3):123-139
[15]BEZABIH M,PELLIKAAN W F,TOLERA A,et al. Evaluation of n-alkanes and their carbon isotope enrichments as diet composition markers[J]. Animal,2011,5(1):57-66
[16]MARTíNEZ J M,MORRIS S T,PARKINSON T J. Estimation of herbage intake of Angus heifers from growth rate and milk production selection lines[J]. New Zealand Journal of Agricultural Research,2010,53(1):29-35
[17]POMPANON F,DEAGLE B E,SYMONDSON W O C,et al. Who is eating what:Diet assessment using next generation sequencing[J]. Molecular Ecology,2012,21(8):1931-1950
[18]TABERLET P,COISSAC E,POMPANON F,et al. Towards next-generation biodiversity assessment using DNA metabarcoding[J]. Molecular Ecology,2012,21(8):2045-2050
[19]MONTERROSO P,GODINHO R,OLIVEIRA T,et al. Feeding ecological knowledge:The underutilised power of faecal DNA approaches for carnivore diet analysis[J]. Mammal Review,2019,49(2):97-112
[20]郭艷萍,張浩,趙新鋼,等. DNA宏條形碼技術在食草動物食性研究中的應用[J]. 生物技術通報,2021,37(3):252-260
[21]ZHAO X Y,JIANG H M,XU N,et al. Molecular diet analysis of common cranes (Grus grus) under supplementary feeding based on fecal DNA metabarcoding[J]. Ecosphere,2023,14(8):e4631
[22]TRAUGOTT M,T380a13b097294811510cdf86ee4ee4beHALINGER B,WALLINGER C,et al. Fish as predators and prey:DNA-based assessment of their role in food webs[J]. Journal of Fish Biology,2021,98(2):367-382
[23]VISSER F,MERTEN V J,BAYER T. Deep-sea predator niche segregation revealed by combined cetacean biologging and eDNA analysis of cephalopod prey[J]. Science Advances,2021,7(14):eabf5908
[24]VAN DER REIS A L,LAROCHE O,JEFFS A G,et al. Preliminary analysis of New Zealand scampi (Metanephrops challengeri) diet using metabarcoding[J]. PEERJ,2018,6:e5641
[25]BROWN D S,JARMAN S N,SYMONDSON W O C. Pyrosequencing of prey DNA in reptile faeces:Analysis of earthworm consumption by slow worms[J]. Molecular Ecology Resources,2012,12(2):259-266
[26]CABODEVILLA X,GóMEZ-MOLINER B J,ABAD N,et al. Simultaneous analysis of the intestinal parasites and diet through eDNA metabarcoding[J]. Integrative Zoology,2022,18(3):399-413
[27]韓婷婷,商正昊妮,袁帥,等. 草甸草原春季不同生境東北鼢鼠(Myospalax psilurus)的食性特征及差異研究[J]. 草地學報,2024,32(10):3167-3175
[28]STAPLETON T E,WEINSTEIN S B,GREENHALGH R,et al. Successes and limitations of quantitative diet metabarcoding in a small,herbivorous mammal[J]. Molecular Ecology Resources,2022,22(7):2573-2586
[29]王月君. 人工草場不同飼養模式對灘羊羊肉脂肪酸及瘤胃微生物組成的影響[D]. 北京:中國農業大學,2020:3-15
[30]BRADLEY B J,STILLER M,DORAN-SHEEHY D M,et al. Plant DNA sequences from feces:Potential means for assessing diets of wild primates[J]. American Journal of Primatology,2007,69(6):699-705
[31]GUO Y P,ZHANG H,CHEN W Q,et al. Herbivore-diet analysis based on Illumina Miseq Sequencing:The potential use of an ITS2-barcoding approach to establish qualitative and quantitative predictions of diet composition of Mongolian sheep[J]. Journal of Agricultural and Food Chemistry,2018,66(37):9858-9867
[32]李貞,彭思嘉,左淑賢,等. 人工草場不同飼養模式對羔羊小腸脂肪消化和肝臟脂質代謝相關基因表達的影響[J]. 動物營養學報,2022,34(3):1920-1930
[33]DEAGLE B E,CHIARADIA A,MCINNES J,et al. Pyrosequencing faecal DNA to determine diet of little penguins:Is what goes in what comes out?[J] Conservation Genetics,2010,11(5):2039-2048
[34]DEAGLE B E,THOMAS A C,MCINNES J C,et al. Counting with DNA in metabarcoding studies:How should we convert sequence reads to dietary data?[J] Molecular Ecology,2019,28(2):391-406
[35]TRAN B T,KIM K Y,HEO J S,et al. Determination of the Pacific oyster Magallana gigas (Crassostrea gigas) diet composition in two aquaculture farms by fecal DNA metabarcoding[J]. Aquaculture,2022(552):738042
[36]ALBERDI A,RAZGOUR O,AIZPURUA O,et al. DNA metabarcoding and spatial modelling link diet diversification with distribution homogeneity in European bats[J]. Nature Communications,2020,11(1):1154
[37]SHAO X N,LU Q,LIU M Z,et al. Generalist carnivores can be effective biodiversity samplers of terrestrial vertebrates[J]. Frontiers in Ecology and the Environment,2021,19(10):557-563
[38]WANG Q Y,WANG Z C,ZHENG K D,et al. Assessing the diet of a predator using a DNA metabarcoding approach[J]. Frontiers in Ecology and Evolution,2022,10:902412
[39]NSUBUGA A M,ROBBINS M M,ROEDER A D,et al. Factors affecting the amount of genomic DNA extracted from ape faeces and the identification of an improved sample storage method[J]. Molecular Ecology,2004,13(7):2089-2094
(責任編輯 劉婷婷)

