基于16S rRNA測序分析敲除基因ZBED6后巴馬豬腸道微生物菌群的變化
摘 要: 旨在探究在巴馬豬中ZBED6基因敲除之后腸道菌群的變化和肌肉表型的關聯。本研究以5月齡野生型和ZBED6敲除型廣西巴馬小型豬為研究對象,按性別和基因型分成3組進行比較,分別是野生型公豬比野生型母豬(公WT:母WT)、野生型母豬比敲除型母豬(母WT:母KO)、野生型公豬比敲除型公豬(公WT:公KO),利用16S rRNA基因的高通量測序技術分析各腸段微生物組成。結果表明:1)在巴馬豬中,大腸的菌群多樣性顯著高于小腸;2)與雌性野生型豬相比,雄性野生型豬瘦肉率更高,回腸蘇黎世桿菌屬、盲腸鏈球菌屬、直腸消化鏈球菌屬在雄性野生型豬中顯著富集,回腸乳桿菌屬在雌性野生型豬中顯著富集;3)敲除ZBED6之后,豬的肌肉生長增加。腸道菌群組成顯示,母豬的回腸中乳桿菌含量顯著下降,盲腸中SMB53的含量顯著上升。敲除ZBED6后,公豬直腸中普雷沃菌屬和瘤胃球菌屬的含量顯著上升,盲腸中SMB53的含量顯著上升。結果提示,蘇黎世桿菌屬、鏈球菌屬、消化鏈球菌屬和乳桿菌屬在腸道內的差異可能會影響能量代謝從而促進雄性野生型豬的肌肉生長;敲除了ZBED6之后,豬的盲腸中SMB53豐度的顯著增加可能是導致肌肉增多的一個因素。
關鍵詞: 巴馬豬;ZBED6;腸道微生物;SMB53
中圖分類號:S828.2
文獻標志碼:A
文章編號:0366-6964(2024)10-4302-09
收稿日期:2024-03-15
基金項目:國家自然科學基金青年科學基金項目(32202621)
作者簡介:徐 成(1999-),男,江蘇無錫人,碩士,主要從事畜禽資源分子評價研究,E-mail: 1326843913@qq.com
*通信作者:王丹丹,主要從事功能基因挖掘與利用研究,E-mail:wangdd1993@126.com
Changes in Gut Microbiota after ZBED6 Knockout were Analyzed Based on 16S rRNA
Sequencing
XU" Cheng, TIAN" Wenjie, MA" Yuehui, WANG" Shengnan, JIANG" Lin, WANG" Dandan*
(Institute of Animal Science, Chinese Academy of Agricultural Sciences, Beijing 100193," China)
Abstract:" The aim of this study was to investigate the association between changes in gut microbiota and muscle phenotype in ZBED6-knockout Bama pigs. In this study, 5-month-old wild-type and ZBED6-knockout Guangxi Bama mini-pigs were divided into 3 groups based on sex and genotypes: wild-type boars compared with wild-type sows (male WT: female WT), wild-type sows compared with knock-out sows (female WT: female Ko), and wild-type boars compared with knock-out boars (male WT: male Ko). The microbial composition of each intestinal segment was analyzed by high-throughput sequencing of 16S rRNA gene. The results showed that: 1) The community diversity of large intestine in Bama pigs was significantly higher than that of small intestine; 2) Compared with female wild-type pigs, male wild-type pigs have higher lean meat percentage. Turicibacter in ileum, Streptococcus in cecum and Peptostreptococcaceae in rectum were found to be concentrated in male wild-type pigs, while Lactobacillus in ileum was found to be concentrated in female wild-type pigs; 3) After knockout of ZBED6, muscle growth increased in pigs. The content of Lactobacillus in ileum of sows was significantly decreased and SMB53 in cecum was significantly increased. After knockout of ZBDE6, Prevotella and Ruminococcus increased significantly in boar rectum, and SMB53 increased significantly in cecum. The results suggest that the differences in abundance of Turicibacter, Streptococcus, Peptostreptococcaceae and Lactobacillus in the gut may affect energy metabolism and promote muscle growth in male wild-type pigs; After ZBED6 knockout, an increase abundance in SMB53 in the cecum of pigs may be a factor contributing to muscle gain.
Key words: Bama pig; ZBED6; gut microbes; SMB53
*Corresponding author: WANG Dandan, E-mail:wangdd1993@126.com
豬是最重要的肉食來源之一,約4 000萬年前,豬科動物在歐洲被發現,家豬是由野豬多點馴化起源而來的[1]。肌肉一直是家畜育種最重要的性狀,腸道微生物的組成與多樣性有助于維持肌肉的正常生長和代謝能力。腸道微生物群可以影響骨骼肌的病理生理學,調節肌肉蛋白質沉積和肌肉功能,這種機制被稱為腸-肌軸[2-3]。如普雷沃菌屬(Prevotella)、丁酸單胞菌(Butyricimonas)和阿克曼菌屬(Akkermansia)等菌屬均影響肌肉的生長發育[4-6]。
榮昌豬和約克夏豬腸道微生物存在顯著差異。與大白豬相比,榮昌豬的瘦肉率更高。將2種豬的腸道微生物通過糞菌移植方式傳遞給無菌小鼠后,受體小鼠表現出與供體豬相似的代謝表型。與移植約克夏豬糞便的小鼠相比,移植榮昌豬糞便的小鼠體脂重具有增加的趨勢,肌肉中的慢肌纖維比例提高,腓腸肌肌纖維直徑和橫截面積具有減小的趨勢[7]。在兩項與衰老相關的肌肉力量損失的試驗中,研究人員給年輕的小鼠補充植物乳桿菌TWK10,不僅可以增強了年輕小鼠的肌肉力量,而且阻止了老年小鼠與衰老相關的肌肉力量損失[8]。另外一個試驗中作者添加了副干酪乳桿菌PS23,可以減緩小鼠衰老導致的肌肉質量和力量的喪失,維持肌肉中較高水平的活性氧和線粒體水平[9]。在過去幾十年,由于對家豬瘦肉性狀進行強烈的選擇,最終使得家豬的肌肉量增加和脂肪沉積減少。ZBED6(zinc finger BED domain-containing protein 6), 作為IGF2的轉錄抑制因子,通過抑制IGF2的表達抑制肌肉生長發育。ZBED6最初被發現在歐洲野豬、皮特蘭豬和大白豬之間的雜交中[10]。IGF2中一個單堿基突變消除了與ZBED6的結合,從而影響了豬肌肉生長、脂肪沉積和心臟大小;在進化過程中,ZBED6基因的2個DNA結合區域在26種胎盤哺乳動物中顯示出接近 100%的氨基酸保守性,證明ZBED6具有重要的功能[11-14]。Younis等[15]構建了敲除ZBED6小鼠模型,發現敲除ZBED6后IGF2的表達增高導致了肌肉的增多;Liu等[16]檢查了ZBED6單敲除小鼠的肌肉表型,發現敲除后提高了小鼠的瘦肉重、瘦肉率和肌纖維面積,表明ZBED6有劑量依賴效應;Wang等[17]利用CRISPR/Cas9技術獲得ZBED6敲除巴馬豬模型,與小鼠的表型一致,ZBED6敲除巴馬豬肌肉含量增大,瘦肉率增多。近年來,ZBED6還被發現和胰島功能、心肌發育等有關[18-19]。但是ZBED6是否能介導豬腸道微生物的改變和肌肉表型的關聯還不得而知。
1 材料與方法
1.1 試驗材料
ZBED6-KO巴馬小型豬由本團隊前期通過CRISPR/Cas9技術獲得,目前飼養在中國科學院動物研究所附屬養豬場。在整個試驗期間,這些豬可以自由地獲得商業豬飼料和水。選取野生型5月齡公豬3頭、ZBED6敲除型5月齡公豬3頭、野生型5月齡母豬5頭和ZBED6敲除型5月齡母豬7頭共18頭,對試驗豬進行安樂死和屠宰,瘦肉從左側屠體解剖和稱重。瘦肉率由左側胴體瘦肉重與胴體重的比值決定。收集豬小腸(十二指腸、空腸、回腸)、大腸(盲腸、結腸、直腸)組織進行取樣,一份樣品先于液氮中速凍,后將樣品置于-80 ℃長期保存。基因敲除豬的制備如前文所述[17]。
1.2 DNA 抽提與PCR擴增
根據E.Z.N.A.? soil試劑盒說明書進行總DNA抽提,DNA濃度和純度利用NanoDrop2000進行檢測,利用1%瓊脂糖凝膠電泳檢測DNA提取質量;用338F(5′-ACTCCTACGGGAGGCAGCAG-3′)和806R(5′-GGACTACHVGGGTWTCTAAT-3′)引物對V3-V4可變區進行PCR擴增。
1.3 文庫構建與Illumina Miseq 測序
使用2%瓊脂糖凝膠回收PCR產物,利用AxyPrep DNA Gel Extraction Kit (Axygen Biosciences, Union City, CA, USA)進行純化,Tris-HCl洗脫,2%瓊脂糖電泳檢測。利用QuantiFluorTM-ST (Promega, USA)進行檢測定量。根據Illumina MiSeq平臺(Illumina, San D-iego,USA)標準操作規程將純化后的擴增片段構建PE 2*300的文庫。構建文庫步驟: 1)連接“Y”字形接頭; 2)使用磁珠篩選去除接頭自連片段; 3)利用PCR擴增進行文庫模板的富集; 4)氫氧化鈉變性, 產生單鏈DNA片段。利用Illumina公司的Miseq PE300平臺進行測序。
1.4 數據質控與物種注釋
分析過程主要是用Qiime2軟件完成的。用qiime tools import 插件,將原始序列fastq文件,導入為可進行QIIME2后續處理的文件格式。然后運用QIIME2 dada2插件進行質控,修剪,去噪,拼接,以及去除嵌合體后,得到了最終的特征序列表格。接著,運用QIIME2 feature-classifier插件將ASV的代表序列比對到預先訓練好的13_8版本99%相似度的GREENGENES數據庫(根據338F/806R引物對將數據庫修剪到V3-V4的區域),得到了物種的分類信息表。之后用QIIME2 feature-table插件剔除了所有污染性的線粒體和葉綠體序列[20-22]。
1.5 差異分析
ANCOM、ANOVA、Kruskal Wallis、LEfSe和DEseq2等方法被用于鑒定分組和樣本間豐度有差異的細菌[23]。
1.6 多樣性分析
使用QIIME2 core-diversity插件計算多樣性矩陣。特征序列水平Alpha多樣性指數包括observed OTUs、 Chao1、香農指數和Faith’s phylogenetics diversity指數被用于評估樣本本身的多樣性程度。Beta多樣性指數包括Bray Curtis, unweighted UniFrac and weighted UniFrac指數被用于評估樣本之間的微生物群落結構差異性,并隨后用PCoA和NMDS 圖來進行展示。應用R軟件包“mixOmics”,偏最小二乘判別分析(PLS-DA)作為一種有監督的判別分析統計方法,被用于揭示微生物群落與樣品類別之間的關系模型來實現對樣品類別的預測[24-25]。
2 結 果
2.1 測序結果分析
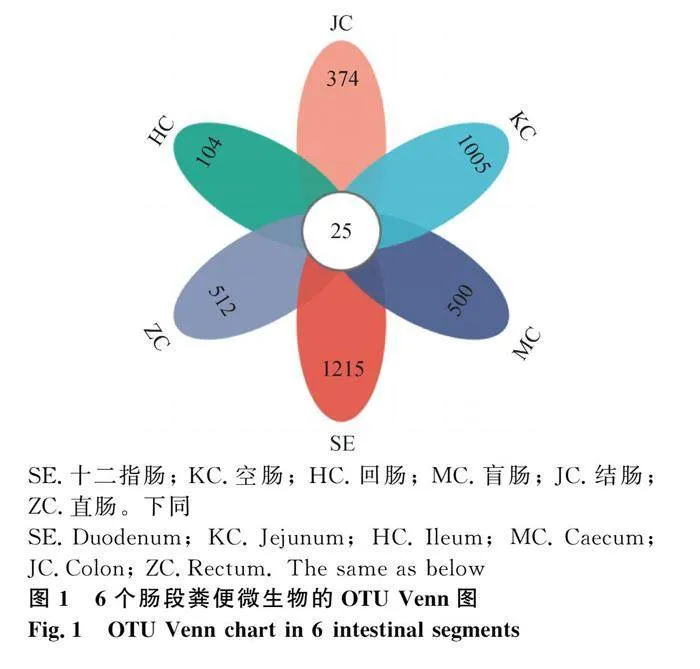
本研究通過16S rRNA的V3-V4測序評估了十二指腸(SE)、空腸(KC)、回腸(HC)、盲腸(MC)、結腸(JC)和直腸(ZC)共6個腸段中的腸道微生物群結構。在108個樣本中檢測到的6 373 728個讀數被DADA2軟件注釋為6 518個OTUs。對6個腸段注釋到的微生物進行交集分析,結果顯示有25個OTUs在6個腸段中都有豐度(圖1)。
2.2 Alpha多樣性分析和Beta多樣性分析
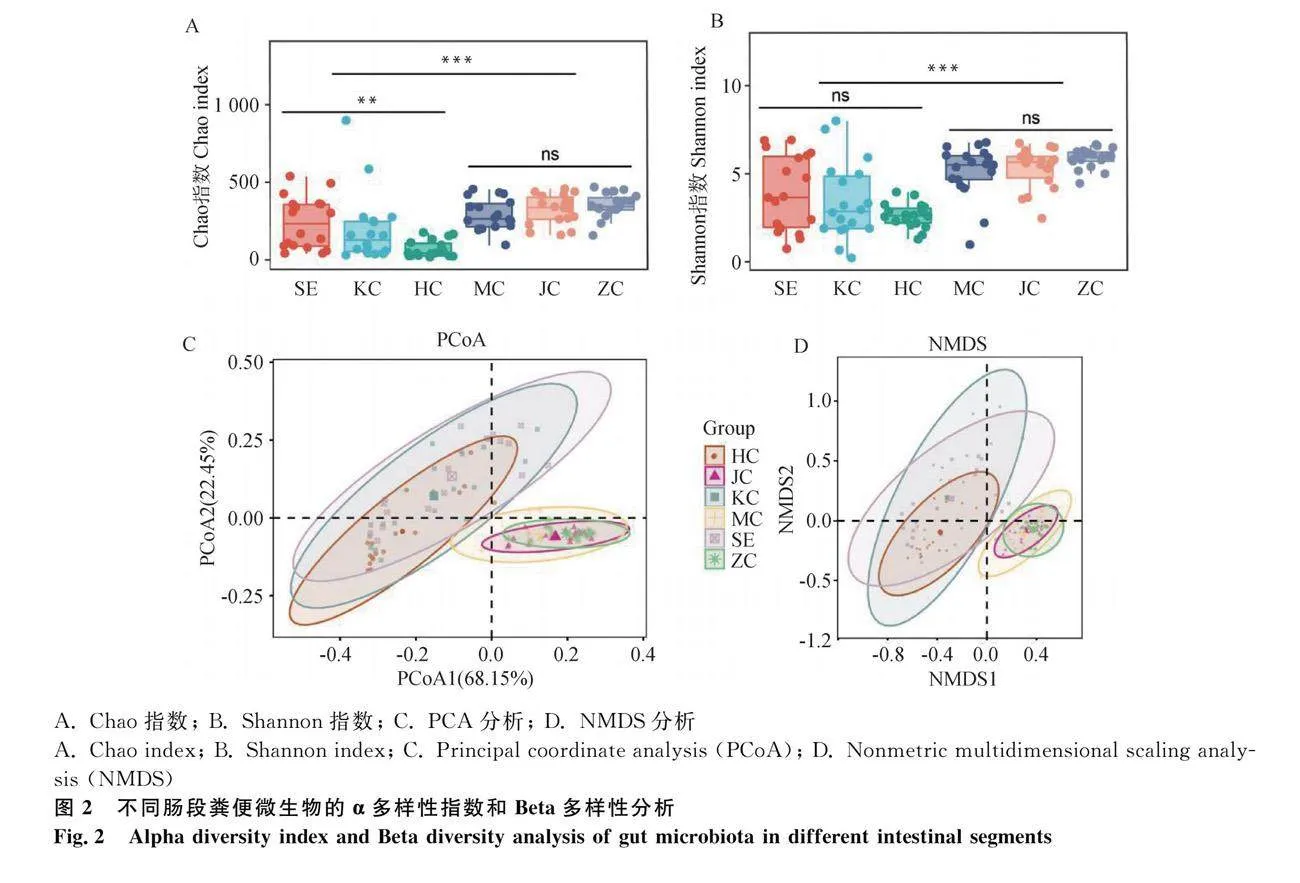
Chao1指數可反映樣品微生物的豐富度,估計群落中實際存在的物種數,Shannon指數可反映樣品微生物的多樣性。結果與OTU數量一致,大腸群落多樣性顯著高于小腸(圖2A、B)。PCA分析是基于屬水平的群落組成結構進行的分析,進化中聚類相近的物種距離越近。結果表明,大腸(盲腸、結腸、直腸)微生物聚類效果類似,小腸(十二直腸、空腸、回腸)微生物聚類效果類似(圖2C)。使用R對基于UniFrac距離矩陣分別進行NMDS分析,通過二維排序圖描述群落樣本的結構分布,NMDS分析也得出類似的結論(圖 2D)。
2.3 分類組成分析
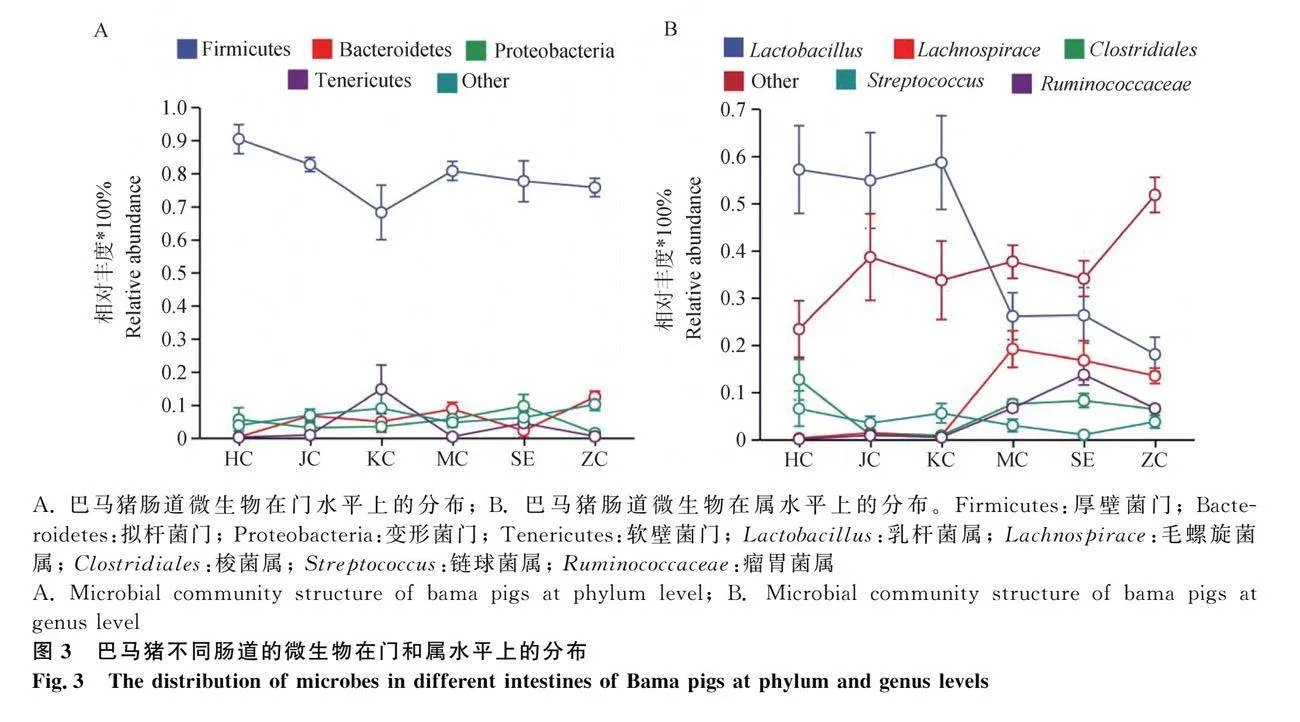
在門和屬分類水平上對樣品進行分類學統計。巴馬豬大腸細菌門以厚壁菌門(盲腸81%,結腸83%,直腸76%)和擬桿菌門(盲腸9%,結腸7%,直腸12%)為優勢物種,變形菌門(盲腸6%,結腸3%,直腸1%)次之。在小腸中,厚壁菌門和變形菌門占優勢,其中厚壁菌門(十二指腸占79%,空腸占69%,回腸占91%)的富集程度最高,變形菌門(十二指腸占9%,空腸占3%,回腸占5%)的富集程度相對較高。但是,擬桿菌只表現出相對豐度(十二指腸2%,空腸5%,回腸0%)(圖3A)。在屬水平上,乳酸桿菌在大腸和小腸中的相對豐度分別為24% 和59% ;毛螺旋菌在大腸和小腸中的相對豐度分別為16%和1%;瘤胃球菌在大腸和小腸中的相對豐度中分別為9%和0.3% (圖3B)。
2.4 野生型巴馬豬公母表型差異和菌群組成的關系
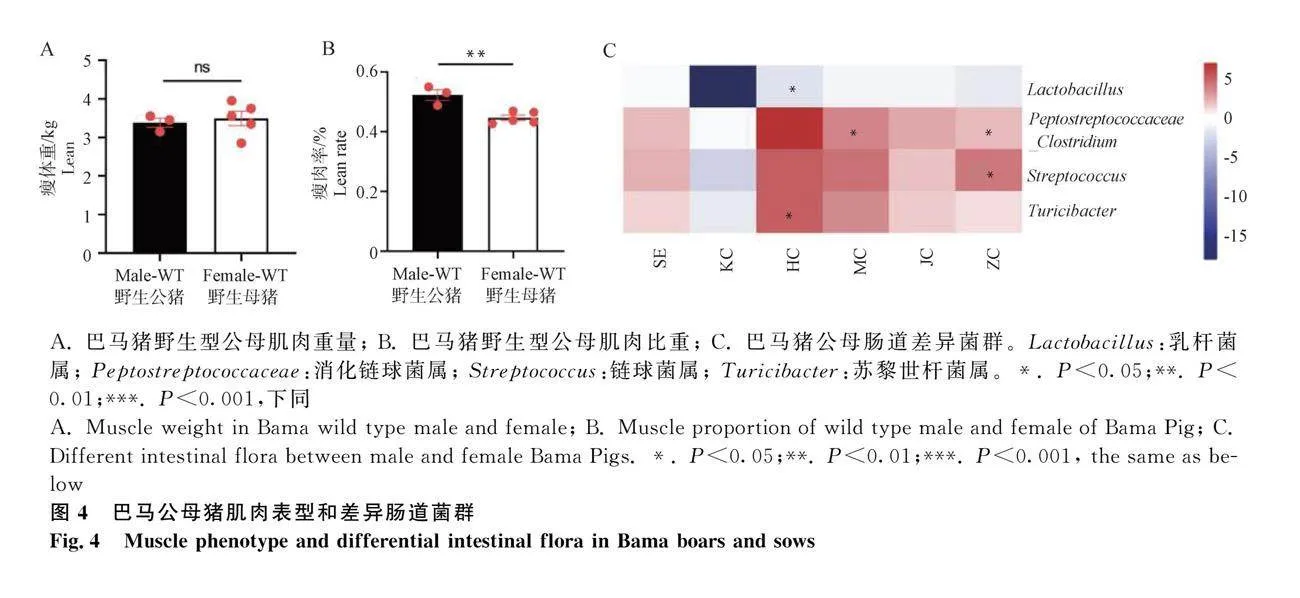
從解剖的表型來看,野生型公豬的肌肉占比顯著高于野生型母豬(圖4 A、B)。將所有樣品分成三組進行比較,找出與表型相關聯的差異菌群。三組分別是(公WT:母WT)、(公WT:公KO)、(母WT:母KO)。對野生型公母豬菌群分類組成進行差異分析,檢測到8個微生物至少在一個腸段中顯著差異(但是只有4個是豐度比較高的);它們分別是蘇黎世桿菌屬(Turicibacter)、鏈球菌屬(Streptococcus)、消化鏈球菌屬(Peptostreptococcaceae)和乳桿菌屬(Lactobacillus)。如圖4C所示,蘇黎世桿菌屬(Turicibacter)在公豬回腸中高豐度,乳桿菌屬(Lactobacillus)在公豬回腸低豐度。鏈球菌屬(Streptococcus)和消化鏈球菌屬(Peptostreptococcaceae)在公豬盲腸、直腸中高豐度。
2.5 基因敲除之后巴馬豬表型差異和菌群組成的關系
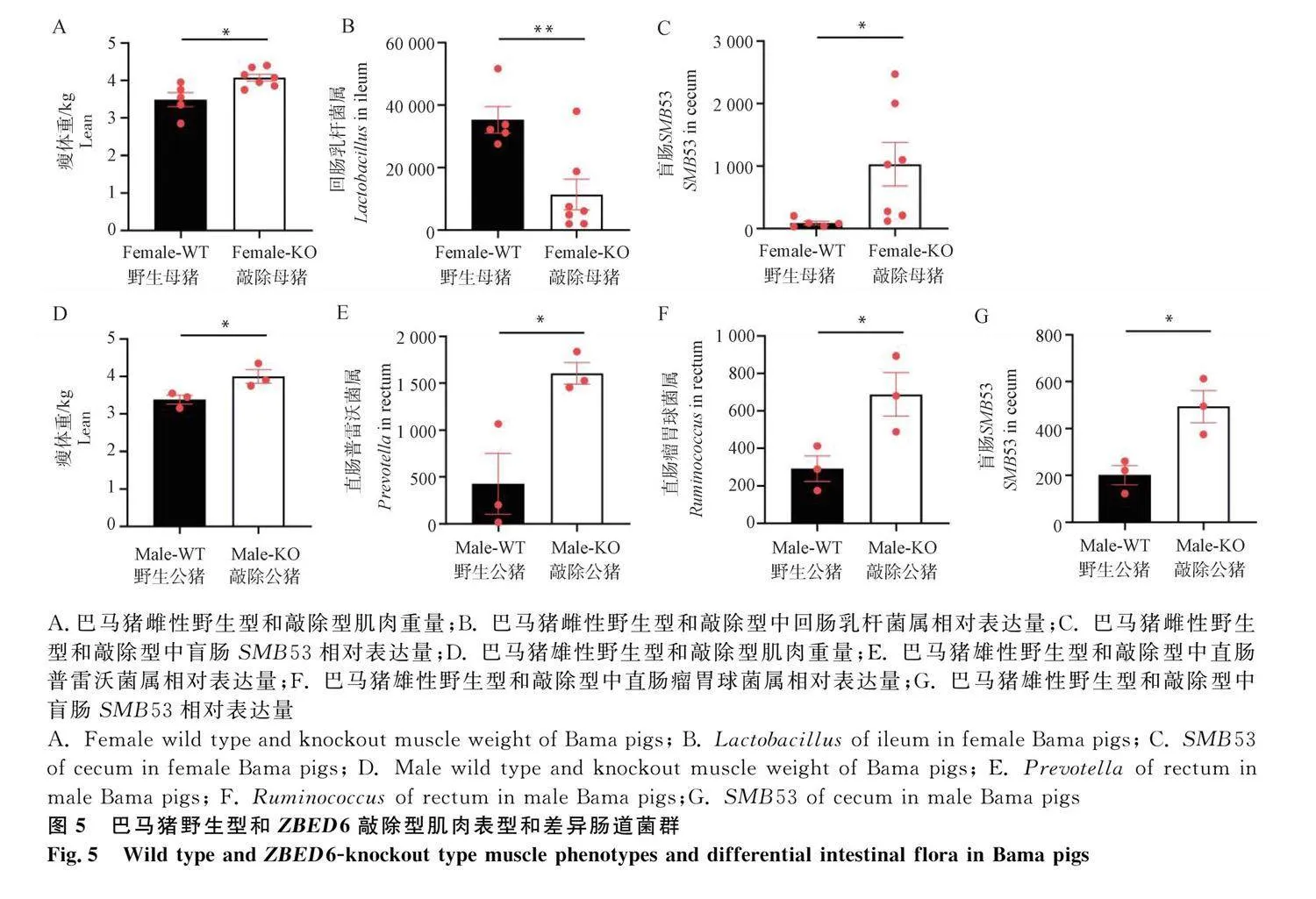
敲除ZBED6后,肌肉生長增加(圖5A、D)[17]。雌性WT豬和雌性KO豬之間有5種不同的OTU,但只有乳桿菌(Lactobacillus)和SMB53顯示高豐度。細菌組成顯示,乳桿菌(Lactobacillus)在雌性WT豬回腸中富集,而SMB53在雌性KO豬的盲腸中富集(圖5B、C)。雄性WT豬和KO豬之間有8種不同的OTU,但只有普雷沃菌屬(Prevotella)、瘤胃球菌屬(Ruminococcus)和SMB53顯示較高的豐度。普雷沃菌屬(Prevotella)和瘤胃球菌屬(Ruminococcus)在直腸中富集(圖5E、F),SMB53在KO豬盲腸中富集(圖5G)。
3 討 論
哺乳動物的腸道是一個密集的、動態的、高度復雜的微生物群落,不同的菌群結構能直接或間接的影響表型。在本研究中,利用前期構建的敲除ZBED6巴馬小型豬模型,對其不同腸段進行了16S rRNA基因測序。結果表明,與人類和小鼠一樣,巴馬小型豬的腸道微生物群也以厚壁菌門和擬桿菌門為主[26]。與野生型母豬相比,公豬瘦肉率較高,其中蘇黎世桿菌屬(Turicibacter)在野生型公豬回腸中顯示高豐度。蘇黎世桿菌屬(Turicibacter)是一種革蘭陽性厭氧菌,能利用可發酵碳水化合物產生大量乳酸[27]。在嚙齒類動物小腸和人類糞便微生物群中,蘇黎世桿菌屬(Turicibacter)可達到20%[28]和0.5%[29]的相對豐度。一項研究運動與腸道菌群相關性的試驗對C57BL/6J小鼠腸道微生物組進行了分析,發現在隨意輪流運動6周后,直腸和糞便中的蘇黎世桿菌屬(Turicibacter)減少[30]。
此外,豬只進食生馬鈴薯淀粉會增加大腸內的蘇黎世桿菌屬(Turicibacter)[31]。說明蘇黎世桿菌屬(Turicibacter)可能會影響采食進而影響肌肉表型。因此推測,與野生型母豬相比,公豬可能是由于腸道高豐度的蘇黎世桿菌屬(Turicibacter)表現出更高的瘦肉率。乳桿菌(Lactobacillus)在調節動物免疫、維持細菌穩態和身體健康、輔助消化、提高生長豬生長速度等方面的有重要作用[32-33]。有研究表明乳桿菌(Lactobacillus)能顧通過腸道-肌肉軸調節與年齡相關的肌肉損傷的發生和進展[34]。乳桿菌(Lactobacillus)在回腸中富集,與野生型母豬相比,野生型公豬中的乳桿菌(Lactobacillus)顯著減少;敲除型母豬中的乳桿菌(Lactobacillus)顯著減少,表明乳桿菌(Lactobacillus)的減少對應著肌肉量的增加,暗示乳桿菌(Lactobacillus)可能通過改變代謝從而影響肌肉表型。敲除ZBED6之后,公豬直腸中普雷沃菌屬(Prevotella)、瘤胃球菌屬(Ruminococcus)豐度增加,這兩種菌通常作為膳食纖維的降解者[35],普雷沃菌屬(Prevotella)可增強多糖的發酵,是阿拉伯木聚糖和低聚果糖中丙酸短鏈脂肪酸的高級生產者[36],公豬直腸中的高豐度可能增強肌肉對糖類的吸收。SMB53作為野生型公母豬和敲除型公母豬共有的差異菌,顯著富集于小腸。SMB53屬于梭菌科,這個家族的大多數成員有能力消耗腸道黏液和植物來源的糖類如葡萄糖[37-38]。在豬成長過程中,SMB53先增加后減少[39],在一項豬基因組和腸道微生物關系的研究中,發現MALRD1基因的內含子區域有一個SNP和SMB53有很強的關聯性,MALRD1基因參與膽汁酸合成調控,能夠修飾腸道微生物,ZBED6作為轉錄因子,也可能存在修飾腸道微生物的可能[40]。也有文獻報道發現2型糖尿病小鼠盲腸中SMB53富集,與糖脂肪代謝密不可分[41]。在一項減重手術的研究中發現,SMB53和體重減輕呈正相關[42]。總之,本研究表明,ZBED6不僅僅作為轉錄因子,從體內調控肌肉和脂肪的改變,同時也影響著腸道菌群,為轉基因育種提供了新思路。
4 結 論
本研究采用微生物16S rRNA基因高通量測序,對野生型廣西巴馬小型豬和ZBED6敲除型廣西巴馬小型豬的腸道微生物的組成變化進行分析。結果發現:敲除ZBED6后,母豬的回腸中乳桿菌(Lactobacillus)含量下降,盲腸中SMB53的含量上升;敲除ZBED6后,公豬的直腸中普雷沃菌屬(Prevotella)和瘤胃球菌屬(Ruminococcus)的含量上升,盲腸中SMB53的含量上升。敲除了ZBED6之后,增加了公母豬的盲腸中SMB53的豐度,可能是導致肌肉增多的一個因素。本研究將瘦肉型轉基因豬和腸道微生物相關聯,為優質性狀的選育提供了參考。
參考文獻(References):
[1] LARSON G,DOBNEY K,ALBARELLA U,et al.Worldwide phylogeography of wild boar reveals multiple centers of pig domestication[J].Science,2005,307(5715):1618-1621.
[2] GROSICKI G J,FIELDING R A,LUSTGARTEN M S.Gut microbiota contribute to age-related changes in skeletal muscle size,composition,and function:biological basis for a gut-muscle axis[J].Calcif Tissue Int,2018,102(4):433-442.
[3] TICINESI A,LAURETANI F,MILANI C,et al.Aging gut microbiota at the cross-road between nutrition,physical frailty,and sarcopenia:is there a gut-muscle axis?[J].Nutrients,2017,9(12):1303.
[4] PETERSEN L M,BAUTISTA E J,NGUYEN H,et al.Community characteristics of the gut microbiomes of competitive cyclists[J].Microbiome,2017,5(1):98.
[5] LIU Z H,LIU H Y,ZHOU H B,et al.Moderate-intensity exercise affects gut microbiome composition and influences cardiac function in myocardial infarction mice[J].Front Microbiol,2017,8:1687.
[6] LAMOUREUX E V,GRANDY S A,LANGILLE M G I.Moderate exercise has limited but distinguishable effects on the mouse microbiome[J].mSystems,2017,2(4):e00006-17.
[7] 嚴鴻林.腸道微生物及其與營養互作對豬骨骼肌表型及代謝的調控[D].成都:四川農業大學,2018.
YAN H L.Regulation of porcine skeletal muscle-phenotypes and metabolism by gut microbiota and its interaction with nutrition[D].Chengdu:Sichuan Agricultural University,2018.
[8] LEE C C,LIAO Y C,LEE M C,et al.Lactobacillus plantarum TWK10 attenuates aging-associated muscle weakness,bone loss,and cognitive impairment by modulating the gut microbiome in mice[J].Front Nutr,2021,8:708096.
[9] LEE M C,HO C S,HSU Y J,et al.Live and heat-killed probiotic Lactobacillus paracasei PS23 accelerated the improvement and recovery of strength and damage biomarkers after exercise-induced muscle damage[J].Nutrients,2022,14(21):4563.
[10] JEON J T,CARLBORG ?,T?RNSTEN A,et al.A paternally expressed QTL affecting skeletal and cardiac muscle mass in pigs maps to the IGF2 locus[J].Nat Genet,1999,21(2):157-158.
[11] VAN LAERE A S,NGUYEN M,BRAUNSCHWEIG M,et al.A regulatory mutation in IGF2 causes a major QTL effect on muscle growth in the pig[J].Nature,2003,425(6960):832-836.
[12] JUNGERIUS B J,VAN LAERE A S,TE PAS M F W,et al.The IGF2-intron3-G3072A substitution explains a major imprinted QTL effect on backfat thickness in a Meishan×European white pig intercross[J].Genet Res,2004,84(2):95-101.
[13] MARKLJUNG E,JIANG L,JAFFE J D,et al.ZBED6,a novel transcription factor derived from a domesticated DNA transposon regulates IGF2 expression and muscle growth[J].PLoS Biol,2009,7(12):e1000256.
[14] HAYWARD A,GHAZAL A,ANDERSSON G,et al.ZBED evolution:repeated utilization of DNA transposons as regulators of diverse host functions[J].PLoS One,2013,8(3):e59940.
[15] YOUNIS S,SCH?NKE M,MASSART J,et al.The ZBED6-IGF2 axis has a major effect on growth of skeletal muscle and internal organs in placental mammals[J].Proc Natl Acad Sci U S A,2018,115(9):E2048-E2057.
[16] LIU L,WANG S N,TIAN W J,et al.Effect of ZBED6 single-allele knockout on the growth and development of skeletal muscle in mice[J].Biology (Basel),2023,12(2):325.
[17] WANG D D,PAN D K,XIE B C,et al.Porcine ZBED6 regulates growth of skeletal muscle and internal organs via multiple targets[J].PLoS Genet,2021,17(10):e1009862.
[18] WANG X,JIANG L,WALLERMAN O,et al.Transcription factor ZBED6 affects gene expression,proliferation,and cell death in pancreatic beta cells[J].Proc Natl Acad Sci U S A,2013,110(40):15997-16002.
[19] WANG S N,TIAN W J,PAN D K,et al.A comprehensive analysis of the myocardial transcriptome in ZBED6-Knockout Bama Xiang Pigs[J].Genes (Basel),2022,13(8):1382.
[20] BOKULICH N A,KAEHLER B D,RIDEOUT J R,et al.Optimizing taxonomic classification of marker-gene amplicon sequences with QIIME 2′s q2-feature-classifier plugin[J].Microbiome,2018,6(1):90.
[21] CALLAHAN B J,MCMURDIE P J,ROSEN M J,et al.DADA2:high-resolution sample inference from Illumina amplicon data[J].Nat Methods,2016,13(7):581-583.
[22] DIXON P.VEGAN,a package of R functions for community ecology[J].J Veg Sci,2003,14(6):927-930.
[23] LOVE M I,HUBER W,ANDERS S.Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2[J].Genome Biol,2014,15(12):550.
[24] MANDAL S,VAN TREUREN W,WHITE R A,et al.Analysis of composition of microbiomes:a novel method for studying microbial composition[J].Microb Ecol Health Dis,2015,26:27663.
[25] ROHART F,GAUTIER B,SINGH A,et al.mixOmics:an R package for ′omics feature selection and multiple data integration[J].PLoS Comput Biol,2017,13(11):e1005752.
[26] MO J L,GAO L,ZHANG N,et al.Structural and quantitative alterations of gut microbiota in experimental small bowel obstruction[J].PLoS One,2021,16(8):e0255651.
[27] BROWNE H P,FORSTER S C,ANONYE B O,et al.Culturing of ′unculturable′ human microbiota reveals novel taxa and extensive sporulation[J].Nature,2016,533(7604):543-546.
[28] LYNCH J B,GONZALEZ E L,CHOY K,et al.Gut microbiota Turicibacter strains differentially modify bile acids and host lipids[J].Nat Commun,2023,14(1):3669.
[29] MARTíNEZ I,STEGEN J C,MALDONADO-GóMEZ M X,et al.The gut microbiota of rural papua new guineans:composition,diversity patterns,and ecological processes[J].Cell Rep,2015,11(4):527-538.
[30] CHEN L H,CHANG S S,CHANG H Y,et al.Probiotic supplementation attenuates age-related sarcopenia via the gut-muscle axis in SAMP8 mice[J].J Cachexia Sarcopenia Muscle,2022,13(1):515-531.
[31] ISAACSON R,KIM H B.The intestinal microbiome of the pig[J].Anim Health Res Rev,2012,13(1):100-109.
[32] 張 賀,徐榮瑩,蘇 勇,等.單胃動物腸道微生物研究進展[J].動物營養學報,2020,32(10):4674-4685.
ZHANG H,XU R Y,SU Y,et al.A review:gut microbiota in monogastric animals[J].Chinese Journal of Animal Nutrition,2020,32(10):4674-4685.(in Chinese)
[33] 計 偉,謝春艷,趙艷平,等.乳酸菌的生物學功能及其在母豬和仔豬生產中的應用[J].動物營養學報,2018,30(11):4320-4326.
JI W,XIE C Y,ZHAO Y P,et al.Biological functions of Lactobacillus and its application in Sows′ and Piglets′ production[J].Chinese Journal of Animal Nutrition,2018,30(11):4320-4326.(in Chinese)
[34] ALLEN J M,BERGMILLER M E,PENCE B D,et al.Voluntary and forced exercise differentially alters the gut microbiome in C57BL/6J mice[J].J Appl Physiol (1985),2015,118(8):1059-1066.
[35] GOMEZ A,PETRZELKOVA K J,BURNS M B,et al.Gut microbiome of coexisting BaAka pygmies and bantu reflects gradients of traditional subsistence patterns[J].Cell Rep,2016,14(9):2142-2153.
[36] TETT A,PASOLLI E,MASETTI G,et al.Prevotella diversity,niches and interactions with the human host[J].Nat Rev Microbiol,2021,19(9):585-599.
[37] SUN Y,SU Y,ZHU W Y.Microbiome-metabolome responses in the cecum and colon of pig to a high resistant starch diet[J].Front Microbiol,2016,7:779.
[38] WST P K,HORN M A,DRAKE H L.Clostridiaceae and Enterobacteriaceae as active fermenters in earthworm gut content[J].ISME J,2011,5(1):92-106.
[39] LONG C X,WU J Q,TAN Z J,et al.Different intestinal microbiota with growth stages of three-breed hybrid pig[J].BioMed Res Int,2022,2022:5603451.
[40] CRESPO-PIAZUELO D,MIGURA-GARCIA L,ESTELLé J,et al.Association between the pig genome and its gut microbiota composition[J].Sci Rep,2019,9(1):8791.
[41] HORIE M,MIURA T,HIRAKATA S,et al.Comparative analysis of the intestinal flora in type 2 diabetes and nondiabetic mice[J].Exp Anim,2017,66(4):405-416.
[42] FARIA S L,SANTOS A,MAGRO D O,et al.Gut microbiota modifications and weight regain in morbidly obese women after Roux-en-Y gastric bypass[J].Obes Surg,2020,30(12):4958-4966.
(編輯 郭云雁)

