基于葉綠體基因rbcL和trnL-F序列的河北省野生苔草屬親緣關系分析

摘要:為研究河北省野生苔草屬(Carex)植物的親緣關系,本研究以43種河北省野生苔草及國外引種的27份苔草種質為試驗材料,克隆了葉綠體基因rbcL和trnL-F序列,進行遺傳多樣性及親緣關系分析,并構建了系統發育樹。結果表明,從70份苔草屬植物中成功克隆到的rbcL和trnL-F序列數量分別為67條、60條,測序成功率分別為95.7%、85.7%,經正反序列拼接對齊獲得序列長度分別為716 bp、976 bp,并上傳至NCBI獲得登錄號。二者合并得到59條序列,長度為1692 bp。rbcL,trnL-F以及合并序列分別定義了43,56,58個單倍型。基于合并序列的59份種質間遺傳距離為0~0.0421,平均遺傳距離為0.0142,整體劃分為3大類,與形態學分類基本相符。trnL-F序列有望開發為DNA條形碼,以期為苔草屬的分類鑒定、系統發育等研究提供依據。
關鍵詞:苔草屬;rbcL;trnL-F;親緣關系;遺傳多樣性
中圖分類號:Q37""" 文獻標識碼:A""""" 文章編號:1007-0435(2024)09-2728-09
收稿日期:2024-01-15;修回日期:2024-04-06
基金項目:河北省苔草屬植物種質資源收集與新種質創制項目(21326348D)資助
作者簡介:
周欣瑩(1999-),女,漢族,河北保定人,碩士研究生,主要從事園林植物遺傳育種與栽培研究,E-mail: 1240260550@qq.com;*通信作者Author for correspondence, E-mail: 123360961@qq.com; liudongyun0505@163.com
doi:10.11733/j.issn.1007-0435.2024.09.006
引用格式:
周欣瑩, 董彥娜, 邱玉鵬,等.基于葉綠體基因rbcL和trnL-F序列的河北省野生苔草屬親緣關系分析[J].草地學報,2024,32(9):2728-2736
ZHOU Xin-ying, DONG Yan-na, QIU Yu-peng,et al.Genetic Relationship Analysis of Wild Carex Species in Hebei Province Based on Sequences of Chloroplast Genes rbcL and trnL-F[J].Acta Agrestia Sinica,2024,32(9):2728-2736
Genetic Relationship Analysis of Wild Carex Species in Hebei Province
Based on Sequences of Chloroplast Genes rbcL and trnL-F
ZHOU Xin-ying1, DONG Yan-na2, QIU Yu-peng1, SUN Hao-nan1, LIU Ya-wei1,
LI Yan1*, LIU Dong-yun1*
(1. Collage of Landscape and Travel, Hebei Agricultural University, Baoding, Hebei Province 071000, China;
2. Shijiazhuang Information Engineering Vocational College, Shijiazhuang, Hebei Province 050000, China)
Abstract:To study the genetic relationships of wild Carex plants in Hebei Province,43 species of wild Carex plants in Hebei Province and 27 germplasms of Carex introduced from abroad were used as experimental materials. The chloroplast genes rbcL and trnL-F sequences were cloned for genetic diversity and phylogenetic analyses. And a phylogenetic tree was constructed. The results showed that the number of rbcL and trnL-F sequences successfully cloned from 70 plants of the genus Carex was 67 and 60,with sequencing success rates of 95.7% and 85.7%,respectively. The sequence lengths obtained after forward and backward sequence splicing and alignment were 716 bp and 976 bp, respectively and were uploaded to NCBI to obtain the GenBank accession number. The rbcL and trnL-F sequences combination obtained 59 sequences with a length of 1 692 bp. The rbcL,trnL-F and combined sequence define 43,56 and 58 haplotypes. Based on the combined sequence,the genetic distance between 59 germplasms is 0~0.0421,with an average genetic distance of 0.0142. Based on NJ phylogenetic tree of combined sequence,59 germplasms could be divided into 3 groups,which was basically consistent with morphological classification. The trnL-F sequence is expected to be developed as DNA barcodes,in order to provide basis for the classification,identification and phylogenetic research of Carex.
Key words:Carex; rbcL;trnL-F;Genetic Relationship;Genetic Diversity
莎草科(Cyperaceae)苔草屬(Carex)植物種類豐富,在世界范圍分布廣泛,全球約有2000種,我國約有500種,其中,河北省內約有45種[1-3]。苔草屬植物春季返青早、生長期長,具有耐寒、耐旱、耐瘠薄、耐踐踏等特點,且自繁能力及適應性強,有一定觀賞價值,能夠作為替代傳統草坪的優質地被植物[4]。同時,苔草屬植物也在生態修復方面發揮著重要的作用[5]。根據形態學分類,苔草屬植物可分為二柱苔草亞屬(Carex Subg. Vignea)、復序苔草亞屬(Carex Subg. Indocarex)和苔草亞屬(Carex Subg. Carex),二柱苔草亞屬和苔草亞屬的分布較為廣泛,而復序苔草亞屬主要分布在長江以南地區[7-8],河北省野生苔草屬于苔草亞屬和二柱苔草亞屬。眾多學者發現,苔草屬具有形態復雜、高度變異等特點,因此,苔草屬內的分類工作是一大難題[9-10]。近年來,隨著對苔草屬研究的深入,國內外不斷發現苔草屬新物種,此外,苔草屬的遺傳背景復雜、親緣關系不明確等原因也限制了引種和育種工作的進行,阻礙了進一步推廣和開發利用。
葉綠體有一套完整的遺傳系統,其獨立的遺傳信息被稱為葉綠體基因組(cpDNA),大多數植物遵循母系遺傳,總體保守性較高,多用于植物種間遺傳信息分析[11]。cpDNA編碼區和非編碼區在進化速率上存在差異,非編碼區的進化速率更快,系統發育信息含量更多[12]。因此,在進行系統發育研究時,需要考慮所選基因序列的進化速率,篩選合適的基因序列[13]。常用于植物系統發育研究的葉綠體基因序列有rbcL,rpl16,rpoC1,trnL-F,trnT-L,psbA-trnH等[14-16],其不僅可以用于構建系統發育樹還可進一步開發為DNA條形碼。Oda等[17]基于多個葉綠體基因和核基因序列(ITS)數據進行了分子系統發育分析,了解日本苔草屬Rarae組種間關系,并結合其形態特征進一步建立了該組的分類檢索表。Starr等[18]利用葉綠體基因開發了DNA條形碼,發現matK條形碼可區分47%的試驗材料,并通過該片段發現新物種。Yano等[19]基于atpB-rbcL,trnT-trnL,trnL內含子和trnL-trnF序列,分析了C. conica及其近緣種的分子地理學關系,在258份材料中得到了16個單倍型,將其分為日本東部和西部兩大地理結構類型。
野生苔草屬植物經過長期自然進化,其遺傳背景復雜,極大地影響研究和開發利用。河北省有著豐富的苔草屬種質資源,對其進行系統性的調查與引種后,發現部分種質的適應性強,且株型、葉色觀賞價值較高,有待進一步開發為優質的草坪地被或觀賞草。本研究以43種河北省野生苔草及國外引種的27份種質為試材,克隆和分析了其葉綠體基因rbcL和trnL-F序列,以此進行了親緣關系和遺傳多樣性分析,并進一步鑒定了種質資源,旨在從分子角度上為苔草屬的分類鑒定、DNA條形碼等研究提供理論依據,推動苔草屬植物的開發利用。
1" 材料方法
1.1" 試驗材料
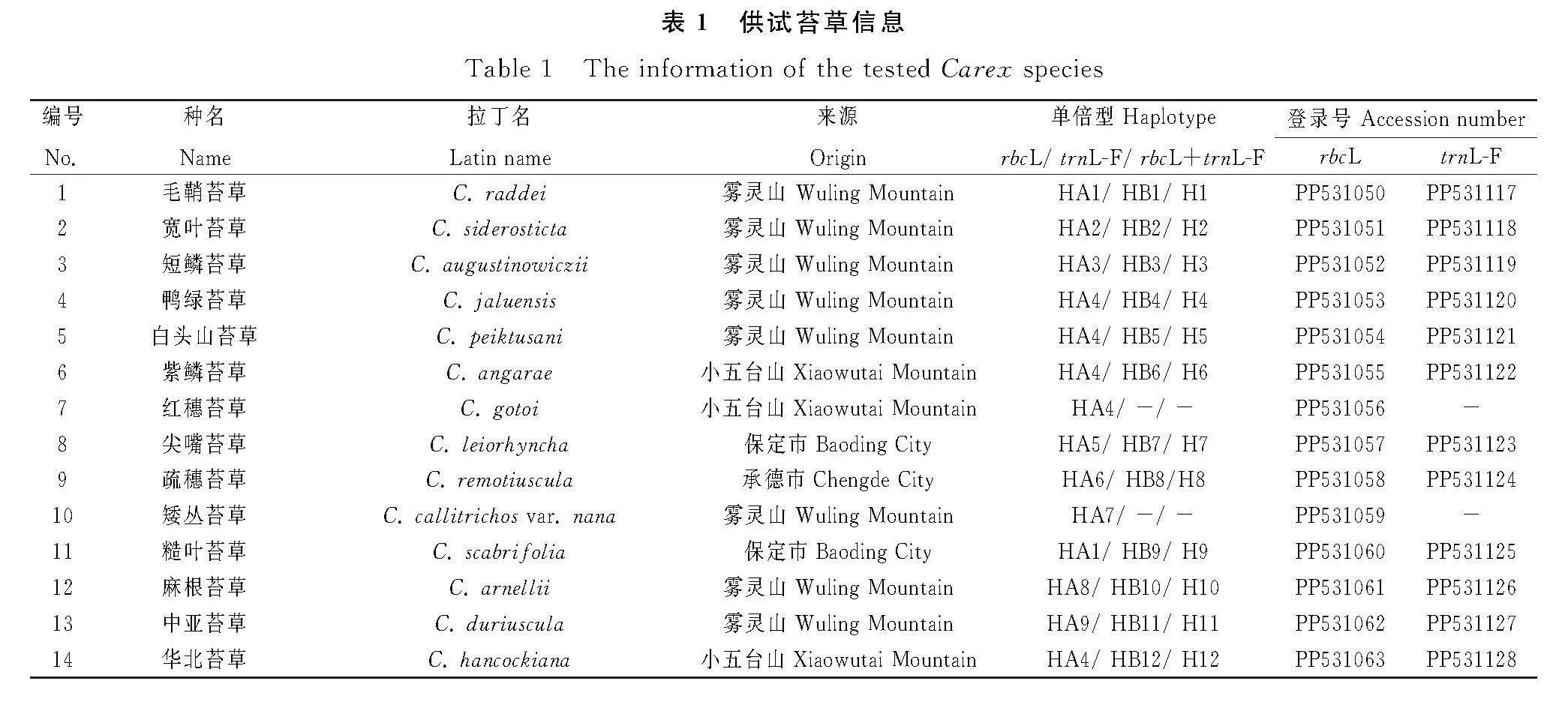
供試材料包括43種河北省野生苔草(根據《河北省植物志》及《內蒙古植物志》鑒定),采自保定市洪崖山、承德市霧靈山、張家口市小五臺山等地,以及27份國外引種的苔草種質,共計70份種質。引種收集后統一栽植于河北農業大學試驗基地,詳見表1。
1.2" 試驗方法
1.2.1" DNA提取和PCR擴增" 使用DNA提取試劑盒(CW0531S,康為世紀)提取70份苔草樣本的DNA用Nano Drop微量分光光度計進行DNA純度檢測并使用1.2%的瓊脂糖凝膠進行電泳檢測。使用ddH2O將DNA母液稀釋至80 ng·μL-1,-20℃保存。
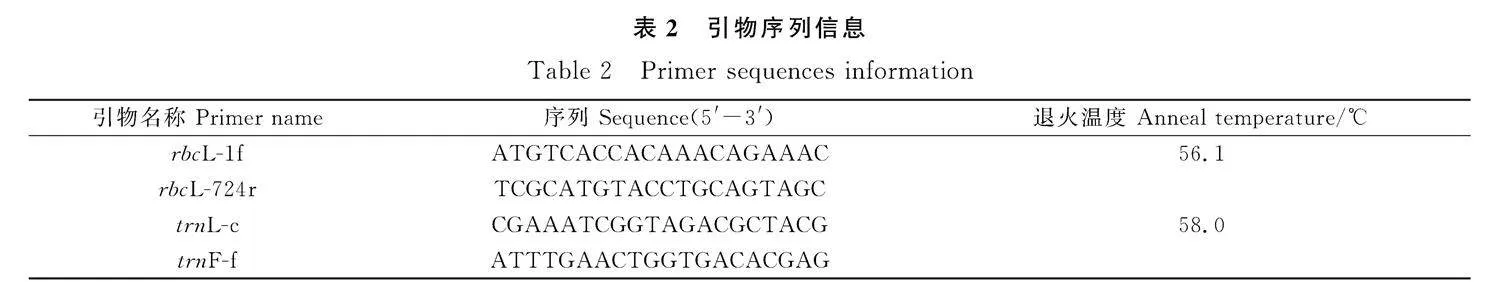
rbcL序列使用通用引物rbcL-1f、rbcL-724r[20],trnL-F間隔區序列使用通用引物trnL-c、trnF-f[21],詳情見表2。
PCR擴增體系采用20 μL體系:1 μL DNA模板,1 μL上下游引物(10 μmol·L-1),10 μL 2×Super Pfx Master Mix,7 μL ddH2O。
擴增程序:98℃預變性3 min,98℃變性10 s,退火30 s,72℃延伸1 min,35個循環,72℃延伸10 min,4℃保存。
1.2.2" 擴增產物的回收與純化" PCR產物采用1.2%的瓊脂糖凝膠電泳進行檢測,待條帶分離后使用膠回收試劑盒(DC3511,BIOMIGA)進行回收、純化,送上海生物公司進行雙向測序。
1.2.3" 數據分析" 利用BioEdite 7對雙向峰圖進行校對、拼接,去除兩端峰圖雜亂、質量較低的序列,將拼接后的序列作為查詢序列輸入到GenBank數據庫中,利用NCBI-BLAST相似性比對工具進行序列比對。利用MEGA 7軟件對序列的基本特點進行統計分析,包括了位點長度、簡約信息位點(Parsimony information sites,Ps)、變異位點(Variable sites,Vs)、保守位點(Conservedsites,C),堿基組成(Nucleotide composition)等,采用鄰接法(Neighbor-Joining,NJ)進行建樹。基于K2P(Kimura 2-parameter model)雙參數模型計算遺傳距離。利用DnaSP 5.0統計序列的特征信息和變異位點信息,Network 10.0基于MJ算法構建單倍型網絡圖。最終以同為莎草科的具芒碎米莎草(Cyperus microiria登錄號MN205054、KR869862)為外類群,構建系統發育樹。
2" 結果與分析
2.1" PCR擴增結果
PCR擴增產物經1.2%瓊脂糖凝膠電泳檢測,所有樣品的rbcL、trnL-F序列目的條帶分別在750~1000 bp、1000~1500 bp,擴增出的條帶清晰、明亮,無明顯雜帶,可用于后續的測序試驗。部分條帶結果見圖1,2。
2.2" rbcL、trnL-F序列特征分析
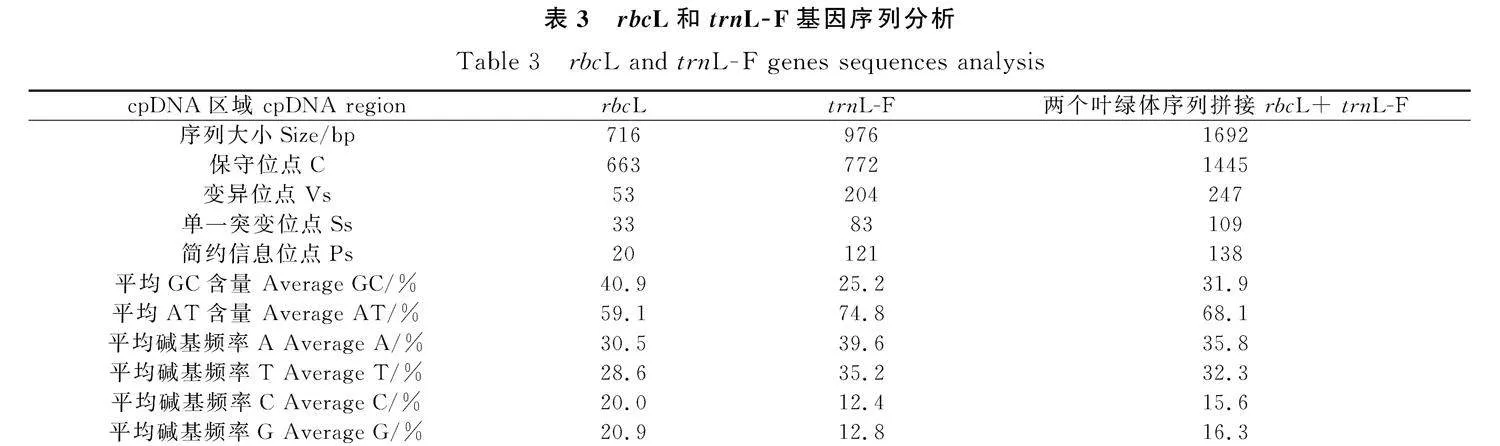
將rbcL和trnL-F序列測序結果進行分析,得到了67條、60條序列信息,測序成功率分別為95.7%,85.7%。正反序列經拼接并去除兩端不準確側翼序列后,分別獲得長度為716 bp、976 bp。
序列基本信息見表3。獲得的rbcL和trnL-F序列合并后得到59條序列,長度為1692 bp。核苷酸構成分析結果顯示,rbcL和trnL-F序列堿基A+T平均含量均高于G+C含量,rbcL、trnL-F及合并序列的平均堿基頻率為A=30.5%,39.6%,35.8%,T=28.6%,35.2%,32.3%;C=20.0%,12.4%,15.6%;G=20.9%,12.8%,16.3%。trnL-F及合并序列的平均GC含量低于rbcL序列,但變異位點、單一突變位點及簡約信息位點均高于rbcL序列。將所測得苔草屬植物rbcL和trnL-F序列提交到NCBI,并獲得登錄號為PP531050~PP531176(表1)。
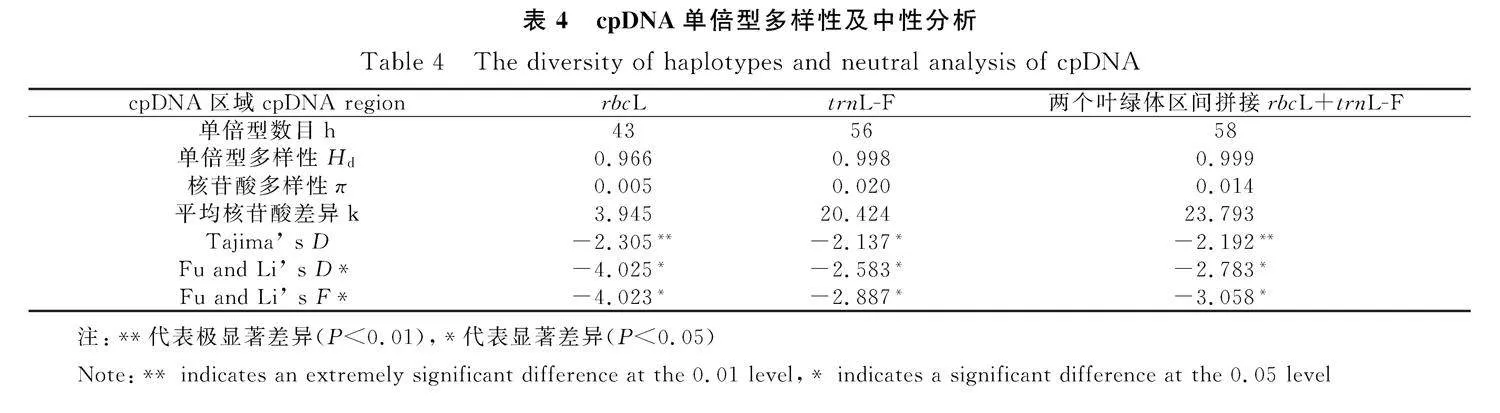
2.3" rbcL、trnL-F單倍型多樣性分析
對rbcL、trnL-F以及合并序列進行多態性分析,詳見表4。rbcL、trnL-F序列的單倍型數目(h)為43,56個,合并序列的單倍型數目為58個,多于rbcL和trnL-F序列的單倍型數目。苔草屬植物基于單一序列和合并序列分析的單倍型多態性(Hd)均大于0.5,核苷酸多態性(π)均高于0.005,合并序列的單倍型多態性高于單一序列。
通過Tajima’ s D和Fu and Li’ s值來檢驗rbcL、trnL-F序列在物種水平的進化過程中是否遵循中性進化模型,得到的Tajima’ s D值分別為-2.305,-2.137,-2.192,Fu and Li’ s D*值為-4.025,-2.583,-2.783,Fu and Li’ s F*值為-4.023,-2.887,-3.058,結果均顯著,不遵循中性進化模型,合并序列分析的35種河北省野生苔草屬植物Tajima’ s D和Fu and Li’ s值均為負值且結果顯著,也不遵循中性進化模型。
通過DnaSP軟件對苔草rbcL、trnL-F兩個序列聯合分析,共得到58個單倍型,利用Network軟件構建其單倍型網絡圖,見圖3。大多單倍型僅包含一個種質,其中矮生苔草和直穗苔草共享一個單倍型,同為H47。
2.4" 遺傳距離分析
基于rbcL、trnL-F以及合并序列分析的遺傳距離分別為0~0.027,0~0.053,0~0.042,平均遺傳距離分別為0.006,0.021,0.014。rbcL序列中鴨綠苔草、白頭山苔草、紫鱗苔草、紅穗苔草、華北苔草等的遺傳距離最小。trnL-F序列中異鱗苔草和灰脈苔草、藍葉苔草和‘藍辛格’、矮生苔草和直穗苔草、索蘭德苔草和三裂苔草遺傳距離最小。基于rbcL和trnL-F序列結合分析,矮生苔草和直穗苔草遺傳距離最小。
2.5" 基于rbcL、trnL-F序列的苔草屬植物親緣關系分析
使用MEGA軟件對苔草屬植物rbcL、trnL-F合并序列建樹,以具芒碎米莎草(C. microiria)為外類群,構建NJ系統發育樹(圖4),59份苔草屬種質可劃分為3類,第Ⅰ類包含寬葉苔草和毛緣寬葉苔草‘寶藏島’,二者葉片較寬,通常為披針形至寬條形,小穗均為兩性,4~10個,雄雌順序,苞片成佛焰苞狀,柱頭3個,屬于苔草亞屬寬葉苔草組(Sect. Siderostictae)。第Ⅱ類包含10種苔草,其中白穎苔草、中亞苔草、尖嘴苔草、砂鉆苔草、疏穗苔草、卵囊苔草屬于二柱苔草亞屬,棕櫚葉苔草、棕櫚葉苔草‘小矮人’、阿巴拉契亞苔草和多刺苔草分類地位暫不明確。二柱苔草亞屬小穗多數,全部為兩性,無柄,雌雄順序或雄雌順序,聚集在小穗頂端,柱頭通常2個。其中尖嘴苔草、卵囊苔草、中亞苔草、白穎苔草小穗通常為雄雌順序,且密集為圓柱狀花序;疏穗苔草小穗為雌雄順序;砂鉆苔草為小穗單性,雌雄異株,花序長圓狀卵形。其余47個材料均屬于第Ⅲ類,包含苔草亞屬和分類地位不明確的種質,如藍葉苔草、全裂苔草等國外引種的種質。其中形態分類學黑穗苔草組(Sect. Atratae)、指狀苔草組(Sect. Digitatae)下材料聚在一起,親緣關系更近。除未觀察到小穗的種質,其余材料小穗1個至多數,單性或單性與兩性兼有,小穗單個或多個生于苞片腋內,具柄,少數柄很短或近無柄,柱頭通常3個。
3" 討論
3.1" rbcL、trnL-F序列特征
在實際應用中,常將cpDNA中進化速率不一的序列結合來區分屬下不同物種[22]。本研究發現,在兩個葉綠體序列中,trnL-F的突變總數、變異位點、單一突變位點、平均核苷酸多樣性等均高于rbcL。合并序列中247個變異位點,109個單一突變位點,138個簡約信息位點,共58個單倍型,單倍型間聯系密切,通過研究各種單倍型間的關系能夠了解其種群遺傳變異及進化關系。合并序列中變異位點、單倍型數目等指標高于單一序列,證明多個序列拼接后具有豐富的遺傳信息,與Kim等[23-24]的研究結果一致。張仕蘭等[25]利用rbcL和trnL-F等5個葉綠體基因對海三棱藨草[× Bolboschoenoplectus mariqueter (Tang amp; F. T. Wang) Tatanov]及其近緣種的分類進行研究,trnL-F的變異位點少于rbcL,變異程度較低。本研究中trnL-F的變異位點多于rbcL,變異程度較高,表現為較高的遺傳多樣性,這與rbcL和trnL-F序列在鳶尾(Iris tectorum)[26]中表現相似,因此,葉綠體基因序列的變異程度可能與物種密切相關。
rbcL序列相對保守,種間變異小,因其強大的通用性被廣泛用于植物系統發育研究中,trnL-F為cpDNA的非編碼區,其進化速率快,片段兩端存在保守序列,適用于分類階元較低的類群間的系統發育研究[27-28]。Starr等[18]在使用引物rbcL-a_f和rbcL-a_r對苔草屬的rbcL序列進行擴增和測序,發現該序列的獲得率僅為18%,本研究采引物rbcL-1f和rbcL-724r對rbcL序列進行擴增和測序,序列的獲得率為95.8%,但序列變異率低,不能對苔草屬種質進行有效區分,與Clerc-Blain等[29]的研究結果一致。無論是單一序列還是多序列組合對苔草屬植物的鑒別率均不超過60%[18],本研究將rbcL和trnL-F片段組合,對物種的鑒別率較高,將進化速率不一的片度進行結合分析,可以提供更多的有效信息點,從而提高研究結果的準確性和可靠性[30]。
3.2" rbcL、trnL-F單倍型多樣性
rbcL和trnL-F合并序列將59份種質定義為58個單倍型,較古老的單倍型通常位于聚類簇內部,而稀有單倍型通常位于網絡分支末端,H1位于單倍型網絡分支的中心位置,則認為該單倍型可能為原始單倍型。中性檢驗的結果為負值,說明研究的群體可能在歷史上經歷過突然擴張或者受到了定向選擇,在分子水平導致進化的原因可能是突變壓力或遺傳漂變[31-32]。葉綠體單倍型網絡圖中的各單倍體呈現的星狀輻射也能夠說明這一現象。矮生苔草和直穗苔草均屬于H47,二者分別屬于苔草亞屬中沼生苔草組、苔草組,其形態較相似,葉片較硬,扁平,小穗3~5個,上部2~3個為雄小穗,接近生,其余為雌小穗,但矮生苔草有粗壯而堅韌的匍匐根狀莖。矮生苔草分布于沿海地區的海邊沙地,采集于河北省秦皇島市黃金海岸,直穗苔草分布于沼澤地、河邊及河邊潮濕地,采集于河北省唐山市曹妃甸湖區域,二者的采集地環境氣候較為相似,地理距離近,可能導致其被定義為同一種葉綠體單倍型,有待結合核基因序列進一步研究。
3.3" 遺傳多樣性與親緣關系
遺傳多樣性的高低可以通過遺傳距離的大小來判斷,遺傳距離越大說明親緣關系越遠,表明其遺傳多樣性越高,反之,遺傳距離越小,說明遺傳多樣性越低。59份苔草屬材料的遺傳距離為0~0.0421,平均遺傳距離為0.0142,親緣關系較近,遺傳多樣性較低,與白羊草(Bothriochloa ischaemum)[33]種質遺傳多樣性的研究結果一致,可能與自身繁育系統的特性有關。無性繁殖、長期的自交或近交會降低種群的遺傳多樣性,根據現有報道來看,苔草屬植物的繁殖方式為營養繁殖和種子繁殖,由于苔草屬小穗頂生雄小穗、雄雌順序或雌雄順序的結構特征以及開花習性,導致散粉后花粉幾乎全部落在雌花柱頭上,極大的增加了自交的概率[34-35]。此外,環境、氣候、人為干擾等外部因素也會間接影響植物的遺傳多樣性[36]。
除分類不明確的材料外,本研究中苔草材料屬于苔草亞屬和二柱苔草亞屬,59份苔草屬種質被劃分為3大類,聚類結果與形態學分類基本相符,與Villaverde等[37]對苔草屬重新分類的結果一致,將寬葉苔草組從苔草亞屬中獨立出來,成為寬葉亞屬,但目前我國還是基于形態學分類將苔草屬植物分為3個亞屬:苔草亞屬、二柱苔草亞屬、復序苔草亞屬。而基于ISSR[38]對河北省野生苔草屬植物進行親緣關系分析,將53份供試材料分為6大類,并不能將亞屬級別的材料進行區分,且與傳統形態學分類存在一定分歧,也與本研究基于葉綠體基因rbcL和trnL-F序列的聚類結果存在不同,推測可能是存在核質沖突,但也可能是該種標記的缺陷或人工讀帶誤差等導致的。從自展值來看,苔草亞屬中黑穗苔草組短鱗苔草、紫喙苔草與華北苔草、白頭山苔草、紫鱗苔草的自展值低于50%,有待結合更多序列來進行種間的區分與鑒定,而部分苔草品種間的自展值高,可以進一步區分同一種的不同品種,如芒髯苔草品種‘香草冰’ ‘冰舞’ ‘永恒光輝’和大島苔草品種‘金芯’ ‘銀邊’ ‘黃金發絲’,有望作為區分苔草品種的DNA條形碼。綜合來看,應采用形態學和分子標記相結合的方式對苔草屬植物進行分類和鑒定。
目前,苔草屬中應用較多的為苔草亞屬和二柱苔草亞屬中的種質,已有不同種質被開發利用,如青綠苔草、澇峪苔草、白穎苔草等。除此之外,還發現這兩個亞屬中披針葉苔草、早春苔草、矮叢苔草、疏穗苔草等植物葉長葉寬適中、株型圓整,觀賞效果好,而寬葉亞屬中寬葉苔草、‘寶藏島’葉片更寬大,株型整齊,整體較美觀。這些苔草適應性強,在華北地區可以露地越冬越夏,維護成本低,但目前應用的較少,可以開發為園林地被或觀賞草。利用誘變育種或依據其親緣關系進行雜交育種等多種方法,進行品種改良和新品種選育,進一步選育出具有良好觀賞性的種質。
4" 結論
葉綠體基因rbcL、trnL-F序列以及二者合并序列分析均顯示苔草屬植物具有一定的遺傳多樣性。經裁剪拼接得到的苔草的rbcL和trnL-F序列分別為716 bp和976 bp,上傳至NCBI獲得登錄號。rbcL序列較為穩定,trnL-F序列具有更豐富的變異,兩條序列拼接長度為1692 bp,序列合并后定義了58種單倍型。59份種質劃分為3類,與形態學分類有一定相關性,首次在河北省苔草屬中驗證了寬葉亞屬的地位。trnL-F序列定義了56個單倍型,有望作為苔草屬植物鑒定的DNA條形碼。本研究為苔草屬植物在分類鑒定、系統發育及開發利用等方面提供了理論依據。
參考文獻
[1]" 張玉,王麗媛,王繼豐,等. 基于文獻計量學的苔草屬植物研究進展[J]. 黑龍江科學,2018,9(20):17-19
[2]" TENG K,TENG W J,WEN H F,et al. PacBio single-molecule long-read sequencing shed new light on the complexity of the Carex breviculmis transcriptome[J]. BMC Genomics,2019,20(1):789
[3]" 河北省植物智編輯委員會. 河北植物志III[M]. 石家莊:河北科學技術出版社,1991:408-439
[4]" 張全剛,孫建華,郝愛,等. 淺談苔草在園林綠地中的應用[J]. 農業與技術,2022,42(24):127-129
[5]" 豆豆,李紅. 黃河源區退化草地褐鱗苔草種植技術及對土壤改良效果研究[J]. 甘肅農業大學學報,2022,57(5):171-180
[6]" 中國科學院中國植物委員會. 中國植物志.第十二卷[M]. 北京:科學出版社,2000:56-519
[7]" 劉凌云,范希峰,滕珂,等. 苔草屬植物遺傳多樣性研究進展[J]. 草地學報,2021,29(6):1148-1157
[8]" 冷建紅,樓爐煥,魏琦,等. 薹草屬植物研究進展及園林應用前景研究[J]. 北方園藝,2011(6):196-201
[9]" SHEKHOVTSOV S V,SHEKHOVTSOV I N,PELTEK S E. Phylogeny of Siberian species of Carex sect. Vesicariae based on nuclear and plastid markers[J]. Nordic Journal of Botany,2012,30(3):343-351
[10]LVEILL-BOURRET,TIENNE,STARR J R,FORD B A. Why are there so many sedges? Sumatroscirpeae,a missing piece in the evolutionary puzzle of the giant genus Carex (Cyperaceae)[J]. Molecular Phylogenetics and Evolution,2018,119:93-104
[11]PERDEREAU A,KLAAS M,BARTH S,et al. Plastid genome sequencing reveals biogeographical structure and extensive population genetic variation in wild populations of Phalaris arundinacea L. in north-western Europe.[J].Global Change Biology Bioenergy,2017,9(1):46-56
[12]姜棟柱. 基于葉綠體基因組和ITS序列的姜屬植物系統發育研究[D]. 荊州:長江大學,2023:3-4
[13]AMAR M H,MAGDY M,WANG L,et al. Peach chloroplast genome variation architecture and phylogenomic signatures of cpDNA introgression in Prunus[J]. Canadian Journal of Plant Science,2019,99(6):885-896
[14]苗藝明,黃松殿,覃永華,等. 基于三個葉綠體基因序列的廣西盾翅藤遺傳多樣性分析[J/OL]. http://kns.cnki.net/kcms/detail/46.1068.S.20231020.1334.006.html,2023-10-20/2024-03-19
[15]龍炳宏,蔣向輝,宋榮,等. DNA條形碼在黃精屬藥用植物鑒定與遺傳多樣性分析中的應用[J]. 植物科學學報,2022,40(4):533-543
[16]錢志瑤,饒海,曹旭林,等. 黔產寬葉纈草葉綠體trnL-F,matK基因遺傳關系的分析[J]. 中國實驗方劑學雜志,2018,24(5):36-42
[17]ODA J,FUSE S,YAMASHITA J,et al. Phylogeny and Taxonomy of Carex (Cyperaceae) in Japan I. C. sect. Rarae[J]. Acta Phytotaxonomica et Geobotanica:APG,2019(2):69-85
[18]STARR J R,NACZI R F C,CHOUINARD B N. Plant DNA barcodes and species resolution in sedges (Carex,Cyperaceae)[J]. Molecular Ecology Resources,2010,9(s1):151-163
[19]YANO O,IKEDA H,HOSHINO T. Phylogeography of the Japanese common sedge,Carex conica complex (Cyperaceae),based on chloroplast DNA sequence data and chromosomal variation[J]. American Journal of Botany,2010,97(8):1365-1376
[20]KRESS W J,ERICKSON D L,SHIU S H. A Two-Locus Global DNA Barcode for Land Plants:The Coding rbcL Gene Complements the Non-Coding trnH-psbA Spacer Region[J]. Plos One,2007,2(6):e508
[21]TABERLET P,GIELLY L,PAUTOU G,et al. Universal primers for amplification of three non-coding regions of chloroplast DNA[J]. Plant Molecular Biology,1991,17(5):1105-1109
[22]孫浩男,李蓉,王鑫,等. 基于三種分子標記的栽培金雞菊遺傳多樣性及親緣關系分析[J]. 草地學報,2022,30(11):2922-2936
[23]KIM Y,SHIN J,OH D R,et al. Comparative Analysis of Complete Chloroplast Genome Sequences and Insertion-Deletion (Indel) Polymorphisms to distinguish five vaccinium species[J]. Multidisciplinary Digital Publishing Institute,2020,11(9):927
[24]王崇,焦春海,楊新筍,等. 基于葉綠體基因trnL-trnF、trnH-psbA和trnT-trnL序列的甘薯種質遺傳多樣性分析[J]. 南方農業學報,2021,52(6):1536-154
[25]張仕蘭,王嶼岑,劉文亮. 海三棱藨草及其近緣種的DNA條形碼研究[J]. 廣西植物,2024,44(4):699-709
[26]蔣喻林. 基于trnL-F、rbcL基因的四川省鳶尾屬植物系統分類進化研究[D]. 成都:四川農業大學,2016:31-32
[27]朱斌,胡利娟,田忠靜,等. 基于葉綠體基因組序列對50種菊科植物親緣關系分析[J]. 貴州師范大學學報(自然科學版),2021,39(2):15-25
[28]王霞,劉艷,賈秀秀,等. 矩鐮莢苜蓿與近緣種花苜蓿的DNA條形碼篩選[J]. 草地學報,2023,31(8):2323-2333
[29]CLERC-BLAIN J L,STARR J R,BULL R D,et al. A regional approach to plant DNA barcoding provides high species resolution of sedges (Carex and Kobresia,Cyperaceae) in the Canadian Arctic Archipelago[J]. Molecular Ecology Resources,2010,10(1):69-91
[30]FAZEKAS A J,BURGESS K S,KESANAKURTI P R,et al. Multiple multilocus DNA barcodes from the plastid genome discriminate plant species equally well[J]. Plos One,2008,3(7):e2802
[31]萬誠,李蕾嘉,陳勝,等. 貴州老鷹基于葉綠體DNA的遺傳多樣性分析[J]. 農業與技術,2022,42(15):22-25
[32]趙字陳,趙玉娟,龔洵,等. 龍血樹柴胡的遺傳多樣性及其分布特征研究[J]. 植物科學學報,2022,40(1):1-10
[33]李鈺瑩,董寬虎. 山西白羊草種質資源遺傳多樣性的ISSR分析[J]. 草業學報,2014,23(1):217-222
[34]文亞峰,韓文軍,吳順. 植物遺傳多樣性及其影響因素[J]. 中南林業科技大學學報,2010,30(12):80-87
[35]林鵬,楊虎彪. 吊羅山薹草開花生物學特征及繁育系統研究[J]. 熱帶作物學報,2021,42(3):689-694
[36]BOLTON P E,ROLLINS L A,BRAZILL-BOAST J,et al. Genetic diversity through time and space:diversity and demographic history from natural history specimens and serially sampled contemporary populations of the threatened Gouldian finch (Erythrura gouldiae)[J]. Conservation Genetics,2018,19(3):737-754
[37]VILLAVERDE T,JIMNEZ-MEJAS P,LUCENO M,et al. A new classification of Carex (Cyperaceae) subgenera supported by a HybSeq backbone phylogenetic tree[J]. Botanical Journal of the Linnean Society,2020,194(2):141-163
[38]周欣瑩,耿宇航,賈艷艷,等. 河北省野生苔草屬植物ISSR分子標記開發及親緣關系分析[J]. 草地學報,2023,31(10):2949-2959
(責任編輯" 彭露茜)

