多重PCR檢測食源金黃色葡萄球菌nuc、blaZ和mecA基因方法的建立與應用*
劉書亮,劉冬香,賈仁勇,韓新鋒
金黃色葡萄球菌(Staphylococcus aureus,簡稱Sa)是引起食物中毒的重要致病菌,也是造成人和動物感染的主要病原菌之一。食源耐甲氧西林Sa(Methicillin-resistant S.aureus,MRSA)的污染研究成為世界各國研究的熱點。我國養殖業中廣泛應用抗菌藥,也引起廣泛關注。
由于傳統的細菌分離鑒定、毒素檢測以及耐藥性測定費時費力,敏感性差。PCR方法快速敏感,在Sa的種屬基因鑒定及耐藥性分析中已有不少報告〔1-3〕。由于基因的表達受多種因素調控,基因檢測與表型存在一定差異〔4〕。本研究旨在建立快速鑒定食源Sa(耐熱核酸酶基因,nuc)及檢測Sa對β-內酰胺類藥物耐藥性(β-內酰胺酶基因,blaZ;甲氧西林耐藥基因,mecA)的多重PCR方法,分析食源MRSA污染情況,同時比較表型與基因型的一致性和差異性。現將結果報告如下。
1 材料與方法
1.1 材料
1.1.1 菌株 標準菌株:金黃色葡萄球菌ATCC33591為M RSA 陽性菌、ATCC29213為β-內酰胺酶陽性Sa,由中國農業大學動物醫學院吳聰明博士惠贈;試驗菌株:分離鑒定于成都等地市的豬源、牛源、雞源動物性食品樣品中的 79 株 Sa〔5〕,由四川農業大學食品微生物實驗室保存。
1.1.2 培養基 Mueller-Hinton(M-H)肉湯:購自杭州微生物試劑有限公司;氨芐青霉素:由寶生物工程(大連)公司提供;LB培養基、氨芐青霉素平板(Amp-LB)自配。
1.1.3 抗生素 青霉素類:青霉素G(PEN)、氨芐西林(AMP)、苯唑西林(OXA)、阿莫西林-克拉維酸(AMC);頭孢類:先鋒唑啉(CFZ)、頭孢曲松(CTX)、頭孢噻呋(CEF);均為原藥粉,由課題組統一提供。
1.1.4 分子生物學試劑 溶菌酶、2×Taq PCR Master Mix、DL2000 DNA Marker、GoldviewTMDNA染料均購自天根生化科技有限公司;DNA凝膠回收試劑盒、pMD18-T Simple Vector和感受態細胞E.coli JM109、dNT Pmixture、Taq聚合酶等購自寶生物工程(大連)有限公司;其它化學試劑均為分析純。
1.1.5 主要儀器 MyCyclerPCR儀(Bio-RAD),PowerPac Basic電泳儀及水平電泳槽(Bio-RAD),凝膠成像系統(Bio-RAD),Milli-Q Gradient超純水系統(Millipore公司),B4i-BR4i冷凍離心機(Thermo)等。
1.2 方法
1.2.1 藥敏試驗 按照美國臨床實驗標準委員會(NCCLS)(2007)進行藥液配制,采用NCCLS推薦的肉湯微量稀釋法對Sa進行β-內酰胺類抗生素(組合)藥敏試驗,并進行結果判定。
1.2.2 產β-內酰胺酶試驗 按照趙旺勝等〔6〕的碘量法進行,以ATCC 29213為β-內酰胺酶陽性對照。
1.2.3 多重PCR方法建立
1.2.3.1 引物設計 根據GenBank中登錄的Sa的nuc、blaZ、mecA 基因序列,利用Primer 5.0軟件設計3對特異性引物(表 1),由 invitrogen公司合成。

表1 引物序列和產物大小Table 1 Primer Sequences and product sizes
1.2.3.2 模板制備 將分純的Sa試驗菌株和標準菌株分別接種于 LB肉湯,37℃震蕩培養過夜。取2mL培養物于12 000r/min離心2min,收集菌體。根據唐俊妮〔7〕報道方法提取DNA。
1.2.3.3 單一基因PCR擴增 根據所設計的引物,在相同條件下以標準菌 ATCC33591和ATCC29213的 DNA為模板進行 nuc、blaZ和mecA的單一基因 PCR擴增。PCR反應體系(25μL)的組成 :2 ×Master Mix 12.5μL、Primer 1(10μ mol/L)1μL、Primer 2(10μ mol/L)1μL、Template 2μL、ddH2O 8.5μL。經預實驗獲得的 PCR反應程序為:94℃預變性5min,94℃變性45s,57℃退火50s,72℃延伸1.5min,進行30個循環,72℃延伸10min,PCR產物于4℃保存。
1.2.3.4 多重PCR條件優化 根據單一基因PCR建立多重PCR方法,反應體系仍25μL,其中2×Master Mix 12.5μL,Template 3μL,加入引物后,補 ddH2O至 25μL。逐一對引物濃度、退火溫度、延伸時間和溫度、循環次數進行優化。
1.2.3.5 瓊脂糖凝膠電泳 PCR產物經1%(0.5×TBE)瓊脂糖凝膠電泳后,觀察并成像。
1.2.3.6 擴增產物測序鑒定:回收的擴增產物進行T克隆后由invitrogen公司測序,并進行序列分析。
1.2.4 多重PCR方法檢測試驗菌株 對食源79株Sa提取的DNA進行多重PCR檢測,并與表型比較分析。
2 結果與分析
2.1 單一基因PCR結果 通過優化,退火溫度為57℃時,各引物的單一基因PCR擴增均能獲得較好結果(見圖1),單一基因片段大小與預期結果相符。
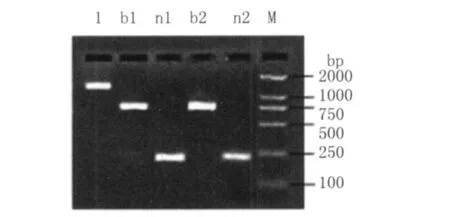
圖1 mecA、blaZ、nuc單一基因 PCR電泳結果Fig.1 PCR products of MecA,blaZ,nuc gene after gel electrophoresis
2.2 多重PCR條件優化 標準菌ATCC33591同時含有nuc、blaZ、mecA 3個基因,以該菌的基因組DNA為模板進行多重PCR條件優化。
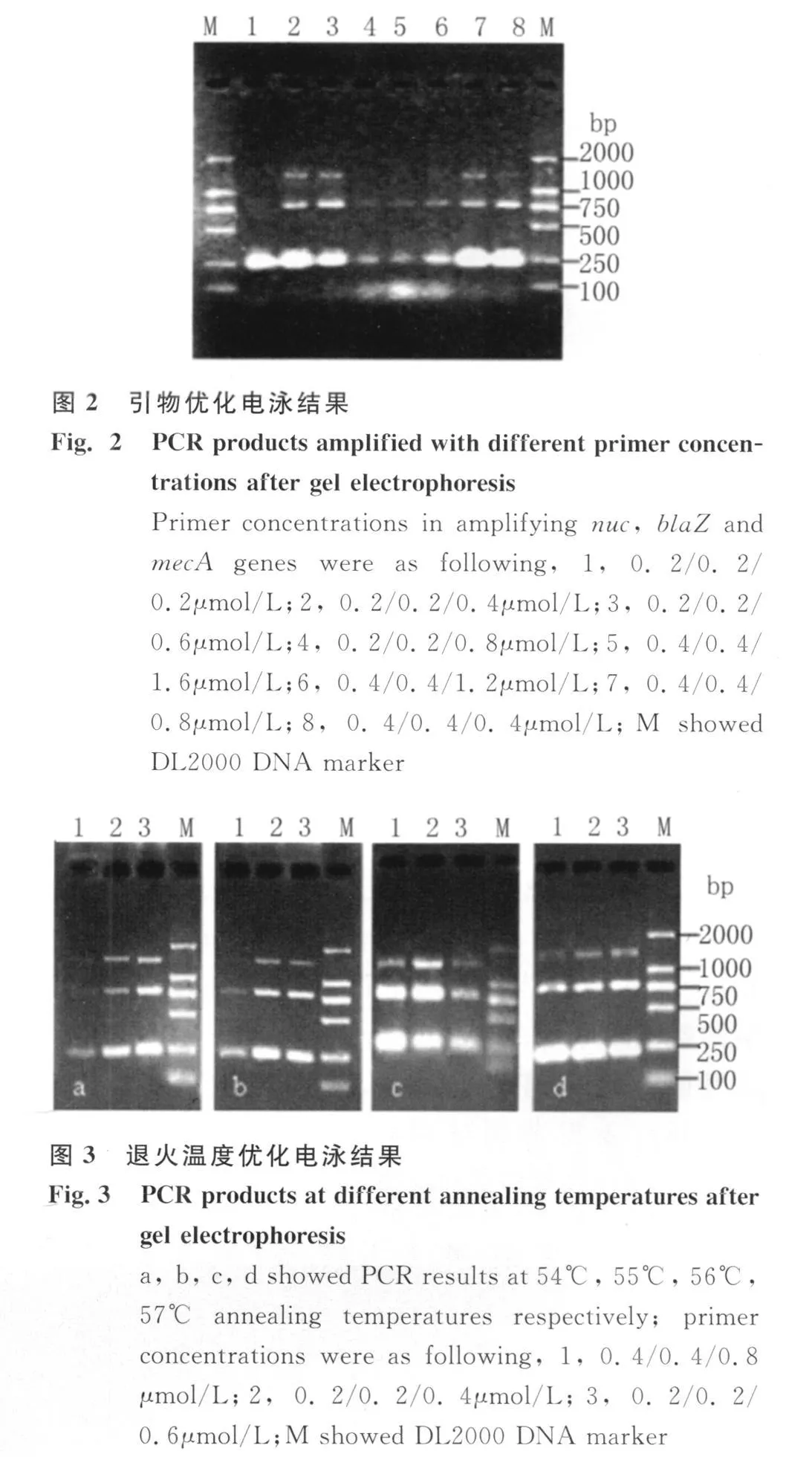
2.2.1 引物優化結果 根據預實驗結果,3對引物等比例混合后,在引物應用濃度均為0.4μ mol/L條件下的擴增結果表明,長片段產物相對較弱,因此,對引物組合nuc/blaZ/mecA設了8個應用濃度梯度,結果 0.2/0.2/0.4μ mol/L,0.2/0.2/0.6μ mol/L,0.4/0.4/0.8μ mol/L條件下均特異擴增出了3個目的片段,且條帶較清晰,見圖2,因此選擇這3個濃度做進一步的PCR條件優化。
2.2.2 退火溫度優化結果 根據引物優化結果,其它條件不變,比較在 54℃、55℃、56℃、57℃退火溫度和3種引物濃度梯度下的擴增結果,最終確定引物組合nuc/blaZ/mecA的濃度是0.2/0.2/0.4μ mol/L,退火溫度為56℃。退火溫度優化結果見圖3。
2.2.3 延伸時間和溫度、循環次數的優化結果 根據對72℃延伸1min、1.5min、2min和68℃延伸2min比較顯示,72℃延伸2min擴增效果最佳,同時反應循環次數優化結果表明,35次循環優越于30次循環反應(見圖4)。因此,最佳的反應程序是:94℃預變性5min后,94℃變性50s,56℃退火50s,72℃延伸2min,進行35個循環,最后以72℃延伸10min。
2.3 基因測序結果與相似性分析 以ATCC33591的DNA為模板,擴增的nuc、blaZ、mecA單一基因產物序列分析表明,該菌株nuc、blaZ、mecA基因PCR產物測序分別得到長 229 bp、787 bp、1459 bp的片段,與設計大小完全符合;測得序列已在NCBI注冊,登錄號分別為 FJ809757、FJ809758、FJ810876,編碼氨基酸序列登錄號分別為ACO37595、ACO37596、ACO24928。采用 BLAST和DNA STAR MegAlign進行基因比對分析。
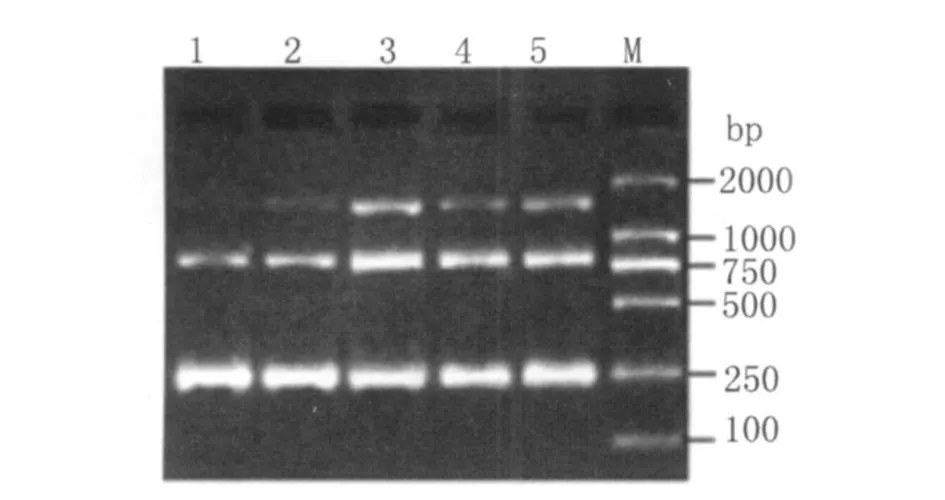
圖4 延伸時間和溫度以及循環數優化電泳結果Fig.4 PCR products with different extension times,temperatures and cycles after gel electrophoresis
ATCC33591的nuc基因(FJ809757)與登錄號EF529596.1(SA strain G8)、CP000730.1(SA subsp.aureus USA300_TCH1516)和EF529608.1(SA strain 2513)等的nuc基因序列有98.7%~100%的相似性,其ACO37595與上述Sa菌株的耐熱核酸酶的氨基酸序列的相似性高達98.7%~100%;而該基因與溶血葡萄球菌(S.haemolyticus)(NC_007168)、表皮葡萄球菌(S.epidermidis)(NC_004461)相似性和編碼相應氨基酸序列(YP_253498.1和NP_764559.1)相似性均較低。
ATCC33591的blaZ基因(FJ809758)與登錄號AB245469.1(SA strain 80s-3)、AY917098.1(SA strain a53)、BX571856.1(SA strain MRSA252)、AJ302698.2(S.haemolyticus)、CP000028.1(S.epidermidis)、(S.intermedius,中間葡萄球菌)等的blaZ基因序列有98.5%~100%的相似性,其ACO37596與上述菌株的β-內酰胺酶的氨基酸序列的相似性達97.7%~100%。
ATCC33591的mecA基因(FJ810876)與登錄號AB425824.1(SA strain JCSC6670)、AY271717.1(SA strain 2314)和CP000046.1(SA subsp.aureus COL)等mecA基因序列有99.7%以上的相似性,其ACO24928與上述菌株的PBP2a氨基酸序列的相似性也達99.2%以上。上述分析進一步表明該方法正確。
2.4 多重PCR方法對試驗菌株檢測結果 采用建立的Sa nuc、blaZ、mecA基因三重 PCR方法,對供試的 79株 Sa進行檢測,結果只有 58株(占73.42%)同時擴增出nuc和blaZ基因,另有19株(占24.68%)只能擴增出nuc基因,所有菌株均未擴增出mecA基因,即無MRSA檢出。該方法能鑒定出77株Sa,鑒定率為97.47%(77/79)。部分菌株的多重PCR檢測結果,見圖5。
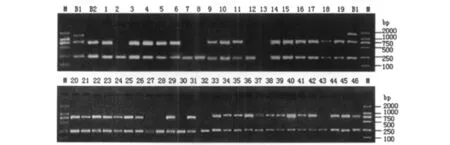
圖5 部分Sa菌株多重PCR檢測結果Fig.5 Multiplex PCR products of partial Sa isolation strains after gel electrophoresis
2.5 PCR檢測結果與表型比較 供試79株菌的菌落在顯色平板上顯粉紅或紫紅色且凝固酶陽性,但只有77株菌nuc基因擴增陽性。試驗菌株基因型與表型符合率為97.47%。
由表2可知,β-內酰胺酶陽性、blaZ 陽性和β-內酰胺酶陰性、blaZ陰性的符合率為60.76%(48/79);對PEN、AMP同時耐藥、blaZ陽性和對PEN、AMP同時敏感、blaZ陰性的菌株占75.95%(60/79),對青霉素類(PEN、AMP)敏感性分析結果、β-內酰胺酶試驗結果和blaZ擴增結果同時符合的菌株占49.37%(39/79)。
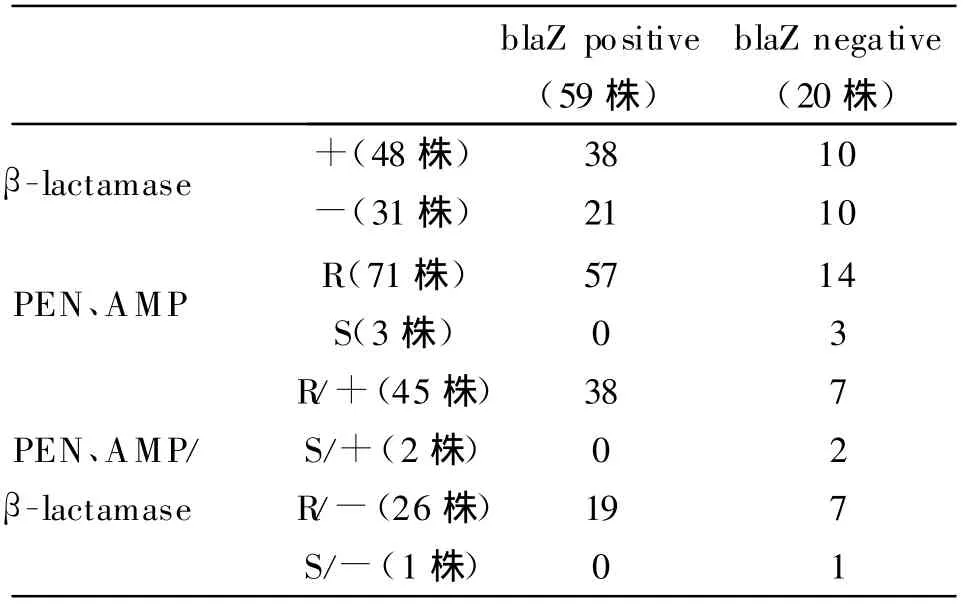
表2 blaZ擴增結果與β-內酰胺酶試驗和對青霉素類敏感性結果對比分析Table 2 Comparison of blaZ gene detection using PCR,β lactamase assay and penicillins sensitivity test
將blaZ擴增結果與菌株對青霉素類藥物的最低抑菌濃度(MICs)進行對比分析,見表3。隨著MICs增加blaZ陽性和blaZ陰性菌株數之間并沒有什么相關性,無論MICs值大小,總有blaZ陽性和blaZ陰性菌株同時存在,blaZ陽性菌株數總是多于blaZ陰性菌株數。
藥敏試驗結果表明,食源79株Sa對測試的3種頭孢菌素均為100%敏感。測定對苯唑西林的敏感性即是代替甲氧西林,苯唑西林藥敏結果顯示菌株的 MICs值在0.03~0.25μ g/mL之間,沒有檢出苯唑西林耐藥的Sa,而79株Sa的mecA PCR檢測結果均為陰性,表明無MRSA檢出。因此,耐甲氧西林的基因與表型檢測結果完全相符。
3 討 論
3.1 檢測Sa的nuc、blaZ和mecA基因多重PCR方法的建立 引物設計是PCR實驗成功的關鍵。本實驗設計的同時擴增nuc、blaZ和mecA基因的3對引物具有特異性。由于三個目標片段的大小差異較大,一定程度上增加了實驗的難度,但通過對擴增體系和擴增參數的逐一優化,成功建立了擴增nuc、blaZ和mecA基因的三重PCR方法。該方法簡便、快捷、準確,不僅能快速鑒定Sa,也能檢測MRSA,更主要的是還能一定程度闡明Sa對β-內酰胺類抗生素的耐藥機制,這點優越于其他多重 PCR方法〔1-3〕。正由于擴增的 nuc、blaZ和mecA 基因目標片段的大小差異較大,還有望結合其它耐藥基因、腸毒素基因進行多基因的同步擴增。
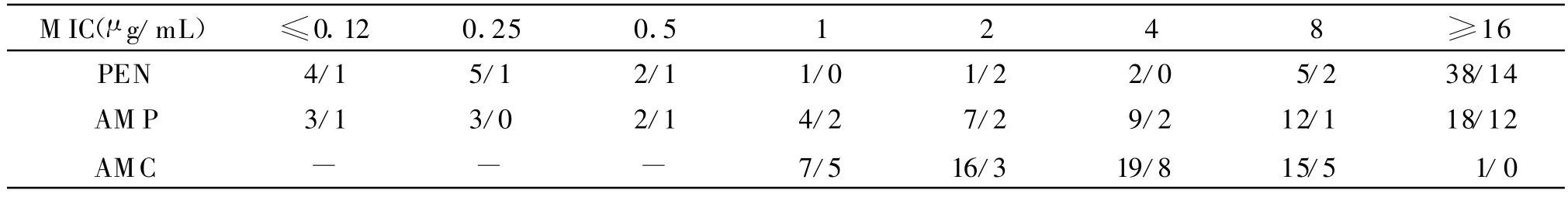
表3 不同MICs值條件下的blaZ陽性/blaZ陰性菌株數比較Table 3 Comparison of blaZ positive or negative strains at different MICs
3.2 Sa的nuc、blaZ和mecA基因檢測結果與表型結果分析 Sa產生耐熱核酸酶使菌落在含有甲苯胺藍的顯色平板上顯紅色,這是Sa重要的表型特征。Sa 的 nuc 基因〔8-9〕、血漿凝固酶(coa)基因〔10〕、16S rRNA〔11〕和耐藥輔助(femA)基因〔12〕常被作為種特異性基因,尤其是 nuc〔8-9〕、f emA 基因〔12〕具有很高的種特異性。本實驗也表明nuc基因與其表型具有較高的符合率為97.47%。
耐藥性的產生與攜帶耐藥相關基因有關。Sa對β-內酰胺類耐藥主要有兩種機制:一是產生β-內酰胺酶,破壞β-內酰胺環從而對青霉素類耐藥,由blaZ基因編碼;二是耐甲氧西林的菌株獲得了mecA基因,編碼新的青霉素結合蛋白 PBP2a〔13〕。目前臨床報道的90%Sa都產生β-內酰胺酶,但Desj Simeoni報道豬肉生產鏈中分離的葡萄球菌中有63.6%菌株為blaZ陽性〔14〕。本實驗中blaZ擴增陽性率為73.42%,相對較高。β-內酰胺酶試驗結果和對青霉素類的敏感性結果與blaZ擴增結果的符合率分別為60.76%和75.95%,而三個試驗結果(β-內酰胺酶實驗,對青霉素類敏感性和blaZ擴增)同時符合率為49.37%。耐青霉素G、氨芐西林的菌株中也有blaZ擴增陰性的菌株,并且通過與MICs進行比較分析,不同 MICs值下幾乎都有blaZ陽性和blaZ陰性菌株,且blaZ陽性菌株占優勢,敏感性與blaZ擴增結果并沒有必然相關性。這些結果也說明耐藥表型分析和基因型分析各有側重點,因而存在一定差異。其原因可能是,本試驗菌株來源于食品源所受到的藥物選擇壓力低,不同于臨床分離菌株,對青霉素耐藥的機制可能沒被藥物激活,抗性蛋白表達量低。文獻表明對Sa耐青霉素G的表型和基因型比較,采用頭孢硝噻吩直接測定β-內酰胺酶比瓊脂平板稀釋法藥敏試驗能更好的預測blaZ基因的存在,在處于臨界MICs的Sa對青霉素的耐藥性判定中,可能瓊脂稀釋法中使用的耐藥折點值太高,因為使用這種方法40%攜帶blaZ基因的菌株被列為敏感株〔15〕。也有可能對于表型測試陽性而blaZ陰性的菌株還有其它的耐藥機制(如生物膜、主動外排)起了作用。
mecA是甲氧西林耐藥決定子,79株食源Sa的mecA基因檢測和表型耐藥監測結果均為陰性,符合率100%,也表明此次檢測動物性食品源Sa中無MRSA。目前動物性食品中MRSA的擴散傳播較少〔4,16〕,故產生 β-內酰胺酶是對青霉素類耐藥的主要原因,本實驗結果與之一致。
目前,養殖業中仍存在濫用抗生素、食品中耐藥細菌危險性逐步增高,給人類健康造成較大危害,由于食源性Sa呈現出不一樣的耐藥特性,本研究為加強動物性食品源Sa尤其MRSA的耐藥性檢測和連續監測提供了技術保障,掌握其發生變化趨勢,為食品源Sa的安全評價提供有力支持。
〔1〕Pérez-Roth E,Claverie-M artí n F,Villar J,et al.Multiplex PCR fo r simultaneous identification of Staphy lococcus aureus and detection of methicillin and mupirocin resistance〔J〕.Journal of Clinical Microbiology,2001,39(11):4037-4041.
〔2〕陽志強,郭兆彪,宋亞軍,等.用雙重聚合酶鏈快速鑒別葡萄球菌及其甲氧西林耐藥性〔J〕.中華醫院感染學雜志,2003,13(10):916-918.
〔3〕Ardic N,Ozyurt M,Sareyyupoglu B,et al.Investigation of erythromycin and tetracycline resistance genes in methicillin-resistant Staphy lococci〔J〕.International Journal of Antimicrobial Agents,2005,26(3):213-218.
〔4〕Normanno G,Corrente M,La Salandra G,et al.Methicillin-resistant Staphylococcus aureus(M RSA)in foods of animal origin product in Italy〔J〕.International Journal of Food Microbiology,2007,117(2):219-222.
〔5〕劉冬香,劉書亮,王印,等.四川動物性食品中金黃色葡萄球菌的分離鑒定及污染分析〔J〕.食品科學,2009,30(8):251-254.
〔6〕趙旺勝,吳桂榮,劉家喜,等.四種檢測β內酰胺酶方法的實驗比較〔J〕.南京醫科大學學報(中文版),1997,17(6):648.
〔7〕唐俊妮.金黃色葡萄球菌耐熱核酸酶相關基因的功能與特征分析〔D〕.湖北:華中農業大學,2007.
〔8〕Martí n M C,Gonzá lez-Hevia M A,Mendoza M C.Usefulness of a two-step PCR procedure for detection and identification of enterotoxigenic Staphy lococci of bacterial isolates and food samples〔J〕.Food Microbiology,2003,20(5):605-610.
〔9〕Pinto B,Chenoll E,Aznar R,et al.Identification and typing of food-bo rne Staphy lococcus aureusby PCR-based techniques〔J〕.Sy stematic and Applied Microbiology,2005,28(4):340-352.
〔10〕Bag herzadeh Y S,Pourmand M R,Zaeimi Y J.Antibiotic susceptibility patterns and detection of Coa and MecA genes in the Iranian isolates of Staphylococcusaureus〔J〕.International Journal of Infectious Diseases,2008,12(Supplement 1):e271.
〔11〕Gillespie B E,Oliver S P.Simultaneous detection of mastitis pathogens,Staphy lococcus aureus,Streptococcus uberis and Streptococcus agalactiae by multiplex real-time polymerase chain reaction〔J〕.American Dairy Science,2005,88:3510-3518.
〔12〕Hassan S R U,Verma V,Qazi G N.Evaluation of three different molecular markers fo r the detection of Staphy lococcus aureus by polymerase chain reaction〔J〕.Food Microbiology,2008,25(3):452-45.
〔13〕Murphy J T,Walshe R,Devocelle M,et al.A computational model of antibiotic-resistance mechanisms in Methicillin-Resistant Staphylococcusaureus(MRSA)〔J〕.Journal of Theoretical Biology,2008,254(2):284 293.
〔14〕Simeoni D,Rizzotti L,Cocconcelli P,et al.Antibiotic resistance genes and identification of Staphylococci collected from the production chain of swine meat commodities〔J〕.Food Microbiolology,2008,25(1):196-201.
〔15〕Haveri M,Suominen S,Rantala L,et al.Comparison of phenotypic and genotypic detection of penicillin G resistance of Staphy lococcus aureus isolated from bovine intramammary infection〔J〕.Veterinary Microbiology,2005,106(1-2):97-102.
〔16〕Kitai S,Shimizu A,Kawano J,et al.Characterization of methicillin resistant Staphy lococcusaureus isolated from retail raw chicken meat in Japan〔J〕.Journal of Veterinary Medicine Science,2005,67(1):107-110.

